克罗诺杆菌(Cronobacter)是一种无芽孢的革兰氏阴性条件致病菌,主要引起严重的新生儿脑膜炎、败血症及坏死性小肠结肠炎等疾病,死亡率高达40%~80%,患者痊愈后仍会有严重的神经系统后遗症。HEALY B等[1]的报道显示,一些免疫力缺陷的成人尤其是老人也会被感染,会导致如肺炎、脓毒症、脚溃疡、局部感染、骨髓炎、脾脓肿等疾病。但与成年人感染情况相比,5岁以下的儿童发生克罗诺杆菌的感染比例更高[2]。
分类系统将克罗诺杆菌独立为属,包括七个种:阪崎克罗诺杆菌(Cronobacter sakazakii)、苏黎世克罗诺杆菌(Cronobacter turicensis)、丙二酸盐克罗诺杆菌(Cronobacter malonaticus)、莫金斯克罗诺杆菌(Cronobacter muytjensii)、都柏林克罗诺杆菌(Cronobacter dublinensis)、康帝蒙提克克罗诺杆菌(Cronobacter condiment)、尤尼沃斯克罗诺杆菌(C.universalis)[3-4]。克罗诺杆菌存在于食品和环境中,其分离来源较为广泛,从肉类、蔬菜、水果、谷物、牛奶、奶粉替代品、加工奶酪、水[5-7]等食品,草药[8]、香料[9-10]以及家庭灰尘、土壤样本等环境[11],甚至在果蝇[12]的肠道均有分离出该细菌。即食食品[13-14]中的克罗诺杆菌的分离率较高,可见多种食品均存在克罗诺杆菌的污染。有研究发现,新生儿患脑膜炎主要与阪崎克罗诺杆菌克隆复合物CC4有关,而成人感染相关菌株集中在ST7和ST1型,ST12与新生儿坏死性小肠结肠炎有关。因此对克罗诺杆菌分离株进行准确的分型鉴定尤为重要,有利于对疾病爆发进行准确的预防[15-18]。
多位点序列分型(multilocus sequence typing,MLST)方法[19]已经成功地将临床相关的克罗诺杆菌菌株聚集到特定的序列型别(sequence type,ST)或克隆复合体(clonal complex,CC)中,形成稳定的进化谱系。CC型包括3个或3个以上的ST型分型组,较为流行的ST型在细菌群组中占据最核心的位置[20]。克罗诺杆菌具有多种ST型,但许多新生儿脑膜炎病例主要是由阪崎克罗诺杆菌ST4型引起的[21]。2015年,ZENG H等[22]报道了一例由阪崎克罗诺杆菌株引起的新生儿脑膜炎病例,该菌株为新的多药耐药菌株,其序列型为阪崎克罗诺杆菌ST256。武汉市妇幼保健院[23]还从2例婴儿脑膜炎患者体内分离出了多重耐药性菌株,分别为丙二酸盐克罗诺杆菌ST60和阪崎克罗诺杆菌ST83。2016年,南京市某儿童医院[24]从重症肺炎新生儿的痰标本中获得了阪崎克罗诺杆菌ST1型菌株。2016年10年,南京江宁某中学[25]出现了疑似由克罗诺杆菌污染的千张烧肉引起的腹痛,腹泻的食物中毒事件。
本文将对PubMLST数据库中的中国克罗诺杆菌分离株(截至2019年6月28日,下载PubMLST数据库(http://pubmlst.org/cronobacter/)的克罗诺杆菌分离株提交信息,包括分离来源、分离时间和国家、血清型,克隆谱系和ST型等进行分析,利用Excel分析PubMLST数据库中全球克罗诺杆菌的分离来源,分布情况,以及序列分型的多样性,并着重分析中国的克罗诺杆菌的分布情况,对比中国临床株与国外临床株的致病情况,深入比较分离株的遗传多样性,揭示其进化关系,为检测我国食品中的克罗诺杆菌提供理论依据。
1 PubMLST数据库中克罗诺杆菌的全球分布状况
截至2019年6月28日,从PubMLST数据库(http://pubmlst.org/cronobacter/)中下载的克罗诺杆菌分离株信息,筛选出具有完整ST型的分离株2 407株,包含657种ST型和52种CC型。
1.1 地区分布
如表1所示,在PubMLST总数据库中,分离株主要来源是亚洲和欧洲:其中亚洲提交的分离株最多(992株),其中仅中国就提交907株,欧洲(899株)主要包括捷克、爱尔兰、德国、英国和法国提交的菌株;其次为北美洲、大洋洲和南美洲:北美洲(274株)以美国提交的菌株为主;大洋洲(98株)以澳大利亚提交的菌株为主;南美洲(66株)以巴西提交的菌株为主;最少的是非洲(4株)。由此可见,以国家为单位,中国的克罗诺杆菌分离株数量最多,反映了我国学者充分利用PubMLST数据库的积极性和能力,也为本文的分析提供了坚实的数据基础。
表1 全球克罗诺杆菌分离株分布情况
Table 1 Distribution of global Cronobacter isolates

注:“*”表示欧洲其他国家分离株数量小于70株国家的总和。
1.2 分离来源
提交到数据库中的克罗诺杆菌分离株主要来源于食品、临床、环境等。如表2所示,我国提交的食品株最多,为580株,主要包括婴幼儿配方奶粉(188株),香料(97株),食品配料(19株)以及即食食物(88株);爱尔兰提交的环境株最多,为88株;捷克提交的临床株最多,为87株,而我国临床株数量较少,仅有38株。
因克罗诺杆菌主要危害2岁以下婴幼儿,而婴幼儿配方奶粉(powdered infant formula,PIF)是其主要感染途径,其中,PubMLST数据库中PIF分离株的ST型分布如图1所示,ST4和ST1是其中的主要型别,但我国分离株优势型别中还有ST64,ST83,ST12型等;而其他国家的优势型别则包括ST13,ST7,ST297,ST14型,与我国的数据存在一定的差异。ST4型菌株被认为与新生儿脑膜炎高度相关,却是中外婴幼儿配方奶粉中检出率最高的克罗诺杆菌型,但该ST型在中国分离株中占比(19.1%)显著低于国外(27.1%),另一个被认为与新生儿坏死性小肠结肠炎高度相关的致病型ST12(4.8%)却在中国占有较高的比例,但在国外则无ST12,不占优势。
表2 全球克罗诺杆菌分离株主要来源
Table 2 Main sources of global Cronobacter isolates
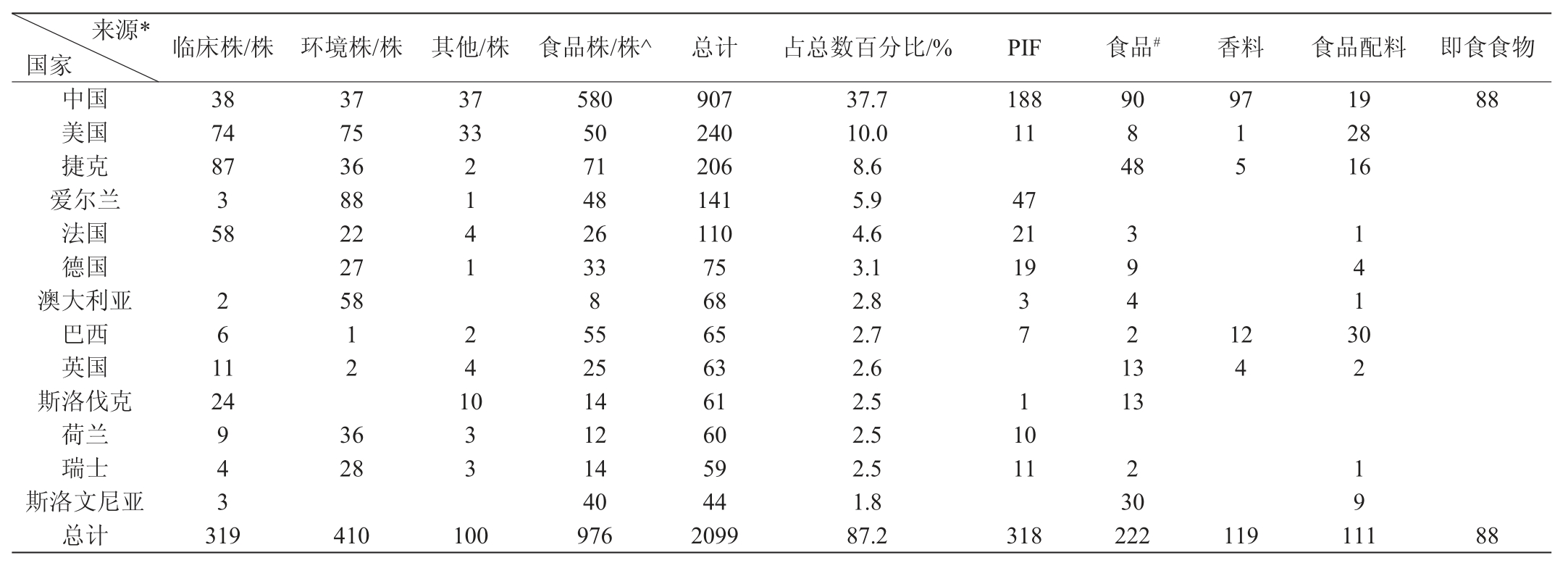
注:“*”表示分离源完全依据数据库中的分类信息;“^”表示食品株是各种食品(PIF即婴幼儿配方奶粉、食品配料、其他食品、即食食物、香料和蔬菜)的总和;“#”表示食品包括面条、方便面、巧克力、冷冻虾、饼干、奶油、太妃糖、酸奶等;表2中分离株少于40株的国家未列出。
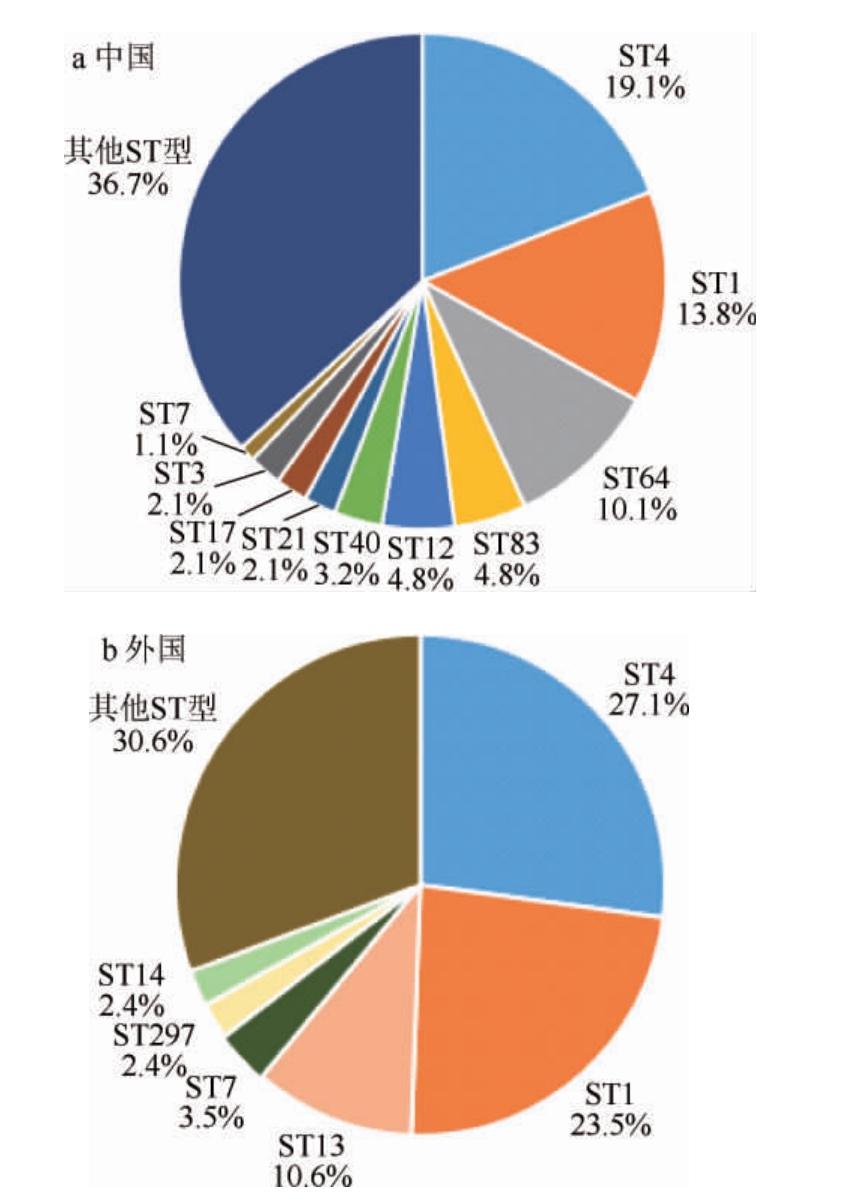
图1 婴幼儿配方奶粉中完整序列型分布
Fig.1 Distribution of complete sequence types in infant milk formula
其他ST型为占比小于2.0%的总和。
2 克罗诺杆菌7个管家基因的保守性与多样性
PubMLST数据库中克罗诺杆菌分离株的等位基因统计结果显示,7个管家基因片段的等位基因值在182~346之间,pps等位基因数目最多,为346个,其等位基因多样性最为丰富;fusA等位基因数目最少,为182个,atpD等位基因数目也较少,为183个,可见这两个管家基因在菌株间差异较小,与其他管家基因序列相比更为保守。
3 中国克罗诺杆菌分析
3.1 中国克罗诺杆菌分离株ST型和CC型分布状况
筛选PubMLST数据库,具有完整ST型的中国分离株有907株,占全球分离株的37.7%,但是我国的ST型和CC型分别为366种(占总数据的55.7%)和43种(占总数据的82.7%),可见中国的克罗诺杆菌分离株相对于其他国家地区分离株具有更为丰富的ST型和CC型。
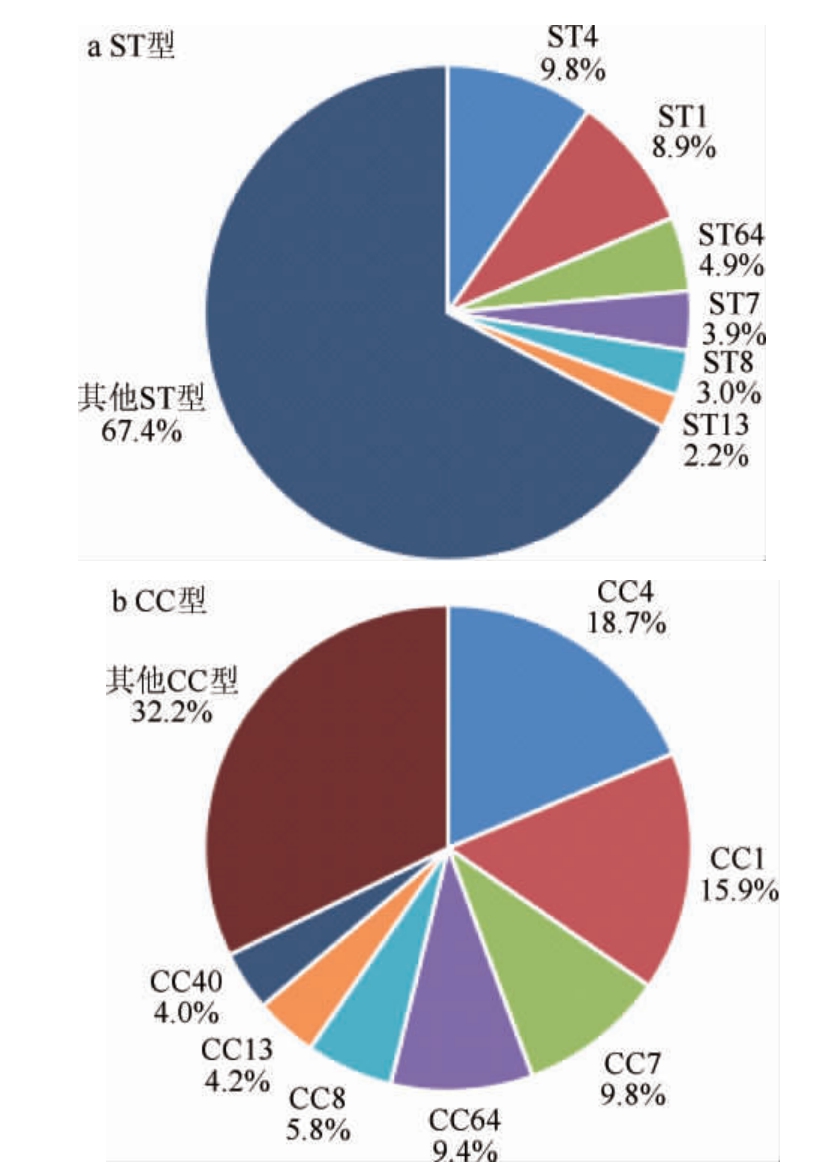
图2 中国分离株完整序列型与克隆复合体型分布
Fig.2 Distribution of complete sequence types and clonal complex types of isolated strains in China
其他ST和CC型为少于20株的总和。
如图2a所示,ST型主要包括ST4(89株),ST1(81株),ST64(44株),ST7(35株),ST8(27株),占总ST型的30.43%;其中ST4,ST1,ST64,ST8均属于阪崎克罗诺杆菌(Cronobacterium sakazakii);ST7属于丙二酸盐克罗诺杆菌(Cronobacter malonaticus)。
如图2b所示,CC型主要包括CC4(103株,以ST4为核心),CC1(88株,以ST1为核心),CC7(54株,以ST7为核心),CC64(52株,以ST64为核心),CC8(32株,以ST8为核心),占总CC型的59.6%。
3.2 中国分离株来源和物种分布情况
表3 中国分离株主要来源和物种分布情况
Table 3 Distribution of main sources and species of isolated strains in China
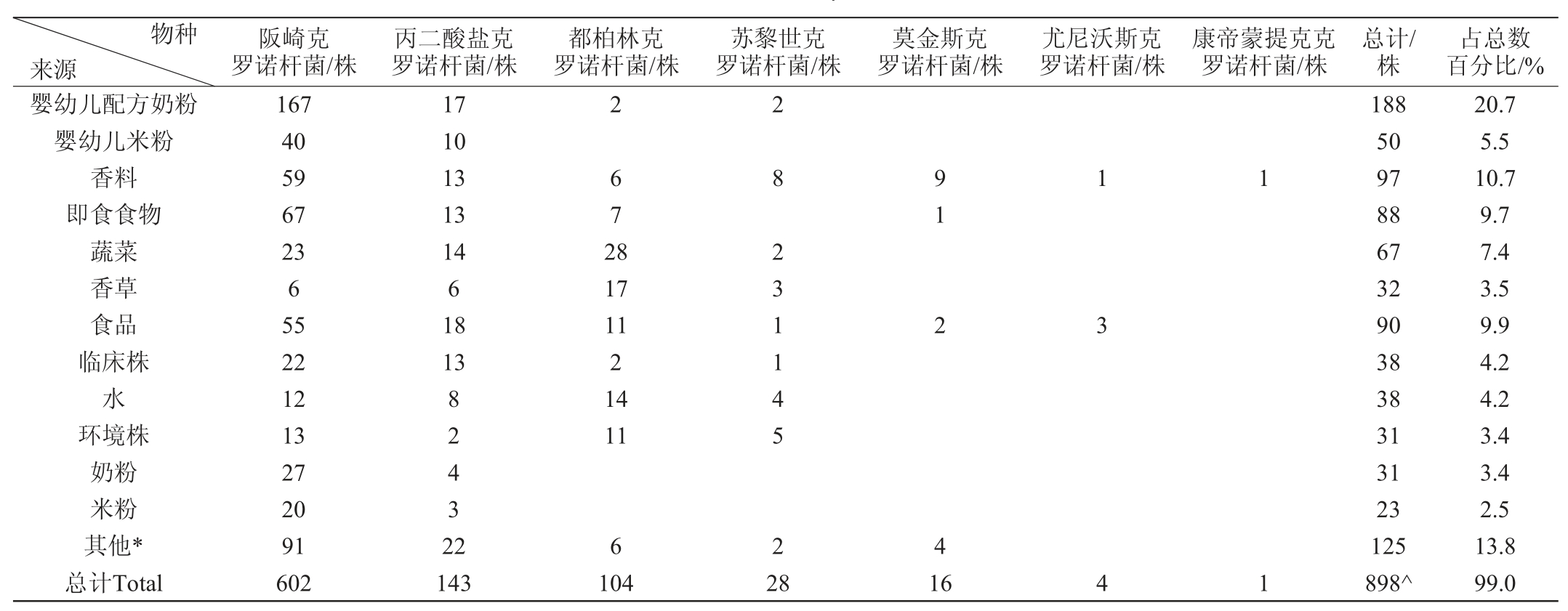
注:“*”表示分离源(≤20株)的菌株数量总和;“^”表示数据库中备注其他菌株的分离源未列出。
如表3所示,中国分离株主要来源于食品,包括婴幼儿配方奶粉、香料、食品配料、即食食品以及蔬菜等。婴幼儿配方奶粉中的分离株最多(188株),临床分离株则较少(38株);克罗诺杆菌属7个种均存在于中国分离株中,其中阪崎克罗诺杆菌602株,丙二酸盐阳性克罗诺杆菌143株以及都柏林克罗诺杆菌104株的数量最多。而前两者在我国临床分离株中占比却高达92.1%。
水和环境分离株分别有38和31株,占比仅为8.9%,这与在我国克罗诺杆菌主要与食品安全特别是婴幼儿食品安全高度相关,对其生态分布的研究不多有关。但也说明食品原料生产及加工环境中该菌的遍在性,污染途径多样,杜绝其污染的难度不小。
3.3 阪崎克罗诺杆菌ST4,ST12及丙二酸盐克罗诺杆菌ST7型的分布以及来源分析
英国学者发现新生儿脑膜炎病例主要与阪崎克罗诺杆菌ST4型相关,中国ST4型分离株有89株,分布广泛,如表4所示,主要来源为婴幼儿配方奶粉、即食食品、奶粉、香料,同时在婴儿面条、冰淇淋、乳清蛋白粉、蔬菜等中也有分离出,但临床分离株非常少(2株)。
阪崎克罗诺杆菌ST12型与新生儿坏死性小肠结肠炎有关,中国ST12型分离株有14株,主要来源于婴幼儿配方奶粉、婴幼儿配方米粉、奶粉、即食食品以及香料中,未见临床分离株。
表4 中国克罗诺杆菌ST4、ST12型与ST7型来源
Table 4 Sources of Cronobacter ST4,ST12 and ST7 in China
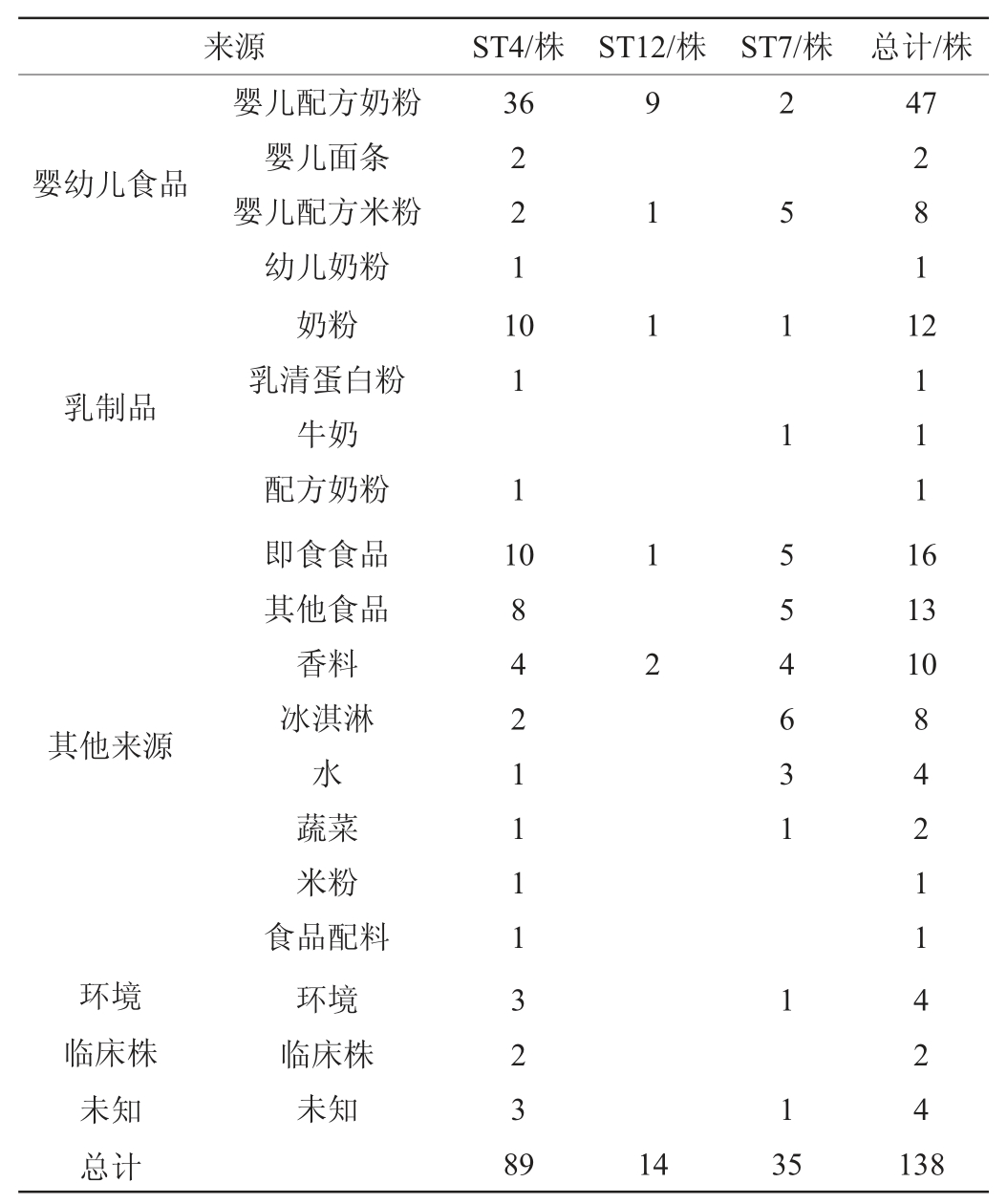
丙二酸盐克罗诺杆菌中ST7型被认为与成年人感染相关,中国ST7型分离株共有35株,主要来源于冰淇淋、即食食品、婴幼儿配方米粉、水,从奶粉、香料、蔬菜、牛奶中也有分离出,但未见临床分离株。
3.4 国内外中国临床分离株比较
对PubMLST数据库国外临床株ST型分布进行统计结果表明,国外临床株有365株,明确致病的菌株有53株,提交国家包括法国(29株),美国(17株),以色列(3株),新西兰(2株),英国(1株),巴西(1株)。明确致病的ST4菌株有39株,占所有明确致病临床株的73.6%,特别在严重致死感染中占比极为突出,如在导致坏死性小肠结肠炎菌株中占到83.3%,在脑膜炎、脑脓肿等脑部疾病致病株中占比70.0%,在其他如菌血症、腹泻等严重感染中占比55.5%。所以有学者明确指出ST4型与新生儿脑膜炎之间具有显著相关性[21]。
PubMLST数据库国外ST21分离株主要分离于成年人粪便,肺标本以及腹膜液;ST148(1株)分离自老年人血液中;ST60主要为成年人临床株,但无进一步的临床信息;ST256曾从丝光绿蝇消化道中分离出,但未出现临床株;ST201无临床株,仅有一株从食品配料中分离出。国外与婴幼儿脑病相关的CC4型菌株(16株),包括ST4,ST107,ST109;其他ST型如ST1,ST307,ST494也会导致脑病;1998年以色列新生儿感染阪崎克罗诺杆菌病例中,从患败血症脑膜炎并发症新生儿血液中分离出ST83菌株[26-27];ST12会导致坏死性小肠结肠炎;ST8则与腹泻有关;在法国婴儿重症监护室爆发克罗诺杆菌感染案例中,分离出3株ST13,两株分离自无症状新生儿,一株分离自具有消化问题的新生儿[28]。值得引起注意的是都柏林克罗诺杆菌ST80和ST88曾引起病人脓肿;苏黎世克罗诺杆菌ST19也曾引起新生儿致死性脑膜炎[29];尤尼沃斯克罗诺杆菌ST59与腿部感染有关[30]。国外[31]曾报道两例由克罗诺杆菌引起的血性腹泻案例与我国报道的腹泻事件症状略有不同。
我国临床分离株(38株)主要为阪崎克罗诺杆菌(22株)和丙二酸盐克罗诺杆菌(13株),同时出现了都柏林克罗诺杆菌(2株),苏黎世克罗诺杆菌(1株)。与国外上传的数据相比,我国明确致病菌株为阪崎克罗诺杆菌(13株)与丙二酸盐克罗诺杆菌(5株),与脑膜炎有关的临床株有ST256(1株),ST60(2株),ST83(2株),没有ST4型;与肺炎相关的临床株为ST1(3株);未注明病症的患者分离株有10株,包括ST1(1株),ST4(2株),ST13(2株),ST201(3株),ST21(1株),ST148(1株)。ST4菌株只有2株而且未注明致病情况,仅占临床分离株的5.3%,我国也没有任何临床分离株为ST7型,与国外情况存在极大差异。
4 结束语
MLST数据库中目前具有2407株完整ST型克罗诺杆菌菌株,分离源主要是亚洲和欧洲的国家,非洲,大洋洲等地区的克罗诺杆菌分离株较少,这可能是不同地区的检测手段和重视程度不同。目前我国的克罗诺杆菌食品分离株较多,分布较为广泛,除婴幼儿食品、即食食品、香料、蔬菜外,冰淇淋、糖果等也有分离株。阪崎克罗诺杆菌ST1和ST4的食品分离株占比较大,且分离自婴幼儿配方奶粉的ST4型(36株)和ST1型(26株)较多,与新生儿坏死性小肠结肠炎相关的ST12菌株也占较高的比例,虽然我国关于婴幼儿配方奶粉中克罗诺杆菌检测的工作开展范围较广,但LIU H等[7,32]从饮用水中也检测到了克罗诺杆菌,可见克罗诺杆菌的污染可能会通过婴幼儿配方奶粉以外的途径传播,并且我国香料中克罗诺杆菌检出率也相当高,而且为避免挥发常在加热杀菌后才加入,使其成为克罗诺杆菌的污染载体。HU J等[33]从环境和土壤中检测出克罗诺杆菌,环境也可能是感染克罗诺杆菌的因素,应当引起重视。
从物种分布来看,我国分离株主要包括阪崎克罗诺杆菌(602株),丙二酸盐克罗诺杆菌(143株)和都柏林克罗诺杆菌(104株)。我国临床株中除阪崎克罗诺杆菌和丙二酸盐克罗诺杆菌,也出现了都柏林克罗诺杆菌和苏黎世克罗诺杆菌,国外曾出现过苏黎世克罗诺杆菌引发新生儿致死性脑膜炎,因而克罗诺杆菌属其他种的致病性也应当引起足够的重视。另外,克罗诺杆菌以非典型食物中毒感染闻名,YONG W等[36]发现阪崎克罗诺杆菌不仅会影响婴幼儿和免疫力低下成人,还可能作为食源性急性肠胃炎的病原体影响健康的成年人。
我国分离株中ST4,ST12和ST7型菌株较多,但主要是食品分离株,临床株较少(ST1为4株,ST4为2株),ST7无临床分离株。而数据库中除了以色列之外其他亚洲国家均未见临床株,我国临床株也较少(38株),而我国菌株总数量在数据库中占比高达37.7%,致病型菌株却仅占9.4%。且我国有明确致病信息和型别的信息与欧美国家的致病型差异也很大:仅有ST256,ST60以及ST83与新生儿脑膜炎相关;中国的文献中仅有1例克罗诺杆菌ST83致死的案例报道。PubMLST数据库中提交的907株克罗诺杆菌中国分离株信息也证实我国学者的科研能力和对克罗诺杆菌的重视程度,因此可能是我国的克罗诺杆菌与其他国家地域的菌株在系统进化和毒力上具有差异,或者与该细菌的感染对象-人种差异有关。
[1]HEALY B,COONEY S,O'BRIEN S,et al. Cronobacter(Enterobacter sakazakii):an opportunistic food borne pathogen[J].Foodborne Pathogens Dis,2010,7(4):339-350.
[2]PATRICK M E,MAHON B E,GREENE S A,et al.Incidence of Cronobacter spp.infections,United States,2003-2009[J].Emerg Infect Dis,2014,20(9):1520-1523.
[3]JOSEPH S,SONBOL H,HARIRI S,et al.Diversity of the Cronobacter genus as revealed by multilocus sequence typing[J]. J Clin Microbiol,2012,50(9):3031-3039.
[4]![]() O,FORSYTHE S.Cronobacter spp.as emerging causes of healthcare-associated infection[J].J Hosp Infect,2014,86(3):169-177.
O,FORSYTHE S.Cronobacter spp.as emerging causes of healthcare-associated infection[J].J Hosp Infect,2014,86(3):169-177.
[5]FRIEDEMANN M. Enterobacter sakazakii in food and beverages(other than infant formula and milk powder)[J]. Int J Food Microbiol,2007,116(1):1-10.
[6]HOCHEL I,RUZICKOVA H,KRASNY L,et al.Occurrence of Cronobacter spp.in retail foods[J].J Appl Microbiol,2012,112(6):1257-1265.
[7]LIU H,YANG Y,CUI J,et al.Evaluation and implementation of a membrane filter method for Cronobacter detection in drinking water[J].FEMS Microbiol Lett,2013,344(1):60-68.
[8]GARBOWSKA M,BERTHOLD-PLUTA A,STASIAK-ROZANSKA L.Microbiological quality of selected spices and herbs including the presence of Cronobacter spp.[J].Food Microbiol,2015,49:1-5.
[9]BAUMGARTNER A,GRAND M,LINIGER M,et al.Detection and frequency of Cronobacter spp.(Enterobacter sakazakii)in different categories of ready-to-eat foods other than infant formula[J].Int J Food Microbiol,2009,136(2):189-192.
[10]LIU M,HU G,SHI Y,et al.Contamination of Cronobacter spp.in Chinese retail spices[J].Foodborne Pathogens Dis,2018,15(10):637-644.
[11]MOLOY C,CAGNEY C,O'BRIEN S,et al.Surveillance and characterisation by pulsed-field gel electrophoresis of Cronobacter spp.in farming and domestic environments,food production animals and retail foods[J].Int J Food Microbiol,2009,136(2):198-203.
[12]MRAMBA F,BROCE A B,ZUREK L.Vector competence of stable flies,Stomoxys calcitrans L.(Diptera:Muscidae),for Enterobacter sakazakii[J].J Vector Ecol,2007,32(1):134-139.
[13]XU X,LI C,WU Q,et al.Prevalence,molecular characterization,and antibiotic susceptibility of Cronobacter spp.in Chinese ready-to-eat foods[J].Int J Food Microbiol,2015,204:17-23.
[14]VASCONCELLOS L,CARVALHO C T,TAVARES R O,et al.Isolation,molecular and phenotypic characterization of Cronobacter spp.in readyto-eat salads and foods from Japanese cuisine commercialized in Brazil[J].Food Res Int,2018,107:353-359.
[15]JOSEPH S,FORSYTHE S.Predominance of Cronobactersakazakii sequence type 4 in neonatal infections[J].Emerg Infect Dis,2011,17(9):1713-1715.
[16]FORSYTHE S J,DICKINS B,JOLLEY K A.Cronobacter,the emergent bacterial pathogen Enterobacter sakazakii comes of age;MLST and whole genome sequence analysis[J].BMC Genomics,2014,15(1):1121.
[17]HANA S,SUSAN J,CATHERINE M,et al.Multilocus sequence typing of Cronobacter spp.from powdered infant formula and milk powder production factories[J].Int Dairy J,2013,30(1):1-7.
[18]KUCEROVA E,JOSEPH S,FORSYTHE S.The Cronobacter genus:ubiquity and diversity[J].Qual Assur Saf Crop Foods,2011,3(3):104-122.
[19]SUSAN J,FORSYTHE S J.Insights into the emergent bacterial pathogen Cronobacter spp.generated by multilocus sequence typing and analysis[J].Front Microbiol,2012,3:397.
[20]徐海燕.不同地区自然发酵乳中乳酸乳球菌多位点序列分型研究[D].呼和浩特:内蒙古农业大学,2013.
[21]HARIRI S,JOSEPH S,FORSYTHE S J. Cronobacter sakazakii ST4 strains and neonatal meningitis,United States[J].Emerg Infect Dis,2013,19(1):175-177.
[22]ZENG H,LEI T,HE W,et al.Novel multidrug-resistant Cronobacter sakazakii causing meningitis in Neonate,China,2015[J]. Emerg Infect Dis,2018,24(11):2121-2124.
[23]CUI J H,YU B,XIANG Y,et al.Two cases of multi-antibiotic resistant Cronobacter spp.infections of infants in China[J].Biomed Environ Sci,2017,30(8):601-605.
[24]SHI L,LIANG Q,ZHAN Z,et al.Co-occurrence of 3 different resistance plasmids in a multi-drug resistant Cronobacter sakazakii isolate causing neonatal infections[J].Virulence,2018,9(1):110-120.
[25]夏广金,董建云,韩宁生,等.一起疑似阪崎肠杆菌引起的学校食源性疾病的流行病学调查[J].中国食品卫生杂志,2018,30(3):299-303.
[26]BAR-OZ B,PREMINGER A,PELEG O,et al.Enterobacter sakazakii infection in the newborn[J].Acta Paediatr,2001,90(3):356-358.
[27]BLOCK C,PELEG O,MINSTER N,et al.Cluster of neonatal infections in Jerusalem due to unusual biochemical variant of Enterobacter sakazakii[J].Eur J Clin Microbiol Infect Dis,2002,21(8):613-616.
[28]CAUBILLA-BARRON J,HURRELL E,TOWNSEND S,et al.Genotypic and phenotypic analysis of Enterobacter sakazakii strains from an outbreak resulting in fatalities in a neonatal intensive care unit in France[J]. J Clin Microbiol,2007,45(12):3979-3985.
[29]ESSERS B,BAENZIGER O,HUISMAN TA,et al.Neonatal sepsis with Enterobacter sakazakii in premature twins[J]. Swiss Med Wkly,2006,136(suppl 151):22S.
[30]JOSEPH S,CETINKAYA E,DRAHOVSKA H,et al.Cronobacter condimenti sp.nov.isolated from spiced meat,and Cronobacter universalis sp.nov.a species designation for Cronobacter sp.genomospecies 1,recovered from a leg infection,water and food ingredients[J].Int J Syst Evol Micr,2012,62(Pt 6):1277-1283.
[31]FLORES J P,MEDRANO S A,SÁNCHEZ J S,et al.Two cases of hemorrhagic diarrhea caused by Cronobacter sakazakii in hospitalized nursing infants associated with the consumption of powdered infant formula[J].J Food Protect,2011,74(12):2177-2181.
[32]VASCONCELLOS L,MEDEIROS V D M,ROSAS C D O,et al.Occurrence of total coliforms,Escherichia coli and Cronobacter species in commercially available 20 L bottled drinking water sold in Rio de Janeiro State,Brazil[J].Lett Appl Microbiol,2019,69(6):431-437.
[33]HU J,LI X,DU X,et al.Identification and characterization of Cronobacter strains isolated from environmental samples[J].Curr Microbiol,2019,76(12):1467-1476.
[34]MARSZEWSKA K,CZERWICKA M,FORSYTHE S J,et al.Structural studies of O-polysaccharide isolated from Cronobacter sakazakii sequence type 12 from a case of neonatal necrotizing enterocolitis[J]. Carbohydr Res,2015,407:55-58.
[35]MASOOD N,MOORE K,FARBOS A,et al.Draft genome sequence of the earliest Cronobacter sakazakii sequence type 4 strain,NCIMB 8272[J].Genome Announc,2013,1(5):e00782-13.
[36]YONG W,GUO B F,SHI X C,et al.An investigation of an acute gastroenteritis outbreak:Cronobacter sakazakii,a potential cause of foodborne illness[J].Front Microbiol,2018,9:2549.