酱香型白酒是中国白酒的五大香型之一,其代表性特征是酱香突出、酒体醇厚、回味悠长和空杯留香等[1]。酱香型白酒的经典酿造工艺为高温制曲、重阳下沙、两次投粮、九次蒸煮、八次加曲发酵、七次取酒。在其开放式生产过程中,依赖于来自大曲、酒醅和窖泥的丰富微生物菌群的功能和作用,完成酱酒生产过程。研究发现,大曲、酒醅和窖泥中的微生物菌群结构具有高度的复杂性和特殊性。在茅台高温大曲中,微生物菌群以嗜热性细菌为主,少量为霉菌[2];泸州老窖酒醅中的优势菌群随着发酵的进行由酿酒酵母(Saccharomyces cerevisiae)演化为耐酸的乳酸菌等[3];在兰陵特曲的窖泥微生物群落相关研究中发现,发酵中后期厌氧细菌占据优势地位[4],暗示细菌菌群对白酒产量和品质起着重要的作用。
依赖传统的分离培养方法,对微生物种类进行鉴定比较有限。同时,鉴于一些微生物的不可培养性,高效全面地解析酿酒微生物的组成和变化始终面临挑战[5-6]。目前,免培养的分子生物学技术广泛应用于微生物多样性的研究,基于高通量测序(high-throughput sequencing,HTS)的宏基因组研究适用于白酒酿造过程中复杂微生物体系的研究。高通量测序不仅具有高通量、高覆盖、高准确率的特点,还可以全面地定性和定量的揭示样品微生物菌群组成和多样性信息[7-8]。
因此,本研究以酱香型郎酒大曲、酒醅和窖泥为研究对象,运用高通量测序技术解析其细菌菌落结构和多样性,旨在能够全面准确地分析酱香型郎酒在高温制曲、高温堆积和多次发酵取酒的生产过程中不同材料中的细菌菌落结构和差异,为建立酱香型郎酒微生物信息数据库和优化酱香型郎酒酿造工艺和提升品质提供一定的理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
本实验所有样品均采集自四川省古蔺郎酒厂有限公司生产车间,高温大曲样品为储存一个月的新制成品曲,整块大曲经粉碎后混合取样约100 g;酒醅样品为第四轮次堆积中的酒醅,于堆积酒醅中心点附近取样约100 g;窖泥于窖池底部采样约100 g。上述样品采集后均转移至无菌聚乙烯袋中,并迅速置于冰盒中运回,之后于-80 ℃超低温冰箱中保藏。
1.1.2 试剂
PowerSoil脱氧核糖核酸(deoxyribonucleic acid,DNA)Isolation Kit:美国MO Bio公司;微生物高通量测序建库试剂盒:北京百迈克公司;Phusion HF MM高保真聚合酶链式反应(polymerase chain reaction,PCR)酶(2 U/μL):美国BioLabs公司;![]() PCR Purification Kit:德国Qiagen公司;Tru-
PCR Purification Kit:德国Qiagen公司;Tru-![]() DNA PCR-Free Sample Preparation Kit:美国Illumina公司;细菌16S rRNA V3-V4区PCR扩增引物(338F:5'-ACTCCTACGGGAGGCAGCA-3';806R:5'-GGACTACHVGGGTWTCTAAT-3'):北京六合华大基因科技有限公司。
DNA PCR-Free Sample Preparation Kit:美国Illumina公司;细菌16S rRNA V3-V4区PCR扩增引物(338F:5'-ACTCCTACGGGAGGCAGCA-3';806R:5'-GGACTACHVGGGTWTCTAAT-3'):北京六合华大基因科技有限公司。
1.2 仪器与设备
Illumina HiSeq 2500测序仪:美国Illumina公司;S1000 PCR仪、CFX96荧光定量PCR仪、Gel DocTM XR+凝胶成像系统:美国Bio-Rad公司;DYY-8C水平电泳槽:北京六一仪器厂;VORTEX-3旋涡混匀器:德国IKA公司;Allegra X-22R低温冷冻离心机:德国Beckman公司;BT25S准微量分析天平:日本SANYO公司;BioPhotometer Plus核酸蛋白测定仪:德国Eppendorf公司;NanoDrop 2000超微量分光光度计:美国Thermo公司。
1.3 实验方法
1.3.1 大曲、酒醅和窖泥菌群基因组DNA的提取及PCR扩增
首先于液氮中研磨样品成粉末状,准确称取0.25 g各样品至PowerBead Tubes中,加入裂解液Solution C1后,3 200 r/min涡旋振荡15 min,经碳化硅研磨珠的机械力和裂解液的化学反应共同作用下,充分裂解微生物,之后按照PowerSoil DNA Isolation Kit说明书分别提取大曲、酒醅和窖泥的基因组DNA,采用超微量分光光度计测定含量后,以其为模板,采用引物338F和806R对细菌的16S rRNA V3-V4区序列进行PCR扩增,PCR扩增程序:98 ℃预变性1 min;98 ℃变性10 s,50 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃再延伸5 min。PCR扩增体系(20 μL):2×Phusion Master Mix 10 μL,正向及反向引物(10 μmol/L)各1 μL,模板DNA 10 ng,补双蒸水(ddH2O)至20 μL。PCR扩增产物经1.8%琼脂糖凝胶电泳检测。
利用![]() PCR Purification Kit对PCR扩增产物进行纯化。利用
PCR Purification Kit对PCR扩增产物进行纯化。利用![]() DNA PCR-Free Sample Preparation Kit制备测序文库并添加接头序列,经过NanoDrop 2000测定文库质量后,利用Illumina HiSeq 2500测序平台进行高通量测序。
DNA PCR-Free Sample Preparation Kit制备测序文库并添加接头序列,经过NanoDrop 2000测定文库质量后,利用Illumina HiSeq 2500测序平台进行高通量测序。
1.3.2 生物信息学分析
利用FLASH[9]对测序获得的原始数据进行拼接,使用Trimmomatic软件[10]对拼接得到的序列质量进行过滤,采用UCHIME软件[11]去除嵌合体,获得高质量的Tags序列。利用UCLUST软件[12]在相似性97%的水平上对序列进行聚类,以所有序列数的0.005%作为阈值过滤操作分类单元(operational taxonomic unit,OTU)[13]。基于Silva[14]和RDP[15]对OTU进行分类学注释分析,同时计算该OTU在各样品中的相对含量。利用ClustalW2软件[16]以邻接(neighbor-joining,NJ)法构建系统进化树,利用Mothur软件[17]对样品进行Alpha多样性分析。
2 结果与分析
2.1 菌群基因组DNA的提取及PCR扩增
采用PowerSoil DNA Isolation Kit提取郎酒大曲、酒醅和窖泥菌群的基因组DNA,以其为模板PCR扩增细菌的16S rRNA V3-V4区序列,结果见图1。由图1可知,PCR扩增产物均在430 bp左右,与预期结果相符。

图1 郎酒大曲、酒醅和窖泥细菌16S rRNA V3-V4PCR扩增产物琼脂糖凝胶电泳结果
Fig.1 Results of agarose gel electrophoresis of PCR products for 16S rRNA V3-V4of bacteria in Langjiu Daqu,fermented grains and pit mud
M为DNA Marker。
2.2 样品测序数据处理结果
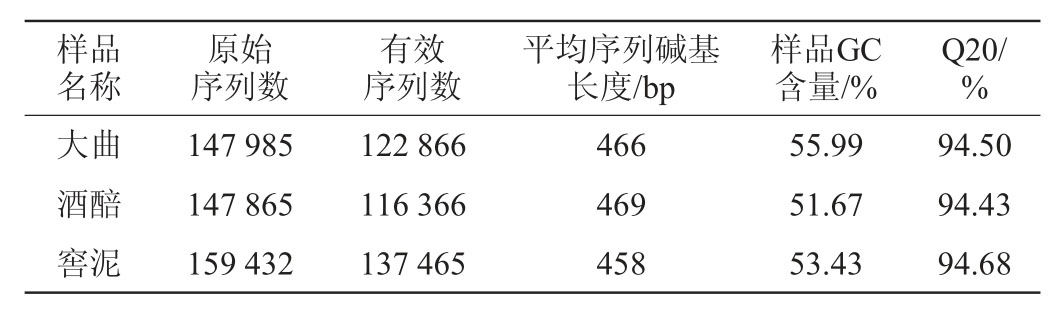
郎酒大曲、酒醅和窖泥中细菌16S rRNA V3-V4区序列的测定结果见表1。由表1可知,从3种样品中共检测出455 282条原始序列,过滤后最终共得到376 697条有效序列,平均每个样品125 566条有效序列。去除引物和barcode后,大曲、酒醅和窖泥样品中细菌16S rRNA V3-V4区序列的平均碱基长度分别为466 bp、469 bp和458 bp,3种样品的Q20值(Q20为质量值≥20的碱基占总碱基数的百分比)均高于94%,说明数据质量较高。
表1 郎酒大曲、酒醅和窖泥样品测序数据处理结果统计
Table 1 Statistics of sequencing data processing results of Langjiu Daqu,fermented grains and pit mud samples
2.3 细菌菌群的多样性分析
由图2可知,随着测序数列的增加,3种样品的OTU及香农指数均呈先急速增加,后趋于平缓的趋势,说明测序数量合理。
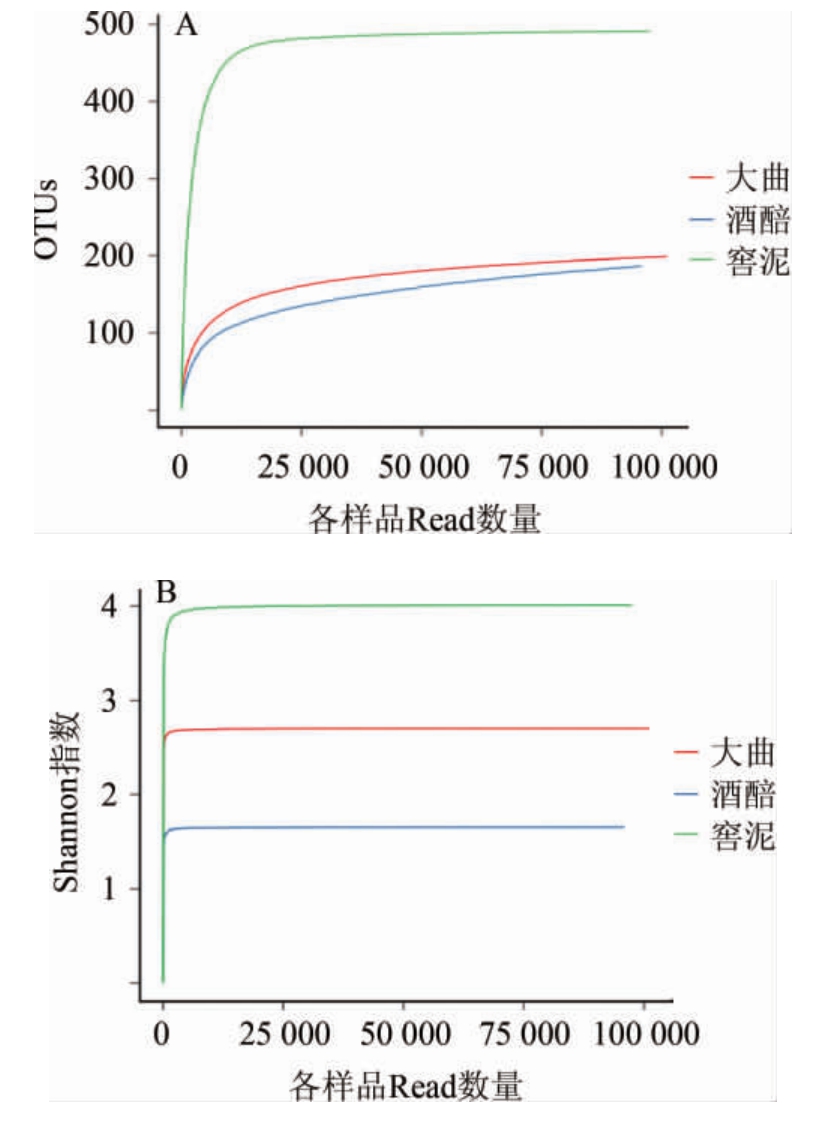
图2 郎酒大曲、酒醅和窖泥细菌菌群的稀释性曲线(A)及香农指数曲线(B)
Fig.2 Rarefaction curve (A) and Shannon index curve (B) of bacterial community in Langjiu Daqu,fermented grains and pit mud
在97%的相似度水平下,对376 697条有效序列进行聚类分析,利用QIIME划分OTU,并基于OTU对3种样品中细菌的Alpha多样性(ACE指数、Chao1指数、Simpson指和Shannon指数)进行分析,结果见表2。
由表2可知,郎酒大曲、酒醅和窖泥样品中的OTU数分别为199个、187个和491个。窖泥中细菌菌群的ACE指数和Chao1最大,丰富度最高,大曲和酒醅的细菌菌群丰富度相近。窖泥中细菌菌群的Simpson指数最小,Shannon指数最大,多样性最高,而酒醅中细菌菌群的多样性最低。
表2 郎酒大曲、酒醅和窖泥样品中细菌Alpha多样性分析结果
Table 2 Results of Alpha diversity analysis of bacteria in Langjiu Daqu,fermented grains and pit mud samples

进一步对3种样品中细菌菌群的构成进行比较分析并绘制韦恩(Venn)图,结果见图3。由图3可知,3种样品共有的OTU数为104个,大曲和酒醅共有的OTU数为14个,大曲和窖泥共有的OTU数为43个,酒醅和窖泥共有的OTU数为18个,而大曲、酒醅和窖泥特有的OTU数分别为34个、51个和322个。结合Alpha多样性分析结果,多样性最丰富的窖泥中存在322个特有的OTU,凸显窖泥微生物菌群的复杂性和特殊性。
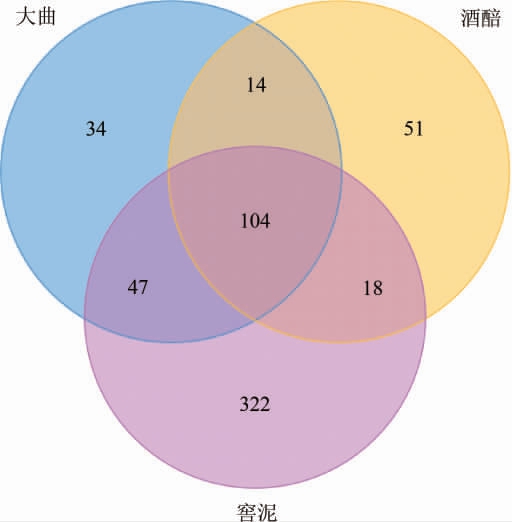
图3 郎酒大曲、酒醅和窖泥样品中OTU的Venn图
Fig.3 Venn diagram of OTU in Langjiu Daqu,fermented grains and pit mud samples
2.4 细菌菌群结构分析
在OTU划分的基础上,选取不同OTU中丰度最高的序列作为该OTU的代表序列,将代表序列与微生物参考数据库进行比对分析,对郎酒大曲、酒醅和窖泥中的细菌进行分类学鉴定,结果见表3。
由表3可知,大曲中的细菌被鉴定为7个门101个属。酒醅中的细菌被鉴定为9个门78个属。窖泥中的细菌被鉴定为11个门186个属,但仍有部分序列经过比对无法鉴定到已知的属和种。细菌属的相对含量见图4,将相对含量≥1%的属定义为优势细菌属。
表3 郎酒大曲、酒醅和窖泥样品的各分类学地位数量统计
Table 3 Quantitative statistics of taxonomic status of Langjiu Daqu,fermented grains and pit mud samples

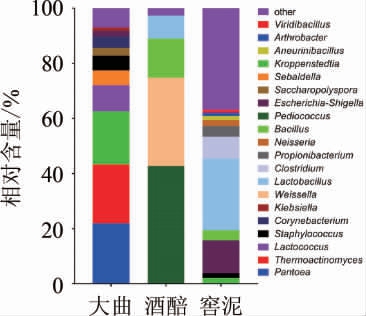
图4 基于属水平郎酒大曲、酒醅和窖泥中的细菌菌群结构分析
Fig.4 Analysis of bacterial community structure in Langjiu Daqu,fermented grains and pit mud based on genus level
other为相对含量<1%的菌属以及未得到分类学注释的OTU之和。
由图4可知,大曲中的优势细菌属主要隶属于泛菌属(Pantoea)(21.9%)、高温放线菌属(Thermoactinomyces)(21.4%)、克罗彭施泰特氏菌属(Kroppenstedtia)(19.4%)、乳球菌属(Lactococcus)(9.4%)、塞巴鲁德氏菌属(Sebaldella)(5.4%)、棒状杆菌属(Corynebacterium)(4.1%)、葡萄球菌属(Staphylococcus)(5.4%)、糖多孢菌属(Saccharopolyspora)(2.8%)、埃希氏菌-志贺氏菌属(Escherichia-Shigella)(2.1%)和克雷伯氏菌属(Klebsiella)(1.2%)。大曲作为白酒发酵中微生物的主要来源,研究其中主要产风味微生物的种类和数量对于提高白酒的品质具有重要意义。本研究发现,酱香型郎酒高温大曲中优势细菌属为泛菌属、高温放线菌属、克罗彭施泰特氏菌属等,其中,泛菌属(Pantoea)菌群作为重要的具有代谢和发酵类型的化能异养菌,在酿造行业中多次被报道,包括在芝麻香型白酒高温大曲中作为第一优势细菌存在[18],同时在古井贡酒大曲[19]、剑南春大曲[20]、张弓老酒中温曲[21]等中均被鉴定为主要菌群;高温放线菌属(Thermoactinomyces)与郎酒高温大曲的制作工艺具有密切关系,本研究中发现的优势菌种包括普通高温放线菌(Thermoactinomyces vulgaris)和甘蔗高温放线菌(Thermoactinomyces sacchari),二者不仅具有产淀粉酶、纤维素酶和果胶酶等能力,还可以生成具有酱味的吡嗪类物质和土霉异味的二甲萘烷醇等挥发性产物[22-23]。因此,推测在郎酒高温大曲中占据较高比例的高温放线菌具有潜在的提供丰富香味物质和风味物质前体的功能,后续对其进行分离筛选和产酶研究。
酒醅中的优势细菌属主要隶属于片球菌属(Pediococcus)(42.7%)、魏斯氏菌属(Weissella)(32.1%)、芽孢杆菌属(Bacillus)(14.2%)和乳杆菌属(Lactobacillus)(8.3%)。酒醅在堆积过程中不仅利用来自大曲中已存在嗜热细菌,还会吸附来自于空气和生产环境中的微生物,同时在堆积过程中可再次对优势菌群进行富集,利于酒醅发酵条件的稳定,增加酒醅中风味物质的种类和含量,说明酒醅中可能具有区别于大曲的独特菌群成分。本研究发现,酱香型郎酒的酒醅中的优势菌群包括片球菌属的乳酸片球菌(Pediococcus acidilactici)、魏斯氏菌属的类肠膜魏斯氏菌(Weissella paramesenteroides)、芽胞杆菌属的凝结芽孢杆菌(Bacillus coagulans)、乳杆菌属的植物乳杆菌(Lactobacillus plantarum),上述菌群均具有乳酸菌的功能。其中,Weissella paramesenteroides产生的乳酸可与乙醇反应生成白酒风味的重要组分乳酸乙酯,其他乳酸菌Bacillus coagulans和Lactobacillus plantarum作为优势菌种,亦常见于其他白酒研究中[24-26]。上述研究揭示种类丰富的乳酸菌在郎酒酒醅中占绝对优势地位,其占细菌总量的90%以上,包括Pediococcus、Weissella、Bacillus和Lactobacillus 4个属。
窖泥中的优势细菌属主要隶属于Lactobacillus(26.1%)、Escherichia-Shigella(12%)、梭菌属(Clostridium)(7.9%)、丙酸杆菌属(Propionibacterium)(3.9%)、芽孢杆菌属(Bacillus)(3.6%)、奈斯氏菌属(Neisseria)(42.7%)、Kroppenstedtia(2.2%)、葡萄球菌属(Staphylococcus)(1.7%)、解硫胺素芽胞杆菌属(Aneurinibacillus)(1.4%)、节杆菌属(Arthrobacter)(1.3%)和绿芽胞杆菌属(Viridibacillus)(1%)。窖泥作为酱香型白酒发酵环境中的重要物质,其厌氧环境下富集的功能微生物可产生多种对酒体有贡献的风味成分[27]。经过长期驯化和演替而逐渐形成的窖泥微生物菌群极为复杂,目前的应用集中于窖泥微生物的复合应用,据报道可有效提高原酒优质品率等[5]。但窖泥环境中可培养的细菌含量极低,大量的细菌不能培养或者难以培养,传统的分离培养方法用于研究窖泥微生物菌群具有较大局限性,因此,利用高通量测序分析窖泥细菌菌群具有重要的意义。本研究发现,窖泥细菌菌群的丰富度和多样性远高于大曲和酒醅。同时,窖泥中的优势菌种多为未培养未鉴定菌种,其功能特征缺少文献报道,可能与其存在的厌氧环境具有密切关系,现有常见培养系统均不能支持其优势菌种的培养和研究。值得注意的是,本研究中发现酱香型郎酒窖泥中的主体细菌是嗜酸乳杆菌(Lactobacillus acetotolerans)(25.9%),与其在白云边酒[28]、五粮液[29]、芝麻香型白酒[30]、古井贡酒[19]和泸型酒[25]窖泥中作为优势菌群的发现相一致,说明郎酒窖泥是提供酿酒过程中Lactobacillus acetotolerans的主要来源。杜海等[30]研究发现,Lactobacillus对于糊精的利用能力较强,可保障其在窖池发酵中后期葡萄糖较少情况下维持生长,与其可在窖泥中作为主体菌具有密切关联。鉴于本研究中发现郎酒窖泥中存在大量的未鉴定的未培养细菌,推测随着窖池发酵的进程,由于酸度和乙醇含量的升高,可利用还原糖含量的减少,窖池内形成一个相对极端的发酵环境,导致窖泥中的细菌菌群具有独特的代谢特征,难以在常规条件下筛选分离培养,但其作为能够产生多种风味物质并提供乙醇和多种有机酸等的菌群,探究其核心细菌菌群的群落结构和代谢变化特征是后续研究工作的重难点。
3 结论
通过高通量测序从郎酒大曲、酒醅和窖泥样品中收集的有效序列的平均碱基长度分别为466 bp、469 bp和458 bp;在97%相似度水平下聚类分析得到各样品的OTU数分别为199个、187个和491个,共有的OTU数为104个。3种样品在细菌菌群组成和多样性上存在较大差异,其中窖泥中微生物丰富度和多样性最高,大曲中细菌菌群的多样性远高于酒醅。窖泥中主要优势细菌属为乳杆菌属(Lactobacillus)(26.1%)、埃希氏菌-志贺氏菌属(Escherichia-Shigella)(12%)、梭菌属(Clostridium)(7.9%)等,嗜酸乳杆菌(Lactobacillus acetotolerans)作为主体菌,同时存在多种未培养菌群;大曲中主要优势菌属为泛菌属(Pantoea)(21.9%)、高温放线菌属(Thermoactinomyces)(21.4%)、克罗彭施泰特氏菌属(Kroppenstedtia)(19.4%)和乳球菌属(Lactococcus)(9.4%)等,与高温大曲的制作工艺密切关联;酒醅中主要优势细菌属为片球菌属(Pediococcus)(42.7%)、魏斯氏菌属(Weissella)(32.1%)、芽孢杆菌属(Bacillus)(14.2%)和乳杆菌属(Lactobacillus)(8.3%),乳酸菌占据绝对优势地位。本研究为建立酱香型郎酒微生物信息数据库,优化酿造工艺和提升酒品品质提供一定的理论依据。
[1]杨国华,邱树毅,黄永光.酱香白酒生产中产香微生物研究[J].中国酿造,2011,30(4):24-27.
[2]郭坤亮.茅台酒酿造微生物的生物多样性成因及研究价值的探讨[J].酿酒,2002,29(2):36-38.
[3]ZHANG W X,QIAO Z W,SHIGEMATSU T,et al.Analysis of the bacterial community in Zaopei during production of Chinese Luzhou-flavor liquor[J].J I Brewing,2005,111(2):215-222.
[4]施安辉.浓香型白酒发酵过程窖中微生物区系的分析[J].酿酒,1986,13(4):24-29.
[5]赵爽,杨春霞,窦屾,等.白酒生产中酿酒微生物研究进展[J].中国酿造,2012,31(4):5-10.
[6]侯小歌,王俊英,李学思,等.浓香型白酒糟醅及窖泥产香功能菌的研究进展[J].微生物学通报,2013,40(7):1257-1265.
[7]SHOKRALLA S,SPALL J L,GIBSON J F,et al.Next-generation sequencing technologies for environmental DNA research[J]. Mol Ecol,2012,21(8):1794-1805.
[8]王兴春,杨致荣,王敏,等.高通量测序技术及其应用[J].中国生物工程杂志,2012,32(1):109-114.
[9]MAGOC T,SALZGERG S L.FLASH:fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics,2011,27(21):2957-2963.
[10]BOLGER A M,LOHSE M,USADEL B.Trimmomatic:a flexible trimmer for Illumina sequence data[J].Bioinformatics,2014,30(15):2114-2120.
[11]EDGAR R C,HAAS B J,CLEMENTE J C,et al.UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics,2011,27(16):2194.
[12]EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[13]BOKULICH N A,SUBRAMANIAN S,FAITH J J,et al.Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J].Nat Methods,2012,10(1):57-59.
[14]QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J].Nucleic Acids Res,2012,41(D1):D590-D596.
[15]WANG Q,GAMITY G.M,TIEDJE J M,et al.Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].Appl Environ Microbiol,2007,73(16):5261-5267.
[16]LARKIN M A,BLACKSHIELDS G,BROWN N,et al.Clustal W and Clustal X version 2.0[J].Bioinformatics,2007,23(21):2947-2948.
[17]SCHLOSS P D,WESTCOTT S L,RYABIN T,etal.Introducing mothur:open-source,platform-independent,community-supported software for describing and comparing microbial communities[J].Appl Environ Microbiol,2009,75(23):7537-7541.
[18]姚粟.芝麻香型白酒高温大曲细菌群落多样性研究[D].北京:北京林业大学,2013.
[19]张会敏.古井贡酒微生物群落结构及其与主要风味物质的关联研究[D].哈尔滨:哈尔滨工业大学,2017.
[20]刘孟华,唐清兰,徐姿静,等.基于宏基因组学的剑南春大曲细菌多样性分析[J].酿酒,2016(5):30-33.
[21]刘延波,赵志军,陈黄曌,等.高通量测序技术分析浓香型白酒中温曲和高温曲的细菌群落结构[J].现代食品科技,2018(5):229-235.
[22]王芙蓉.酱香型大曲中高温放线菌的筛选及其产酶条件的优化[D].贵阳:贵州大学,2017.
[23]罗小叶.酱香型大曲高温放线菌的特性研究[D].贵阳:贵州大学,2017.
[24]邢敏钰,杜海,徐岩.芝麻香型白酒发酵过程中乳酸菌多样性及其演替规律[J].微生物学通报,2018,45(1):19-28.
[25]栗连会.泸型酒酒醅中乳酸菌和乳酸降解菌的多样性和代谢特性[D].无锡:江南大学,2016.
[26]刘凡,周新虎,陈翔,等.洋河浓香型白酒发酵过程酒醅微生物群落结构解析及其与有机酸合成的相关性[J].微生物学报,2018,58(12):2087-2099.
[27]ZHAO J S,ZHENG J,ZHOU R Q,et al.Microbial community structure of pit mud in a Chinese strong aromatic liquor fermentation pit[J]. J I Brewing,2012,118(4):356-360.
[28]黄莹娜,熊小毛,胡远亮,等.基于PCR-DGGE 和高通量测序分析白云边酒窖泥细菌群落结构与多样性[J].微生物学通报,2017,44(2):375-383.
[29]赵东,郑佳,彭志云,等.高通量测序技术解析五粮液窖泥原核微生物群落结构[J].食品与发酵工业,2017(9):5-12.
[30]杜海,邢敏钰,徐岩.芝麻香型白酒酿造过程中乳酸菌分离及其碳源利用特征[J].食品与发酵工业,2018,44(1):13-18.