中国白酒是世界六大著名蒸馏酒之一,是中国食品工业的重要组成部分,在中国传统文化中占有独特的地位[1]。大曲以小麦、大麦、豌豆等为原料,经自然接种发酵而成,不仅是白酒酿造过程中的酒化剂与糖化剂,也是必不可少的生香剂,均含有大量的用于大分子水解和代谢的微生物和酶,在传统的白酒酿造过程中起着至关重要的作用[2-3]。研究人员对比研究强化大曲和传统大曲时发现,强化大曲可以显著提高白酒生产的效率,使白酒中酯类及芳香族化合物得到显著提高,进而影响白酒的风味[4-5];其中中低温大曲淀粉酶活性与多样性最高,而在高温大曲中蛋白酶起主导作用[6]。
利用传统的微生物分离培养方法与分子生态学研究技术对包括酱香型白酒[7-10]、浓香型白酒[5,11-12]及清香型白酒[13-14]等不同香型的白酒大曲微生物群落结构及其多样性进行探究发现:不同香型的白酒大曲微生物种类与群落组成之间存在着显著的差异,由于微生物种群组成的多样性使得微生物来源的各类酶系也极为复杂,其中包括蛋白酶、淀粉酶等许多重要酶系。不同微生物来源的酶,由于其酶学特性的不同,在白酒酿造过程中所起的作用也不尽相同[15],这是不同白酒风味类型形成的重要影响因素之一[1]。相关研究指出在白酒酿造过程中,芽孢杆菌对酿酒酵母的生长几乎不造成影响,但可以通过提高芳香族化合物的含量以及大曲中酶的含量与活性进而促进白酒风味的形成[16-17]。
芝麻香型白酒是中国传统的白酒香型之一,是以芝麻香为主体,兼具浓、清、酱三种香型风味,这种香型的白酒也是以高粱、小麦、麸皮等为主要原料,经传统固态发酵、蒸馏、贮存、勾调而成,因酒中主体香气与焙炒芝麻的香气类似,故称为芝麻香型白酒[18]。高温制曲是芝麻香型白酒酿造的特点之一,也是芝麻香型白酒特殊风味形成的关键环节之一,对保障该香型白酒的生产与品质发挥着重要作用[19]。芝麻香白酒高温大曲的发酵温度一般保持在45 ℃以上,制曲过程中最高温度可达65 ℃,高温制曲不仅可以提供用于芝麻香型白酒发酵的各种微生物和酶,还可以形成包括呋喃、吡喃、吡嗪和醛类等在内的多种风味物质成分[10,20]。目前,研究发现与芝麻香型白酒酿造相关的芽孢杆菌属种类主要涉及枯草芽孢杆菌(Bacillus subtilis)、嗜热芽孢杆菌(Bacillus thermophilus)、腊状芽孢杆菌(Bacillus cereus)、地衣芽孢杆菌(Bacillus licheniformis)、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)等,其中以嗜热芽孢杆菌为主[21-22]。
菌株Q2B1是前期在芝麻香型白酒高温大曲微生物群落结构与多样性的研究中分离获得的,并发现以其为代表菌株的微生物类群是供试大曲样品的优势菌[23]。芝麻香主体香是在醛、醇、酸、酯达到一定比例后再辅以一些杂环化合物以及含硫化合物而形成的复合香[24],而这些风味物质的形成必须通过降解原料中蛋白质、淀粉等在内的多种大分子物质并经多步化学反应得到,因此芝麻香型白酒酿造过程中相关酶类物质的产生对于这种复合香的形成具有很重要的意义。本研究以菌株Q2B1为研究对象,利用分子生物学鉴定方法明确其菌种分类学地位,并对其产生蛋白酶和淀粉酶的能力与活性进行检测,还利用全基因组学测序与分析技术富集与探寻与该菌株产生蛋白酶和淀粉酶的能力相关的功能基因与代谢通路,以期在基因组水平揭示其产酶相关的分子机制。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
贝莱斯芽孢杆菌(Bacillus velezensis)Q2B1:本实验室在芝麻香型白酒大曲中分离并保存。
1.1.2 化学试剂
葡萄糖(分析纯)、3,5-二硝基水杨酸(dinitrosalicylic acid,DNS)(分析纯):北京化工厂;可溶性淀粉(生化试剂):北京西陇化工有限公司;脱脂奶粉:黑龙江省完达山乳业股份有限公司;聚合酶链式反应(polymerase chain reaction,PCR)Mix、脱氧核糖核酸(deoxyribonucleic acid,DNA)Marker:北京全式金生物技术公司;细菌基因组提取试剂盒:天根生化科技(北京)有限公司;PCR引物由北京诺赛生物公司合成。
1.1.3 培养基
LB液体固培养基:北京陆桥技术股份有限公司。
1.2 仪器与设备
FTC-3000P型实时荧光定量PCR仪:加拿大Funglyn Biotech公司;GelDocEZ型凝胶成像系统:美国Bio-Rad公司;DYY-8C型电泳仪:北京六一生物科技有限公司;BPH-9402精密恒温培养箱:上海一恒科学仪器有限公司;VS-840-1超净工作台:上海博迅实业有限公司;YX-280A手提式压力蒸汽灭菌器:合肥华泰集团有限公司。
1.3 实验方法
1.3.1 菌株Q2B1的分子生物学鉴定
利用细菌基因组提取试剂盒提取菌株Q2B1基因组DNA,并以提取获得的DNA为模板,采用16S rRNA基因扩增引物27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R(5'-GGTTACCTTGTTACGACTT-3')[25]以及gyrA基因扩增引物BS-gyrA-F(5'-CAGTCAGGAAATGCGTACGTCTT-3')和BS-gyrA-R(5'-CAAGGTAATGCTCCAGGCATTGCT-3')[26]。16S rRNA和gyrA基因扩增条件为94 ℃,5 min;94 ℃,1 min,55 ℃,1 min,72 ℃,1.5 min,30个循环;72 ℃,10 min。扩增产物经1.0%琼脂糖凝胶电泳检测后,由北京诺赛基因组研究中心有限公司完成测序。测序结果在EzBioCloud数据库[27]和GenBank数据库中进行比对分析,利用CLUSTAL_X v1.8对菌株Q2B1及其若干近缘种的16S rRNA和gyrA基因序列进行比对与长度均一化处理[28],并利用MEGA 5软件的邻接(neighbor-joining,N-J)法构建上述两个基因的系统发育树与进行系统发育分析[29]。
1.3.2 菌株Q2B1最适生长温度的测定
设置4 ℃、20 ℃、28 ℃、37 ℃、45 ℃、52 ℃、60 ℃、65 ℃共8个温度梯度,将菌株Q2B1接种于LB培养基平板中并分别置于不同温度梯度中培养48 h,观察最终各平板有无菌落产生及菌落大小来确定其最适的生长温度。
1.3.3 菌株Q2B1产淀粉酶与蛋白酶活性定性检测
将菌株接种在含1%淀粉LB琼脂培养基平板上,置于30 ℃培养48 h后,在培养基平板上滴加适量碘液,由于碘液可使添加可溶性淀粉的培养基变蓝,因此若菌落周围呈现透明圈,则表示该菌株产生淀粉酶并分解平板中的淀粉;将菌株接种在含1%的脱脂奶粉的LB琼脂培养基平板上,置于30 ℃培养48 h,由于培养基添加脱脂奶粉使平板含有大量的蛋白质,呈现乳白色,若菌落周围出现透明圈,则表示该菌株能够产生蛋白酶分解平板中脱脂奶粉的蛋白成分。
1.3.4 菌株Q2B1产淀粉酶与蛋白酶活性定量检测
葡萄糖标准曲线的绘制:配制葡萄糖溶液质量浓度分别为0.2mg/mL、0.4mg/mL、0.6mg/mL、0.8mg/mL、1.0mg/mL,在试管中精确加入0.5 mL标准葡萄糖糖溶液和1.5 mL DNS试剂,在沸水中煮沸15 min,之后冷却,并加入10.5 mL蒸馏水,充分摇匀后,在波长550 nm条件下测吸光度值A。以葡萄糖含量(x)为横坐标,以吸光度值(y)A550nm为纵坐标,绘制葡萄糖标准曲线,得到标准曲线回归方程为y=0.701 1x+0.100 7,相关系数R2为0.998 03。
菌株Q2B1淀粉酶活力的定量测定参照DNS法进行[30]:1 mL适当稀释的酶液加入9 mL的1%淀粉缓冲液(pH5.5)中,50 ℃恒温水浴中反应20 min,迅速吸取0.5 mL反应液加入1.5 mL DNS试剂中,终止反应,在波长520 nm条件下测定吸光度值。按照葡萄糖标准曲线回归方程计算生成还原糖(以葡萄糖计)含量。淀粉酶活力单位定义为1 mL粗酶液(或1 g固体发酵物)在pH 5.0、50 ℃的条件下,1 h水解1.0%淀粉液生成1 μmol葡萄糖为1个酶活力(U/mL)。菌株Q2B1蛋白酶活力按照GB/T 23527—2009《蛋白酶制剂》中的酸性蛋白酶检测方法进行定量测定,蛋白酶的活力单位定义为在一定pH值(pH 3.6或7.0)和40 ℃温度条件下,每分钟水解酪蛋白产生1 μg酪氨酸定义为一个蛋白酶活力单位[30](U/mL)。
1.3.5 菌株Q2B1的全基因组测序与生信分析
将基因组DNA经Covaris破碎仪随机打断成长度为350 bp的片段,经末端修复和加A尾后在片段两端分别连接接头制备DNA文库,文库构建完成后,先使用Qubit 2.0进行初步定量,随后使用Agilent 2100对文库的进行检测,符合预期后,使用定量实时聚合酶链反应(quantitative-real time-polymerase chain reaction,Q-RT-PCR)方法对文库的有效浓度(2 nmol/L)进行准确定量,以保证文库质量。库检合格,根据文库的有效浓度及数据产出需求进行Illumina HiSeq PE150测序。测序结果经过碱基质量分布和kmer分布分析等质量控制后,低质量的测序读长(low quality reads)被过滤掉;采用spade程序对质控后得到的可用测序数据进行基因组序列拼装[31],并对拼接后数据进行评价,最终选取>500 bp的scaffold序列进行后续分析。后续生信分析主要涉及非编码核糖核酸(ribonucleic acid,RNA)预测[32-33]、蛋白编码基因的预测[34]、蛋白编码基因的基因本体(gene ontology,GO)和直源同源群集(clusters of orthologous groups,COG)注释[35],以及蛋白编码基因的《京都基因和基因组百科全书》(kyoto encyclopedia of genes and genomes,KEGG)注释[36]等,并通过GO与KEGG注释富集与探寻与该菌株产生蛋白酶和淀粉酶的能力相关的功能基因与代谢通路。
2 结果与分析
2.1 菌株Q2B1的分子生物学鉴定结果
菌株Q2B1的16S rRNA和gyrA基因扩增产物全长分别为1 429 bp和949 bp,并将序列提交至GenBank,登录号分别为MK629006和MK642700。通过菌株Q2B1与其近缘菌种模式菌株的16S rRNA基因序列的比对分析显示,Q2B1与Bacillus siamensis KCTC 13613T的序列相似性最高,为99.79%;Q2B1与Bacillus属中16S rRNA基因序列相似度高于99.30%的其他近缘菌种模式菌株依次为贝莱斯芽孢杆菌(Bacillus velezensis)CR-502T(99.78%)、Bacillus subtilis subsp.stercoris D7XPN1T(99.68%)、枯草芽孢杆菌枯草亚种(Bacillus subtilis subsp.subtilis)NCIB 3610T(99.65%)、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)DSM 7T(99.51%)、中村芽孢杆菌(Bacillusnakamurai)NRRLB-41091T(99.51%)、特基拉芽孢杆菌(Bacillustequilensis)KCTC13622T(99.44%)、枯草芽孢杆菌沙漠亚种(Bacillus subtilis subsp.inaquosorum)KCTC 13429T(99.44%)和死亡谷芽孢杆菌(Bacillus vallismortis)DV1-F-3T(99.37%)。利用上述近缘菌种模式菌株并选择嗜盐杆菌(Halobacillushalophilus)DSM2266T(HE717023)作为外群,构建菌株Q2B1与模式菌株的16S rRNA和gyrA基因系统发育树,结果见图1和图2。

图1 基于16S rRNA序列菌株Q2B1的系统发育树
Fig.1 Phylogenetic tree of strain Q2B1 based on 16S rRNA sequences
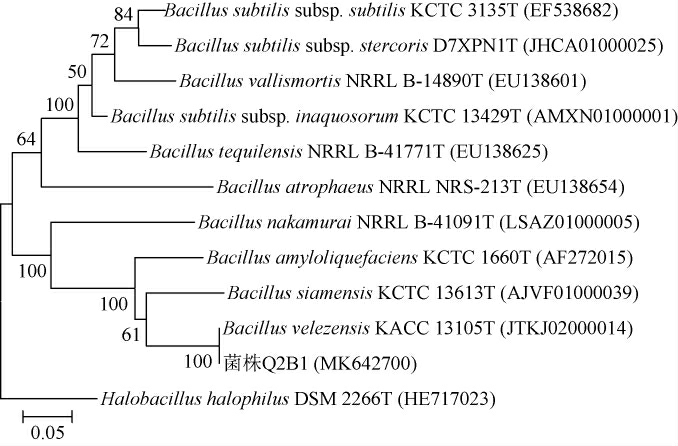
图2 基于gyrA基因菌株Q2B1的系统发育树
Fig.2 Phylogenetic tree of strain Q2B1 based on gyrA gene
由图1可知,16S rRNA基因系统发育分析确定菌株Q2B1归属为芽孢杆菌属(Bacillus);由图2可知,通过gyrA基因系统发育分析,结果显示菌株Q2B1与Bacillus velezensis CR-502T在系统发育树中聚在了一个单独的分支,可将其鉴定为贝莱斯芽孢杆菌(Bacillus velezensis)。
2.2 菌株Q2B1在不同温度条件下生长状况
菌株Q2B1接种于LB培养基平板,分别置于4 ℃、20 ℃、28 ℃、37 ℃、45 ℃、52 ℃、60 ℃、65 ℃共8个温度梯度条件下培养48 h后观察菌株菌体在培养基上生长情况,结果见表1。由表1可知,菌株Q2B1能够在试验设定的28~52 ℃温度范围生长,最适生长温度为45 ℃。结果表明,菌株Q2B1可以在芝麻香型白酒高温大曲制曲过程中在较宽的温度范围随制曲温度的逐步升高而得到富集与积累,但到达制曲温度的峰值(60~65 ℃)附近,菌体的生长受到了限制。
表1 不同温度条件下菌株Q2B1的生长状况
Table 1 Growth status of strain Q2B1 under different temperature conditions
注:“-”表示不生长;“+”表示生长弱;“++”表示生长状况良好;“+++”表示生长旺盛。
2.3 菌株Q2B1产淀粉酶与蛋白酶活性定性定量检测结果
菌株Q2B1接种于含1%的淀粉和脱脂奶粉的LB琼脂培养基平板,置于30 ℃培养48 h,结果见图3。由图3可知,菌株Q2B1能够在含有淀粉和脱脂奶粉的LB平板上生长良好,由于菌株在生长过程中产生能够分解蛋白质和可溶性淀粉的酶,因此明显观察到加入脱脂奶粉的平板上菌体周围产生了透明圈,加入可溶性淀粉的平板在滴加碘液后菌体周围形成明显的透明圈。菌株Q2B1具有产淀粉酶与蛋白酶的能力,其酶活力测定结果分别为5.852 U/mL和26.770 U/mL,这也说明这株菌很可能对芝麻香型白酒风味的形成产生。

图3 菌株Q2B1产淀粉酶(A)和蛋白酶(B)活性的效果
Fig.3 Effect of amylase (A) and protease (B) activity by strain Q2B1
2.5 菌株Q2B1的全基因组测序与分析结果
基于基因组测序结果的生物信息学分析结果见图4。由图4可知,菌株Q2B1基因组全长为3 475 602 bp,注释获得基因总数3 749个,GC含量为46.43%;rRNA与tRNA基因数目经过预测分别为10个和80个;通过蛋白编码基因的GO和COG注释,发现参与菌株代谢过程和催化活性,尤其是与能量产生与转换、碳水化合物转运和代谢与催化过程相关的基因数目明显较高。此外,蛋白编码基因的KEGG注释结果也能够反映出菌株Q2B1在生物合成与参与代谢等方面相关的代谢通路与反应网络相对集中,其中代谢通路中注释获得的与氨基酸和碳水化合物代谢相关的基因数量最多,分别为176和198个,这与该菌株具有产蛋白酶和淀粉酶活力的实验结果相符合。通过基因注释发现,在菌株Q2B1基因组中编码蛋白酶的基因包括多种肽酶(谷氨酰氨肽酶、甲硫氨酰氨肽酶、PfpI肽酶、Gpr肽酶、AprX肽酶、RseP肽酶、IspA肽酶、HtpX肽酶等)、转肽酶、信号肽酶、D-Ala-D-Ala羧肽酶、氨基甲酰磷酸合成酶、γ-聚谷氨酸水解酶、D-氨基肽酶、γ-谷氨酰转移酶、谷氨酸合成酶、胶原蛋白水解酶和氨基脱氧胆酸盐合成酶等编码基因;基因组中编码淀粉酶的基因包括α-淀粉酶和麦芽糖淀粉酶等编码基因。这些注释获得的编码基因及其相关的代谢通路能够在基因组水平揭示菌株Q2B1具有合成蛋白酶和淀粉酶能力的分子机制。

图4 菌株Q2B1的KEGG代谢通路注释图
Fig.4 Annotated map of KEGG metabolic pathway of strain Q2B1
3 结论
本研究在前期关于芝麻香型白酒高温大曲微生物群落结构与多样性的研究基础上,以分离自供试大曲样品的绝对优势菌代表菌株Q2B1为研究对象,通过分子鉴定技术将菌株Q2B1鉴定为贝莱斯芽孢杆菌(Bacillus velezensis),并通过平板实验检测到该菌具有产生蛋白酶和淀粉酶的活性,其淀粉酶与蛋白酶活力分别为5.852 U/mL和26.770 U/mL;同时利用全基因组学测序与分析技术富集与探寻得到其产生蛋白酶和淀粉酶的能力相关的功能基因与部分代谢通路,菌株Q2B1基因组全长为3 475 602 bp,通过基因注释发现在菌株Q2B1基因组中存在肽酶、转肽酶、羧肽酶、氨基甲酰磷酸合成酶等蛋白酶编码基因,以及α-淀粉酶和麦芽糖淀粉酶等淀粉酶编码基因,实现在菌株Q2B1基因组水平揭示其产酶相关的分子机制。
[1]LIU H L,SUN B G.Effect of fermentation processing on the flavor of Baijiu[J].J Agr Food Chem,2018,66(22):5425-5432.
[2]DU H,WANG X S,ZHANG Y H,et al.Exploring the impacts of raw materials and environments on the microbiota in Chinese Daqu starter[J].Int J Food Microbiol,2019,297:32-40.
[3]YI Z L,JIN Y L,XIAO Y,et al.Unraveling the contribution of high temperature stage to jiang-flavor Daqu,a liquor starter for production of Chinese Jiang-flavor Baijiu,with special reference to metatranscriptomics[J].Front Mic,2019,10:472.
[4]WU Z Y,ZHANG W X,ZHANG Q S,et al.Developing new sacchariferous starters for liquor production based on functional strains isolated from the pits of several famous Luzhou-flavor liquor brewers[J]. J I Brewing,2012,115(2):111-115.
[5]HE G Q,HUANG J,ZHOU R Q,et al.Effect of fortified Daqu on the microbial community and flavor in Chinese Strong-flavor liquor brewing process[J].Front Mic,2019,10:56.
[6]LIU J J,CHEN J Y,FAN Y,et al.Biochemical characterization and dominance of different hydrolases in different types of Daqu-a Chinese industrial fermentation starter[J].J Sci Food Agr,2018,98(1):113-121.
[7]WU Q,CHEN L Q,XU Y.Yeast community associated with the solid state fermentation of traditional Chinese Maotai-flavor liquor[J].Int J Food Microbiol,2013,166(2):323-330.
[8]WANG Q,ZHANG H X,LIU X.Microbial community composition associated with Maotai liquor fermentation[J].J Food Sci,2016,81(6):M1485-M1494.
[9]WANG W Y,LIU R L,SHEN Y,et al.The potential correlation between bacterial sporulation and the characteristic flavor of Chinese Maotai liquor[J].Front Mic,2018,9:1435.
[10]WANG M Y,YANG J G,ZHAO Q S,et al.Research progress on flavor compounds and microorganisms of Maotai flavor Baijiu[J].J Food Sci,2019,84(1):6-18.
[11]ZOU W,ZHAO C Q,LUO H B.Diversity and function of microbial community in Chinese Strong-flavor Baijiu ecosystem:A review[J].Front Mic,2018,9:671.
[12]WANG X S,DU H,XU Y.Source tracking of prokaryotic communities in fermented grain of Chinese Strong-flavor liquor[J].Int J Food Microbiol,2017,244:27-35.
[13]LI X R,MA E,YAN L Z,et al.Bacterial and fungal diversity in the traditionalChineseliquorfermentationprocess[J].Int J Food Microbiol,2011,146(1):31-37.
[14]LI R Y,ZHENG X W,ZHANG X,et al.Characterization of bacteria and yeasts isolated from traditional fermentation starter(Fen-Daqu)through a 1H NMR-based metabolomics approach[J].Front Mic,2018,76:11-20.
[15]申孟林,张超,王玉霞,等.白酒大曲微生物酶系研究进展[J].中国酿造,2017,37(4):7-11.
[16]MENG X,WU Q,WANG L,et al.Improving flavor metabolism of Saccharomyces cerevisiae by mixed culture with Bacillus licheniformis for Chinese Maotai-flavor liquor making[J].J Ind Microbiol Biot,2015,42(12):1601-1608.
[17]WANG P,WU Q,JIANG X J,et al.Bacillus licheniformis affects the microbial community and metabolic profile in the spontaneous fermentation of Daqu starter for Chinese liquor making[J].Int J Food Microbiol,2017,250:59-67.
[18]WU J H,HUO J Y,HUANG M Q,et al.Structural characterization of atetrapeptide from sesame flavor-type Baijiu and its preventive effects against AAPH-induced oxidative stress in HepG2 cells[J]. J Agr Food Chem,2017,65(48):10495-10504.
[19]YAO S,LIU Y,LI H,et al.Bacterial communities during the process of high-temperature Daqu production of roasted sesame-like flavour liquor[J].J I Brewing,2015,121(3):440-448.
[20]ZHANG C L,AO Z H,CHUI W Q,et al.Characterization of the aroma-active compounds in Daqu:A tradition Chinese liquor starter[J].Eur Food Res Technol,2012,234(1):69-76.
[21]李环宇.芝麻香型白酒工艺探究[D].济南:山东轻工业学院,2010.
[22]WU Q,LING J,XU Y.Starter culture selection for making Chinese sesame-flavored liquor based on microbial metabolic activity in mixedculture fermentation[J]. Appl Environ Microbiol,2014,80(14):4450-4459.
[23]刘晓昆,陈文浩,刘洋.芝麻香型白酒高温大曲制曲过程中产酶细菌的分离与鉴定[J].食品与发酵工业,2019,45(17):54-59.
[24]高传强.芝麻香型白酒风味物质及其生物活性研究[D].武汉:湖北工业大学,2017.
[25]STACKEBRANDT E,GOODFELLOW M.Nucleic acid techniques in bacterial systematic[M].New York:John Wiley and Sons,1991:115-175.
[26]QIU Z C,LU X M,LI N Y,et al.Characterization of garlic endophytes isolated from the black garlic processing[J]. Microbiologyopen,2018,7(1):doi:10.1002/mbo3.547.
[27]CHUN J,LEE J H,JUNG Y,et al.EzTaxon:a web-based tool for the identification of prokaryotes based on 16S ribosomal RNA gene sequences[J].Int J Syst Evol Micr,2007,57(10):2259-2261.
[28]THOMPSON J D,GISON T J,PLEWNIAK F,et al.The CLUSTAL X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res,1997,25:4876-4882.
[29]TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Mol Biol Evol,2011,28(10):2731-2739.
[30]周景祥,王桂芹,余涛.蛋白酶和淀粉酶活性检测方法探讨[J].中国饲料,2011(11):23-24.
[31]BANKEVICH A,NURK S,ANTIPOV D,et al.SPAdes:a new genome assembly algorithm and its applications to single-cell sequencing[J]. J Comput Biol,2012,19(5):455-477.
[32]LASLETT D,CANBACK B.ARAGORN,a program to detect tRNA genes and tmRNA genes in nucleotide sequences[J].Nucleic Acids Res,2004,32(1):11-16.
[33]LAGESEN K,HALLIN P,RØDLAND E A,et al.RNAmmer:consistent andrapidannotationofribosomalRNAgenes[J].Nucleic Acids Res,2007,35(9):3100-3108.
[34]SEEMANN T.Prokka:rapid prokaryotic genome annotation[J].Bioinformatics,2014:30(14):2068-2069.
[35]FINN R D,BATEMAN A,CLEMENTS J,et al.Pfam protein families database[J].Nucleic Acids Res,2004,42(Database issue):D222-D230.
[36]MORIYA Y,ITON M,OKUDA S,et al.KAAS:an automatic genome annotation and pathwayreconstruction server[J].Nucleic Acids Res,2007,35(suppl 2):W182-W185.