白酒发酵是由多种微生物共同参与,在开放性的环境中进行的一个复杂过程[1]。窖泥的质量可直接决定酒质的优劣,归根到底对白酒发酵过程起决定性作用的是窖泥微生物[2]。微生物以空间位置相对固定的窖泥作为栖息地[3],且经过酿酒环境长时间的驯化,已经形成了一个较为稳定的微生物群落,并形成了一批具有产酒、产香功能的微生物,合理开发利用这些微生物,对白酒品质的提升有重大意义[4]。
目前关于窖泥微生物菌落结构的研究方法主要有聚合酶链式反应(polymerase chain reaction,PCR)克隆文库技术、PCR-变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)技术、聚合酶链式反应单链构象多态性(PCR-single strand conformation polymorphism,PCR-SSCP)分析、荧光原位杂交(fluorescence in situ hybridization,FISH)技术、磷脂脂肪酸(phosphor lipid fatty acid,PLFA)指纹图谱技术和高通量测序(high throughput sequencing,HTS)技术。其中高通量测序技术,又称“下一代”测序(next generation sequencing,NGS)技术,能够深度测序,是新一代测序技术。与其他方法相比,具有规模更大,通量更高,耗时越短等优势,可以快捷方便的读取样品中复杂的微生物结构,是分析复杂多菌种样品的首选方法,已被广泛应用于动植物科学、医学、土壤学等领域[5]。目前,关于采用高通量测序技术研究窖泥微生物群落的研究已有报道,王莉等[6]采用高通量测序技术对酱香型窖池窖底微生物进行了研究,发现随着窖底泥的不断驯化,乳杆菌科(Lactobacillaceae)、梭菌科(Clostridiaceae)和瘤胃菌科(Ruminococcaceae)等厌氧微生物逐渐成为主要优势种群;雷振河[7]采用高通量测序技术对清香型大曲和酒醅中的微生物进行了测定,对比二者在微生物群落结构上的差异,得出大曲中优势原核微生物和真核微生物的种类;邓杰等[8-9]利用高通量测序技术研究不同窖龄的泸州老窖窖泥中细菌及古菌群落的分布情况;刘延波等[10]利用高通量测序技术分析浓香型白酒中温曲和高温曲的细菌群落结构,并从中温曲中首次检测到小球菌属(Pediococcus)、泛菌属(Pantoea)、迈勒吉尔霉菌属(Melghirimyces)等微生物,丰富了大曲中的微生物种类;吕锡斌等[11]运用高通量测序技术分析了酱香型白酒下糙沙轮次微生物多样性,确定了下糙沙的主要微生物种类,反映了不同阶段微生物群落特点。
本研究采用高通量测序技术对浓香型白酒窖泥的微生物群落进行全面的研究,分析浓香型白酒窖泥细菌的主要构成,并通过主成分分析(principal component analysis,PCA)对窖泥细菌的影响进行评价,有助于更好的了解浓香型窖泥微生物工作机理,为建立浓香型白酒窖泥微生物信息数据库、提升浓香型白酒质量提供理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
窖泥样品:取自川南某白酒厂3个同期建造车间中的9口窖池,且窖池连续生产10年以上,取样部位为每口窖底中心部位,取得窖泥样品后将其编号为1~9,其中1~3、4~6、8~9为分别同一车间不同窖池窖泥。所取样品用塑封袋密封后,迅速置于低温取样盒中冷藏运回,并在-20 ℃条件下低温冷冻保存。
1.1.2 试剂
PowerSoil 脱氧核糖核酸(deoxyribonucleic acid,DNA)Isolation Kit:美国MOBIO公司;Gel Extraction Kit:北京康为世纪生物科技有限公司;Quant-iT PicoGreen DNA Kit:美国nvitrogen公司;无水乙醇(分析纯):成都市科龙化工试剂厂;Buffer、MgCl2、Ex Taq酶(5 U/μL)、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs)、DNA Marker:日本Takara公司;引物:上海生工生物有限公司;高通量测序试剂:美国Roche公司。
1.2 仪器与设备
DK-S28恒温水浴锅:上海一恒科学仪器有限公司;SORVALLStratos高速冷冻离心机、Thermo 900超低温冰箱:美国Thermo Scientific公司;TGL-16G台式高速离心机:上海实验仪器厂有限公司;Minisubcell水平电泳仪、GelDoc 2000凝胶成像系统、MyCycler PCR仪:美国Bio-Rad公司;D-160均质机:美国Scilogex公司;GS junior高通量测序仪:美国Roche公司;Cary Eclipse荧光分光光度计:安捷伦科技(中国)有限公司。
1.3 方法
1.3.1 窖泥总DNA提取
采用PowerSoil DNA Isolation Kit提取窖泥微生物总DNA,采用1%的琼脂糖凝胶电泳检测其完整性,DNA样品于-20 ℃保存[12]。
1.3.2 PCR扩增
以提取的窖泥微生物总DNA为模板,采用正向引物968FMID1~9和反向引物1401RMID1进行PCR扩增。PCR扩增体系:10×Buffer 2.5 μL,25 mmol/L dNTP 2 μL,MgCl22μL,10μmol/L正反向引物各0.5μL,5U/μLExTaq酶0.5μL,DNA模板0.3 μL,双蒸水(ddH2O)16.7 μL。PCR扩增条件:94 ℃预变性3 min;94 ℃变性15 s,56 ℃退火30 s,72 ℃延伸90 s,共30个循环;72 ℃再延伸10 min。PCR扩增产物经1%琼脂糖凝胶检测,检测合格后送去测序,测序时通过正向引物上的数字标签区分不同样品。
1.3.3 PCR扩增产物纯化
采用Gel Extraction Kit对PCR扩增产物进行回收、纯化以除去引物二聚体,采用1%琼脂糖凝胶电泳进行检测。采用Quant-iT PicoGreen Kit对纯化后的PCR扩增产物进行定量[13]。
1.3.4 高通量测序
以定量好的PCR扩增产物为模板进行微乳滴PCR(emulsion PCR,emPCR),emPCR扩增体系:Mol.Bio.Grade Water 410μL,Additive515μL,AmpMix270μL,AmpPrimer80μL,Enzyme Mix 70 μL,Ppiase 2 μL。emPCR扩增程序:94 ℃预变性4 min;94 ℃变性30 s,58 ℃退火4.5 min,68 ℃延伸30 s,共50个循环,10 ℃保温。采用高通量测序技术对emPCR扩增产物进行测序。
1.3.5 数据处理
通过Cope V1.2.3.3软件进行序列拼接及过滤后利用Mothur软件以平均邻近聚类算法在97%的相似度水平下对窖泥样品中细菌的操作分类单位(operational taxonomic unit,OTU)进行划分[14]。利用SPSS 20.0软件对窖泥样品的细菌属进行主成分分析(PCA)。
2 结果与分析
2.1 测序数据长度及序列有效性分析
以9个窖泥样品的DNA为模板,采用带有特殊标签的引物进行PCR扩增,PCR扩增产物经纯化后,以其为模板进行emPCR扩增,emPCR扩增产物碱基长度在450 bp左右。采用高通量测序技术对emPCR扩增产物进行测序,通过标签信息区分不同样品。本次测序共获得298 405条序列,序列碱基平均长度为321 bp,序列碱基长度分布见图1。由图1可知,大部分序列的碱基长度集中在450 bp左右,序列碱基长度与实验设计碱基长度基本一致。经过Cope V1.2.3.3软件筛选过滤掉低质量序列和重复序列,最终获得有效序列66 322条,有效序列的碱基长度在450 bp左右。
2.2 窖泥中细菌门的分类
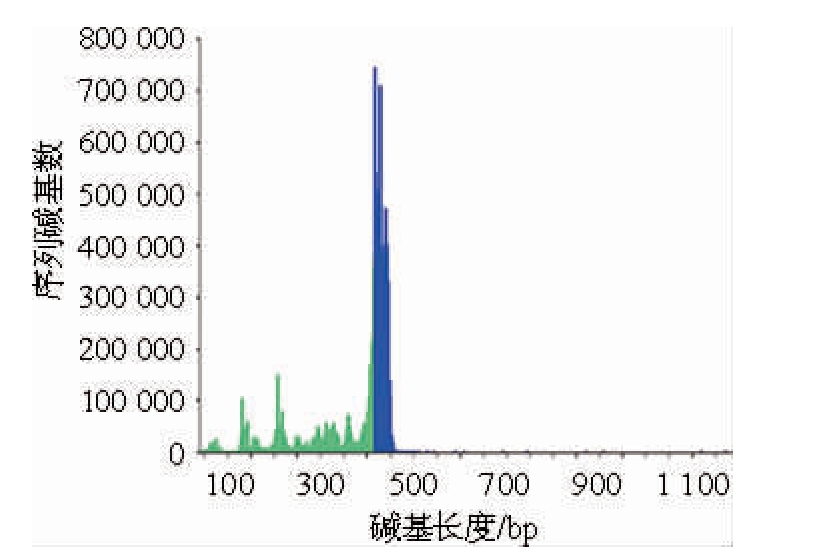
图1 测序序列碱基长度的分布
Fig.1 Distribution of base length of sequencing sequences
9个窖泥样品共获得2 050个可操作分类单元,其中1、2、3号窖泥样品的OTU数分别是249、297、492个;4、5、6号窖泥样品的OTU数分别是195、61、305个;7、8、9号窖泥样品的OTU数分别是178、63、210个。采用Mothur软件对以上数据分析处理,获得不同细菌门分类,结果见图2。
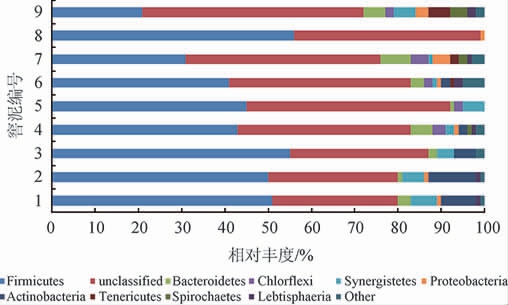
图2 不同窖泥样品中细菌群落门的分类
Fig.2 Classification of bacterial community in different pit mud samples based on phylum
由图2可知,9个窖泥样品中共划分出9个门,分别是厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、绿弯菌门(Chlorflexi)、互养菌门(Synergistetes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、无壁菌门(Tenericutes)、螺旋体门(Spirochaetes)和球孢菌门(Lebtisphaeria)。9个门中既有厌氧菌又有好氧菌,也有兼性厌氧菌,说明白酒发酵并不是处于单一严格厌氧的环境中。在每个窖泥样品的细菌群落中,Firmicutes占主导地位,其占30%~55%;该类菌具有细胞壁结构,并且大多数能够产生内生孢子抵抗极端环境。Bacteroidetes占1%~7%,其中窖泥样品4和9的Bacteroidetes数量较为接近,Bacteroidetes的大多数细菌寄生在动物肠道内,有部分属于病原菌。在窖泥样品1、2、3中未发现有Chlorflexi的存在,其他窖泥样品中,Chlorflexi占2%~4%。Synergistetes有降解氨基酸能力,部分为专性厌氧,且能耐60 ℃的高温;Synergistetes在不同窖泥样品都有分布,占比为1%~6%。Proteobacteria是细菌中最大的一个门,大多数具有固氮能力,兼性或专性厌氧,也包括很多病原菌;窖泥样品中Proteobacteria占1%~4%。Actinobacteria大多数为好氧菌,少部分厌氧,可以产生抗生素;窖泥样品7、8、9中未发现有Actinobacteria的存在,且在其他不同窖泥样品中所占比例差异较大,为2%~11%。Tenericutes和Spirochaetes在不同窖泥样品中所占比例基本相同,分别占1%~5%和1%~4%。Lebtisphaeria在不同窖泥样品中的占比较为一致,都在1%左右。
2.3 窖泥中细菌属的分类
在窖泥样品中细菌门分类的基础上,进一步对窖泥样品中的细菌属进行分类,结果见图3。
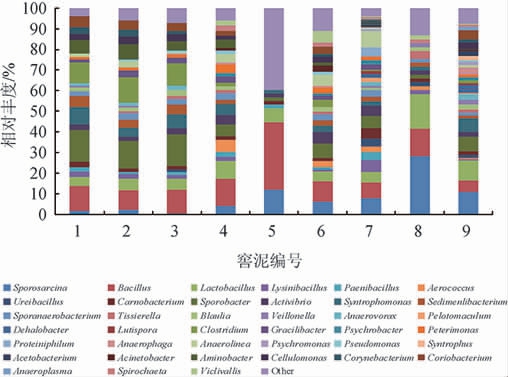
图3 不同窖泥样品中细菌群落属的分类
Fig.3 Classification of bacterial community in different pit mud samples based on of genus
由图3可知,9个窖泥样品中共获得39个细菌属,各个细菌属在不同窖泥中的分布有以下特点:八叠球菌属(Sporobacter)在窖泥样品1、2、3中占15%左右,而在其他窖泥中所占比例为6%左右;Sporobacter能在厌氧条件下代谢产生有机酸和芳香族化合物[15-16]。芽孢杆菌属(Bacillus)在所有窖泥样品中都存在,其中在5号窖泥中占比最高达到32.76%。红蝽杆菌属(Coriobacterium)是一种厌氧菌[17],在窖泥样品1、2、3中占5%左右,在其他窖泥样品中占2%~3%。梭菌属(Clostridium)在窖泥样品1、2、3中所占比例较高,约为12%;其次是窖泥样品4和6,约占3%;窖泥样品7、8、9中最少,只占1%;梭菌属(Clostridium)是一类严格厌氧菌,能够代谢产生有机酸和醇[18]。纤维素单胞菌属(Cellu-lomonas)在窖泥样品1、2、3中约占4%,在其他窖泥中约占2%左右;纤维素单胞菌能够产生纤维素酶[19],能够提高酿酒原料的利用率。氨基杆菌属(Aminobacter)在1、2、3号窖泥中占比最高,约占6%,4、6号窖泥中约占3%,7、8、9号窖泥中最少,不到1%,氨基杆菌属能够在厌氧条件下降解氨基酸[20]。棒杆菌属(Corynebacterium)在1、2、3号窖泥中约占3%,在其他窖泥中占不到1%。芽孢八叠球菌属(Sporosarcina)在1、2、3号窖泥中所占比最少,占2%左右,4、6号窖泥中占6%左右,8、9号窖泥中所占比最高约10%。相同车间窖泥中芽孢八叠球菌属(Sporobacter)、Coriobacterium、Clostridium、Cellulomonas、Sediminibacter、Aminobacter、Corynebacterium、Sporosarcina所占比例基本相同。脱卤素杆菌属(Dehalobacter)、醋杆菌属(Acetobacter)、互营菌属(Syntrophus)、冷单胞菌属(Psychromonas)只在7、8、9号窖泥中发现有一定比例的分布,而在其他窖泥中均未检测出。
2.4 窖泥中细菌主成分分析
对9个窖泥样品中的39个细菌属进行主成分分析,结果见表1。由表1可知,成分1、成分2、成分3、成分4的特征值>5,分别为10.414、10.094、7.634、5.446,累积贡献达到83.97%。张文彤[21]在《IBM SPSS数据分析与挖掘实战案例精粹》中说方差贡献率达到50%就可以酌情接受。为了统计方便,本研究选取方差贡献率最大的前两个主成分(累计方差贡献率达到51.27%)进行讨论分析。因此按照成分1和成分2对窖泥中的细菌属进行分析,结果见图4。细菌属的主成分载荷值矩阵见表2。
表1 细菌属的主成分特征值
Table 1 Principal components eigenvalues of bacteria
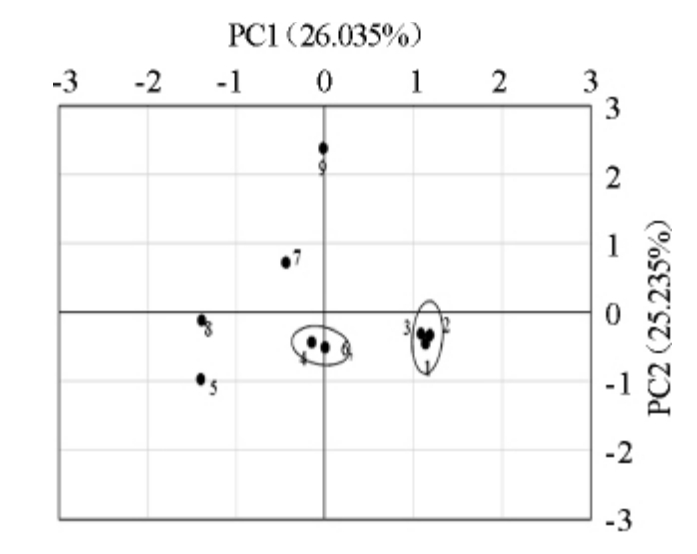
图4 按主成分1和主成分2划分的窖泥样品分布图
Fig.4 Distribution map of pit mud samples according to PC1 and PC2
由图4可知,窖泥样品1、2、3,4、6,7、9分别聚为一组,基本按不同车间窖泥进行划分,窖泥样品5、8聚为一组,分析原因可能是测序序列都较少,微生物覆盖率较低的缘故。
表2 细菌属的主成分载荷值矩阵
Table 2 Principal components load matrix of bacteria

由表2可知,主成分1中,载荷值较高的细菌属为Sporobacter、Coriobacterium、Clostridium、Cellulomonas、Sediminibacter、Aminobacter、Corynebacterium、Sporosarcina,载荷值分别为0.974、0.924、0.919、0.913、0.856、0.855、0.814、-0.808。其中,Sporosarcina对主成分1产生负向影响,其余为正向影响。主成分2中,载荷值较高的细菌属为厌氧原体属(Anaeroplasma)、Dehalobacter、Acetobacter、Syntrophus、Psychromonas,载 荷值分别为0.970、0.970、0.970、0.970、0.881,都为正向影响。
3 结论
本实验主要通过高通量测序法分析了浓香型白酒不同窖泥样品中的细菌群落结构。通过高通量测序共获得66 322条有效序列,2 050个OTU。其中窖泥样品1、2、3平均获得351个OTU,窖泥样品4、5、6平均获得214个OTU,窖泥样品7、8、9平均获得153个OTU。根据OTU,9个不同窖泥样品中共分为9个门类,其中窖泥样品1、2、3中共有6个门类,窖泥样品4、5、6中共有9个门类,窖泥样品7、8、9中共有8个门类。9个门类分别为厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、绿弯菌门(Chlorflexi)、互养菌门(Synergistetes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、柔壁菌门(Tenericutes)、螺旋体门(Spirochaetes)和Lebtisphaeria,其中Firmicutes占主导地位。在细菌门分离的基础上,9个窖泥样品中共获得39个细菌属。通过主成分分析(PCA)发现,其中13个细菌属对窖泥来源划分的贡献度较高。相同车间窖泥中Sporobacter、Coriobacterium、Clostridium、Cellulomonas、Sediminibacter、Aminobacterium、Corynebacterium、Sporosarcina所占基本相同。Dehalobacter、Acetobacter、Syntrophus、Psychromonas只在7、8、9号窖泥中发现有一定比例的分布,而在其他窖泥中均未检测出,说明相同生产环境的窖泥微生物群落结构基本是一致的。
[1]张宿义.泸型酒技艺大全[M].北京:中国轻工业出版社,2011:18-32.
[2]王艳丽,孟雅静,何宏魁,等.不同品质浓香型窖泥理化因子与细菌总量的相关性[J].食品与发酵工业,2019,45(3):89-94.
[3]程伟,吴丽华,徐亚磊,等.浓香型白酒酿造微生物研究进展[J].中国酿造,2014,33(3):1-4.
[4]张文学,乔宗伟,向文良,等.中国浓香型白酒窖池微生态研究进展[J].酿酒,2004(2):34-38.
[5]MIKKELSEN M,ROCKENBAUER E,W?CHTER A.Application of full mitochondrial genome sequencing using 454 GS FLX pyrosequencing[J].Foren Sci Int Genet Sup,2009,2(1):518-519.
[6]王莉,王亚玉,王和玉,等.酱香型白酒窖底泥微生物组成分析[J].酿酒科技,2015(1):12-15.
[7]雷振河.采用高通量测序技术分析清香型白酒酿造微生物[J].食品与发酵工业,2015(9):164-167.
[8]邓杰,黄治国,卫春会,等.基于高通量测序的浓香型白酒窖池细菌群落结构分析[J].现代食品科技,2015,31(7):57-62.
[9]邓杰,卫春会,罗惠波,等.基于高通量测序的浓香型白酒窖池古菌群落结构分析[J].现代食品科技,2015,31(8):213-218.
[10]刘延波,赵志军,陈黄曌,等.高通量测序技术分析浓香型白酒中温曲和高温曲的细菌群落结构[J].现代食品科技,2018,34(5):229-235.
[11]吕锡斌,吴耀领,郝飞,等.高通量测序技术分析酱香型白酒下糙沙轮次微生物多样性的研究[J].酿酒科技,2019(2):85-95.
[12]ZHANG W X,QIAO Z W,SHIGEMATSU T,et al.Analysis of the bacterial community in Zaopei during production of Chinese Luzhou-flavor liquor[J].J Inst Brew,2005,111(2):215-222.
[13]SCHEIRLINCK I,VAN DE MEULEN R,VAN SCHOOR A,et al.Influence of geographical origin and flour type on diversity of lactic acid bacteria in traditional Belgian sourdoughs[J].Appl Environ Microbiol,2007,73(19):6262-6239.
[14]王勇,周森,魏金旺.基于高通量测序技术分析牛栏山大曲微生物多样性[J].中国酿造,2019,38(2):58-61.
[15]JEONG H,LIM Y H,SEKIGUCHI Y,et al. Anaerosporobacter mobilis gen.nov.,sp.nov.,isolated from forest soil[J].Int J Syst Evol Micr,2007,57(8):1784-1787.
[16]GRECHMORA I,BKC F M,OLLIVIER B,et al.Isolation and characterization of Sporobacter termitidis gen.nov.,sp.nov.,from the Digestive Tract of the Wood-Feeding Termite Nasutitermes lujae[J].Int J Syst Bacteriol,1996,46(2):512-518.
[17]HAAS F,KÖNIG H.Coriobacterium glomverans gen.nov.,sp.nov.from the intestinal tract of the red soldier bug[J].Int J Syst Bacteriol,1988,38(4):382-384.
[18]张善亭,史燕,张淑丽,等.丁酸梭菌的研究应用进展[J].生物技术通报,2013(9):27-33.
[19]郭爱莲,杨琳,刘梅,等.产黄纤维单胞菌纤维素酶的培养条件[J].西北大学学报,1999(6):575-577.
[20]BAENA S,FARDEAU M L,LABAT M,et al. Aminobacterium mobile sp.nov.,a new anaerobic amino-acid-degrading bacterium[J].Int J Syst Evol Micr,2000,50(1):259-264.
[21]张文彤,钟云飞.IBM SPSS 数据分析与挖掘实战案例精粹[M].北京:清华大学出版社,2013:98-120.