海藻糖(trehalose)是由两分子葡萄糖以α-1,1-糖苷键连接而成的非还原性双糖,广泛存在于细菌、酵母、丝状真菌、植物、昆虫、无脊椎动物等生物体体内[1-2]。海藻糖化学性质稳定,具有非还原性、保湿性、热酸稳定性、抗冻结、干燥性等物化性质,对生物体及生物大分子有很好的非特异性保护作用,因此,海藻糖被广泛应用于食品加工业、医药业、农业、生化制品业和化妆品产业中[3-5]。在食品工业中,海藻糖作为食品添加剂能够防止淀粉老化、蛋白质变性和脂类物质的氧化变质;保持食品原有的色泽、风味;也可用于食品、水果、蔬菜的保鲜[6-9]。因此,海藻糖的市场需求量很大。
海藻糖在生物体内的合成途径主要有3种[10]:6-磷酸海藻糖合成酶(trehalose-6-phosphate synthase,TPS)/6-磷酸海藻糖磷酸酯酶(trehalose-6-phosphate phosphatase,TPP)途径,在自然界中分布最为广泛,在真核生物、真细菌和古生菌中都有发现[11-12];麦芽寡糖基海藻糖合成酶(maltooligosyl trehalose synthase,MTSase)/麦芽寡糖基海藻糖水解酶(maltooligosyl trehalose trehalohydrolase,MTHase)途径,主要存在于细菌和古生菌中[13-14];海藻糖合酶(trehalose synthase,TreS)途径[15-16]。与其他海藻糖合成途径相比,TreS途径具有一定的优势,只需要一种酶一步反应就能够获得海藻糖,而且其底物为更加廉价的麦芽糖。
目前,国内外研究者已从分支杆菌(Mycobacterium smegmatis)[17]、红球菌(Rhodococcus opacus)[15]、嗜热放线菌(Thermobifida fusca)[16]等不同的微生物中克隆、表达了海藻糖合酶基因,并研究了重组海藻糖合酶的性质[18-20]。同时,探究其蛋白结构及催化机理[21-23]。但由于海藻糖合酶催化双向反应,不能完全将麦芽糖转化成海藻糖,因此,寻找转化率高的海藻糖合酶具有十分重要的意义。
本实验室前期已从云南昆明磷矿粉中分离得到一株可以产生海藻糖的昆明假单胞菌(Pseudomonas kunmingensis)HL22-2[24],本研究以其为研究对象,采用热不对称交错聚合酶链式反应(thermal asymmetric interlaced-polymerase chain reaction,Tail-PCR)从该菌株中克隆海藻糖合酶基因HL22-2TreS,将该基因与表达载体pETM3C连接形成重组载体,在大肠杆菌(Escherichia coli)BL21(DE3)pLysS中进行异源表达,通过Ni-NTA柱纯化重组酶HL22-2TreS,并对其酶学性质进行分析,为工业利用其将廉价的麦芽糖转化成附加值更高的海藻糖提供理论基础。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株及质粒
昆明假单胞菌(Pseudomonas kunmingensis)HL22-2(CGMCC 1.12273,DSM 25974):本实验室分离保藏;Escherichia coli DH5α:宝生物工程(大连)有限公司;Escherichia coli BL21(DE3)pLysS:北京康为世纪生物科技有限公司;pGEM-T载体:美国Promega公司;pETM3C载体:美国Invitrogen公司。
1.1.2 主要试剂
限制性内切酶BamH I(10 U/μL)、Not I(20 U/μL)、T4脱氧核糖核酸(deoxyribonucleic acid,DNA)连接酶(5 U/μL):美国ThermoFisherScientific公司;LATaq酶(5U/μL)、Genome Walking Kit:日本TaKaRa公司;质粒小量抽提试剂盒、DNA快速纯化/回收试剂盒:北京鼎国昌盛生物技术有限公司;细菌基因组DNA抽提试剂盒:美国Axygen公司;麦芽糖和海藻糖(纯度≥99%):德国Merk公司;异丙基硫代-β-D-半乳糖苷(isopropyl-β-D-thiogalactoside,IPTG):上海百赛生物技术有限公司;胰蛋白胨、酵母提取物(均为生化试剂):英国Oxoid公司。
1.1.3 引物
本研究中所用的引物及其序列见表1,引物由软件Primer Premier 5.0设计,由北京六合华大基因科技有限公司合成。
表1 本研究中所用的引物
Table 1 Primers used in this study

注:标有下划线的碱基序列为酶切位点。
1.1.4 培养基
LB培养基:酵母提取物5 g,NaCl 10 g,蛋白胨10 g,蒸馏水1 L,pH 7.2,121 ℃高压蒸气灭菌20 min。
优化肉汤(superoptimalbroth,SOB)培养基:蛋白胨20g,酵母提取物5 g,NaCl 0.5 g,KCl 0.2 g,MgCl21 g,蒸馏水1 L,pH自然,121 ℃高压下蒸汽灭菌20 min。
分解抑制型优化肉汤(super optimal broth with catabolite repression,SOC)培养基:在SOB培养基中加入用0.22 μm滤膜过滤除菌后的1 mol/L葡萄糖20 mL,混匀,pH 7.0。
1.2 仪器与设备
ST8R台式高速冷冻离心机:赛默飞世尔科技(中国)有限公司;T6新世纪紫外可见分光光度计:北京普析通用仪器有限责任公司;GF254硅胶板:青岛海洋化工有限公司;T-Gradient&ThermoblockO型PCR扩增仪:德国Biometra公司;DYY-6C核酸电泳仪:北京六一仪器厂;Agilent1200高效液相色谱(high performance liquid chromatography,HPLC)仪:美国Agilent公司;JYD-650超声细胞破碎仪:上海之信仪器有限公司;Ni-NTA柱:安诺伦(北京)生物科技有限公司。
1.3 实验方法
1.3.1 昆明假单胞菌HL22-2海藻糖合酶基因的克隆
采用Tail-PCR克隆菌株海藻糖合酶基因,具体方法如下:
(1)根据昆明假单胞菌的海藻糖合酶的两段保守序列,设计引物TreS-MF/MR。采用细菌基因组提取试剂盒提取昆明假单胞菌HL22-2的基因组DNA,以其为模板进行PCR扩增。PCR扩增体系:LA Taq(5 U/μL)聚合酶0.5 μL,基因组DNA模板0.5 μL,引物TreS-MF和TreS-MR各0.6 μL,脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphates,dNTPs)0.6 μL,10×buffer 3 μL,超纯水24.2 μL。PCR扩增条件:94 ℃预变性4 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸35 s,共30个循环;72 ℃再延伸10 min。
(2)通过PCR扩增获得一段DNA片段,测序。根据获得的序列设计引物PU-1、PU-2、PU-3和PD-1、PD-2、PD-3,用Genome Walking Kit进行Tail-PCR,获得DNA片段两端的序列。
(3)根据序列分析,获得昆明假单胞菌HL22-2海藻糖合酶基因全长序列,设计引物TreS-F/R,以基因组DNA为模板,PCR扩增得到昆明假单胞菌HL22-2海藻糖合酶基因HL22-2TreS。PCR扩增体系:LA Taq聚合酶0.2 μL,基因组DNA模板0.5μL,引物TreS-F和TreS-R各0.6μL,dNTPs0.6μL,2×GC buffer 15 μL,超纯水12.5 μL。PCR扩增条件:94 ℃预变性4 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1.5 min,共30个循环;72 ℃再延伸10 min。
将PCR扩增产物纯化,与载体pGEM-T连接,转化至Escherichia coli DH5α感受态细胞中,挑选转化子进行菌落PCR,挑取阳性转化子进行测序,获得昆明假单胞菌HL22-2海藻糖合酶基因HL22-2TreS序列信息。采用SignalP4.1server(http://www.cbs.dtu.dk/services/SignalP/)预测信号肽序列,采用Clustal X 2.0对海藻糖合酶的氨基酸序列进行比对[25]。
1.3.2 昆明假单胞菌HL22-2海藻糖合酶基因HL22-2TreS的表达
分别将纯化的昆明假单胞菌HL22-2海藻糖合酶基因HL22-2TreS、载体pETM3C用BamH I和Not I进行双酶切,回收目的片段、T4 DNA连接酶连接,转化至Escherichia coli DH5α感受态细胞中,挑选转化子进行菌落PCR,挑取阳性转化子进行测序。将正确的重组质粒转化至Escherichia coli BL21(DE3)pLysS感受态细胞中,挑选转化子进行菌落PCR,采用终浓度20%(V/V)的甘油保藏阳性转化子。
种子培养:将阳性转化子接入装有5 mL LB培养基(含有100 μg/mL氨苄青霉素和34 μg/mL氯霉素)的20 mL试管中,于37 ℃、220 r/min条件下过夜培养。
诱导培养:将过夜培养好的菌株按1%(V/V)的接种量转接到装有100 mL LB培养基(含有100 μg/mL氨苄青霉素和34 μg/mL氯霉素)的250 mL三角瓶中,于37 ℃、220 r/min条件下振荡培养。待菌液OD600nm值为0.6时,将三角瓶置于冰上10 min左右,加入终浓度为300 nmol/L的IPTG,迅速将其转至16 ℃、220 r/min条件下进行低温诱导培养12 h,离心,收集菌体。将菌体重悬在20 mmol/L磷酸钠缓冲液中(pH 7.4,含有终浓度500 mmol/L NaCl和20 mmol/L咪唑),超声破碎细胞、离心,上清液即为粗酶液[26]。
1.3.3 昆明假单胞菌HL22-2海藻糖合酶的纯化
用20 mmoL/L磷酸钠缓冲液(pH 7.4,含有终浓度500mmol/LNaCl和20mmol/L咪唑)平衡Ni-NTA柱,10mL粗酶液加入到平衡好的镍柱,静置1h,然后分别用含50mmol/L、90 mmol/L、200 mmol/L、500 mmol/L咪唑的磷酸钠缓冲液(pH 7.4)分步洗脱,每一步的洗脱组分都测定酶活力,并进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate polyacrylamide gel electrophoresis,SDS-PAGE)分析,最后将有酶活力的单一条带的组分合并,即为纯化的酶液[27]。采用Bradford法测定酶蛋白的浓度[28]。
1.3.4 海藻糖合酶活力的测定
在80 μL用20 mmol/L磷酸钠缓冲液(pH 7.4)配制的150 mmol/L的麦芽糖溶液中加入20 μL不同稀释的酶液,混匀,37 ℃水浴30 min,100 ℃煮沸10 min。采用HPLC定量检测海藻糖及麦芽糖的含量[18]。HPLC条件:色谱柱为ZORBAX NH2(4.6 mm×250 mm,5 μm),流动相为乙腈∶去离子水=75∶25(V/V),检测器为示差检测器,流速为1.0 mL/min。采用不同浓度的标准海藻糖绘制标准曲线,然后根据标准曲线回归方程计算样品中海藻糖和麦芽糖的转化率,进而得出海藻糖合酶活力。
海藻糖合酶酶活力定义:在37 ℃、pH 7.4条件下,每分钟生成1 μmol海藻糖所需的酶量定义为1个酶活单位(U)。
1.3.5 海藻糖合酶对底物的转化
在80 μL用20 mmol/L磷酸钠缓冲液(pH 7.4)配制的150 mmol/L的麦芽糖溶液或海藻糖溶液中加入20 μL酶液,混匀,37 ℃水浴保温30 min,100 ℃煮沸10 min。取反应液1 μL进行薄层层析(thin layer chromatography,TLC)[18]。层析板为TLC Silica gel 60 F254 20 cm×20 cm,层析液为n-正丁醇∶吡啶∶水(4∶6∶1,V/V)。
1.3.6 海藻糖合酶的酶学性质
最适反应温度:在80 μL用20 mmol/L磷酸钠缓冲液(pH 7.4)配制的150 mmol/L的麦芽糖溶液中加入20 μL酶液,混匀,分别在20 ℃、30 ℃、40 ℃、50 ℃、60 ℃、70 ℃、80 ℃条件下保温30 min,然后将离心管在100 ℃煮沸10 min以终止反应,测定海藻糖合酶活力,并以最高酶活力为100%,计算相对酶活。温度稳定性:先分别将海藻糖合酶在20 ℃、30 ℃、40℃、50℃、60℃、70℃、80℃条件下保温30 min,然后在37 ℃条件下测定海藻糖合酶活性,并以最高酶活力为100%,计算相对酶活。
最适反应pH值:用不同pH值的缓冲液(甘氨酸-盐酸缓冲液(pH 2.0、pH 3.0),乙酸-乙酸钠缓冲液(pH 4.0、pH 5.0),磷酸氢二钠-磷酸二氢钠缓冲液(pH 6.0、pH 7.0、pH 8.0),甘氨酸-氢氧化钠缓冲液(pH 9.0、pH 10.0))溶解麦芽糖,在80 μL 150 mmoL/L的麦芽糖溶液中加入20 μL分别用不同pH值缓冲液稀释的酶液,混匀,在37 ℃条件下测定海藻糖合酶活力。并以最高酶活力为100%,计算相对酶活。pH值稳定性:分别将海藻糖合酶用不同pH的缓冲液稀释,37 ℃保温1 h,然后取20 μL处理后的酶液加入80 μL用20 mmol/L磷酸钠缓冲液(pH 7.4)配制的150 mmol/L的麦芽糖溶液中,混匀,在37 ℃条件下测定海藻糖合酶活力,并以最高酶活力为100%,计算相对酶活。
金属离子对海藻糖合酶活力的影响:先将纯化的3 U海藻糖合酶和用20 mmol/L磷酸钠缓冲液(pH 7.4)溶解的不同金属离子(1 mmol/L和10 mmol/L)在37℃条件下保温30 min,以未处理的藻糖合酶为对照,在37 ℃条件下检测海藻糖合酶活力,并采用SPSS22.0软件对数据进行统计学分析。
Km值的测定:参照文献[29]测定麦芽糖和海藻糖的Km值。
2 结果与分析
2.1 昆明假单胞菌HL22-2海藻糖合酶基因HL22-2TreS的克隆和序列分析
以昆明假单胞菌HL22-2基因组DNA为模板,以TreSMF/MR为引物,克隆出一个碱基长度为654 bp的DNA片段,测序。根据获得的序列设计引物PU-1、PU-2、PU-3和PD-1、PD-2、PD-3,采用Genome Walking Kit进行Tail-PCR,获得DNA片段两端的序列。根据序列分析,获得昆明假单胞菌HL22-2海藻糖合酶基因HL22-2TreS全长序列,设计引物TreS-F/R,以基因组DNA为模板,PCR扩增得到昆明假单胞菌HL22-2海藻糖合酶基因HL22-2TreS,结果见图1。

图1 昆明假单胞菌HL22-2海藻糖合酶基因HL22-2TreS
Fig.1 Trehalose synthase gene HL22-2TreS of Pseudomonas kunmingensis HL22-2
由图1可知,HL22-2TreS基因全长为3 336 bp(GenBank accession number:MF543128),编码1 111个氨基酸,其翻译的蛋白序列中不含信号肽,预测蛋白质分子质量为126 kDa。
将HL22-2TreS的氨基酸序列与GenBank数据库中的海藻糖合酶序列进行BLAST比对发现,HL22-2TreS的氨基酸序列与假单胞菌(Pseudomonas sp.)来源的海藻糖合酶的氨基酸序列高度相似(与来自于Pseudomonas sp.K35的海藻糖合酶(GenBank:OCX97659.1)的氨基酸相似度达99%),但这些海藻糖合酶的氨基酸序列都是由基因组测序后通过基因注释得出来的,对其功能并没有加以验证。将4个已成功表达并得到功能验证的不同微生物来源的海藻糖合酶的氨基酸序列与HL22-2TreS编码的氨基酸序列进行多重序列比对,结果见图2。
由图2可知,HL22-2TreS与4个不同微生物来源的海藻糖合酶的氨基端序列具有极高的相似度,而羧基端的相似度不高,说明HL22-2TreS确为海藻糖合酶家族成员。

图2 HL22-2TreS与不同微生物来源的海藻糖合酶氨基酸序列同源性比对结果
Fig.2 Results of homology comparison of amino acid sequences between Hl22-2TreS and trehalose synthase from different microorganisms
PkTS为Pseudomonas kunmingsis HL22的TreS;BPTS为类鼻疽伯克氏菌(Burkholderia pseudomallei)Bp1651的TreS(Genbank:ALJ69836.1);AcTS为无色杆菌属(Achromobacter sp.)2789STDY5608621的TreS(Genbank:CUI35560.1);PsTS为Pseudomonas sp.P8005的TreS(Genbank:AFK94626.2);TaTS为水生栖热菌(Thermus aquaticus)ATCC33923的TreS(Genbank:O06458.1)。矩形方框表示已知的TreS保守区;阴影部分表示绝对保守的残基。
2.2 HL22-2TreS基因的表达和重组酶HL22-2TreS的纯化
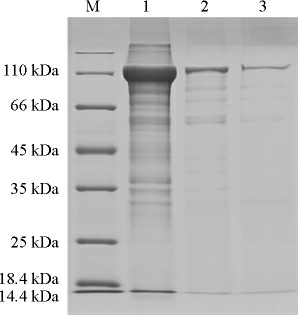
图3 重组酶HL22-2TreS的SDS-PAGE结果
Fig.3 Results of SDS-PAGE of recombinase HL22-2TreS
M为蛋白Marker;1为上清液;2为50 mmol/L咪唑洗脱液;3为90 mmol/L咪唑洗脱液。
将昆明假单胞菌HL22-2菌株的海藻糖合酶基因HL22-2TreS连接到pETM3C质粒上,转化到E.coli BL21(DE3)pLysS菌株中进行诱导表达。通过超声破碎细胞,上清液中海藻糖合酶的比活力为12.8 U/mg,说明HL22-2TreS被成功表达。上清液通过Ni-NTA纯化后,进行SDS-PAGE电泳分析,结果见图3。
由图3可知,经过12 h诱导表达,菌体裂解液的主要蛋白为HL22-2TreS,经Ni-NTA柱纯化后,得到一个分子质量约为126 kDa的条带,与理论分子质量的计算结果相一致,纯化后海藻糖合酶的比活力为38.5 U/mg。
2.3 重组酶HL22-2TreS对不同底物的转化能力
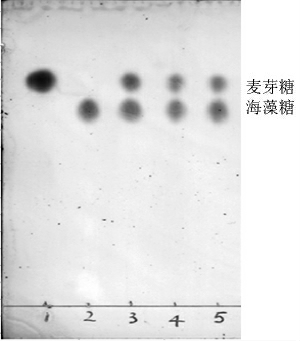
图4 HL22-2TreS对不同底物转化后产物的薄层层析结果
Fig.4 Thin layer chromatography results of reaction products from different substrates by HL22-2TreS
1为麦芽糖标准品;2为海藻糖标准品;3为麦芽糖和海藻糖混合标准品;4为以海藻糖为底物的反应液;5为以麦芽糖为底物的反应液。
由图4可知,HL22-2TreS既能将麦芽糖转化成海藻糖,又能将海藻糖转化成麦芽糖。HPLC定量检测海藻糖和麦芽糖的吸收峰,并通过标准曲线计算得出重组酶HL22-2TreS对麦芽糖和海藻糖的转化率分别为73.2%和52.5%。
2.4 重组酶HL22-2TreS的酶学性质
2.4.1 温度对重组酶HL22-2TreS活力的影响
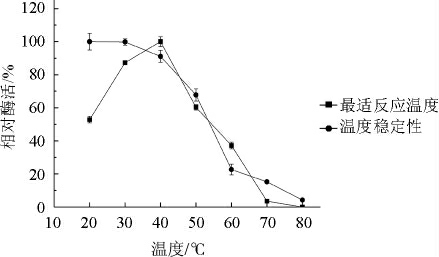
图5 重组酶HL22-TreS的最适反应温度及温度稳定性
Fig.5 Optimum reaction temperature and temperature stability of recombinase HL22-2TreS
由图5可知,当反应温度为40 ℃时,重组酶HL22-2TreS的酶活力最高;当反应温度>70 ℃之后,酶活力基本消失。当重组酶HL22-2TreS在40 ℃以下保温30 min后,酶活力比较稳定,相对酶活力保持在90%以上,在50 ℃保温30 min后,相对酶活力为67.8%,但是,当在高于60 ℃以上温度保温30 min之后,酶活力快速下降。结果表明,重组酶HL22-2TreS的最适反应温度为40 ℃,在20~50 ℃条件下比较稳定。
2.4.2 pH值对重组酶HL22-2TreS活力的影响
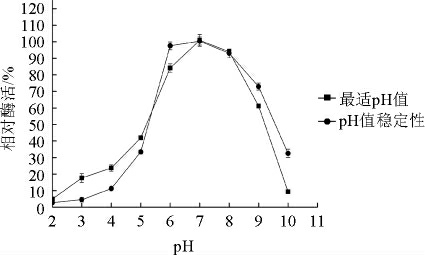
图6 重组酶HL22-TreS的最适pH值及pH值稳定性
Fig.6 Optimum reaction pH and pH stability of recombinase HL22-2TreS
由图6可知,重组酶HL22-2TreS的最适反应pH值为7.0,在pH 6.0~9.0比较稳定。
2.4.3 金属离子对重组酶HL22-2TreS活力的影响
表2 金属离子对重组酶HL22-2TreS酶活力的影响
Table 2 Effect of metal ions on the activity of recombinase HL22-2TreS
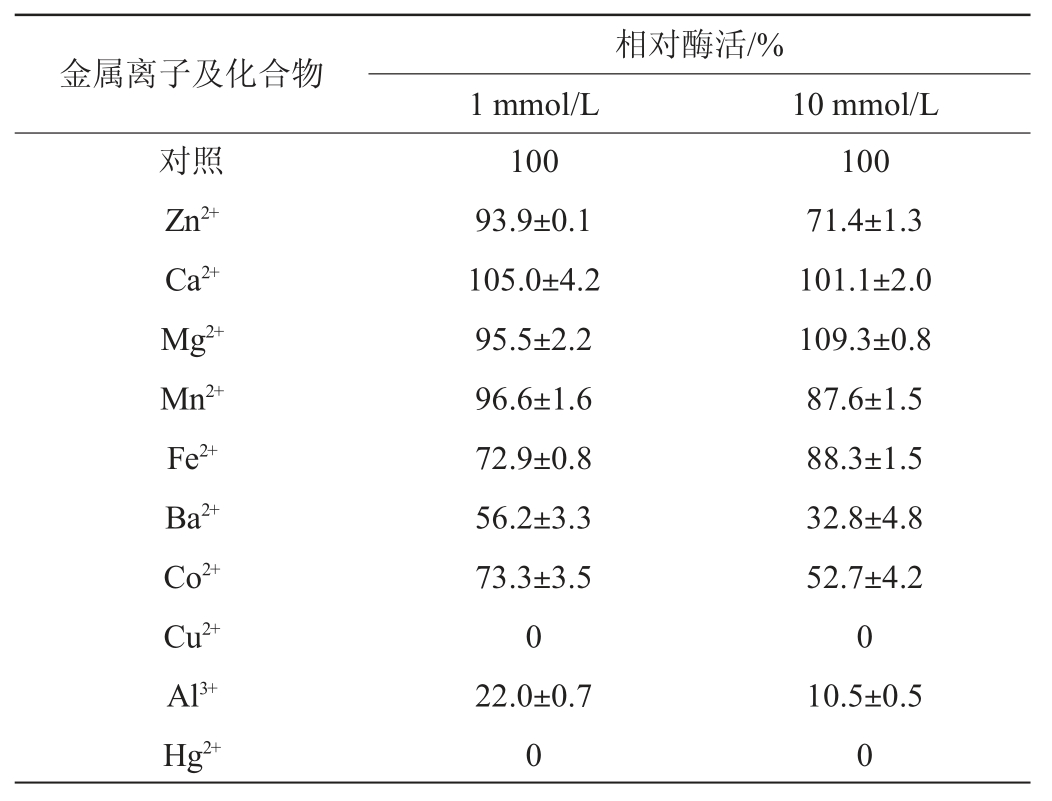
由表2可知,Cu2+、Hg2+、Ba2+及Al3+对海藻糖合酶的活力有强烈的抑制效果,其中Cu2+、Hg2+对酶活力完全抑制;Fe2+、Co2+对海藻糖合酶活力有部分抑制效果,随着离子浓度的升高,Co2+对酶活力的抑制效果更加明显,而Fe2+对酶活力的抑制效果减弱;其他金属离子对酶的活力影响不明显。
2.4.4 重组酶HL22-2TreS对麦芽糖和海藻糖的Km值
HL22-2TreS对麦芽糖和海藻糖的Km值分别为20.6mmol/L和87.5 mmol/L,说明HL22-2TreS对麦芽糖具有更高的亲和性,更容易将麦芽糖转化成海藻糖,使反应更容易朝着合成海藻糖的方向进行。
3 结论
通过Tail-PCR从云南昆明磷矿来源的昆明假单胞菌(Pseudomonas kunmingensis)HL22-2中克隆得到海藻糖合酶基因HL22-2TreS,其基因全长为3 336 bp,编码1 111个氨基酸,氨基酸序列与Genbank数据库中相关的海藻糖合酶序列具有极高的相似性。将该基因与表达载体pETM3C连接后在大肠杆菌(Escherichia coli)BL21(DE3)pLysS中进行异源表达,并通过Ni-NTA柱纯化得到分子质量约126 kDa的海藻糖合酶HL22-2TreS,其最适反应温度和pH值分别为40 ℃和7.0,在温度20~50 ℃及pH值6.0~9.0条件下比较稳定;Cu2+,Hg2+,Ba2+及Al3+对海藻糖合酶的活力有强烈的抑制效果。HL22-2TreS催化的反应是双向进行的,其对麦芽糖和海藻糖的米氏常数(Km)分别为20.6 mmol/L和87.5 mmol/L,对麦芽糖具有更高的亲和性,更容易将麦芽糖转化成海藻糖。因此,利用昆明假单胞菌HL22-2的海藻糖合酶可将廉价的麦芽糖转化成具有更高附加值的海藻糖,具有很高的工业应用潜力。
[1]MISEON P,MITCHELL W J,FATEMEH R.Effect of trehalose and trehalose transport on the tolerance of Clostridium perfringens to environmental stress in a wild type strain and its fluoroquinolone-resistant mutant[J].Int J Microbiol,2016,2016:4829716.
[2]PIWOWAREK K,LIPIN′ SKA E,HAC-SZYMAN′ CZUK E,et al. Propionibacterium spp.-source of propionic acid,vitamin B12,and other metabolites important for the industry[J].Appl Microbiol Biot,2018,102(2):515-538.
[3]FEOFILOBA E P,USOV A L,MYSYAKINA I S.Trehalose:chemical structure,biological functions,and practical application[J].Microbiology,2014,83(3):184-194.
[4]徐天生.海藻糖的性能和应用概述与其在牙膏中的应用研究[J].口腔护理用品工业,2016,26(2):8-12.
[5]靳文斌,李克文,胥九兵,等.海藻糖的特性、功能及应用[J].精细与专用化学品,2015,23(1):30-33.
[6]王羽,云雪艳,张晓燕,等.海藻糖对蛋白质的抗逆保护及其在食品领域中的应用[J].食品科技,2015,40(10):229-232.
[7]宋树鑫,王羽,梁敏,等.海藻糖对冷鲜猪肉的保鲜优势探究[J].食品科技,2016,41(7):260-265.
[8]陈岳祥,赵文革,高军龙.海藻糖在传统山核桃仁制品中的应用探究[J].农村经济与科技,2018,29(6):40-41.
[9]RICHARDS A B,KRAKOWKA S,DEXTER L B,et al.Trehalose:a review of properties,history of use and human tolerance,and results of multiple safety studies[J].Food Chem Toxicol,2002,40(7):871-898.
[10]刘德海,权淑静,解复红,等.海藻糖合成酶产生菌筛选、鉴定及其产酶特性[J].中国酿造,2016,35(9):95-100.
[11]VAN VAECK C,WERA S,VAN DIJCK P,et al.Analysis and modification of trehalose 6-phosphate levels in the yeast Saccharomyces cerevisiae with the use of Bacillus subtilis phosphotrehalase[J].Biochem J,2001,353(1):157-162.
[12]LI Y,ZHANG H,SHENG H,et al.Cloning,expression and characterization of trehalose-6-phosphate phosphatase from a psychrotrophic bacterium,Arthrobacter strain A3[J].World J Microb Biot,2012,28(8):2713-2721.
[13]曲茂华,张凤英,何名芳,等.海藻糖生物合成及应用研究进展[J].食品工业科技,2014,35(16):358-362.
[14]NAKADA T,MARUTA K,TSUSAKI K,et al.Purification and properties of a novel enzyme,maltooligosyl trehalose synthase,from Arthrobacter sp.Q36[J].Biosci Biotech Biochem,1995,59(12):2210-2218.
[15]YAN J,QIAO Y,HU J,et al.Cloning,expression and characterization of a trehalose synthase gene from Rhodococcus opacus[J].Protein J,2013,32(3):223-229.
[16]WEI Y T,ZHU Q X,LUO Z F,et al.Cloning,expression and identification of a new trehalose synthase gene from Thermobifida fusca genome[J].Acta Biochim Biophys Sinica,2004,36(7):477-484.
[17]PAN Y T,KOROTH EDAVANA V,JOURDIAN W J,et al.Trehalose synthase of Mycobacterium smegmatis:purification,cloning,expression,properties of the enzyme[J].Eur J Biochem,2004,271(21):4259-4269.
[18]GAO Y,XI Y,LU X L,et al.Cloning,expression and functional characterization of a novel trehalose synthase from marine Pseudomonas sp.P8005[J].World J Microb Biot,2013,29(11):2195-2206.
[19]PANEK A,PIETROW O,FILIPKOWSKI P,et al.Effects of the polyhistidine tag on kinetics and other properties of trehalose synthase from Deinococcus geothermalis[J].Acta Biochim Pol,2013,60(2):163-166.
[20]ZHU Y,WEI D,ZHANG J,et al.Overexpression and characterization of a thermostable trehalose synthase from Meiothermus ruber[J].Extremophiles,2010,14(1):1-8.
[21]MIAH F,KOLIWER-BRANDL H,REJZEK M,et al.Flux through trehalose synthase flows from trehalose to the alpha anomer of maltose in Mycobacteria[J].Chem Biol,2013,20(4):487-493.
[22]ZHANG R,PAN Y T,HE S,et al.Mechanistic analysis of trehalose synthase from Mycobacterium smegmatis[J].J Biol Chem,2011,286(41):35601-35609.
[23]王一雯,马焕,权淑静,等.海藻糖合酶的结构和催化机制研究进展[J].中国酿造,2018,37(3):14-18.
[24]XIE F,MA H,QUAN S,et al. Pseudomonas kunmingensis sp.nov.,an exopolysaccharide-producing bacterium isolated from a phosphate mine[J].Int J Syst Evol Micr,2014,64(Pt2):559-564.
[25]LARKIN M A,BLACKSHIELDS G,BROWN N P,et al.Clustal W and Clustal X version 2.0[J].Bioinformatics,2007,23(21):2947-2948.
[26]张靖坤.海藻糖合酶在毕赤酵母中的外源表达[D].北京:北京化工大学,2016.
[27]LAEMMLI U K.Cleavage of structural protein during the assembly of the head of bacteriophage T4[J].Nature,1970,227(5259):680-685.
[28]BRADFORD M M.A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J].Anal Biochem,1976,72(1-2):248-254.
[29]XIE F H,QUAN S J,LIU D H,et al.Purification and characterization of a novel α-amylase from a newly isolated Bacillus methylotrophicus strain P11-2[J].Process Biochem,2014,49(1):47-53.