生物胺是一类含氮的低分子质量碱性有机化合物的总称,普遍存在于中国传统发酵食品中,过量摄入会引起不良反应。在发酵食品中,具有氨基酸脱酸酶活性的微生物对生物胺的形成至关重要[1-2]。常见的产生物胺的微生物有肠杆菌属(Enterobacter)、肠球菌属(Enterococcus)、乳酸杆菌属(Lactobacillus)、明串珠菌属(Leuconostoc)、链球菌属(Streptococcus)、酒球菌属(Oenococcus)、梭菌属(Clostridium)和假单胞菌属(Pseudomonas)等,其中乳酸菌是产生物胺的主要菌种[3-4]。而发酵肉制品中生物胺的产生主要与微球菌属(Micrococcus)和葡萄球菌属(Staphylococcus)有关,其中肉葡萄球菌(Staphylococcus carnosus)和鱼发酵葡萄球菌(Staphylococcus piscifermentans)具有高氨基酸脱羧酶活性,能够产生尸胺、2-苯乙胺、腐胺和组胺[5-7]。MARTUSCELLI M等[6]从香肠中分离得到51株木糖葡萄球菌(Staphylococcus xylosus),发现其中的21株有体外氨基酸脱羧酶活性,只有7株菌株的酪胺、亚精胺和精胺产量>10 mg/kg,未检测到组胺的产生[6]。
目前,发酵食品中生物胺含量主要通过高效液相色谱(highperformanceliquidchromatography,HPLC)法进行检测,检测方法的前处理较为繁琐,耗时长。近年来有学者利用分子生物学的方法检测微生物中产生物胺的相关酶的基因判断其是否产生某种特定的生物胺。此方法具有快速、可靠、不依赖于培养等优点,并可在生物胺形成之前,检测出产生物胺的基因,因此可分析出潜在的产生物胺危害[8-11]。不同种类的发酵肉制品中生物胺种类和含量有一定差别[12-13]。其中,酪胺、尸胺和组胺报道较多。酪胺是由酪氨酸脱羧酶(tyrosine decarboxylase,TDC)催化酪氨酸脱羧形成的;尸胺是由赖氨酸在赖氨酸脱羧酶(lysine decarboxylase,IDC)的催化下脱羧形成的;组胺是毒性最大的生物胺,由组氨酸脱羧酶(histidine decarboxylase,HDC)催化组氨酸脱羧形成的。
本研究以云南腊肠为研究对象,首先,采用高通量测序技术研究发酵肉制品中细菌群落结构的多样性。然后利用传统培养分离法及双层显色培养基对发酵肉制品中产生物胺的细菌进行分离筛选,并基于16S rRNA序列进行鉴定。最后,利用聚合酶链式反应(polymerase chain reaction,PCR)扩增与测序的方法对细菌中产组胺、酪胺和尸胺的相关基因进行特异性筛选,以期建立发酵肉制品中生物胺的快速筛选机制,为基因检测技术在发酵食品安全性检测中的应用奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
云南腊肠:云南市场购买。
1.1.2 培养基
MRS液体培养基:蛋白胨10.0 g,牛肉膏10.0 g,葡萄糖20.0 g,酵母浸粉5.0 g,柠檬酸氢二铵2.0 g,无水乙酸钠5.0 g,磷酸氢二钾2.0 g,吐温80 1.0 mL,MgSO4·7H2O 0.58 g,MnSO4·4H2O 0.2 g,蒸馏水1 000 mL,调节pH值至6.80。121 ℃高压灭菌15 min。
MRS固体培养基:在MRS液体培养基的基础上添加20.0 g琼脂。121 ℃高压灭菌15 min。
双层显色下层培养基[14]:蛋白胨5.0 g,酵母浸粉5.0 g,牛肉膏5.0 g,NaCl 2.5 g,葡萄糖0.5 g,吐温80 1.0 mL,MgSO40.4 g,MnSO40.03 g,K2HPO42.0 g,柠檬酸三铵2.0 g,CaCO30.1 g,FeSO40.04 g,维生素B1(vitamin B1,VB1)0.01 g,磷酸吡哆醛0.05 g,琼脂20.0 g,色氨酸、精氨酸、赖氨酸、酪氨酸、苯丙氨酸、组氨酸各5.0 g,蒸馏水1 000 mL,pH值5.2。双层显色上层培养基:溴甲酚紫0.06 g,琼脂20.0 g,蒸馏水1 000 mL,pH值5.2。121 ℃高压灭菌15 min,以不加氨基酸的双层显色培养基为对照。
1.1.3 引物
PCR引物见表1[10,15-17],由北京美吉桑格生物医药科技有限公司合成。
表1 本研究所用引物序列
Table 1 Primer sequences used in the study

1.1.4 化学试剂
脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒、KOD高保真酶、引物、DNA Marker:上海生工生物工程有限公司;Axy Prep DNA凝胶回收试剂盒:德国AXYGEN公司;Quant-iTPicoGreen荧光定量试剂盒:美国Life Technologies公司;TruSeq DNA建库试剂盒、MiSeq上机试剂盒:美国Illumina公司。FastPfu Polymerase(5.0 U)、EX Taq酶(5.0 U)、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)、5×FastPfu buffer、10×EX Taq Buffer:日本TaKaRa公司。
1.2 仪器与设备
ZDX35BI灭菌锅:上海申安医疗器械厂;DNP-9082电热恒温培养箱:上海精宏实验设备有限公司;H1650-W医用离心机:湖南湘仪离心机仪器有限公司;SW-CJ-2D超净工作台:苏州净化设备有限公司;MG96+型PCR仪:杭州郎基科学仪器有限公司;JY04S-3C凝胶成像仪、JY300C电泳系统:北京君意电泳设备有限公司;HH-2恒温水浴锅:上海比郎仪器有限公司;625-00203730XL测序仪:美国ABI公司;810R台式高速冷冻离心机:德国Eppendorf公司;SpectraMax M5酶标仪:美国MolecularDevice公司;Miseq基因组测序仪:美国Illumina 公司;QuantiFluorTM-ST蓝色荧光定量系统:美国Promega公司。
1.3 实验方法
1.3.1 发酵肉制品中细菌群落结构分析
(1)PCR扩增细菌16S rRNA V3-V4区序列
采用DNA提取试剂盒提取腊肠的总DNA,以其为模板,采用上游引物338F和下游引物806RPCR扩增细菌16SrRNA的V3-V4区序列。PCR扩增体系:5×FastPfu buffer 4.0 μL,2.5 mmol/L dNTPs Mix 2.0 μL,上下游引物(5 μmol/L)各0.8 μL,FastPfu Polymerase 0.4 μL,BSA 0.2 μL,模板DNA 10.0 ng,加双蒸水(ddH2O)补至终体积为20 μL。PCR扩增条件:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,共27个循环;72 ℃再延伸10 min。使用Axy Prep DNA凝胶回收试剂盒切胶回收PCR扩增产物,采用2%琼脂糖凝胶进行电泳检测。
(2)文库构建和高通量测序
根据电泳结果,对PCR扩增产物进行定量分析,采用TruSeq DNA建库试剂盒构建文库。采用Miseq平台双端PE 300测序策略,使用上机试剂盒将文库加入Miseq测序仪进行测序。
(3)生物信息学分析和数据分析
对原始数据进行过滤拼接处理后,得到优化序列。在97%的相似水平下,利用vsesion 7.1 Usearch(http://drive5.com/uparse/)软件平台,划分可操作分类单元(operational taxonomicunit,OTU),并挑取OTU的代表序列。采用RDPclassifier贝叶斯算法,设置置信度阈值为0.7,基于Silva数据库(http://www.arb-silva.de)对OTU代表序列进行分类学分析,并在属水平上统计样品的细菌群落组成。
1.3.2 发酵肉制品中产生物胺细菌的筛选与鉴定
(1)细菌的分离纯化
取腊肠1.0 g于9.0 mL无菌生理盐水中,充分振荡,用无菌生理盐水梯度稀释(10-1、10-2、10-3、10-4、10-5、10-6、10-7),并涂布于MRS固体培养基上,37 ℃条件下培养48 h。根据菌落形态、大小、颜色等挑取单菌落,-20 ℃保藏。
(2)产生物胺细菌的筛选
将分离纯化的菌株接种到双层显色下层培养基上,37℃条件下培养48 h,无菌条件下,在下层培养基上倒一层上层培养基(50 ℃),显色,并在5 min内记录实验结果。以不接种菌株的培养基作为空白对照,显紫色的为阳性(产生物胺菌),不变色即黄色为阴性(不产生物胺菌)[18-19]。
(3)产生物胺细菌的鉴定
将产生物胺菌株接种到MRS液体培养基中,37 ℃条件下培养12 h,取1 mL菌液,10 000 r/min离心1 min,弃上清,取菌体沉淀,参照DNA提取试剂盒的步骤提取基因组DNA,1%琼脂糖凝胶电泳检测,于-20 ℃条件下保存。以提取的DNA为模板,采用引物27F及1492R进行PCR扩增。PCR扩增体系:10×Ex Taq buffer 5.0 μL,2.5 mmol/L dNTPs Mix 4.0 μL,上下游引物各2.0 μL,Ex Taq酶0.5 μL,模板DNA 2.0 μL,ddH2O补至终体积为50μL。PCR扩增条件:94℃预变性3min;94℃变性30s,54℃退火30s,72℃延伸1.5min,共24个循环;72 ℃再延伸10 min。PCR扩增产物经1%琼脂糖凝胶电泳检测合格后,送去北京美吉桑格生物医药科技有限公司测序,测序结果与美国国立生物技术信息中心(national center for biotechnology information,NCBI)的Genbank数据库进行BLAST比对搜索。
1.3.3 产生物胺细菌功能基因的扩增及测序
选择产生物胺能力较强的菌株(选择双层显色培养基中紫色较深的菌株),进行功能基因检测。PCR扩增:以提取的细菌基因组DNA为模板,采用表1中的引物PCR扩增ldc、hdc和tdc基因片段。PCR扩增体系:10×Ex Taq Buffer 5.0 μL,2.5 mmol/L dNTPs Mix 4.0 μL,上下游引物各2.0 μL,Ex Taq酶0.5 μL,模板DNA 2.0 μL,ddH2O补至终体积为50 μL。PCR扩增条件:95 ℃预变性5 min;95 ℃变性45 s,hdc基因48 ℃退火45 s、ldc基因50 ℃退火45 s、tdc基因50 ℃退火30 s,72 ℃延伸2 min,共35个循环;72 ℃再延伸5 min。PCR扩增产物经1%琼脂糖凝胶电泳检测。
系统进化分析:将PCR扩增产物送去北京美吉桑格生物医药科技有限公司测序,测序结果与NCBI的Genbank数据库进行BLAST比对搜索,选取与序列具有同源性该菌属内有代表性的多种模式菌株基因序列,利用MEGA7.0软件中的邻接(neighbor joining,NJ)法构建系统进化树,其中Bootsdrap 1 000次检验进化树置信度。
2 结果与分析
2.1 发酵肉制品中微生物群落结构分析
通过高通量测序技术分别对发酵肉制品样本中细菌的16S rRNA V3-V4区序列进行测序,测序结果经过滤拼接处理后得到细菌序列72 835条,样品质控Q20>96%,Q30>89%(Q20和Q30是碱基质量值,其中Q20为碱基识别出错率为1/100,碱基识别精度为0.99;Q30为碱基识别出错率为1/1 000,碱基识别精度为0.999),发酵肉制品样品稀释曲线见图1。
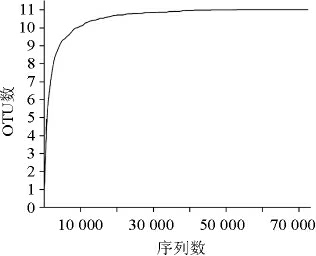
图1 发酵肉制品中细菌多样性测序稀释曲线
Fig.1 Rarefaction curve for the bacteria diversity sequencing of fermented meat sample
由图1可知,随着测序数量的增加,OUT数先急剧增加后趋于平缓,说明测序质量和测序深度均满足实验需求,可用于后续结果分析。在97%的相似水平下对OTU进行分类学分析,结果见图2。
由图2可知,发酵肉制品样本中细菌98.59%为厚壁菌门(Firmicutes)的葡萄球菌属(Staphylococcus)。传统发酵肉制品的加工过程均有微生物参与,但由于不同发酵肉制品的不同原料、地区来源、加工工艺、贮藏条件等使最终产品中微生物组成有较大差别。我国传统发酵肉制品中较常见的微生物有肠细菌(Enterobacteria)、乳酸菌、葡萄球菌(Staphylococcus)和嗜冷杆菌(Psychrobacter)的某些种,国外的发酵香肠中分离出的微生物主要有肠细菌、乳酸菌、葡萄球菌、肉杆菌(Carnobacterium)以及酵母菌的某些种[20-21]。
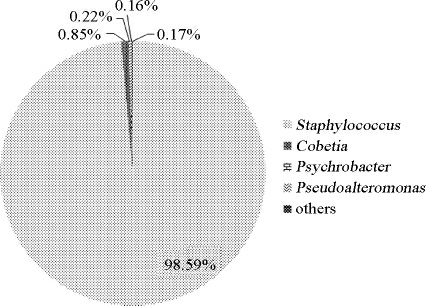
图2 基于属水平发酵肉制品中细菌群落结构
Fig.2 Bacterial community structure in fermented meat based on genus level
2.2 发酵肉制品中产生物胺细菌的筛选与鉴定
通过传统培养分离方法从发酵肉制品中共分离得到52株细菌,通过双层显色培养基从中筛选到43株产生物胺的阳性菌株。通过PCR扩增获得43株菌株的16S rRNA序列,在NCBI的Genbank数据库中进行比对,结果见表2。
表2 发酵肉制品中产生物胺细菌16S rRNA序列比对结果
Table 2 Alignment results of 16S rRNA sequences of biogenic amines-producing bacteria in fermented meat

续表

由表2可知,发酵肉制品中产生物胺的细菌主要为表皮葡萄球菌(Staphylococcus epidermidis)。有报道称,葡萄球菌在发酵肉制品中主要产生蛋白酶和脂肪酶,对发酵肉制品的风味形成和色泽稳定起重要作用[22]。另外,有报道称,表皮葡萄球菌可产生生物胺氧化酶,对已产生的生物胺进行降解,从而降低生物胺的积累[20]。在本研究中,表皮葡萄球菌可能对发酵肉制品的品质有促进作用,同时也是产生物胺的主要微生物。
2.3 发酵肉制品中产生物胺细菌功能基因检测
2.3.1 赖氨酸脱羧酶编码基因ldc的确定与分析
在产生物胺微生物的分离过程中,利用的是双层显色培养基法。在显色过程中,不同产生物胺的微生物显示紫色的能力有显著差别,选择产紫色较深的微生物即生物胺产生能力较强的微生物(菌株5、8、10、12、21、31和45),进行功能基因检测。采用革兰氏阳性菌的赖氨酸脱羧酶引物LDC1和LDC2 PCR扩增以上7株菌株的ldc基因,结果见图3。
由图3可知,7株细菌PCR扩增后均出现阳性条带。PCR扩增产物经测序及序列比对显示,7株细菌的ldc基因序列与表皮葡萄球菌赖氨酸脱羧酶基因序列的一致性>97%。选取同源性较高的该菌属内有代表性的多种模式菌株的ldc基因序列构建系统进化树,结果见图4。
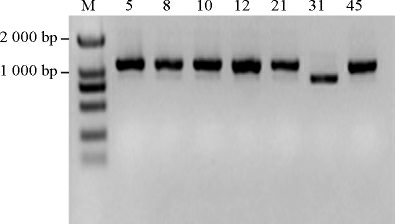
图3 发酵肉制品中产生物胺细菌赖氨酸脱羧酶基因的PCR扩增产物
Fig.3 PCR products of lysine decarboxylase gene of biogenic amines-producing bacteria in fermented meat
M为DL2000 DNA Marker。下同。
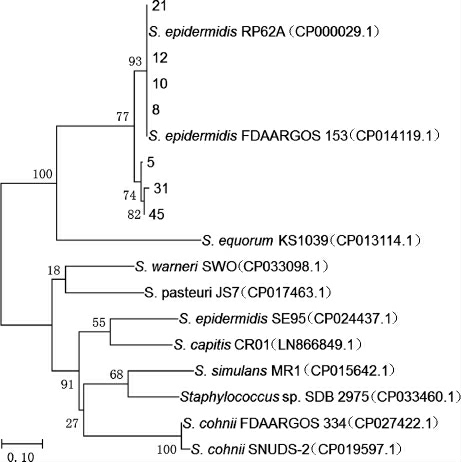
图4 基于赖氨酸脱羧酶基因产生物胺细菌的系统进化树
Fig.4 Phylogenetic tree of biogenic amines-producing bacteria based on lysine decarboxylase gene
由图4可知,同一菌种来源的赖氨酸脱羧酶相似性较高,因此,本研究中采用的引物及PCR扩增条件可用于表皮葡萄球菌中赖氨酸脱羧酶基因的检测。
2.3.2 组氨酸脱羧酶编码基因hdc的确定与分析
采用革兰氏阳性菌的组氨酸脱羧酶引物HDC1和HDC2 PCR扩增菌株5、8、10、12、21、31和45的hdc基因,结果见图5。
由图5可知,7株细菌PCR扩增后均出现阳性条带,PCR扩增产物经测序及序列比对显示,7株细菌的hdc基因序列与表皮葡萄球菌的组氨酸脱羧酶基因序列的一致性>98%。选取同源性较高的该菌属内有代表性的多种模式菌株的hdc基因序列构建系统进化树,结果见图6。
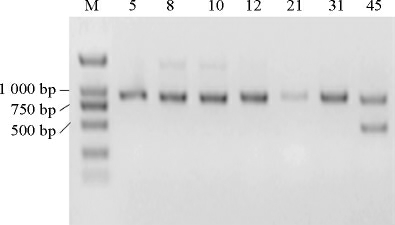
图5 发酵肉制品中产生物胺细菌组氨酸脱羧酶基因的PCR扩增产物
Fig.5 PCR products of histidine decarboxylase gene of biogenic amines-producing bacteria in fermented meat
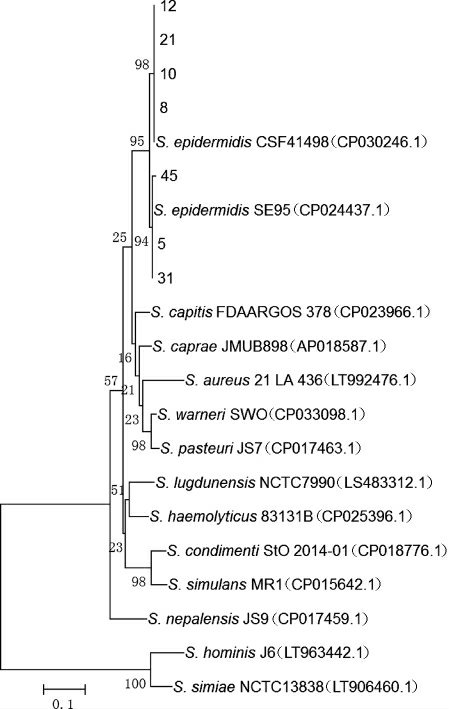
图6 基于组氨酸脱羧酶基因产生物胺细菌的系统进化树
Fig.6 Phylogenetic tree of biogenic amines-producing bacteria based on histidine decarboxylase gene
由图6可知,不同菌种来源的组氨酸脱羧酶的相似性较高。因此,本研究中采用的引物及PCR扩增条件不仅可用于表皮葡萄球菌中组氨酸脱羧酶基因的检测,还可扩展到其他菌种来源的组氨酸脱羧酶基因检测。
2.3.3 酪氨酸脱羧酶编码基因tdc的确定与分析
利用酪氨酸脱羧酶引物TDC1和TDC2 PCR扩增菌株5、8、10、12、21、31和45的酪氨酸脱羧酶基因,结果见图7。
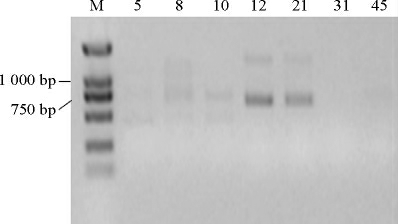
图7 发酵肉制品中产生物胺细菌酪氨酸脱羧酶基因的PCR扩增产物
Fig.7 PCR products of tyrosine decarboxylase gene of biogenic amines-producing bacteria in fermented meat
由图7可知,只有菌株12和21 PCR扩增后出现了阳性条带。阳性条带经测序及序列比对显示,上述菌株基因序列与表皮葡萄球菌酪氨酸脱羧酶基因序列的一致性>98%。由于本研究中采用的引物扩增效果较差,因此,后期需进行PCR扩增引物的筛选以及扩增条件的改变,以找到适合表皮葡萄球菌酪氨酸脱羧酶基因检测的引物及条件。
3 结论
通过高通量测序技术对发酵肉制品中细菌的16S rRNA V3-V4区序列进行测序。结果表明,发酵肉制品中细菌主要为厚壁菌门(Firmicutes)的葡萄球菌属(Staphylococcus)。通过传统培养分离法从发酵肉制品中共分离得到52株细菌,利用双层显色培养基从中筛选出43株产生物胺细菌,且主要为表皮葡萄球菌(Staphylococcus epidermidis),与细菌多样性结果一致。对7株高产生物胺的表皮葡萄球菌的赖氨酸脱羧酶、组氨酸脱羧酶和酪氨酸脱羧酶编码基因进行PCR扩增,结果表明,7株表皮葡萄球菌均含有基因hdc和ldc,为两种生物胺的快速检测提供理论基础。
[1]LADERO V,CALLES-ENRIQUEZ M,FERNANDEZ M,et al,Toxicological effects of dietary biogenic amines[J].Curr Nutr Food Sci,2010,6(2):145-156.
[2]HALASZ A,ÁGNES B,SIMON-SARKADI L,et al,Biogenic amines and their production by microorganisms in food[J].Trends Food Sci Tech,1994,5(2):42-49.
[3]孙玥晖,刘姝韵,吴宝森,等.发酵肉制品中生物胺及产生菌株研究进展[J].食品安全质量检测学报,2017,8(1):131-135.
[4]LINARES D M,BEATRIZ D R,VICTOR L,et al,Factors influencing biogenic amines accumulation in dairy products[J].Front Microbiol,2012,3:180.
[5]MONTEL M C,MASSON F,TALON R.Comparison of biogenic amine content in traditional and industrial French dry sausages[J].Sci Aliment,1999,19(2):247-254.
[6]MARTUSCELLI M,CRUDELE M A,GARDINI F,et al,Biogenic amine formation and oxidation by Staphylococcus xylosus strains from artisanal fermented sausages[J].Lett Appl Microbiol,2010,31(3):228-232.
[7]TEODOROVIC V,BUNCIC S,SMILJANIC D,et al.A study of factors influencinghistamineproductioninmeat[J].Fleischwirtschaft,1994,74(2):181-183.
[8]JEUNE C L,LONVAUD-FUNEL A,BRINK B T,et al.Development of a detection system for histidine decarboxylating lactic acid bacteria based on DNA probes,PCR and activity test[J].J Appl Microbiol,2010,78(3):316-326.
[9]MARCOBAL A,RIVAS B D L,MORENO-ARRIBAS M V,et al.Identification of the ornithine decarboxylase gene in the putrescine-producer Oenococcus oeni BIFI-83[J].Fems Microbiol Lett,2010,239(2):213-220.
[10]LUCAS P,WOLKEN W O,LOLKEMA J,et al.Histamine-producing pathway encoded on an unstable plasmid in Lactobacillus hilgardii 0006[J].Appl Environ Microbiol,2005,71(3):1417-1424.
[11]SHAKILA R J,VASUNDHARA T S,KUMUDAVALLY K V,et al.A comparison of the TLC-densitometry and HPLC method for the determination of biogenic amines in fish and fishery products[J].Food Chem,2001,75(2):255-259.
[12]SHALABY A R.Significance of biogenic amines to food safety and human health[J].Meat Res,2005,29(7):675-690.
[13]SILVA M V D,PINHO O,FERREIRA I,et al.Production of histamine and tyramine by bacteria isolated from Portuguese vacuum-packed coldsmoke fish[J].Food Control,2002,13(6):457-461.
[14]卢士玲,李开雄,徐幸莲,等.传统香肠中产生物胺肠细菌和乳酸菌分离方法的研究[J].食品与发酵工业,2012(8):47-52.
[15]HAJIME T,BON K,MIWAKO Y,et al.Cloning and sequencing of the histidine decarboxylase genes of gram-negative,histamine-producing bacteria and their application in detection and identification of these organisms in fish[J].Appl Environ Microbiol,2003,69(5):2568-2579.
[16]LUCAS P,LONVAUD-FUNEL A.Purification and partial gene sequence of the tyrosine decarboxylase of Lactobacillus brevis IOEB 9809[J].Fems Microbiol Lett,2002,211(1):85-89.
[17]WOLKEN W A M,LUCAS P M,ALINE L F,et al,The mechanism of the tyrosine transporter TyrP supports a proton motive tyrosine decarboxylation pathway in Lactobacillus brevis[J].J Bacteriol,2006,188(6):2198-2206.
[18]李志军.食品中生物胺及其产生菌株检测方法研究[D].青岛:中国海洋大学,2007.
[19]RUIZ-CAPILLAS C,MORAL A.Production of biogenic amines and their potential use as quality control indices for hake(Merluccius merluccius,L.)stored in ice[J].J Food Sci,2010,66(7):1030-1032.
[20]冉春霞,陈光静.我国传统发酵肉制品中生物胺的研究进展[J].食品与发酵工业,2017,43(3):285-294.
[21]SANTOS M H S.Biogenic amines:their importance in foods[J].Int J Food Microbiol,1996,29(2-3):213-231.
[22]李志军,吴永宁,薛长湖.生物胺与食品安全[J].食品与发酵工业,2004,30(10):84-91.