浓香型白酒质量与白酒的品质有密切关系,窖泥的性状通常由经验丰富的工作人员根据窖泥的感官性状以及窖池的产酒率和优品率进行判断,缺乏科学依据,且由微生物引起的窖泥的感官变化具有延滞性,不能及时呈现窖泥性状的变化。以己酸为前体物质的己酸乙酯是浓香型白酒中最重要的香气成分,在已有的文献报道中,克氏梭菌(Clostridium kluyveri)是浓香型白酒中主要的产己酸微生物之一[1-4],因此对窖泥中克氏梭菌以及己酸循环系统中关键基因的表达量研究,对实现科学养窖护窖,提高白酒品质具有重要意义。
近年来,基于基因组学的测序技术使人们更加全面地了解环境样本中微生物多样性组成,转录组学(transcriptomics)、蛋白组学(protemics)、代谢组学(metabolomics)和流量组学(fluxomics)等技术的兴起,可以更加系统更加深入地剖析环境样本中微生物基因转录、信号转导、蛋白表达以及代谢关系等[5-6]。其中,转录组广义上是指在某一特定生理条件或者环境下,一个细胞、组织或者生物体中所有核糖核酸(ribonucleic acid,RNA)总和,包括信使RNA(messenger RNA,mRNA)、核糖体RNA(ribosome,rRNA)、转运RNA(transfer RNA,tRNA)以及非编码RNA(non-coding RNA),狭义上是特指细胞中转录出来的所有mRNA的总和。
目前,对窖泥己酸的研究主要停留在对窖泥中的己酸菌进行分离鉴定,并利用传统的培养方法研究其产酸条件[7-9],但是关于其代谢机理的研究报道较少,而且固态混合发酵比液体纯种发酵方式更为复杂,液态发酵结果不一定能在窖泥发酵中再现。因此,本研究采用宏转录组技术主要研究产己酸关键酶基因在老化窖泥中的表达,并对老化窖泥中的己酸代谢途径进行分析,研究结果有利于为提升白酒品质奠定理论基础,同时也为窖泥己酸的机理研究提供参考。
1 材料与方法
1.1 材料与试剂
1.1.1 窖泥样品
老化窖泥样品:取自四川某酒厂,取样人员佩戴无菌无酶手套、口罩,窖池打开后,迅速取样,充分混合,放入液氮罐。样品由液氮罐运回,-80 ℃冰箱冷冻保存备用。
1.1.2 化学试剂
E.Z.N.A 土壤核糖核酸(RNA)提取试剂盒:美国OMEGAbio-Tek 公司;Trizol 试剂:上海生工生物工程股份有限公司;互补脱氧核糖核酸(complementary deoxyribonucleicacid,cDNA)第一链合成试剂盒:美国Thermo Fermentas公司;质粒小提试剂盒:天根生化科技有限公司;SYBRGreen实时聚合酶链式反应(polymerase chain reaction,PCR)Master Mix:日本TOYOBO公司;核酸染料、DNA Marker、MarkerⅢ:上海生工生物工程股份有限公司。
土壤核糖核酸(RNA)提取试剂盒:美国OMEGAbio-Tek 公司;Trizol 试剂:上海生工生物工程股份有限公司;互补脱氧核糖核酸(complementary deoxyribonucleicacid,cDNA)第一链合成试剂盒:美国Thermo Fermentas公司;质粒小提试剂盒:天根生化科技有限公司;SYBRGreen实时聚合酶链式反应(polymerase chain reaction,PCR)Master Mix:日本TOYOBO公司;核酸染料、DNA Marker、MarkerⅢ:上海生工生物工程股份有限公司。
1.2 仪器与设备
LightCycler Nan荧光定量聚合酶链式反应仪:瑞士Roch公司;MyCyclerTMThermal Cycler PCR仪:美国Bio-Rad公司;DYY-5电泳仪:北京六一生物科技有限公司;JY04S-3C凝胶成像仪:北京君意设备有限公司;MDF-86低温保存箱:安徽中科都菱商用电器股份有限公司;Legend Micro 17R离心机:美国Thermo Scientific公司;TA2004 电子天平:上海良平仪器仪表有限公司。
Nan荧光定量聚合酶链式反应仪:瑞士Roch公司;MyCyclerTMThermal Cycler PCR仪:美国Bio-Rad公司;DYY-5电泳仪:北京六一生物科技有限公司;JY04S-3C凝胶成像仪:北京君意设备有限公司;MDF-86低温保存箱:安徽中科都菱商用电器股份有限公司;Legend Micro 17R离心机:美国Thermo Scientific公司;TA2004 电子天平:上海良平仪器仪表有限公司。
1.3 方法
1.3.1 样品总RNA的提取
采用E.Z.N.A. 土壤RNA 抽提试剂盒提取窖泥总RNA,实验所使用的离心管、枪头等均无菌无酶,所有试剂均为RNA专用试剂,实验在封闭环境中进行,具体步骤详见说明书,将提取的总RNA 置于-80 ℃保存备用。
土壤RNA 抽提试剂盒提取窖泥总RNA,实验所使用的离心管、枪头等均无菌无酶,所有试剂均为RNA专用试剂,实验在封闭环境中进行,具体步骤详见说明书,将提取的总RNA 置于-80 ℃保存备用。
1.3.2 老化窖泥宏转录组数据库构建
老化窖泥样品的宏转录组测序由上海派森诺生物公司完成。
提取窖泥样品的总RNA后,去除rRNA,对mRNA进行富集并以mRNA为模板合成互补脱氧核糖核酸(cDNA)双链。采用全转录鸟枪法(whole genome shotgun,WGS),基于Illumina HiSeq X-ten高通量测序平台对构建的文库进行双端测序。测序后,采用在线网站FastQC(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)对测序产生的原始数据进行质量控制,同时完成对高质量序列的筛查和过滤。采用在线网站SortMeRNA(http://bioinfo.lifl.fr/RNA/sortmerna/)[10],将筛查后的有效序列集与软件自带的rRNA序列数据库进行比对,剔除rRNA序列。对样品的mRNA序列采用在线软件Trinity(http://trinityrnaseq.github.io/)[11]从头组装拼接,将拼接后的样品与所有转录本进行比对,并把注释到同一蛋白的转录本归并为Unigene。将Unigene数据集与美国国立生物技术信息中心蛋白数据库(National Center for Biotechnology Information Non-redundant protein collection,NCBI-NR)(ftp://ftp.ncbi.nih.gov/blast/db/)、京都基因与基因组百科全书数据库(Kyoto ENcyclopedia of Genes and Genomes,KEGG)(http://www.genome.jp/kegg/)和碳水化合物活性酶数据库(Carbohydrate-Active enZymes Database,CAZy)(http://www.cazy.org/)比对,分别获得物种注释、KEGG注释、EggNOG注释、CAZy注释及各等级功能注释表达量分布[12-15]。
2 结果与分析
2.1 老化窖泥己酸代谢途径及关键酶基因物种来源分析

图1 老化窖泥中己酸合成途径
Fig.1 Biosynthesis pathway of caproic acid in aged pit mud
由图1可知,在窖泥中己酸的生成途径中,丙酮酸被丙酮酸氧化还原酶EC 1.2.7.1(0.033 2%)或2-氧酸氧化还原酶EC 1.2.7.11(0.025 4%)催化降解为乙酰辅酶A,在乙酰辅酶A-C-乙酰转移酶EC 2.3.1.9(0.062 4%)的作用下催化生成乙酰乙酰辅酶A,再经由乙酰乙酰辅酶A还原酶EC 1.1.1.36(0.005 3%)、3-羟酰基辅酶A脱氢酶EC 1.1.1.35(0.056 5%)、3-羟基丁酰辅酶A 脱氢酶EC 1.1.1.157(0.011 6%)催化生成3-羟基丁酰基辅酶A,经由烯酰辅酶A水合酶EC 4.2.1.17(0.033 8%)或3-羟基丁酰辅酶A脱水酶EC 4.2.1.55(0.000 8%)生成巴豆酰辅酶A,再经由反式-2-烯酰辅酶A还原酶EC 1.3.1.44(0.002 8%)催化生成丁酰基辅酶A。而乙酰辅酶A和丁酰基辅酶A被C-酰基转移酶EC 2.3.1.16(0.032 3%)催化生成3-氧代己酰辅酶A,再经3-羟酰基辅酶A脱氢酶EC 1.1.1.35、β-羟酰辅酶A脱氢酶EC 1.1.1.211(0.002 6%)、烯酰辅酶A水合酶EC 4.2.1.17、N-乙酰-γ-谷氨酰-磷酸还原酶EC 1.2.1.38(0.008 2%)的共同作用下催化生成己酰基辅酶A,从而进一步产生己酸。
丁酸是生产己酸的前体物质,HU L等[16]验证了克氏梭菌中一种产生丁酸的转录单元crt1-bcd-etfAB1-hbd1-nfnAB,而氧化还原敏感转录调节器(redox-sensing transcriptional regulator,Rex)在碳循环和能量代谢中起重要作用,且可以调控丁酰辅酶A脱氢酶/电子转移黄素蛋白复合物(butyryl coenzyme A dehydrogenase/electronic transfer flavin protein complex,Bcd/EtfAB)和NADPH+氧化还原酶复合体(NADPH+oxidoreductase complex,NfnAB),从而直接或间接控制丁酸合成。将己酸循环关键酶基因3-羟基丁酰辅酶A脱水酶(dehydration of 3-hydroxybutyryl coenzyme A,crt1)、丁酰辅酶A脱氢酶(butyryl CoA dehydrogenase,bcd)、电子转移黄素蛋白A1(electronic transfer flavin protein complex A1,etfA1)、电子转移黄素蛋白B1(electronic transfer flavin protein complex B1,etfB1)、NADP依赖性3-羟基丁酰辅酶A脱氢酶(NADP-dependent 3-hydroxybutyrate coenzyme A dehydrogenase,hbd1)、NADPH+氧化还原酶A(NADPH+oxidoreductase A,nfnA)和NADPH+氧化还原酶B(NADPH+oxidoreductase B,nfnB)注释到本批次窖泥样品转录本中,发现基因hbd1与图1中乙酰乙酰辅酶A还原酶EC 1.1.1.36、3-羟酰基辅酶A脱氢酶EC 1.1.1.35、3-羟基丁酰辅酶A脱氢酶EC 1.1.1.157在己酸生成中功能相似,基因crt1与图1A中烯酰CoA水合酶EC 4.2.1.17和3-羟基丁酰CoA脱水酶EC 4.2.1.55功能相似,而基因bcd1、etfB1、etfA1、nfnA和nfnB则等同于图1B中的乙酰-CoA C-酰基转移酶EC 2.3.1.16。基于此,挑选出己酸代谢系统中关键酶基因EC 1.1.1.36、EC 1.1.1.35、EC 1.1.1.157、EC 4.2.1.17、EC 4.2.1.55和EC2.3.1.16,并将其注释到物种门水平来源,拟找出老化窖泥中的功能微生物与特征微生物。

图2 老化窖泥中编码己酸合成途径关键酶基因在微生物门水平相对丰度
Fig.2 Relative abundance of key enzyme gene involved in caproic acid biosynthesis at phylum level
由图2可知,EC 1.1.1.36主要来源于变形菌门(Proteobacteria)(52.45%)和浮霉菌门(Planctomycetes)(1.96%);EC 1.1.1.35主要来源于细菌中的变形菌门(Proteobacteria)(42.85%)、蓝藻菌门(Cyanobacteria)(9.15%)、拟杆菌门(Bacteriodetes)(0.90%)和放线菌门(0.13%)以及真核生物界中的链型植物门(Streptophyta)(2.30%)、节肢动物门(Arthropoda)(0.96%)以及子囊菌门(Ascomycota)(0.32%);EC 1.1.1.157主要来源于变形菌门(Proteobacteria)(20.67%)、厚壁菌门(Firmicutes)(11.76%)、拟杆菌门(Bacteriodetes)(3.02%)以及放线菌门(Actinobacteria)(2.54%);EC 4.2.1.17主要来源于变形菌门(Proteobacteria)(50.10%)、放线菌门(Proteobacteria)(0.91%)、厚壁菌门(Firmicutes)(0.56%)以及真核生物界的链型植物门(Streptophyta)(7.47%);EC 4.2.1.55主要来源于变形菌门(Proteobacteria)(42.85%)、蓝藻菌门(Cyanobacteria)(9.15%)、拟杆菌门(Bacteriodetes)(0.90%)以及真核生物中的链型植物门(Streptophyta)(2.30%)、节肢动物门(Arthropoda)(0.96%)以及子囊菌门(Ascomycota)(0.32%)等;EC 2.3.1.16主要来源于变形菌门(Proteobacteria)(14.48%)、芽单胞菌门(Gemmatimonadetes)(0.22%)以及脊索动物门(Chordata)(1.55%)。由此说明变形菌门在老化窖泥的己酸合成途径中起重要作用,其次为厚壁菌门、拟杆菌门、放线菌门、蓝藻菌门,而真核生物在己酸合成途径中具有一定贡献,但关于其在窖泥中具体作用的研究较少。
将挑选出己酸代谢系统中关键酶基因EC 1.1.1.36、EC 1.1.1.35、EC 1.1.1.157、EC 4.2.1.17、EC 4.2.1.55和EC 2.3.1.16注释到老化窖泥中物种种水平来源,结果见图3。由图3可知,EC 1.1.1.36主要来源于变形菌门中的食酸菌属(Acidovorax sp.)JS42(3.92%)和Gemmata sp.SH-PL17(1.96%);EC 1.1.1.35主要来源于真核域的支链淀粉菌(Amyelois transitella)(4.31%)以及厚壁菌门中的普通变形杆菌(Proteus vulgaris)(9.34%)、脱卤厌氧杆菌(Anaeromyxobacter dehalogenans)(1.12%);EC 1.1.1.157主要来源于库兹涅佐夫脱硫菌(Desulfotomaculum kuznetsovii)(11.76%)、脱卤厌氧杆菌(Anaeromyxobacter dehalogenans)(8.11%)、玫瑰微囊藻(Minicystis rosea)(7.95%)、阿克苏恩斯庞蒂巴杆菌(Pontibacter akesuensis)(1.59%)、黄杆菌科细菌(Flavobacteriaceae bacterium)MAR_2010_188(1.43%)、车前孢属(Plantactinospora sp.)KBS50(1.27%)等;EC 4.2.1.17主要来源于脱卤厌氧杆菌(Anaeromyxobacter dehalogenans)(7.58%)等;EC 4.2.1.55来源于自养黄杆菌(Xanthobacter autotrophicus)(11.11%);EC 2.3.1.16主要来源于脱卤厌氧杆菌(Anaeromyxobacter dehalogenans)(10.39%)等。
由图3可知,未注释到通常在窖泥中关注到的己酸菌[17-19],而EC 1.1.1.35、EC 1.1.1.157、EC 4.2.1.17以及EC 2.3.1.16均可在脱卤厌氧杆菌(Anaeromyxobacter dehalogenans)编码得到,脱卤厌氧杆菌(Anaeromyxobacter dehalogenans)是土壤中的一类重要的功能微生物,可在一定程度上避免大气循环中的氮损失[20],但是未见其与窖泥性状关系的报道。因此,说明固态混合发酵不同于液态发酵,在纯培养条件下己酸菌的代谢产物可由固态发酵中其他非己酸菌混合作用产生,上述菌株未被关注到的原因可能是在特定的筛选条件无法筛选未被认识的菌株,因此也进一步说明传统的可培养方法并不能全面解析环境微生物的多样性。虽然在以往的研究中,未见窖泥中关于脱卤厌氧杆菌(Anaeromyxobacter dehalogenans)的报道,但是基于本研究中转录组的数据注释,推测脱卤厌氧杆菌(Anaeromyxobacter dehalogenans)可能是窖泥中一类重要的功能微生物,还有待进一步研究。
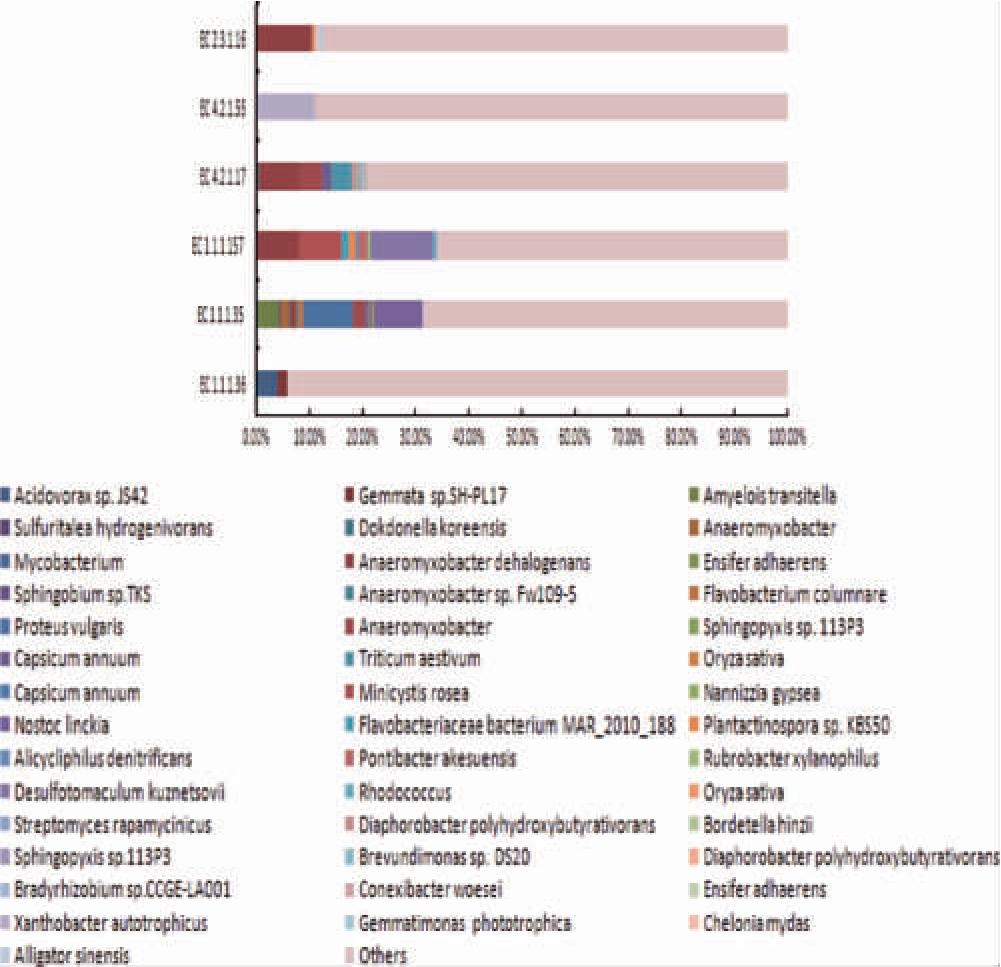
图3 老化窖泥中编码己酸合成途径关键酶基因在微生物种水平相对丰度
Fig.3 Relative abundance of key enzyme gene involved in caproic acid biosynthesis at species level
除了上述与关键酶基因crt1、bcd、etfA1、etfB1、hbd1、nfnA和nfnB相似的基因外,根据图1,还有可催化生成丁酰基辅酶A的反式-2-烯酰辅酶A还原酶EC 1.3.1.44(0.002 8%),催化生成3-羟基己酰基辅酶A的β-羟酰辅酶A脱氢酶EC1.1.1.211(0.002 6%)以及催化生成己酰辅酶A的N-乙酰-γ-谷氨酰-磷酸还原酶EC1.2.1.38(0.008 2%)。将其注释到微生物物种,结果见图4。
由图4可知,在微生物门水平上,EC1.3.1.44主要来源于螺旋体门(Spirochaetes)(9.09%)以及变形菌门(Proteobacteria)(6.36%);EC1.1.1.211 主要来源于链型植物门(Streptophyta)(91.36%);而EC 1.2.1.38 主要来源于脊索动物门(Chordata)(5.15%)。而在微生物属水平,EC 1.3.1.44 主要来源于密螺旋体属(Treponema)(9.09%)和欧文氏菌属(Erwinia)(6.36%);EC 1.1.1.211 主要来源于小麦属(Triticum)(46.91%)、辣椒属(Capsicum)(42.59%)和稻属(Oryza)(1.85%);EC 1.2.1.38 主要来源于鲱属(Clupea)(5.15%)。
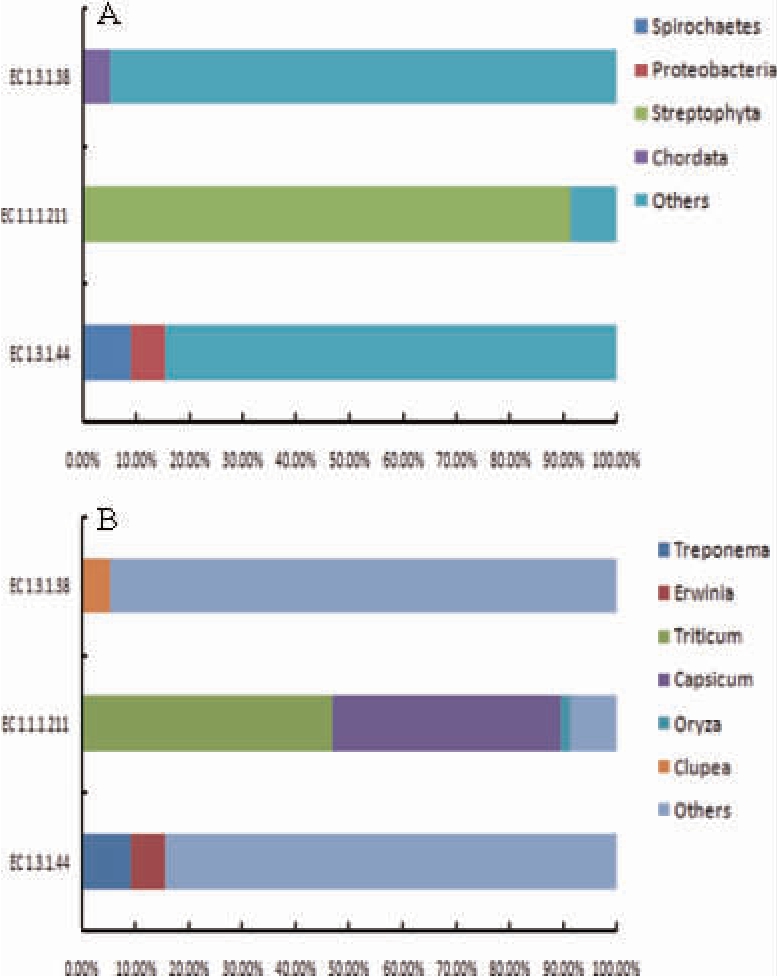
图4 老化窖泥中编码己酸合成途径关键酶基因在微生物门(A)和属(B)水平的相对丰度
Fig.4 Relative abundance of key enzyme gene involved in caproic acid biosynthesis at phylum level (A) and genus level (B)
2.2 老化窖泥己酸相关代谢途径分析
对乙酸代谢、丁酸代谢、乳酸代谢、丙酮酸代谢以及糖酵解途径进行综合分析,筛选出代谢通路中的关键酶基因,并对其注释到物种来源,结果见图5。由图5可知,在丁酸代谢途径中,丙酮酸通过丙酮酸氧化还原酶EC1.2.7.1(0.033 2%)和2-氧代酸氧化还原酶EC1.2.7.11(0.025 4%)转化为乙酰辅酶A。丙酮酸降解生成乙酰辅酶A后,经乙酰辅酶A-C-乙酰转移酶EC2.3.1.9(0.062 4%)催化生成乙酰辅酶A(图1),其后再参与到图1中的代谢通路。同时,醋酸盐和乙酰辅酶A之间则可以通过丙酸辅酶A转移酶EC2.8.3.1(0.001 8%)或乙酸辅酶A转移酶EC2.8.3.18(0.001 8%)实现其相互转化。乙酰磷酸酯(acetyl phosphate,Acetyl-P)是醋酸盐的底物之一,可以被乙酰磷酸酶EC3.6.1.7(0.005 7%)转化为醋酸盐,同时可以通过磷酸乙酰转移酶EC2.3.1.8(0.003 7%)实现与乙酰辅酶A之间的相互转化。而在醋酸盐和乙醛之间,可以通过乙醛脱氢酶EC1.2.1.3(0.077 3%)、乙醛脱氢酶(aldehyde dehydrogenase,ALDH)EC1.2.1.5(0.001 1%)或者醛脱氢酶EC1.2.1.-(0.001 3%)实现相互转化。
由图5可知,D-乳酸和L-乳酸之间相互转化的关键酶乳酸消旋酶EC 5.1.2.1在老化窖泥中并未检测到,而根据本实验室前期的研究结果,在老熟窖泥的转录本中也未检测到乳酸消旋酶[21],推测不同性状窖泥的某种共有理化特性导致了乳酸消旋酶mRNA 的降解、抑制了乳酸消旋酶的表达。L-乳醛是L-乳酸的前体物质,将其转化为L-乳酸的乳醛脱氢酶EC 1.2.1.22在老化窖泥中检测到0.000 196%,但并未注释到物种。丙酮酸与L-乳酸可通过L-乳酸脱氢酶EC 1.1.1.27(0.003 1%)相互转化,同时,L-乳酸可以经由另一种L-乳酸脱氢酶EC 1.1.2.3(0.001 2%)催化降解为丙酮酸。磷酸烯醇丙酮酸是丙酮酸的前体物质,可以通过丙酮酸激酶EC 2.7.1.40(0.042 8%)实现转化。由此可知,丙酮酸是窖泥中乙酸代谢、丁酸代谢、乳酸代谢以及己酸代谢重要的中间产物。
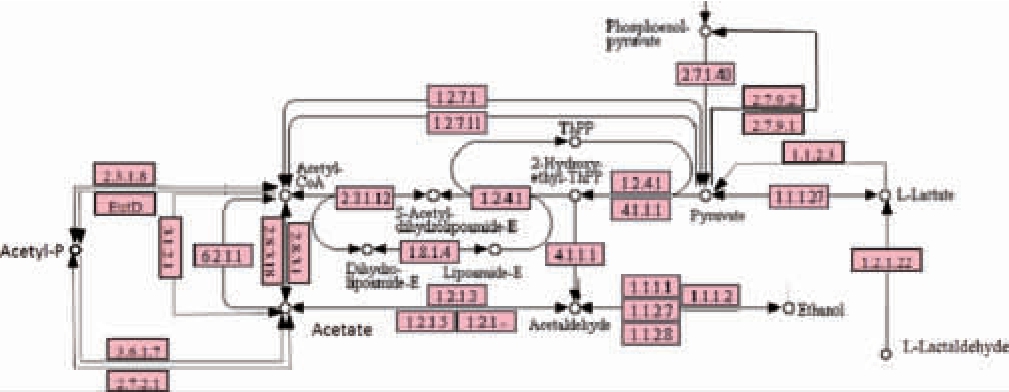
图5 老化窖泥代谢图
Fig.5 Metabolic map of aged pit mud
3 结论
本研究采用宏转录组技术分析了老化窖泥中功能酶基因的表达情况以及己酸代谢途径,通过测序及生物信息学分析,主要得出如下结论:
将产己酸关键基因crt1、bcd、etfA1、etfB1、hbd1、nfnA和nfnB注释到转录本物种来源,发现真核生物在老化窖泥代谢中具有重要作用,几组关键酶基因并未注释到窖泥中关注较多的己酸菌,但是酶基因EC1.1.1.35、EC1.1.1.157、EC4.2.1.17以及EC2.3.1.16均可在脱卤厌氧粘细菌(Anaeromyxobacter dehalogenans)找到,推测脱卤厌氧粘细菌在窖泥中虽然含量不多,但可能是窖泥中一种活性微生物,具有重要功能,同时真核生物和变形菌门在老化窖泥的己酸代谢中,具有重要作用,窖泥中的己酸并非完全由己酸菌合成,而是其他非己酸菌共同作用的结果。
丙酮酸是连接乙酸代谢、丁酸代谢、乳酸代谢以及己酸代谢的重要中间产物。在老化窖泥中,丙酮酸通过丙酮酸氧化还原酶EC 1.2.7.1或者2-氧代酸氧化还原酶EC 1.2.7.11转化为乙酰CoA,但是不能通过丙酮酸甲酸裂解酶EC 2.3.1.54转化丙酮酸,同时老熟和老化窖泥中某些共有特性使乳酸消旋酶EC 5.1.2.1无法表达从而导致D-乳酸和L-乳酸之间在老熟和老化窖泥中不能通过乳酸消旋酶EC 5.1.2.1直接相互转化。
本研究利用宏转录组学的方法探测了窖泥中活性微生物的动态表达,不仅有利于寻找窖泥微生物的差异表达基因,还可以帮助探明浓香型白酒关键风味物质的代谢通路,为浓香型白酒生产企业及时养窖护窖、提高白酒品质提供了理论依据。
[1]MIAO Z J,WANG X C,LIU X N.Isolation and purification of caproic acid bacteria from pit mud&study of its acid-producing capacity[J].Liquor-Making Sci Technol,2012,11:79-81.
[2]嵇翔,徐岩,穆晓清,等.克氏梭菌和酿酒酵母混合培养提高己酸产量[J].食品与生物技术学报,2017,36(9):922-926.
[3]任聪,辜杨,杜海,等.基于新老窖泥的微生物菌群结构判定浓香型白酒生产中的主体己酸菌[J].食品与发酵工业,2018,44(12):12-18.
[4]JUN X.Application of caproic acid bacteria functional liquid in luzhou-flavor liquor production[J].Liquor-Making Sci Technol,2012,1:77-78.
[5]PARK J H,LEE S Y,KIM T Y,et al.Application of systems biology for bioprocess development[J].Trend Biotechnol,2008,26(8):404-412.
[6]SANCHEZ B,RUIZ L,GUEIMONDE M,et al.Omics for the study of probiotic microorganisms[J].Food Res Int,2013,54(1):1061-1071.
[7]吴衍庸,易伟庆.泸酒老窖己酸菌分离特性及产酸条件的研究[J].食品与发酵工业,1986(5):4-9.
[8]熊俐,胡洋,刘俊,等.窖泥己酸菌的分离培养与诱变选育[J].四川理工学院学报(自然科学版),2010,23(3):324-327.
[9]陈翔,王亚庆,邵海燕,等.己酸菌纯种培养与混合培养技术在窖泥生产中的应用[J].酿酒,2009,36(3):35-37.
[10]KOPYLOYA E,NOE L,TOUZET H.SortMeRNA:fast and accurate filtering of ribosomal RNAs in metatranscriptomic data[J].Bioinformatics,2012,28(24):3211-3217.
[11]GRABHERR M G,HAAS B J,YASSOUR M,et al.Full-length transcriptome assembly from RNA-Seq data without a reference genome[J].Nat Biotechnol,2011,29(7):644-652.
[12]KANEHISA M.The KEGG resource for deciphering the genome[J].Nucl Acid Res,2004,32:277-280.
[13]JENSEN L J,JULIEN P,KUHN M,et al.eggNOG:automated construction and annotation of orthologous groups of genes[J]. Nucl Acid Res,2007,36:D250-D254.
[14]CANTAREL B L,COUTINHO P M,RANCUREL C,et al.The Carbohydrate-Active EnZymes database(CAZy):an expert resource for glycogenomics[J].Nucl Acid Res,2009,37:D233-D238.
[15]APWEILER R,BAIROCH A,WU C H,et al.UniProt:the Universal Protein Knowledgebase[J].Nucl Acid Res,2004,32(D1):D115-D119.
[16]HU L,HUANG H,YUAN H,et al.Rex in Clostridium kluyveri is a global redox-sensing transcriptional regulator[J].J Biotechnol,2016,233:17-25.
[17]彭兵,祝熙,李忠奎,等.窖泥高产己酸菌分离鉴定及培养条件优化的研究[J].中国酿造,2016,35(5):43-46.
[18]杨菁瑾,唐雪,杨先超,等.泸州老窖不同窖龄窖泥中己酸菌遗传多样性及系统发育[J].四川农业大学学报,2015,33(2):181-188.
[19]浓香型白酒窖泥产酸菌群培养及新型己酸合成菌的鉴定与特性分析[D].无锡:江南大学,2018.
[20]ONLEY J R,AHSAN S,SANFORD R A,et al.Denitrification by Anaeromyxobacter dehalogenans,a common soil bacterium lacking nitrite reductase genes(nirS/nirK)[J]. Appl Environ Microbiol,2017,84(4):AEM.01985-17.
[21]张倩颖.老熟窖泥分子微生态分析及克氏梭菌己酸合成调控研究[D].成都:四川大学,2017.