脂肪酶(lipase EC3.1.1.3)是一类能催化甘油酯水解的酶的总称,催化效率高,副产物少,反应条件温和,不需要辅酶或辅因子的参与,已在合成反应、生物燃料、制药工业以及洗涤剂、食品、纺织等领域得到广泛应用。脂肪酶不仅能催化甘油酯类水解与合成,还能催化酯化反应、酯交换反应、醇解反应,促进多肽、表面活性剂和药物等的合成反应[1]。微生物脂肪酶的研究始于20世纪初,经过100多年的研究,目前已经成为脂肪酶的主要来源。特别是近几十年来,随着高效筛选技术和先进基因工程技术的应用,已经实现了曲霉、毛霉和假单胞菌的相对高产,并且部分菌株已经实现工业化应用[2]。我国高质量的脂肪酶产生菌种类少,产量低,很多企业不得不依赖价格昂贵的进口脂肪酶,因此筛选和培育高产优势脂肪酶产生菌显得尤为重要[3]。另外,脂肪酶催化的反应类型和条件多种多样,不同类型的脂肪酶应用方向千差万别,因此筛选不同来源的脂肪酶是脂肪酶研究的基础。
微生物脂肪酶的表达主要受环境因子的调节,产脂肪酶微生物需要以油脂和脂肪酸作为碳源生长,因此常用的微生物筛选来源包括油脂加工厂、乳制品工厂以及附近被油脂污染的土壤和水域等[2]。芽孢杆菌是一类潜在的优良脂肪酶生产菌株,目前用于生产脂肪酶的芽孢杆菌有枯草芽孢杆菌[4]、蜡状芽孢杆菌[5]、类芽孢杆菌[6]和地衣芽孢杆菌[7]等。枯草芽孢杆菌所生产的脂肪酶属于胞外酶,便于下游分离纯化,适用于大规模工业化生产[8-9],并且普遍具有较高的碱耐受力[10-11],在洗涤剂和纺织等领域市场需求巨大。本研究从油脂污染水样中分离出一株产脂肪酶菌株,对其进行形态学特征、生理生化试验及分子生物学鉴定,并通过单因素试验和响应面试验对其发酵培养基组分进行优化,旨在提高脂肪酶产量,进一步对其酶学性质研究提供技术参考和实验基础。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
从曲阜市多个食堂和食品厂附近被油脂污染的下水道中采集样品10份。
1.1.2 化学试剂
葡萄糖、酵母膏、磷酸氢二钠、硫酸铵、氯化钠、三水磷酸氢二钾、七水硫酸镁、琼脂粉、蛋白胨、橄榄油、罗丹明B、聚乙烯醇(polyvinyl alcohol,PVA):国药集团化学试剂有限公司。
1.1.3 培养基[12-14]
富集培养基:酵母膏0.2 g/L,氯化钠0.5 g/L,磷酸氢二钠3.5 g/L,磷酸二氢钾1.5 g/L,七水硫酸镁0.5 g/L,橄榄油10 mL,用蒸馏水定容至1 000 mL,121 ℃灭菌20 min。
初筛罗丹明B培养基[14]:硫酸铵0.5 g,氯化钠4 g,磷酸氢二钾1 g,七水硫酸镁0.5 g,琼脂粉15 g,三丁酸甘油脂乳化液100 mL(4%PVA∶三丁酸甘油脂=3∶1,超声乳化),用蒸馏水定容至1 000 mL,121 ℃灭菌20 min后,待温度降至60 ℃时加入1 mL罗丹明B 溶液(质量浓度<10 mg/mL),混匀后倒平板。
初始发酵培养基:葡萄糖5.0 g/L,蛋白陈2 g/L,尿素6.0 g/L,三水磷酸氢二钾1.0 g/L,硫酸铵1 g/L,七水硫酸镁0.5 g/L,三丁酸甘油脂10.0 mL,用蒸馏水定容至1 000 mL,自然pH,115 ℃灭菌15 min。
1.2 仪器与设备
SW-CJ-1D超净工作台:苏州净化设备有限公司;SIGMA 2-16PK通用型离心机:德国Sigma公司;TS-1102立式摇床:上海天呈试验仪器制造有限公司;CL-32L高压蒸汽灭菌锅:日本ALP公司;上海申安医疗器械厂;T100型PCR仪、Mini-SubCell核酸电泳仪:美国BioRad公司;CX23生物显微镜:日本奥林巴斯有限公司。
1.3 方法
1.3.1 菌株的筛选
将采集的样品分别依次进行富集培养、初筛和初始发酵复筛。样品经过3次富集培养后,分别取50 μL菌液上清涂布于罗丹明B培养基平板,罗丹明B能与脂肪酶降解油脂产生的脂肪酸特异性结合,产酶菌落周围会生成玫红色,在波长365 nm紫外灯下呈现橙黄色。将罗丹明B平板放在波长365 nm紫外灯下进行观察,选取橙黄色圈较大的菌株进行分离培养,镜检至无杂菌后作为出发菌种,接种于初始发酵培养基中进行复筛,选出脂肪酶高产菌株[15-17]。
1.3.2 菌种鉴定
根据《常见细菌系统鉴定手册》[18],对所筛选的菌株进行形态学特征和生理生化鉴定。提取目的菌株的基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)为模板,以16S rDNA序列的通用引物进行聚合酶链式反应(polymerase chain reaction,PCR)扩增。扩增产物送至上海博尚生物科技有限公司进行测序,并将得到的序列与GenBank数据库中的序列进行比对分析,使用MEGA-X软件构建系统发育树[19]。
1.3.3 产脂肪酶菌株的种子液的制备及发酵培养
种子液的制备:将1.3.1筛选得到的菌种于无菌条件下,接种一环菌至装液量为50 mL/250 mL的初始发酵培养基中,35 ℃、220 r/min,摇瓶培养12 h,制备成种子液。将种子液按照10%(V/V)装液量接种至为25 mL/250 mL的发酵培养培养基中,发酵液pH=7,35 ℃、220 r/min条件下摇瓶培养40 h。
1.3.4 发酵培养基组分优化
单因素试验:设计单因素试验考察发酵培养基中三丁酸甘油酯添加量(1.2%、1.4%、1.6%、1.8%、2.0%、2.2%、2.4%)、葡萄糖添加量(4 g/L、5 g/L、6 g/L、7 g/L、8 g/L、9 g/L、10 g/L)及尿素添加量(6 g/L、8 g/L、10 g/L、12 g/L、14 g/L、16 g/L、18 g/L)对发酵液中粗脂肪酶活性的影响,确定各因素的中心值及水平。
响应面试验:根据响应面分析法中Box-Behnken Design(BBD)试验设计原理,采用上述单因素试验确定的中心值、低水平和高水平(分别用0、-1、+1表示),以脂肪酶活力(Y)为响应值,对发酵培养基中三丁酸甘油酯添加量(A)、葡萄糖添加量(B)、尿素添加量(C)进行优化,得到最佳发酵条件组合,试验因素与水平见表1。
表1 发酵条件优化响应面试验因素与水平
Table 1 Factors and levels of response surfer experiments for fermentation conditions optimization

1.3.5 发酵液中脂肪酶活力的检测
粗酶液的制备:将发酵液低温离心去除菌体,4 ℃、5 500 r/min条件下离心15 min,除菌体。上清用0.22 μm滤膜过滤,制成粗酶液于4 ℃保存备用。
脂肪酶活力检测:参考国标GB/T 23535—2009《脂肪酶制剂》[20]。脂肪酶活力定义:在pH7.5,40 ℃条件下,每分钟产生1 μmol可滴定脂肪酸所需的酶量为1个酶活力单位(U/mL)。
2 结果与分析
2.1 目的菌株的筛选
经过初筛和富集培养,从10份样品中筛选分离得到4株橙黄色圈较大的菌株,分别编号为U1、U2、U4、U5,再分别进行摇瓶发酵培养,测定各发酵液中产脂肪酶的活力值,结果见表2。由表2可知,菌株U5脂肪酶酶活最高,为3.3 U/mL,其余3株菌脂肪酶酶活较低,均在1.3~2.9 U/mL范围内。因此,选择菌株U5作进一步鉴定。
2.2 菌株U5的鉴定
2.2.1 菌株U5形态学特征
菌株U5的菌落及细胞形态见图1。由图1A可知,在琼脂平板上,菌落乳白色,边缘不规则,不透明,菌落表面粗糙,略有突起。由图1B可知,菌体呈圆杆状,无荚膜,有鞭毛。

图1 菌株U5的菌落(A)及细胞(B)形态
Fig.1 Colony morphology (A) and cell morphology (B) of strain U5
2.2.2 生理生化鉴定
菌株U5能利用甘露醇、柠檬酸盐、葡萄糖,接触酶试验、硝酸盐还原、V-P试验、7%氯化钠生长均为阳性。根据菌株U5的形态特征和生理生化特性,可以初步鉴定菌株为芽孢杆菌属(Bacillus sp.)。
2.2.3 分子生物学鉴定
以菌株U5基因组DNA为模板,采用细菌16S rDNA通用引物进行PCR扩增,PCR产物经1%琼脂糖凝胶电泳分析,结果发现扩增条带约1 500 bp,测序结果见图2。

图2 菌株U5 16S rDNA测序结果
Fig.2 Sequencing results of 16S rDNA of strain U5
由图2可知,扩增片段长1 406 bp,符合细菌16S rDNA序列特征。将扩增序列结果同美国国家生物技术信息中心(national center of biotechnology information,NCBI)中的Genbank核酸数据库进行基本局部比对搜索工具(basic local alignment search tool,BLAST)同源性比对分析,用MEGA-X构建系统发育树,结果见图3。

图3 菌株U5 16S rDNA序列系统发育树
Fig.3 Phylogenetic tree of strain U5 based on 16S rDNA sequence
由图3可知,菌株U5与Bacillus subtilis strain IAM10(MH815091.1)位于同一族群,亲缘关系最近,再结合形态学观察和生理生化试验结果,进一步证实菌株U5为枯草芽孢杆菌(Bacillus subtilis)。
2.3 发酵培养基组分的优化
2.3.1 单因素试验结果
分别对发酵组分中三丁酸甘油酯(1.2%~2.4%)、葡萄糖(4~10 g/L)、尿素(6~18 g/L)的添加量进行单因素试验设计,结果如图4所示。
由图4a可知,随着三丁酸甘油酯添加量的增加,脂肪酶活力呈现先升后降的趋势,当三丁酸甘油酯添加量为1.8%时,脂肪酶活力最高,达到4.0 U/mL,故将1.8%作为响应面设计中三丁酸甘油酯含量的中心点,中心点左、右两侧等距离的因变量1.6%和2.0%作为低水平和高水平。
由图4b可知,随着葡萄糖添加量的增加,脂肪酶活力整体呈现先上升后略微下降的趋势,当葡萄糖添加量为8 g/L时,脂肪酶活力达到最高;葡萄糖添加量>8 g/L后略有下降,故葡萄糖含量的中心点设为8 g/L,低水平和高水平分别为7 g/L和9 g/L。
由图4c可知,随着尿素添加量的增加,脂肪酶活力呈现先上升后下降的趋势,当尿素添加量为10 g/L时,脂肪酶活力达到最高,为3.7 U/mL;尿素添加量>10 g/L脂肪酶活力显著下降,故将10 g/L作为响应面设计中尿素含量的中心点,中心点左、右两侧等距离的因变量8 g/L和12 g/L作为低水平和高水平。
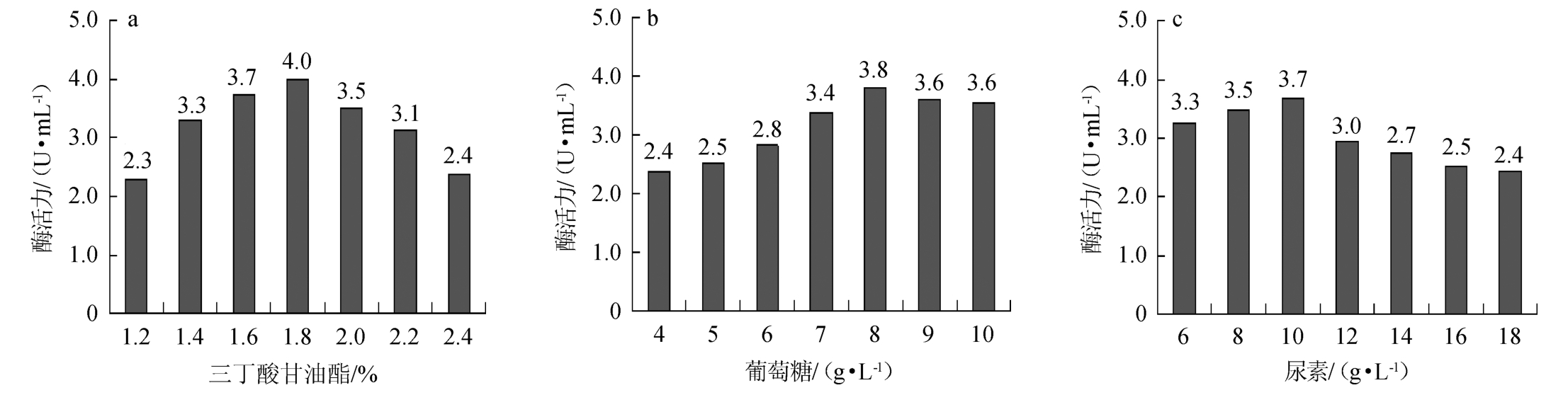
图4 三丁酸甘油(a)、葡萄糖(b)及尿素(c)添加量对脂肪酶活力的影响
Fig.4 Effects of tributyrin (a),glucose (b) and urea (c) addition on lipase activity
2.3.2 Box-Behnken Design试验结果
根据单因素确定的中心点和因素水平,运用DesignExpert 8.0.5b中BBD试验设计原理进行3因素3水平的响应面试验设计,试验设计方案和结果见表3,方差分析见表4。对表3中进行多元回归拟合,建立二阶回归模型,找到最优响应因子水平,多元二次回归方程如下所示:
表3 Box-Behnken试验设计及结果
Table 3 Design and results of Box-Behnken experiments
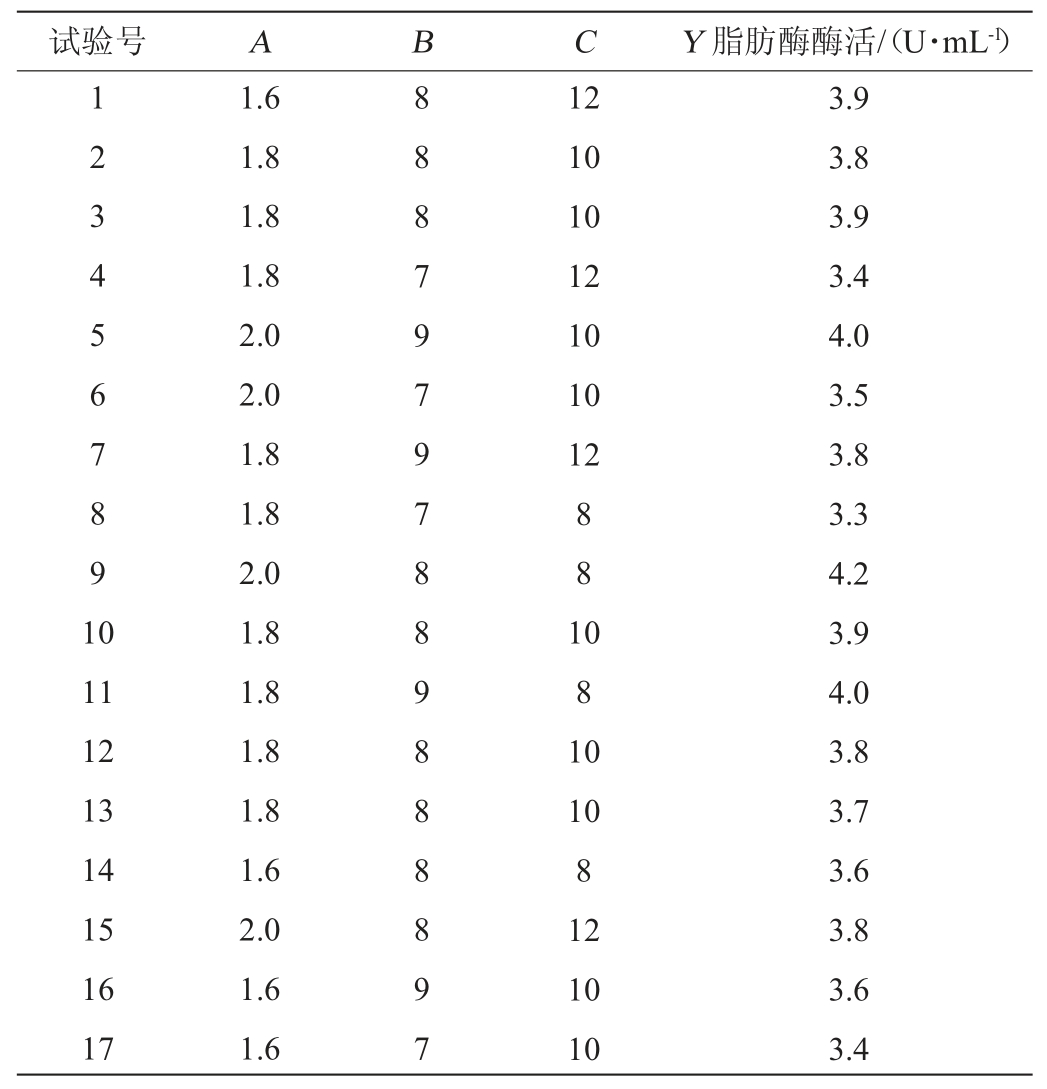
由模型的回归结果分析和方程方差分析可知,该二次模型显著(P=0.000 9<0.05),失拟项不显著(P=0.495 7>0.05),决定系数R2=0.950 1,说明有95.01%的变差能够由该模型解释;变异系数为2.21%,说明变异(偏离)程度较小。由图5可知,各因素间有一定的交互作用,最佳预测点在试验考查范围内。因此,该二阶归模型具有良好的拟合度,能真实地描述各因素与响应值之间的关系,可对菌株U5发酵产脂肪酶条件进行预测和分析。一次项A、B,交互项AC,二次项B2对结果影响极显著(P<0.01)。
表4 响应面试验回归模型方差分析
Table 4 Variance analysis of response surface experiments
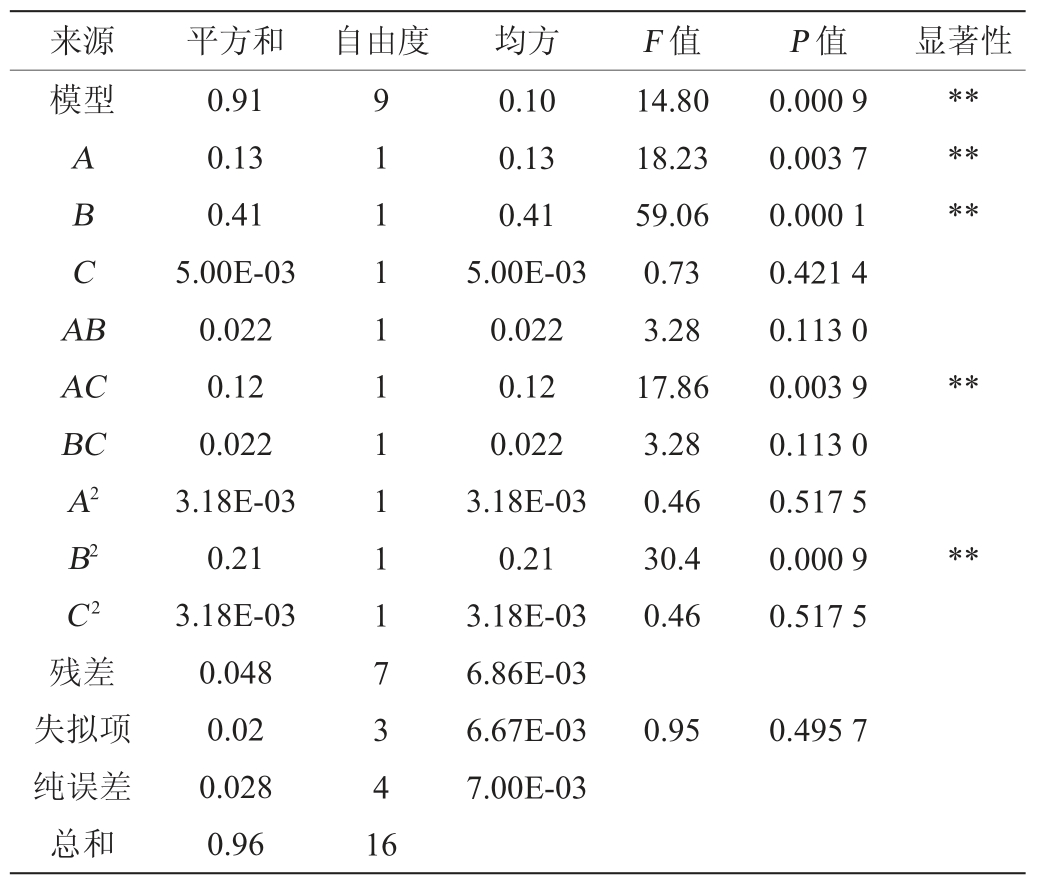
注:“**”表示对结果影响极显著(P<0.01);“*”表示对结果影响显著(P<0.05)。
由回归方程预测得到三丁酸甘油脂添加量为2.00%,葡萄糖添加量为8.84 g/L,尿素添加量为8.00 g/L,发酵获得粗脂肪酶活力预测值为4.36 U/mL。为方便实际培养基配制,选取三丁酸甘油脂添加量为2%,葡萄糖添加量为9 g/L,尿素添加量为8 g/L。在此优化培养基条件下,发酵获得粗脂肪酶活力实际值为4.30 U/mL,与预测值基本接近,说明所建模型拟合良好且可靠。因此通过响应面优化后得到的最佳发酵培养基配方为三丁酸甘油脂2%,葡萄糖9 g/L,尿素8 g/L,蛋白陈2 g,K2HPO4·3H2O 1.0 g/L,(NH4)2SO41.0 g/L,MgSO4·7H2O 0.5 g/L;发酵条件为接种量10%,初始pH值=7,温度35 ℃,转速220 r/min,摇瓶培养40 h。

图5 三丁酸甘油脂、葡萄糖及尿素添加量对发酵产脂肪酶活力影响的响应面及等高线
Fig.5 Response surface plots and contour line of effects of interaction of between tributyrin,glucose and urea addition on lipase activity
2.4 模型验证及分析
为检验模型可靠性和有效性,分别用模型预测的最优培养基配方和原始培养基配方进行摇瓶发酵,每组试验设置3个平行,检测不同取样时间时的脂肪酶活力,结果见图6。由图6可知,脂肪酶活力最高为4.3 U/mL,比未优化前的活力3.7 U/mL提高16.22%。模型的预测值与实际发酵结果十分接近说明模型预测性良好。
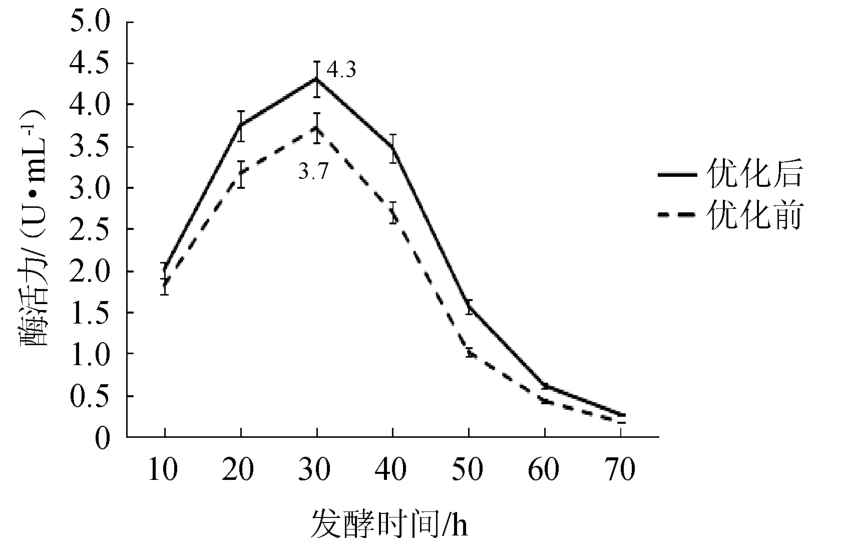
图6 培养基组分优化前后脂肪酶活力的变化
Fig.6 Changes of lipase activity before and after culture medium optimization
3 结论
研究从富油水样中筛选出一株脂肪酶产生菌株U5,经形态观察、生理生化试验及16SrDNA序列分析鉴定其为枯草芽孢杆菌(Bacillussubtilis),利用单因素及响应面试验优化,得到菌株U5发酵产脂肪酶的培养基中最佳配方为三丁酸甘油2%,葡萄糖9 g/L,尿素8 g/L,在此培养基组分下所产脂肪酶活力为4.3 U/mL,比未优化前的酶活力提高16.22%,研究可继续对U5芽孢杆菌发酵产脂肪酶的菌种进行工程化改造、发酵过程控制优化、发酵罐放大试验等方面做进一步探索,逐步提高芽孢杆菌U5产脂肪酶的能力和活力。
[1]NATHALIA S R,BRUNA B P,MAÍSA P P,et al.Biotechnological potential of lipases from Pseudomonas:Sources,properties and applications[J].Process Biochem,2018,75:99-120.
[2]智倩,王彪,薛永常.微生物脂肪酶的研究进展[J].安徽农业科学,2013,41(4):1453-1455.
[3]恽丽红.Burkholderia mana SYBC LI-1 产脂肪酶发酵条件优化及酶学性质研究[D].无锡:江南大学,2008.
[4]WESTERS L,WESTERS H,QUAX W J. Bacillus subtilis as cell factory for pharmaceutical proteins:a biotechnological approach to optimize the host organism[J].Biochim Biophys Acta,2004,1694(1-3):299-310.
[5]袁蕊,王学江,李峰.一株枯草芽孢杆菌的分离鉴定及其发酵条件优化[J].化学与生物工程,2019,36(7):35-38.
[6]DARTOIS V,BAULARD A,SCHANCK K,et al.Cloning,nucleotide sequence and expression in Escherichia coli of a lipase gene from Bacillus subtilis 168[J].Biochim Biophys Acta,1992,1131(3):253-260.
[7]韩雪,童攀.一株产脂肪酶蜡状芽孢杆菌的分离鉴定[J].江汉大学学报(自然科学版),2017,45(1):68-71.
[8]GAO J X,OU X Y,XU P,et al.Cloning,overexpression,and characterization of a novel organic solvent-tolerant lipase from Paenibacillus pasadenensis CS0611[J].Chinese J Catalys,2018,39(5):937-945.
[9]吴程雨,叶飘,杨孝婷.固定化地衣芽孢杆菌胞外脂肪酶的制备[J].浙江理工大学学报(自然科学版),2015,33(5):694-697.
[10]SCHALLMEY M,SINGH A,WARD O P.Developments in the use of Bacillus species for industrial production[J].Canad J Microbiol,2004,50(1):1-17.
[11]VAN POUDEROYEN G,EGGERT T,JAEGER KE,et al.The crystal structure of Bacillus subtilis lipase:a minimal α/β hydrolase fold enzyme[J].J Molecul Biol,2001,309(1):215-226.
[12]宋萍,戚小灵,胡燚,等.响应面法优化枯草芽孢杆菌产脂肪酶的合成培养基[J].中国生物工程杂志,2010,30(8):100-105.
[13]BADOEI-DALFARD A,KARAMI Z.Screening and isolation of an organic solvent tolerant-protease from Bacillus sp.JER02:Activity optimization by response surface methodology[J].J Mol Catal B,2013,89:15-23.
[14]JETTE J F,ZIOMEK E.Determination of lipase activity by a rhodaminetriglyceride-agarose assay[J].Anal Biochem,1994,219(2):256-260.
[15]杨军方.产脂肪酶细菌的筛选和脂肪酶性质研究[D].南京:南京理工大学,2004.
[16]施巧琴.碱性脂肪酶的研究I——菌株的分离和筛选[J].微生物学报,1981,8(3):108-110.
[17]谢清,王瑶,陈尚武,等.高产脂肪酶菌株的筛选鉴定[J].中国农业大学学报,2018,23(5):86-92.
[18]东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社,2001:253-298.
[19]SAM BROOK J,FRITSCH E F,MANIATIS T,et al.Molecular cloning:a laboratory manual 2nd[J].Quart Rev Biol,1989,49(1):411.
[20]刘延波,邢星月,赵志军,等.高产脂肪酶菌株的筛选鉴定及产酶条件优化[J].中国酿造,2019,38(7):54-59.