山西老陈醋在我国有3 000多年的历史,通过固态发酵过程中多种微生物的不断繁殖和演替,产生多种代谢产物,进而赋予产品独特的口感与风味。酒精发酵阶段,糖类物质在微生物及其酶系液化糖化的作用下发酵生成酒精等,醋酸发酵阶段,糖类和醇类物质在醋酸菌、乳酸菌等微生物的共同作用下分解代谢生成乙酸、乳酸等酸类物质,同时产生多种风味物质,形成特有的风味[1]。聂志强等[2-3]发现,乳酸菌在山西老陈醋中丰度较高,且存在于食醋酿造各个阶段,主要包括食窦魏斯氏菌(Weissella cibaria)、干酪乳杆菌(Lactobacillus casei)、瑞士乳杆菌(Lactobacillus helveticus)、植物乳杆菌(Lactobacillus plantarum)、发酵乳杆菌(Lactobacillus fermentum)、乳酸片球菌(Pediococcus acidiactici)、戊糖片球菌(Pediococcus pentosaceus)、鼠李糖乳杆菌(Lactob acillus rhamnosus)等。这些不同种类的乳酸菌产生的乳酸、琥珀酸、苹果酸等有机酸对食醋风味和品质具有重要的作用[4]。反过来,食醋酿造过程中积累的这些有机酸也会对乳酸菌产生胁迫作用。山西老陈醋固态酿造过程中总酸度最高可达5.64 g/100 mL,与之对应的pH在3.5~4.5内波动[3]。因此,研究乳酸菌的耐酸机制,对于深入理解该类微生物在山西老陈醋固态酿造过程中的作用具有重要的科学意义。
目前研究者主要对一些模式微生物(如干酪乳杆菌[5-6]、植物乳杆菌[7-9]、乳酸乳球菌(Lactococcuslactis)[8-9]、鼠李糖乳杆菌[10]、戊糖乳杆菌[11)]在单一酸压力(如盐酸[7-12]和乳酸[13)]下的耐性机制展开了研究。结果发现,乳酸菌的耐酸机制主要包括:维持胞内pH稳态[14-15];改变细胞膜的组成和流动性[16,17];脱氧核糖核酸(deoxyribonucleic acid,DNA)和蛋白质对酸耐受的保护和修复作用[18-19];糖酵解/糖异生、丙酮酸代谢等代谢调控[20-21]。目前,以来源于复杂酸性环境的山西老陈醋的乳酸菌为对象,研究其在多种酸压力下的耐性机制还未见报道。
课题组前期对山西老陈醋固态酿造过程中不同阶段(大曲;酒精发酵1d、4d、7d、10d;醋酸发酵0d、3d、5d、7d;陈酿)的样品总酸度及乳酸菌菌群的耐醋酸性能进行了初步分析。结果表明,醋酸发酵3 d和7 d的样品总酸度分别为5.24 g/100 mL和6.35 g/100 mL,乳酸菌菌群在3%(V/V)醋酸下混合培养后的培养液具有较高的OD600nm值,这暗示该菌群中的乳酸菌具有较高的酸耐受能力[22]。在此基础上,本论文对上述菌群中分离纯化得到的8株乳酸菌分离株(瑞士乳杆菌AAF3-1、干酪乳杆菌AAF3-2、乳酸片球菌AAF3-3、干酪乳杆菌AAF3-5,来源于醋酸发酵3 d;食窦魏斯氏菌AAF7-1、食窦魏斯氏菌AAF7-2、干酪乳杆菌AAF7-4、乳酸片球菌AAF7-5,来源于醋酸发酵7 d)的耐醋酸性能展开进一步分析,筛选获得了耐醋酸性能优良的乳酸片球菌,利用定量实时聚合酶链反应(quantitative real time polymerase chain reaction,qRT-PCR)对蛋白质组学技术初筛得到的在三种酸压力(醋酸、乳酸、盐酸)下表达水平均显著上调的蛋白——乙酰辅酶A羧化酶编码基因的转录水平进行检测。之后,利用过表达技术,探讨该基因对乳酸片球菌耐酸性能的影响,为山西老陈醋固态酿造过程中乳酸菌的耐酸机制和耐酸性能调控研究提供了重要的基础数据。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株和质粒
本实验所用菌株和质粒见表1。
表1 本实验所用菌株和质粒
Table 1 Strains and plasmids used in the experiment

菌株和质粒 特征及用途 来源瑞士乳杆菌(Lactobacillus helveticus)AAF3-1干酪乳杆菌(Lactobacillus casei)AAF3-2乳酸片球菌(Pediococcus acidilactici)AAF3-3干酪乳杆菌(Lactobacillus casei)AAF3-5食窦魏斯氏菌(Weissella cibaria)AAF7-1食窦魏斯氏菌(Weissella cibaria)AAF7-2干酪乳杆菌(Lactobacillus casei)AAF7-4乳酸片球菌(Pediococcus acidilactici)AAF7-5 E.coliMC1061 E.coliMC1061/pNZ8148-acc P.acidilacticiAAF3-3/pNZ8148 P.acidilacticiAAF3-3/pNZ8148-acc pNZ8148 pNZ8148-acc分离自山西老陈醋醋酸发酵3 d分离自山西老陈醋醋酸发酵3 d分离自山西老陈醋醋酸发酵3 d分离自山西老陈醋醋酸发酵3 d分离自山西老陈醋醋酸发酵7 d分离自山西老陈醋醋酸发酵7 d分离自山西老陈醋醋酸发酵7 d分离自山西老陈醋醋酸发酵7 d str.K-12 F-λ-Δ(ara-leu)7697[araD139]B/r Δ(codB-lacI)3|galK16|galE15|e14-mcrA0 relA1|rpsL150(StrR)|spoT1|mcrB1|hsdR2(r-m+)]携带pNZ8148-acc质粒的重组大肠杆菌携带pNZ8148质粒的重组乳酸片球菌携带pNZ8148-acc质粒的重组乳酸片球菌大肠杆菌-乳酸乳球菌穿梭表达载体大肠杆菌-乳酸乳球菌穿梭表达载体,携带acc基因本实验室保藏本实验室保藏本实验室保藏本实验室保藏本实验室保藏本实验室保藏本实验室保藏本实验室保藏天津科技大学罗学刚教授课题组赠予本实验构建本实验构建本实验构建江南大学张娟教授课题组赠予本实验构建
1.1.2 材料与试剂
TransStart Fast Pfu DNA Polymerase酶(2.5 U/μL):北京全式金生物技术有限公司;DL 5000 marker、PrimeScript RT reagent Kit、SYBR Premix ExTaqGC试剂盒:宝生物(大连)有限公司;细菌基因组提取试剂盒(DP302):北京天根生化科技有限公司;Plasmid Mini Kit I、Gel Extraction Kit、Cycle-Pure Kit:美国Omega公司;EastepTM总RNA提取试剂盒:上海普洛麦格生物有限公司;ClonExpress One Step Cloning Kit:南京诺唯赞生物科技有限公司;其余试剂为生化试剂或化学纯试剂,均购自天津市福晨化学试剂厂。
1.1.3 培养基
LB液体培养基:胰蛋白胨10 g,酵母提取物5 g,NaCl 10 g,蒸馏水1 000 mL,调节pH 7.2,121℃灭菌20 min。
LB固体培养基:LB液体培养基中加入20 g/L的琼脂,121℃灭菌20 min。
MRS液体培养基:葡萄糖20 g,蛋白胨10 g,牛肉提取物10 g,酵母提取物5 g,无水乙酸钠5 g,吐温80 1mL,柠檬酸铵2 g,磷酸氢二钾2 g,硫酸镁0.58 g,硫酸锰0.25 g,调节pH至6.2~6.8,蒸馏水1 000 mL,121℃灭菌20 min。
MRS固体培养基:MRS液体培养基中加入20 g/L的琼脂,121℃灭菌20 min。
复苏培养基:MRS液体培养基中加入0.5mol/L的蔗糖,115℃灭菌20 min。
1.2 仪器与设备
GHP-9270恒温培养箱:上海一恒科技有限公司;GI54D高压蒸汽灭菌锅:美国Zealway公司;DYY-2C电泳仪、Gel Doc XR凝胶成像仪:美国Bio-Rad公司;ECM 399高压脉冲电转仪:北京市六一仪器厂;PHS-3C标准pH计:上海梅特勒-托利多仪器有限公司;UVmini-1240紫外分光光度计:上海分析仪器厂;SW-CJ-1FD超净工作台:苏州安泰空气技术有限公司;Bioscreen C全自动生长曲线分析仪:芬兰Bioscreen公司;ABI Step-One Plus Real Time PCR System:美国Applied Biosystems公司。
1.3 方法
1.3.1 乳酸菌菌株的活化和培养
从甘油管中分别吸取菌液,以2%(V/V)接种量接种到装有5 mL MRS培养基的试管中,37℃恒温箱中静置活化培养8h。以2%(V/V)接种量将上述培养液转接入装有新鲜的5 mL MRS液体培养基的试管中,37℃恒温箱中静置进行一级培养,培养8 h后菌体生长进入对数期(OD600nm值≈1.0)。以1%(V/V)的接种量将一级培养液接入装液量为100 mL/250 mL的MRS液体培养基中,37℃恒温箱中静置进行二级培养。对于工程菌株,培养基中需要加入10μg/mL的氯霉素维持质粒的稳定性。
1.3.2 乳酸菌分离株的生长曲线测定
按照1.3.1的方法进行菌株的活化和一级培养,在二级培养初始将OD600nm值调节为0.1,取300 μL培养液接入杀菌消毒的微生物生长板的微孔中,每株菌设三个平行,放入全自动生长曲线分析仪中,设置温度环境37℃,设置菌体浓度检测频率30 min,培养至微生物生长稳定期。
1.3.3 乳酸菌分离株的耐酸性能分析
菌株在适当酸压力下的生长性能分析:按照1.3.1的方法进行菌株的活化和一级培养,在二级培养的初始(OD600nm值调节为0.1)向培养基中加入不同的酸压力(包括2%、3%(V/V)醋酸;1%、2%(V/V)乳酸;pH 5.0、4.0、3.5、3.0(盐酸调节)),37℃恒温箱中静置培养,于不同时间取样,利用紫外分光光度计测定OD600 nm值或者采用平板活菌计数法测定培养24 h时的活菌数,并以不添加压力条件作为空白对照,计算相对活菌数,其计算公式如下:
菌株在高浓度酸压力冲击下的存活性能分析:按照1.3.1的方法进行菌株的活化、一级和二级培养,待二级培养至OD600 nm值≈1.0(约8 h)时收集菌体(5 000 r/min、10 min、4℃),并将其重悬于5 mL MRS液体培养基中。将其分成两份,分别接种到添加压力(包括含5%(V/V)醋酸,3%(V/V)乳酸,pH 2.5(盐酸调节))和不添加压力的装有新鲜的50mLMRS液体培养基的250 mL三角瓶中。37℃静置培养,于不同时间取样,利用平板活菌计数法37℃恒温箱中培养36~48 h,测定活菌数,计算相对活菌数,或稀释不同梯度后拍照观察菌落的存活情况。
1.3.4 基因转录水平分析
按照1.3.3的方法将菌株在5%(V/V)醋酸、3%(V/V)乳酸、pH 2.5(盐酸调节)条件下冲击90 min后,收集菌体(6 000 r/min、4℃、10 min)。按照EastepTM总RNA提取试剂盒的说明书提取总RNA。按照PrimeScript real-time reagent Kit说明书以RNA为模板进行反转录生成cDNA。设计上游引物5'-CTTACCCAGGAATGGATGCTGAATA-3',下游引物5'-CAATTACGCTGATGTATGGTACCCG-3',根据SYBR Premix ExTaqGC试剂盒的说明书,利用ABI Step-One Plus Real Time PCR System仪器进行实时荧光定量PCR。以编码16S rRNA的基因作为内参,通过比较Ct(2-ΔΔCt方法)计算基因相对表达水平,然后归一化至未经压力处理的条件,每个样品设3个平行,结果取平均值,并用标准偏差显示其数据的波动。
1.3.5 重组质粒的构建
(1)克隆载体的线性化
线性化克隆载体由反向PCR扩增制备。上游引物5'-TCTAGAGAGCTCAAGCTTTC-3',下游引物5'-CCATGGTGAGTGCCTCCTTA-3',反应条件:95 ℃、3 min;95 ℃、10s,55℃、15s,72℃、3min30s,30个循环,使用1%的琼脂糖凝胶核酸电泳对PCR产物进行验证。之后加入1μL快切酶DpnI,37℃酶切2 h,进行回收。
(2)目的基因的获得
按照G+细菌基因组DNA提取试剂盒的说明书提取乳酸片球菌AAF3-3的基因组DNA。用核酸分析仪测定DNA的浓度,通过1%琼脂糖凝胶核酸电泳检测,-20℃保存,备用。以基因组为模板,利用上游引物5'-TAAGGAGGCACTCA CCATGGGGATGACAAAAACAGCATATGAAACGG-3',下游引物5'-GAAAGCTTGAGCTCTCTAGACTAAAATTT ACTAAACCGTTCGTGGC-3'扩增基因acc。该方法应用同源重组原理连接,引物由与线性化质粒两端互补的碱基序列(下划线部分)和与目的基因两端互补的碱基序列(其余部分)两部分构成。PCR反应条件见1.3.5(1),使用1%琼脂糖凝胶核酸电泳对PCR产物进行验证。
(3)重组质粒的构建
按照ClonExpress One Step Cloning Kit的说明书配制连接体系。反应条件:37℃、30 min。将连接产物采用化转入E.coliMC1061感受态中。挑取转化子,提取质粒后进行PCR和Hind III单酶切验证。选择条带大小正确的重组质粒送到金唯智生物科技有限公司进行测序。
1.3.6 过表达菌株的构建
参照RODRÍGUEZMC等[23]的方法制备感受态细胞。具体过程为菌株二级培养的初始加入40mmol/L苏氨酸,37℃恒温静置培养8 h至OD600nm值≈1.0,收集菌体(6 000 r/min、4℃、10 min),用含7 mmol/L磷酸钾缓冲液、1 mmol/L氯化镁的0.5 mol/L蔗糖溶液洗涤三次,然后再用2 000 U/mL的溶菌酶重悬菌体,37℃、20 min,重复3次,最后用含10%甘油的0.5 mol/L蔗糖溶液重悬分装。
将测序正确的重组质粒电转(2 kV/cm,5 ms)进入乳酸片球菌AAF3-3的感受态细胞中,复苏培养基中37℃静置培养2 h后涂布于含10 μg/mL氯霉素的MRS固体平板上。37℃恒温培养箱中培养36~48 h后随机挑取转化子,通过质粒PCR和Hind III单酶切进行验证。实验中,以含有空质粒的重组菌株作为对照菌株。
2 结果与分析
2.1 乳酸菌分离株在无压力下的生长性能
山西老陈醋醋酸发酵3d的4株乳酸菌分离株(AAF3-1、AAF3-2、AAF3-3、AAF3-5)和7d的4株乳酸菌分离株(AAF7-1、AAF7-2、AAF7-4、AAF7-5)在无压力下的生长曲线见图1。由图1可知,8株菌的生长速率和生理阶段划分没有明显差异,均存在明显的延滞期(0~2 h)、对数期(2~12 h)、稳定期(12~24 h),培养24 h后的最终OD600nm值在1.2~1.4之间波动。这表明上述8株乳酸菌分离株在无压力下的生长性能没有明显差异。
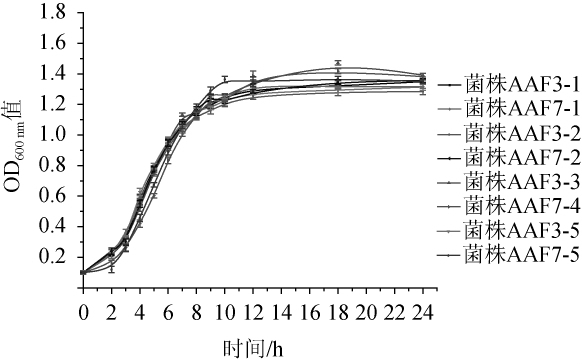
图1 来源于醋酸发酵3 d和7 d的乳酸菌分离株在无压力下的生长曲线
Fig.1 Growth curves of lactic acid bacteria isolated from acetic acid fermentation for 3 d and 7 d under non-stress condition
2.2 耐醋酸性能优良乳酸菌的筛选
2.2.1 耐醋酸性能优良乳酸菌的初筛
8株乳酸菌分离株在2%(V/V)醋酸下的生长性能见图2。由图2(a)可知,8株乳酸菌分离株在2%(V/V)醋酸下没有明显的延滞期,分离株AAF3-3、AAF3-1、AAF3-5不仅具有比其他5个分离株更快的生长速率,而且具有更长的对数期(2~12 h)。分离株AAF3-3、AAF3-1、AAF3-5培养12 h后进入稳定期,菌液OD600nm值分别达到了0.46、0.37、0.33,而其他5个分离株则在培养5h后生长趋于平缓,菌液OD600nm值在0.22~0.28之间波动。为更好的反映菌株在醋酸压力条件下的生长情况,进一步选取上述8株菌培养24 h后的菌液进行活菌数测定并计算相对活菌数。由图2(b)可知,AAF3-3、AAF3-5、AAF3-1的相对活菌数较高,分别为0.19%、0.18%、0.13%,而分离株AAF7-5、AAF3-2、AAF7-1、AAF7-2、AAF7-4的相对活菌数较低(均<0.05%)。综合上述结果,选取AAF3-1、AAF3-3、AAF3-5用于后续复筛。
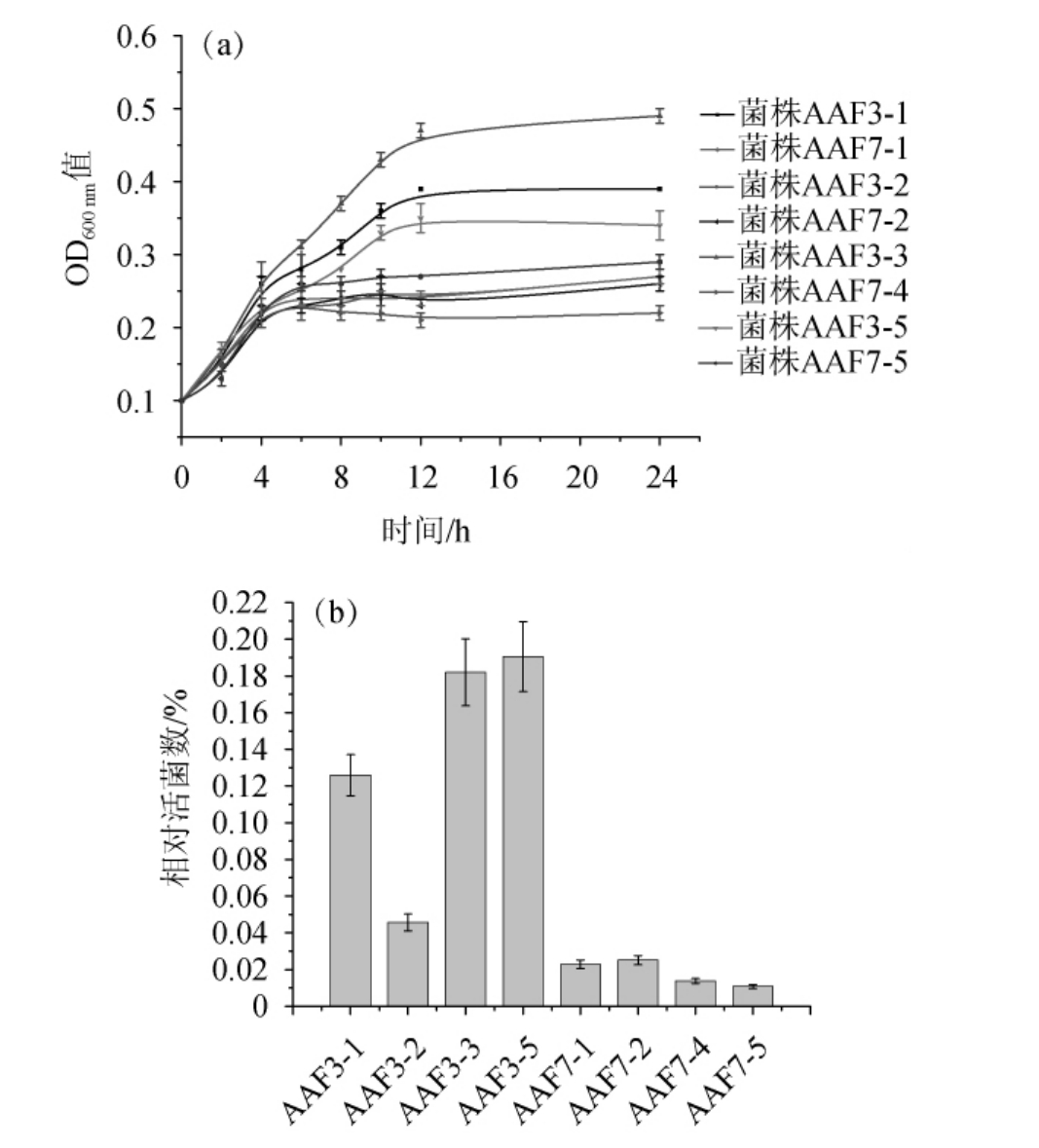
图2 8株乳酸菌株在2%(V/V)醋酸下的生长曲线(a)和24 h时的相对活菌数(b)
Fig.2 Growth curves(a)and relative number of viable bacteria at 24 h(b)of eight lactic acid bacteria under 2%(V/V)acetic acid stress
2.2.2耐醋酸性能优良乳酸菌的复筛
醋酸发酵3 d的总酸度为5.24 g/100 mL,体积比约为3%(V/V)醋酸。因此,进一步考察了AAF3-1、AAF3-3、AAF3-5三个菌株在3%(V/V)醋酸压力的生长性能见图3。由图3(a)可知,AAF3-3培养6 h后菌液的相对活菌数为0.26%,比AAF3-1(0.066%)和AAF3-5(0.14%)分别高出315.01%和98.65%。随着培养时间的延长,三个菌株的相对活菌数均明显下降,其中,AAF3-1和AAF3-5的相对活菌数分别在培养12h和24h后下降为0,而AAF3-3在培养24 h仍保持0.001%的相对活菌数。由图3(b)可知,5%(V/V)醋酸冲击30 min后,AAF3-3的相对活菌数为10.67%,相比AAF3-1(0.016%)和AAF3-5(0.29%)分别高出651.93倍和34.92倍。随着冲击时间的延长,3个菌株的相对活菌数明显下降,其中乳酸片球菌AAF3-3在90 min时仍保持将近0.08%的相对活菌数,而AAF3-1、AAF3-5的相对活菌数在冲击60 min和90 min后分别变为0。因此,3个乳酸菌分离株的耐醋酸性能由高到低排序为AAF3-3>AAF3-5>AAF3-1,选取乳酸片球菌AAF3-3作为后续研究对象。
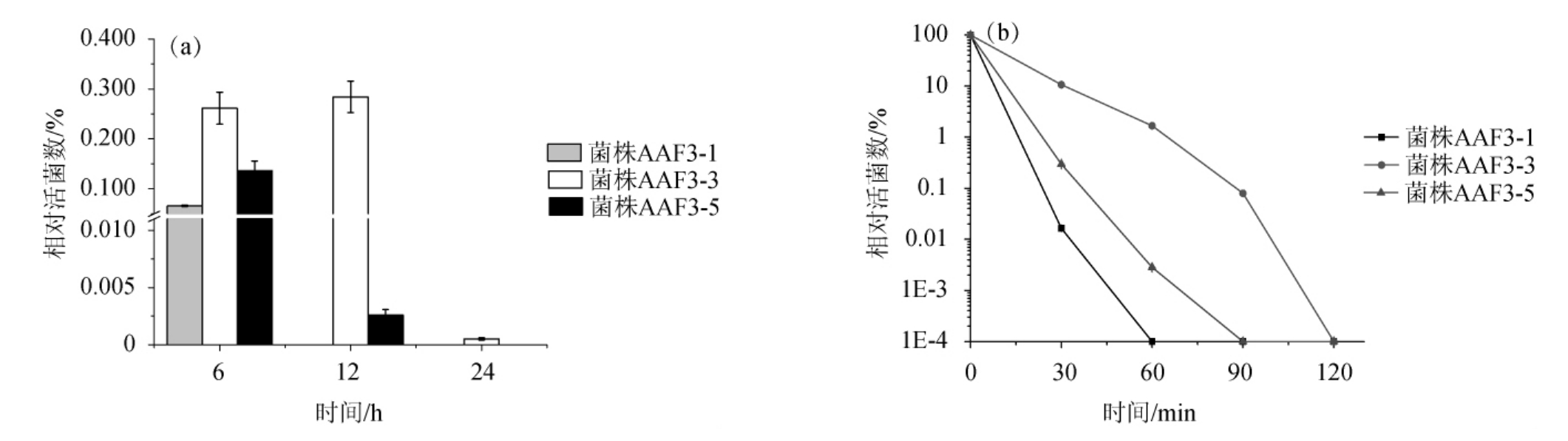
图3 三个乳酸菌分离株在3%(V/V)醋酸压力下的生长性能(a)和5%(V/V)醋酸冲击下的存活性能(b)
Fig.3 Growth performance of three lactic acid isolates under 3%(V/V)acetic acid stress(a)and survival performance shocked by 5%(V/V)acetic acid stress(b)
2.3 乳酸片球菌AAF3-3对其他酸压力的耐受性能
考虑到乳酸片球菌AF3-3是来源于复杂酸环境的食醋固态酿造过程,因此,进一步考察了该菌株对其他酸压力的耐受性能。
2.3.1 乳酸片球菌AAF3-3在乳酸压力下的耐酸性能
由图4(a)可知,菌株AAF3-3在1%(V/V)乳酸下的相对活菌数随着培养时间的延长呈现增长趋势,其中,培养6 h的相对活菌数为5.61%,培养24 h后增长为9.01%,这说明,AAF3-3在1%的乳酸压力下保持良好的生长性能。当乳酸浓度提高至2%(V/V),培养6 h和12 h后的AAF3-3菌液的相对活菌数为0.51%和0.11%,而24 h后下降为0。由图4(b)可知,AAF3-3在3%(V/V)乳酸下冲击30 min后的相对活菌数为29.84%。随着冲击时间的延长,其相对活菌数明显下降,当冲击120 min时,相对活菌数仅为0.0005%。这些结果表明,乳酸片球菌AAF3-3对2%(V/V)和3%(V/V)的乳酸压力表现出一定的耐受性。

图4 乳酸片球菌AAF3-3在1%、2%(V/V)乳酸压力下的生长性能(a)和3%(V/V)乳酸冲击下的存活性能(b)
Fig.4 Growth performance of P.acidilactici AAF3-3 under 1%,2%(V/V)lactic acid stress(a)and survival performance shocked by 3%(V/V)lactic acid stress(b)
2.3.2乳酸片球菌AAF3-3在盐酸压力下的耐酸性能
由图5(a)可知,菌株AAF3-3在pH 5.0下的相对活菌数随着培养时间的延长呈现缓慢上升的趋势。这说明AAF3-3在pH 5.0的酸压力下保持一定的生长能力。当pH降至4.0以下时,随着培养时间的延长,菌株的相对活菌数呈现下降趋势。其中pH 3.5条件下,相对活菌数由6 h的1.08%急剧下降为24 h的0.32%,下降了237.50%。pH 3.0条件下,培养6 h后菌液相对活菌数为0.63%,而培养24h后降为0.0013%。这说明pH<4.0时,盐酸对菌株产生一定的毒害作用。图5(b)表明,AAF3-3在pH 2.5条件下冲击30 min时相对活菌数仍保持在25.98%,而后相对活菌数急剧下降,120 min时仅为0.005 9%。因此,AAF3-3在pH 3.0和pH 2.5的盐酸压力下表现出一定的耐受性。
DONATIEN K等[24]对来源于非洲baobab树种子发酵液中的乳酸片球菌进行了分离纯化,并通过检测菌株在pH 2.5条件下冲击不同时间后的活菌数对其耐酸性能进行了分析。结果表明,野生型乳酸片球菌菌株L87在pH 2.5条件下呈现缓慢上升的趋势,对数活菌数由0 h的6.20逐渐增长至2 h的6.69,之后不再增长,而分离菌株L169在pH 2.5条件下的生长则受到了抑制,其对数活菌数由0 h的6.32下降至4 h的6.08。这些结果和本论文的结果共同说明,乳酸片球菌对酸压力表现出一定的耐受性能。
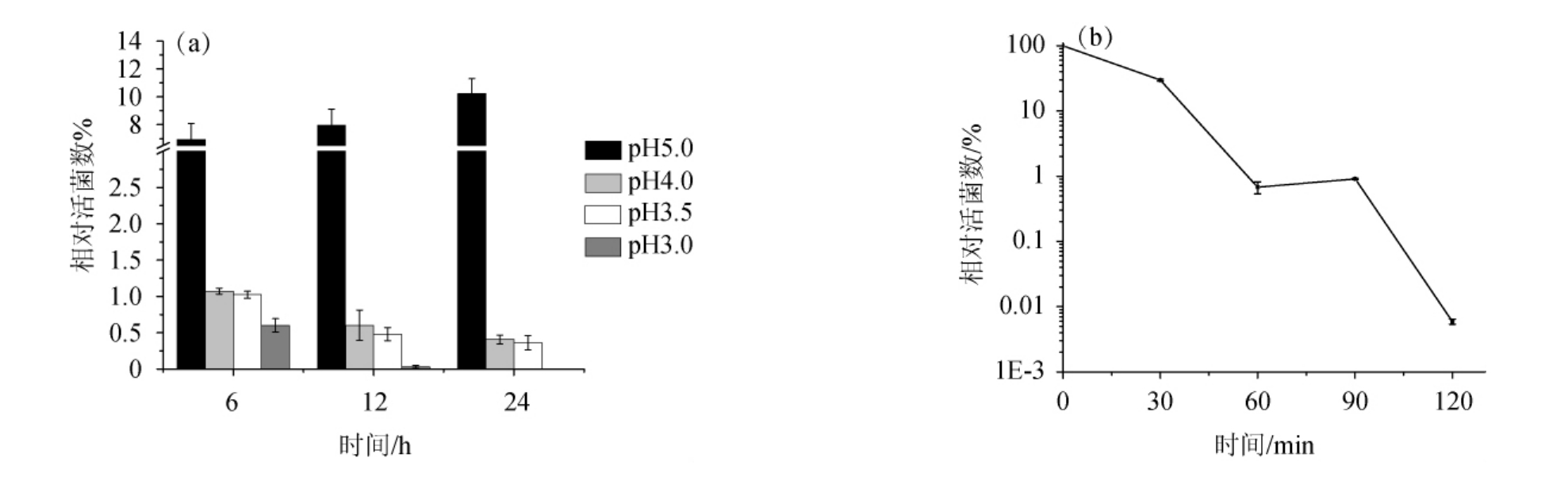
图5 乳酸片球菌AAF3-3在pH 5.0、4.0、3.5、3.0(盐酸调节)下的生长性能(a)和pH 2.5(盐酸调节)下的存活性能(b)
Fig.5 Growth performance ofP.acidilactici AAF3-3 under pH 5.0,4.0,3.5,3.0(adjusted by hydrochloric acid)(a)and survival performance under pH 2.5(adjusted by hydrochloric acid)(b)
2.4 acc在不同种类酸压力下的转录水平
课题前期的蛋白组学工作中,发现在醋酸、乳酸、盐酸三种压力高浓度冲击后,乳酸片球菌AAF3-3中乙酰辅酶A羧化酶(编码基因为acc)的蛋白表达水平较无压力条件下均出现了显著上调,分别为1.26、2.35和2.45倍。乙酰辅酶A羧化酶是催化“乙酰辅酶A+ATP+HCO3-→丙二酰辅酶A+ADP+Pi”反应的生物素酶。此反应制约着脂肪酸合成第一阶段的速度,是脂肪酸合成的限速酶[25],其底物乙酰辅酶A和产物丙二酰辅酶A也分别在丙酮酸代谢和三羧酸循环中扮演重要作用。利用qRT-PCR的方法对acc基因在三种酸压力下的转录水平进行了检测,结果见图6。由图6可知,acc基因在5%(V/V)醋酸、3%(V/V)乳酸、pH 2.5(盐酸)三种压力高浓度冲击下的转录水平均出现了显著上调,分别是菌株在无压力条件下转录水平的4.32倍、2.02倍和1.13倍,这一结果与蛋白组学结果一致,也暗示了该基因在菌株的耐酸机制中发挥着重要作用。
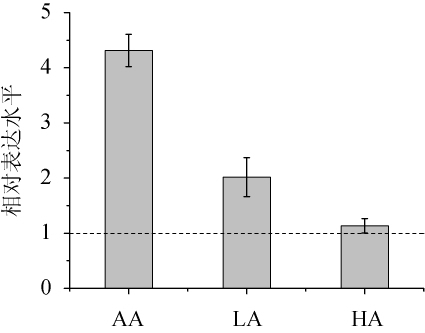
图6 基因acc在三种酸压力冲击下的转录水平
Fig.6 Transcription level ofaccafter shocked by high concentration acid stresses
2.5 过表达菌株P.acidilactici/pNZ8148-acc的构建
如图7(a)所示,重组质粒PCR在770 bp左右出现条带,与基因acc的理论大小768bp相符合。如图7(b)所示,质粒单酶切在4000bp左右出现条带,相比空质粒pNZ8148(3167bp)高出约770 bp,与目的基因acc(768 bp)理论大小相符。测序结果进一步证明,重组质粒pNZ8148-acc构建成功。将验证正确的重组质粒电转入乳酸片球菌菌株中,通过质粒PCR和Hind III单酶切进一步验证过表达菌株P.acidilactici/pNZ8148-acc构建成功。
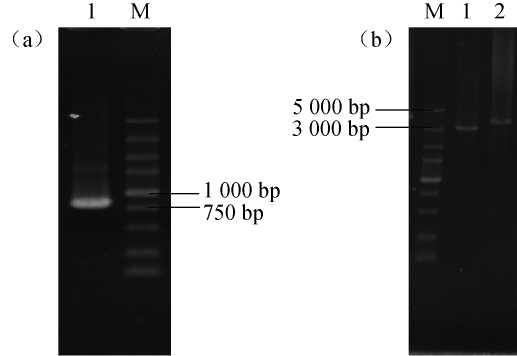
图7 重组质粒PCR(a)和Hind III单酶切(b)验证
Fig.7 Identification of PCR(a)and single restriction enzyme Hind III digestion(b)of recombinant plasmid
M:DL 5 000 bp DNA Marker;(a):Lane 1:PCR产物;(b):Lane 1:pN8148酶切产物,Lane 2:pNZ8148-acc酶切产物
2.6 过表达菌株P.acidilactici/pNZ8148-acc的耐酸性能
对上述构建成功的过表达菌株在适当酸压力下(包括3%(V/V)醋酸、2%(V/V)乳酸、pH 3.0(盐酸调节))的生长性能和高浓度酸冲击下(包括5%(V/V)醋酸,3%(V/V)乳酸和pH 2.5(盐酸调节))的存活性能进行分析。由图8(a)可知,过表达菌株在3%(V/V)醋酸下培养6 h和24 h的相对活菌数分别为12.93%和0.04%,相比对照菌株分别提高了46.10%和24倍。如图9(a)所示,过表达菌株经5%(V/V)醋酸冲击90 min后及冲击180 min后的活菌数仍分别保持在104、103数量级,而对照菌株则分别保持在102数量级及没有明显存活。这些结果表明,acc基因对乳酸片球菌的醋酸耐受性具有正向调控作用。
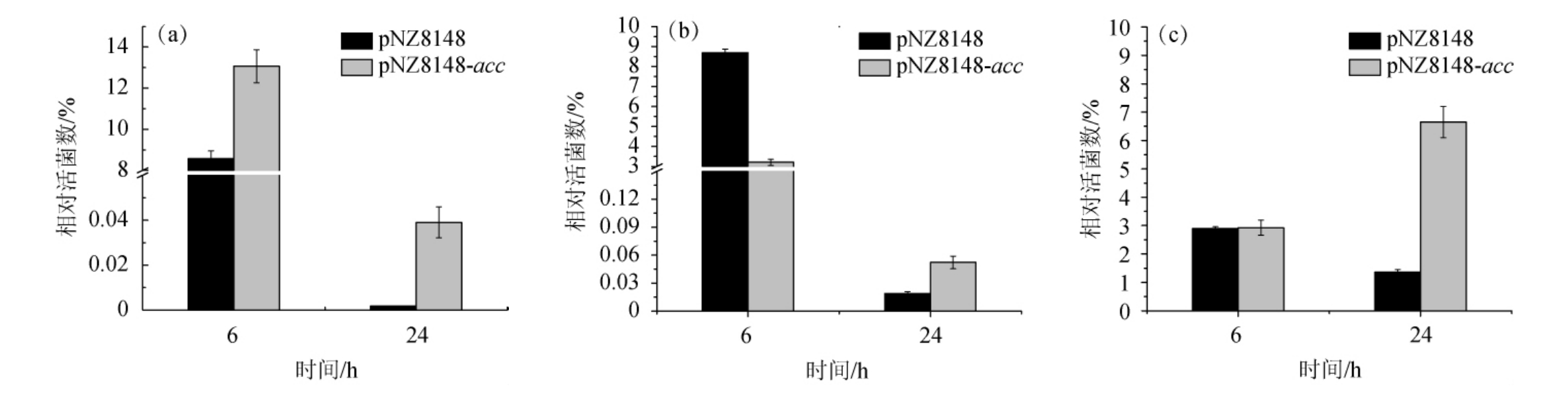
图8 过表达菌株P.acidilactici/pNZ8148-acc在3%(V/V)醋酸压力(a)、2%(V/V)乳酸压力下(b)以及pH 3.0(盐酸调节)下的生长性能(c)
Fig.8 Growth performance ofP.acidilactici/pNZ8148-accunder 3%(V/V)acetic acid stress(a),2%(V/V)lactic acid stress(b)and pH 3.0(adjusted by hydrochloric acid)(c)

图9 过表达菌株P.acidilactici/pNZ8148-acc在3%(V/V)醋酸压力(a)、2%(V/V)乳酸压力下(b)以及pH 3.0(盐酸调节)下的存活性能(c)
Fig.9 Survival performance ofP.acidilactici/pNZ8148-accunder 3%(V/V)acetic acid stress(a),2%(V/V)lactic acid stress(b)and pH 3.0(adjusted by hydrochloric acid)(c)
由图8(b)和图9(b)可知,2%(V/V)乳酸压力下,P.acidilactici/pNZ8148-acc培养6 h时的生长性能不及对照菌株,推测可能是还未适应该环境。而培养24 h后的相对活菌数为0.46%,比对照菌株(0.17%)高出170.59%。在3%(V/V)乳酸下,随着冲击时间的延长,P.acidilactici/pNZ8148-acc在90 min后活菌数比对照菌株高出1个数量级,冲击时间延长到180 min后,过表达菌株仍能保持103的活菌数,而对照菌株的菌落数极少。这表明,基因acc可以明显改善菌株对乳酸压力的耐受性。
由图8(c)可知,P.acidilactici/pNZ8148-acc在pH3.0下培养6 h时与对照菌株生长性能无明显差别,培养24 h的相对活菌数(6.61%)较对照菌株(1.47%)高出349.66%。由图9(c)可知,pH 2.5下冲击90 min后,P.acidilactici/pNZ8148-acc无活菌数,而对照菌株仍保持在103数量级。冲击180 min后P.acidilactici/pNZ8148-acc与对照菌株均未有存活。表明基因acc可以在一定pH范围内改善菌株对盐酸压力的耐受性。
综上所述,acc基因的过表达可以显著改善菌株P.acidilactici/pNZ8148-acc在醋酸、乳酸、盐酸压力下的耐受性能,这证明acc基因是能够影响乳酸片球菌多种耐酸性能的关键基因。XIAN M等[26]通过acc基因的过表达和敲除发现,该基因在大肠杆菌对盐(6%NaCl)和碱压力(pH9)耐受实验中发挥着重要的正向调控作用。本论文的结果与该基因提高大肠杆菌耐受性能的结果是相一致的,这是acc基因在乳酸菌耐酸性能中发挥重要作用的首次报道。
3 结论
本研究从山西老陈醋酿造过程中醋酸发酵阶段筛选出一株对醋酸具有一定耐受性的乳酸片球菌AAF3-3,在培养6 h和24 h后菌液的相对活菌数分别为0.26%和0.001%。该菌株对其他的酸压力(如乳酸、盐酸)也表现出耐受性,其中在3%(V/V)乳酸和pH 2.5(盐酸调节)冲击120 min后相对活菌数分别为0.000 5%和0.005 9%。以该菌株为对象,通过转录水平分析、过表达菌株构建及其耐酸性能发现,基因acc在醋酸、乳酸、盐酸下转录水平均发生明显上调。乙酰辅酶A羧化酶对菌株在多种酸压力(醋酸、乳酸、盐酸)下的耐受性能具有重要的正向调控作用,其中3%(V/V)乳酸冲击180 min后,过表达菌株的存活数仍能达到104以上,而对照菌株没有存活。研究成果为乳酸片球菌耐酸机制的研究和耐酸性能的调控提供了重要的基础数据和理论支持。
[1]曹珊.山西老陈醋醋酸发酵阶段乳酸菌乳酸合成代谢分析[D].天津:天津科技大学,2018.
[2]聂志强,韩玥,郑宇,等.宏基因组学技术分析传统食醋发酵过程微生物多样性[J].食品科学,2013,34(15):198-203.
[3]郭旭凯,杨玲,刘聪,等.山西老陈醋酿造过程中乳酸菌和醋酸菌的分离鉴定及高粱单宁对其生长的影响[J].中国酿造,2018,37(8):37-44.
[4]杜宏福,董爱静,聂志强,等.电子舌分析山西老陈醋固态发酵过程及主要有机酸的预测[J].食品与发酵工业,2015,41(1):196-201.
[5]WU C D,ZHANG J,WANG M,et al.Lactobacillus casei combats acid stress by maintaining cell membrane functionality[J].J Ind Microbiol Biotechnol,2012,39(7):1031-1039.
[6]BROADBENT J R,LARSEN R L,DEIBEL V,et al.Physiological and transcriptional response of Lactobacillus casei ATCC 334 to acid stress[J].J Bacteriol,2010,192:2445-2458.
[7]TIAAN H,SHELLY D,SALOME S.Proteomic profiling of the acid stress response in Lactobacillus plantarum423[J].Proteome Res,2014,13:4028-4039.
[8]TRIP H,MULDER N,LOLKEMA J S.Improved acid stress survival of Lactococcus lactis expressing the histidine decarboxylation pathway of Streptococcus thermophilus CHCC1524[J].J Biol Chem,2012,287(14):11195-11204.
[9]HAO P,LIANG D,CAO L,et al.Promoting acid resistance and nisin yield of Lactococcus lactis F44 by genetically increasing D-Asp amidation level inside cell wall[J].Appl Microbiol Biotechnol,2017,101(15):6137-6153.
[10]KOPONEN J,LAAKSO K,KOSKENNIEMI K,et al.Effect of acid stress on protein expression and phosphorylation in Lactobacillus rhamnosus GG[J].J Proteomics,2012,75(4):1357-1374.
[11]MONTORO B P,BENOMAR N,CABALLERO G,et al.Proteomic analysis of Lactobacillus pentosus,for the identification of potential markers involved in acid resistance and their influence on other probiotic features[J].Food Microbiol,2018,72:31-38.
[12]HEUNIS T,DEANE S,SMIT S,et al.Proteomic profiling of the acid stress response in Lactobacillus plantarum 423[J].J Proteome Res,2014,13(9):4028-4039.
[13]WU C,HE G,ZHANG J.Physiological and proteomic analysis of Lactobacillus casei in response to acid adaptation[J].J Ind Microbiol Biotechnol,2014,41(10):1533-1540.
[14]VOSSENBERG VLCM,DRIESSEN A,ZILLIG W,et al.Bioenergetics and cytoplasmic membrane stability of the extremely acidophilic,thermophilic archae on Picrophilus oshimae[J].Extremophiles,1998,2:67-74.
[15]GOLYSHINA O V,TIMMIS K N.Ferroplasma and relatives,recently discovered cell wall-lacking archaea making a living in extremely acid,heavymetal-richenvironments[J].Environ Microbiol,2005,7:1277-1288.
[16]KULLEN M J,KLAENHAMMER T R.Identification of the pH-inducible,proton-translocating F1F0-ATPase(atpBEFHAGDC)operon of Lactobacillus acidophilusby differential display,gene structure,cloning and characterization[J].Mol Microbiol,1999,33(6):1152-1161.
[17]STREIT F,DELETTRE J,CORRIEU G,et al.Acid adaptation of Lactobacillus delbrueckii subsp.bulgaricus induces physiological responses at membrane and cytosolic levels that improves cryotolerance[J].J Appl Microbiol,2008,105(4):1071-1080.
[18]COTTER P D,HILL C.Surviving the acid test:responses of gram-positive bacteria to low pH[J].Microbiol Mol Biol Rev,2003,67:429-53.
[19]ANGELIS M,BINI L,PALLINI V,et al.The acid-stress response in Lactobacillus sanfranciscensis CB1[J].Microbiology,2001,147:1863-73.
[20]CIARAMELLA M,NAPOLI A,MOSÈ R.Another extreme genome:how to live at pH 0[J].Trend Microbiol,2005,13:49-51.
[21]COTTER P D,GAHAN C G,HILL C.A glutamate decarboxylase system protects Listeriamonocytogenes in gastric fluid[J].Mol Microbiol,2001,40(2):465-475.
[22]牛纪伟.山西老陈醋酿造过程中乳酸菌的分离鉴定与多样性分析[D].天津:天津科技大学,2015.
[23]RODRÍGUEZ M C,ALEGRE M T,MESAS J M.Optimization of technical conditions for the transformation of Pediococcus acidilactici P60 by electroporation[J].Plasmid,2007,58(1):44-50.
[24]DONATIEN K,HAGRETOU S L,MAMOUDOU H D,et al.Acid resistance,bile tolerance and antimicrobial properties of dominant lactic acid bacteria isolated from traditional"maari"baobab seeds fermented condiment[J].Afr J Biotechol,2012,11(5):1197-1206.
[25]李洁琼,郑世学,喻子牛,等.乙酰辅酶A羧化酶:脂肪酸代谢的关键酶及其基因克隆研究进展[J].应用与环境生物学报,2011,17(5):753-758.
[26]XIAN M,ZHAI L,ZHONG N,et al.A novel gene(Aa-accA)encoding acetyl CoA carboxyltransferase α-subunit of Alkalimonas amylolytica N10 enhances salt and alkali tolerance of Escherichia coliand tobacco BY-2 cells[J].Acta Microbiol Sinica,2013,53(8):809-816.