白酒中关键风味物质的微生物溯源是白酒研究的一项重要工作,其研究成果的应用对白酒产量和品质的保障和提升具有明显的作用[1-4]。但白酒酿造过程是开放的多菌种混合固态发酵体系,其复杂的微生物结构和风味组成使得对产目标风味物质微生物的溯源十分困难。目前主要是通过可培养手段大量获得微生物资源并逐个进行功能验证开展溯源研究[5-8],但该过程存在工作量大,目标性差,筛选结果不可预知等不足。
随着生物技术的发展,针对微生物的研究手段已经从细胞水平转向了分子水平的研究[9]。在此过程中关于核酸、蛋白质等生物信息数据大量积累,形成了包含有众多生物资源信息的数据库,依据资源信息类别与研究用途不同,生物信息学数据库可分为15个大类[10]。目前数据库中的生物信息资源已被广泛应用于微生物研究领域,为微生物鉴定、基因水平溯源、化合物合成及致病基因研究提供了有力支持[11-12]。迄今为止,利用生物信息数据资源指导风味物质的微生物溯源尚鲜见报道。
高级醇是我国传统白酒的芳香组分之一,能够衬托酒体酯类香气,但含量过高又会引起酒体辛辣,使人上头易醉[13]。正丙醇作为高级醇的重要组成部分,其含量已成为我国酒精类食品检测的一项重要指标[14]。目前关于白酒中产正丙醇微生物研究主要集中在酵母菌[15-17]与芽孢杆菌[14,18],而白酒中其他微生物代谢产正丙醇的相关研究报道较少。该研究拟利用生物信息数据资源对白酒中产正丙醇微生物进行溯源,并对菌株进行发酵实验验证其产正丙醇能力。通过筛选白酒酿造中高产正丙醇的优势微生物,明确酿造过程中产正丙醇的微生物种类及组成,为白酒中正丙醇的调控奠定研究基础,为后期工艺控制提供理论指导。
1 材料与方法
1.1 材料与试剂
布赫内氏乳杆菌(Lactobacillus buchneri)、面包乳杆菌(Lactobacillus panis)、短乳杆菌(Lactobacillus brevis)、桥乳杆菌(Lactobacillus pontis)、耐酸乳杆菌(Lactobacillus acetotolerans)、戊糖乳杆菌(Lactobacillus pentose):茅台微生物菌种库保存。
正丙醇(色谱纯)、叔戊醇(色谱纯):美国Sigma公司;氯化钠(分析纯):上海国药集团化学试剂有限公司;MRS肉汤培养基:青岛海博生物技术有限公司。
1.2 仪器与设备
KS4000i摇床:德国艾卡公司;380R离心机:德国Hettich公司;LRH-250恒温培养箱:上海飞越实验仪器有限公司;JAR GASSING SYSTEM厌氧培养罐:英国DWS公司;IEC61010-1超净工作台:新加坡艺思高科技有限公司;CL-32L全自动蒸汽灭菌器:日本ALP株式会社;G1888A-7890A-5975C顶空-气相色谱-质谱(headspace-gas chromatographymass spectrometry,HS-GC-MS)联用仪:美国Agilent公司。
1.3 方法
1.3.1 代谢通路与关键酶查询
利用京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)数据库对目标物质进行比对,明确其代谢通路及参与反应的酶,确定目标物质代谢所需关键催化酶。具体步骤:(1)利用KEGG COMPOUND DATABASE(代谢物及其他小分子化合物数据库)检索目标风味物质,并筛选包含目标风味物质的Entry编号选项;(2)通路选项中确定目标风味物质的代谢图谱,并获取该代谢途径的可视化图谱;(3)结合代谢途径所涉及相关酶信息及其文献报道,确定催化反应的关键酶。
1.3.2 关键酶表达微生物查询
通过美国国家生物技术信息中心(national center for biotechnologyinformation,NCBI)数据库获得催化酶微生物信息,确定产目标物质潜在的微生物种属。
1.3.3 乳酸菌发酵液制备
分别挑取不同供试菌株,于50mLMRS肉汤培养基中,37℃静置厌氧培养至对数期,制备种子液备用。分别接种1 mL种子液于MRS肉汤培养基中,37℃静置厌氧培养5~7 d,检测发酵液中正丙醇含量。
1.3.4 正丙醇含量测定
采用顶空-气相色谱-质谱联用仪法测定发酵液中的正丙醇含量,具体方法为:(1)样品前处理:于20 mL顶空瓶中加入2gNaCl和5mL菌体发酵液,加入3 μL叔戊醇乙醇溶液作为内标。(2)顶空条件:孵育室温度70℃,孵育时间10min,振摇频率高,定量环温度100℃,传输线温度130℃,进样体积3mL,GC循环时间22min。(3)气相色谱分析条件:DB-WAX色谱柱(30 m×0.25 mm×0.25 μm),进样口温度230 ℃,分流比30∶1,载气为氦气(He),流速为0.8 mL/min。柱温采用程序升温:起始温度40℃,保持1 min后以1℃/min升温至45℃,然后以35℃/min升温至220℃,保持7 min,运行时间18 min。(4)质谱分析条件:电子电离(electron ionization,EI)源,电子能量70 eV,离子源温度230 ℃,接口温度250℃。扫描质量范围33~350 amu。采集模式:选择离子扫描(selected ion monitoring,SIM),溶剂延迟2 min。SIM参数:组1为1-丙醇,绘图离子为59。(5)正丙醇定性、定量方法:采用标准谱图比对和标准品比对,对目标物进行定性,内标标准曲线法进行定量。
2 结果与分析
2.1 催化产正丙醇关键酶
通过在KEGG代谢网络数据库中对正丙醇产生途径查询,发现其产生途径为丙酸代谢途径(Map00640)中由丙醛代谢产生正丙醇,其代谢通路如图1所示。

图1 正丙醇产生途径
Fig.1 Schematic diagram of 1-propanol biosynthetic pathway
从图1可以看出,在丙酸代谢途径中,正丙醇存在一条代谢通路,为1,2-丙二醇在丙二醇脱水酶的作用下生成丙醛,从而丙醛在1,3-丙二醇脱氢酶和1-丙醇脱氢酶的作用下代谢产生正丙醇。
相关报道指出[19-20],在大肠杆菌(Escherichia coli)和谷氨酸棒状杆菌(Corynebacterium glutamicum)中导入丙二醇脱水酶与醇脱氢酶,能够代谢丙二醇产生正丙醇。因此结合代谢通路确定丙二醇脱水酶、1,3-丙二醇脱氢酶和1-丙醇脱氢酶为催化产生正丙醇的关键酶。
2.2 关键酶表达微生物
通过在NCBI数据库中查询能够表达上述3种关键酶的微生物,在NCBI数据库中能够分别表达上述3种酶的微生物种属,如表1所示。
表1 NCBI中表达关键酶的微生物
Table 1 Microorganisms expressing key enzymes in NCBI

检索信息 丙二醇脱水酶 1,3-丙二醇脱氢酶 1-丙醇脱氢酶检索数细菌属真菌属古菌属105 675 143 7 223 213 7 733 24 0 4 1 6 0 0
发现共计17个微生物属能够同时表达上述3种关键催化酶,具体如表2所示,表2中所示微生物种属为供试菌株的筛选提供范围。
表2 同时表达3种关键酶的微生物属
Table 2 Genera of microorganisms simultaneously expressing 3 key enzymes

属水平微生物类别芽孢杆菌属(Bacillus)柠檬酸杆菌属(Citrobacter)梭菌属(Clostridium)肠杆菌属(Enterobacter)埃希氏菌属(Escherichia)肠球菌属(Enterococcus)真杆菌属(Eubacterium)克雷伯氏菌属(Klebsiella)乳杆菌属(Lactobacillus)李斯特菌属(Listeria)甲烷嗜热杆菌属(Metakosakonia)片球菌属(Pediococcus)假单胞菌属(Pseudomonas)沙门氏菌属(Salmonella)赛氏杆菌属(Serratia)链球菌属(Streptococcus)弧菌属(Vibrio)
2.3 供试菌株选择
针对不同香型白酒微生物研究表明,上述17个微生物属除李斯特菌属(Listeria)和甲烷嗜杆菌属(Metakosakonia)未见报道外,其余15个微生物属分别在不同香型白酒酿造中有检出,其中乳杆菌属(Lactobacillus)与芽孢杆菌属(Bacillus)微生物占绝对优势地位[21-31];且丰度较高的微生物对白酒风味具有明显的贡献作用[27]。柏永昊等[14,18]研究发现,白酒中存在能够代谢产正丙醇的Bacillus属微生物;而关于白酒中Lactobacillus属微生物代谢产正丙醇的研究尚未见报道。为进一步确认白酒酿造过程中正丙醇的优势微生物来源,本研究以Lactobacillus属微生物为研究对象验证其产正丙醇的能力。
挑选6株已保藏且报道的在不同香型白酒酿造过程占优势地位的Lactobacillus属微生物作为实验菌株[32-37],验证其代谢产正丙醇的能力。具体种属为布赫内氏乳杆菌(Lactobacillus buchneri)、面包乳杆菌(Lactobacillus panis)、短乳杆菌(Lactobacillus brevis)、桥乳杆菌(Lactobacillus pontis)、耐酸乳杆菌(Lactobacillus acetotolerans)、戊糖乳杆菌(Lactobacillus pentose),编号分别为Lac1~Lac6。
2.4 乳酸菌产正丙醇结果分析
2.4.1 发酵液中正丙醇含量分析方法的建立
依据HS-GC-MS方法对发酵液进行分析,通过谱库谱图比对和标准品比对,确认可检出内标物质叔戊醇和目标物质正丙醇。采用HS-GC-MS法测得的发酵液的总离子流色谱图见图2。
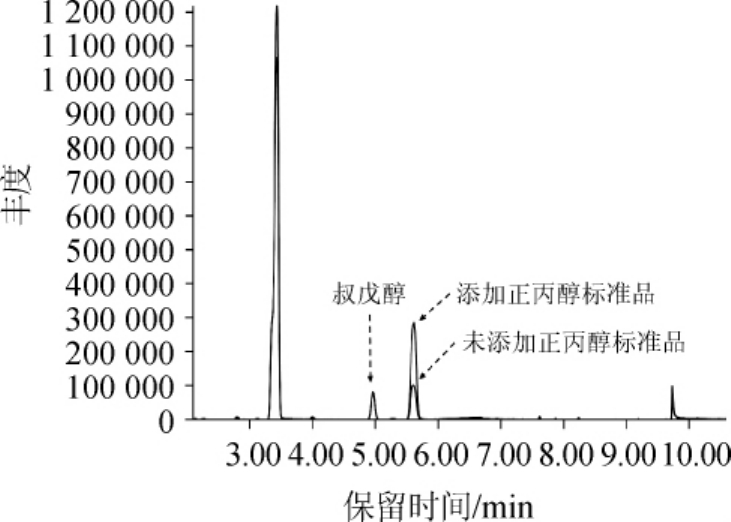
图2 乳杆菌发酵液正丙醇含量HS-GC-MS分析的总离子流图
Fig.2 Total ion chromatogram of fermentation broth of Lactobacillusdetected by HS-GC-MS
将配制好的系列标准溶液进行静态顶空气-相色谱-质谱分析,数据分析后得到正丙醇标准曲线,并以正丙醇与叔戊醇的峰面积比值为x,发酵液中正丙醇含量为y,计算得到该标准曲线的回归方程y=24.965 5x+0.301 1,其线性范围为0.79~159.12 mg/L,线性关系良好(R2=0.999 9),平均加标回收率为102.70%。以信噪比为10为标准,计算得到该方法的定量限为0.386 mg/L。
2.4.2 正丙醇含量分析
将制备好的6株供试菌株种子液1mL分别接种于MRS肉汤培养基中,以不接菌作为空白对照(CK),37℃厌氧发酵5~7 d,其发酵液中正丙醇含量检测结果如图3所示。
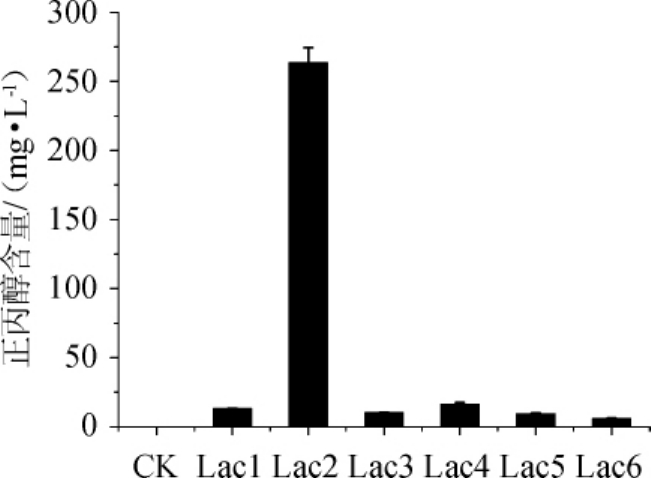
图3 乳杆菌发酵液中正丙醇含量
Fig.3 1-propanol content in fermentation broth ofLactobacillus
由图3可知,接种6株Lactobacillus属微生物的发酵液均有正丙醇的检出,其中面包乳杆菌(Lactobacillus panis)发酵液检出量最高,其含量为263.7 mg/L,是目前已有文献报道的白酒产正丙醇的酵母菌[15]与芽孢杆菌[18]纯菌发酵液中正丙醇含量的3.6倍与4.5倍。该结果表明白酒酿造过程中存在能够代谢产正丙醇的乳杆菌属微生物。
3 结论
本研究阐述了一种目标风味物质微生物溯源的新方法。通过KEGG数据库明确目标风味物质代谢途径及其催化的关键酶;并利用NCBI数据库确定表达关键酶的微生物种类信息,同时对样品进行微生物群落结构分析,对比样品微生物群落结构与表达关键酶微生物信息,将分类地位相同的微生物作为目标菌株;以此微生物种属开展筛选与功能研究。该方法具有工作量小、目的性强等优势,同时也可为其他食品发酵过程中产特征风味物质的微生物溯源提供新思路。
本研究利用上述方法开展白酒中产正丙醇微生物的补充溯源,发现在纯菌发酵条件下,研究的6株乳杆菌属(Lactobacillus)均能代谢生成正丙醇,其中面包乳杆菌(Lactobacillus panis)正丙醇产量最高,为263.7 mg/L,显著高于已报道的白酒酿造过程中酵母菌与芽孢杆菌的正丙醇产量。该研究结果表明,乳酸杆菌是白酒酿造过程中正丙醇的另一重要微生物来源,但由于白酒酿造体系中复杂的微生物组成、发酵环境与营养条件对微生物的代谢产物种类与含量均有重要影响,因此,乳杆菌属微生物在酿造过程中的正丙醇代谢能力及贡献仍需进一步探索,这将为调控白酒中正丙醇的含量奠定理论基础。
[1]XU Y Q,SUN B G,FAN G S,et al.The brewing process and microbial diversityof strong flavour Chinese spirits:a review[J].J Inst Brew,2017,123(1):5-12.
[2]YOU L,WANG T,YANG Z R,et al.Performance of indigenous yeasts in the processing of Chinese strong-flavoured liquor during spontaneous mixed solid-state or submerged fermentation[J].J Inst Brew,2015,121(2):295-303.
[3]FU Z L,SUN B G,LI X T,et al.Isolation and characterization of a high ethyl acetate-producing yeast from Laobaigan Daqu and its fermentation conditions for producing high-qualityBaijiu[J].Biotechnol Biotechnolog Equip,2018,32(5):1218-1227.
[4]ZOU W,YE G B,ZHANGK Z.Diversity,function,and application of Clostridiumin Chinese strong flavorBaijiuecosystem:a review[J].J Food Sci,2018,83(5):1193-1199.
[5]DU H,LIU B,WANG X S,et al.Exploring the microbial origins of,p-cresol and its co-occurrence pattern in the Chinese liquor-making process[J].Int J Food Microbiol,2017,260:27-35.
[6]WU Q,ZHU W A,WANG W,et al.Effect of yeast species on the terpenoids profile of Chinese light-style liquor[J].Food Chem,2015,168:390-395.
[7]ZHAO C Q,YAN X L,YANG S T,et al.Screening ofBacillusstrains from Luzhou-flavor liquor making for high-yield ethyl hexanoate and low-yield propanol[J].LWT-Food Sci Technol,2017,77:60-66.
[8]陈向田.芝麻香型白酒关键细菌的分离与风味功能研究[J].酿酒,2018,45(5):95-96.
[9]AlEJANDRA E Z,ARTURO V P D L,ALEJANDRO S F.The road to metagenomics:from microbiology to DNA sequencing technologies and bioinformatics[J].Front Genet,2015,6(104):348.
[10]贾栋,贾小云,马瑞燕.生物信息学数据库及查询[J].山西农业大学学报(自然科学版),2013,33(6):520-525.
[11]PALLEN M J.Microbial bioinformatics 2020[J].Microbial Biotechnol,2016,9(5):681-686.
[12]ORTIZ-ESTRADAAM,GOLLAS-GALVÁNT,MARTÍNEZ-CÓRDOVA L R,et al.Predictive functional profiles using metagenomic 16S rRNA data:a novel approach to understanding the microbial ecology of aquaculture systems[J].Rev Aquacult,2018:1-12.
[13]谢方安.谈白酒香气成分和作用[J].酿酒,2006,33(5):52-55.
[14]柏永昊,张明春,缪礼鸿.芽孢杆菌对白酒发酵过程中正丙醇含量的影响[J].酿酒科技,2013(11):26-29.
[15]孙神英.不同酵母菌对白酒风味成分的影响[D].武汉:武汉轻工大学,2014.
[16]王霜.产酱香功能芽孢杆菌的筛选及浓酱兼香型白酒复合功能强化曲的研究[D].武汉:武汉轻工大学,2017.
[17]游玲,任羽,王涛,等.酵母对浓香型白酒糟醅中高级醇生成的影响[J].食品与发酵工业,2016,42(2):23-28.
[18]赵长青,徐莎,杨阳,等.浓香型白酒酿造大曲及糟醅中功能芽孢杆菌的筛选[J].食品工业科技,2017,38(7):151-155.
[19]MASTUBARA M,URANO N,YAMADA S,et al.Fermentative production of 1-propanol from D-glucose,l-rhamnose and glycerol using recombinantEscherichia coli[J].J Biosci Bioeng,2016,122(4):421-426.
[20]SIEBERT D,WENDISCH V F.Metabolic pathway engineering for production of 1,2-propanediol and 1-propanol byCorynebacterium glutamicum[J].Biotechnol Biofuel,2015,8(1):91-103.
[21]ZHANG L Q,WU C D,DING X F,et al.Characterisation of microbial communities in Chinese liquor fermentation starters Daqu using nested PCR-DGGE[J].World J Microb Biotechn,2014,30(12):3055-3063.
[22]ZHENG X W,YAN Z,HAN B Z,et al.Complex microbiota of a Chinese"Fen"liquor fermentation starter(Fen-Daqu),revealed by culturedependent and culture-independent methods[J].Food Microbiol,2012,31(2):293-300.
[23]HU X L,DU H,XU Y.Identification and quantification of the caproic acid-producing bacteriumClostridium kluyveriin the fermentation of pit mud used for Chinese strong-aroma type liquor production[J].Int J Food Microbiol,2015,214:116-122.
[24]LIU X,GUO K L,ZHANG H X.Determination of microbial diversity in Daqu,a fermentation starter culture of Maotai liquor,using nested PCR-denaturing gradient gel electrophoresis[J].World J Microb Biotechn,2012,28(6):2375-2381.
[25]LI K,ZHANG Q,ZHONG X T,et al.Microbial diversity and succession in the Chinese Luzhou-flavor liquor fermenting cover lees as evaluated by SSU rRNA Profiles[J].Ind J Microbiol,2013,53(4):425-431.
[26]郭敏,黄永光,邱树毅,等.高通量测序在酱香白酒微生态多样性研究中的应用[J].中国酿造,2017,36(5):146-151.
[27]王鹏,吴群,徐岩.中国白酒发酵过程中的核心微生物群及其与环境因子的关系[J].微生物学报,2018,58(1):142-153.
[28]刘凡,周新虎,陈翔,等.洋河浓香型白酒发酵过程酒醅微生物群落结构解析及其与有机酸合成的相关性[J].微生物学报,2018,58(12):2087-2099.
[29]WANG L,WANG Y Y,WANG D Q,et al.Dynamic changes in the bacterial community in Moutai liquor fermentation process characterized by deep sequencing[J].J Inst Brew,2015,121(4):603-608.
[30]FAN G S,SUN B G,FU Z L,et al.Analysis of physicochemical indices,volatile flavor components,and microbial community of a light-flavor Daqu[J].J Am Soc Brew Chem,2018,76(3):209-218.
[31]YANG F,CHEN L Q,LIU Y F,et al.Identification of microorganisms producing lactic acid during solid-state fermentation of Maotai flavour liquor[J].J Inst Brew,2018.
[32]HONG X T,CHEN J,LIU L,et al.Metagenomic sequencing reveals the relationship between microbiota composition and quality of Chinese rice wine[J].Sci Report,2016,6:26621.
[33]WANG X S,DU H,ZHANG Y,et al.Environmental microbiota drives microbial succession and metabolic profiles during Chinese liquor fermentation[J].Appl Environ Microbiol,2017,84(4):02369-17.
[34]WANG J,ZHONG Q P,YANG Y Y,et al.Comparison of bacterial diversity between two traditional starters and the round-koji-maker starter for traditional cantonese chi-flavor liquor brewing[J].Front Microbiol,2018,9:1053.
[35]CHEN Q P,KANG G T,ZHOU T N,et al.Significant difference of the culturable anaerobic microbial species in GujingTribute pitmud with different ages[J].Mat Sci Eng,2018,394:22-25.
[36]LI X R,MA E B,YAN L Z,et al.Bacterial and fungal diversity in the traditional Chinese liquor fermentation process[J].Int J Food Microbiol,2011,146(1):31-37.
[37]ZHANG W X,QIAO Z W,TORU S,et al.Analysis of the bacterial communityin Zaopei during production of Chinese Luzhou-flavor liquor[J].J Inst Brew,2005,111(2):215-222.