多药耐药病原菌包括耐甲氧西林金黄色葡萄球菌(methicillin-resistantStaphylococcus aureus,MRSA)、耐青霉素肺炎链球菌(penicillin-resistantStreptococcus pneumoniae,PRSP)、耐多药结核分枝杆菌(multiple drugs resistant tuberculosis,MDR-TB)等革兰氏阳性菌和耐碳青霉烯类鲍曼不动杆菌(carbapenem-resistantAcinetobacter baumannii,CRAB)、超广谱β-内酰胺酶(extended spectrum β-lactamases,ESBL)肠杆菌等革兰氏阴性菌[1-2],其介导的感染性疾病是当今世界需要亟待解决的健康安全问题。现有的抗生素对MRSA介导的感染性疾病治疗效果降低或者无效,导致患者死亡率增加,已超过艾滋病和肺结核患者[3-4],美国每年大约有23 000名患者死于MRSA引起的感染[5-6]。因此,针对MRSA开发新的抗生素显得极其重要。
放线菌(actinomycete)是一种能产大量活性次级代谢产物的丝状细菌,临床上2/3的抗生素主要来源于放线菌。已有文献报道[7],放线菌代谢产物中具有生物活性的物质约12000种,其中具有抗生素活性的代谢产物多达10000种,是新型抗生素开发的宝贵资源库。目前,基因筛选的方法已被较多应用于放线菌新的活性次级代谢产物的研究,且基本都是基于聚酮合酶I型(polyketide synthase I,PKS I)、聚酮合酶II型(polyketide synthase II,PKS II)、非核糖体多肽合成酶(nonribosomalpeptidesynthetase,NRPS)和后修饰酶[如卤化酶(halogenase,Hal)]进行的基因筛选[8-9]。LI X G等[10]使用还原黄素腺嘌呤二核苷酸(reduced flavine adenine dinucleotide,FADH2)依赖型卤化酶基因筛选的方法,从163株红树林放线菌中筛选出26株具有抑菌或抑制肿瘤活性的阳性菌株,证明了卤化酶基因筛选的有效性;LU Y等[11-12]从贵州梵净山等地筛选出Hal、NRPS、PKS I阳性链霉菌(Streptomycessp.)FJS31-2,且从中分离出具有抗MRSA、抗肝癌的新化合物zunyimycin A、B、C;杨小燕等[13]利用靶向PKS II的聚合酶链式反应(polymerase chain reaction,PCR)技术从167株海洋放线菌中发现12株PKS II阳性菌株,且从阳性菌株Streptomycessp.PKU-MA00218中发现新的聚酮类天然产物angucyclinone C。
贵州省黔北地区保留了其独特的原生态环境,这些原生态环境中可能蕴藏极为丰富而独特的微生物资源,具有从中开发微生物抗菌药物的巨大潜力,值得进一步研究[9]。因此,本研究以抗MRSA活性为导向,同时基于NRPS、PKSI、PKS II 3种产物骨架合成基因和Hal修饰关键酶基因勘探,对来自黔北地区土壤放线菌进行分离筛选,以期找到具有较好抗MRSA活性的放线菌菌株,为获得可能创制抗MRSA新药的苗头药或先导化合物提供菌株来源。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
土样:32份试验土样采集于贵州遵义凤凰山公园(10份)、赤水丹霞山(12份)、大板水国家森林公园(10份)周边人员活动较少地区、海拔500~1 000 m左右的除地表腐殖质10~20cm的土层,分装于无菌样品采集袋中常温保存运输。
1.1.2 菌株
耐甲氧西林金黄色葡萄球菌(MRSA)31:由本实验室保藏。
1.1.3 培养基
改良高氏1号培养基:可溶性淀粉20g,NaCl0.5g,KNO3 1 g,K2HPO4·3H2O 0.5 g,MgSO4·7H2O 0.5 g,FeSO4·7H2O 0.01 g,玉米汁50 mL,土壤浸出物[14]50 mL,土壤样品细粉10 g,复合维生素浸提物10 mL,琼脂18 g,蒸馏水1 L,pH 7.2,115℃高压灭菌30 min。其中玉米汁的制备:称取新鲜的玉米100 g,加入1 L蒸馏水,用豆浆机搅拌均匀后,滤掉废渣,补加蒸馏水至2 L,经微孔滤膜过滤除菌后于4℃冰箱保存备用;复合维生素浸提物的制备:取21金维他多维元素片20片(0.825 g/片)研磨成粉,入15 mL无水乙醇和35 mL无菌水振荡悬浮,8 000 r/min离心10 min,上清用微孔滤膜过滤除菌后于4℃冰箱保存备用。
土壤浸汁腐殖酸培养基[14]:牛肉膏3 g,蛋白胨10 g,微量元素预混液(ZnSO4·7H2O 2 g/L,FeSO4·7H2O 2 g/L,MnCl2·4H2O 2g/L,CuSO4·5H2O 2 g/L,Na2B4O7·10H2O 2 g/L,(NH4)6Mo7O24·4H2O 2 g/L)0.5 mL,腐殖酸2.0 g,琼脂18 g,土壤浸出物1 L,pH 7.2,121℃高压灭菌30 min。
国际链霉菌计划(internationalStreptomycesproject,ISP)2培养基[15]:葡萄糖4 g,CaCO32 g,麦芽抽提物10 g,酵母抽提物4 g,琼脂15 g,蒸馏水定容至1 L,115℃高压灭菌30 min。
改良的葡萄糖酵母麦芽(glucose yeast malt,GYM)链霉素培养基[16]:葡萄糖4 g,CaCO32 g,麦芽抽提物10 g,酵母抽提物4g,微量元素预混液0.5mL,腐殖酸浸出液10mL,琼脂18 g,蒸馏水定容至1 L,115℃高压灭菌30 min。
GYM液体培养基:葡萄糖4 g,酵母提取物4 g,麦芽提取物10 g,蒸馏水定容至1 L,115℃高压灭菌30 min。
LB液体培养基[16]:胰蛋白胨10 g,酵母提取物5 g,NaCl 10 g,蒸馏水定容至1 L,121℃高压灭菌30 min。LB固体培养基:在LB液体培养基中加入琼脂20 g。
1.1.4 主要试剂
抗生素、抗菌药物储存液(重铬酸钾50 mg/L,制霉菌素20 mg/L,萘啶酮酸20 mg/L,放线菌酮50 mg/L):德国Sigma公司;酚、氯仿等脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂:北京索莱宝科技有限公司;乙酸乙酯、甲醇(均为分析纯):成都科龙化工试剂厂;ExTaq酶(5U/μL)、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)Mixture、TaqBuffer等分子生物学试剂及酶类:宝生物工程(大连)有限公司。
1.2 仪器与设备
BSP-400培养箱:上海博迅医疗生物仪器股份有限公司;E002092超级洁净工作台:苏州市金净净化设备科技有限公司;Direct-Q5UV超纯水机:美国密理博公司;MLS-3781L-PC自动蒸汽灭菌器:日本松下公司;T100快速梯度PCR仪:美国伯乐公司;Gel Doc XR+凝胶成像系统:美国伯乐公司;DYCP-33A电泳仪:北京六一生物科技有限公司。
1.3 方法
1.3.1 菌株的分离纯化
土壤样品预处理:每份样品分别称2 g于无菌研钵中,加入0.2 g CaCO3,用无菌杵研磨成细粉,装入50 mL无菌离心管中,28℃风干2周后,加入30 mL无菌水,置于28℃摇床上150 r/min振荡1 h使土壤充分悬浮,55℃处理20 min,促使放线菌孢子萌发[14]。
菌株的分离纯化:采用梯度稀释法将土壤预混液分别稀释成10-2、10-3和10-43个浓度梯度,振荡混匀,分别吸取不同浓度梯度的土样悬浊液200 μL均匀涂布于4种放线菌分离培养基(改良高氏1号培养基、土壤浸汁腐殖酸培养基、ISP2培养基、改良的GYM链霉素培养基)上,28℃静置培养,随时观察培养基上的菌落生长状态,挑取目的菌株进行分离纯化并保藏。
1.3.2 抗MRSA活性的测定
将MRSA接种于100 mL LB液体培养基中,37℃、200 r/min条件下振荡培养6~8 h,取10 mL菌液与200 mL未凝固的LB培养基混合均匀后倒板。用无菌打孔器挖取直径6 mm的分离菌株于含有MRSA的LB固体培养基上,于37℃培养箱中过夜培养,观察抑菌圈的有无。
1.3.3 菌株的分子生物学鉴定
参照文献[17]提取活性菌株的基因组总DNA,以其为模板,利用细菌16S rRNA通用引物27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R(5'-TACGGCTACCTTGTTACGACTT-3')进行PCR扩增。PCR扩增体系:EXTaq酶0.25μL,10×buffer 5 μL,脱氧核糖核苷三磷酸(dNTP)4 μL,MgCl2 4μL,模板DNA2μL,27F1μL,1492R1μL,双蒸水(ddH2O)32.75μL。PCR扩增条件:94℃预变性5 min;94℃变性30 s,56℃退火30s,72℃延伸90s,共30个循环;72℃再延伸5min。采用1%琼脂糖凝胶电泳检测PCR扩增产物,PCR扩增产物送至上海英骏生物公司完成。测序结果在美国国立生物技术信息中心(nationalcenter for biotechnology information,NCBI)的Genbank数据库中进行Blast同源性搜索,选取同源性较高的模式菌株的16S rRNA序列,采用Mega 6.0软件中的邻接(neighbour joining,NJ)法构建系统进化树[18-19]。
1.3.4 功能基因的勘探
以提取的基因组DNA为模板,利用Hal、PKSⅠ、PKSⅡ、NRPS基因的保守引物(见表1)分别进行PCR扩增。根据不同的引物和扩增片段选择相应的PCR扩增体系[13,20-22],PCR扩增条件[22]:94℃预变性5min;94℃变性30s,56℃退火30 s,72℃延伸1min,共30个循环;72℃再延伸5 min。采用1%琼脂糖凝胶电泳检测PCR扩增产物。
表1 本研究所采用的引物、序列及用途
Table 1 Primers and its sequences and application used in the study

引物序列(5'→3')用途A3F A7R K1F M6R KSαF KSαR Halo-B4-FW Halo-B7-RV GCSTACSYSATSTACACSTCSGG SASGTCVCCSGTSCGGTAS TSAAGTCSAACATCGGBCA CGCAGGTTSCSGTACCAGTA TSGCSTGCTTCGAYGCSATC TGGAANCCGCCGAABCCGCT TTCCCSCGSTACCASATCGGSGAG GSGGGATSWMCCAGWACCASCC NRPS(750 bp)PKS I(1 300 bp)PKS II(700 bp)Hal(550 bp)
2 结果与分析
2.1 菌株分离纯化及抗MRSA分析
从采集的32份土样中共分离到169株放线菌菌株,其中赤水丹霞山土样65株(以CS命名菌株)、凤凰山公园土样45株(以FHS命名菌株)、大板水国家森林公园土样59株(以DBS命名菌株)。采用琼脂扩散法对分离菌株的抗MRSA活性进行测定,部分分离菌株的抗MRSA活性结果见图1。
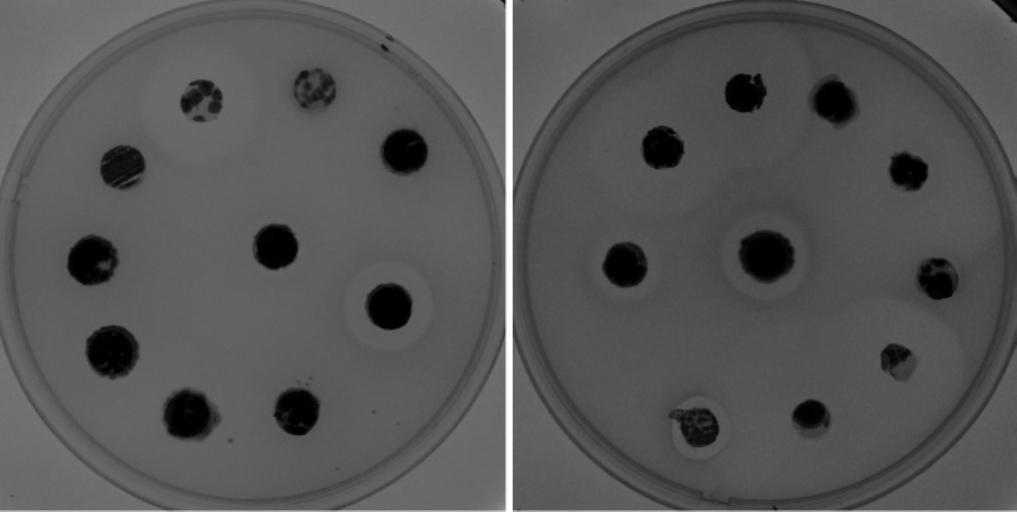
图1 部分分离菌株抗MRSA活性测定结果
Fig.1 Determination results of anti-MRSA activity of partially
isolated strains
由图1可知,不同分离菌株具有不同程度的抗MRSA活性,且分离的169株菌株中有68株具有抗MRSA活性,占总分离菌株的40.24%。因此,以68株活性抗MRSA菌株进行后续的基因组勘探。
2.2 活性菌株的分子生物学鉴定
选取68株活性菌株,利用细菌16S rRNA通用引物27F/1492R进行PCR扩增,PCR扩增产物经琼脂糖凝胶检测,部分结果见图2。由图2可知,PCR扩增产物的碱基长度约1 500 bp,与预期结果相符,PCR扩增产物进行测序。
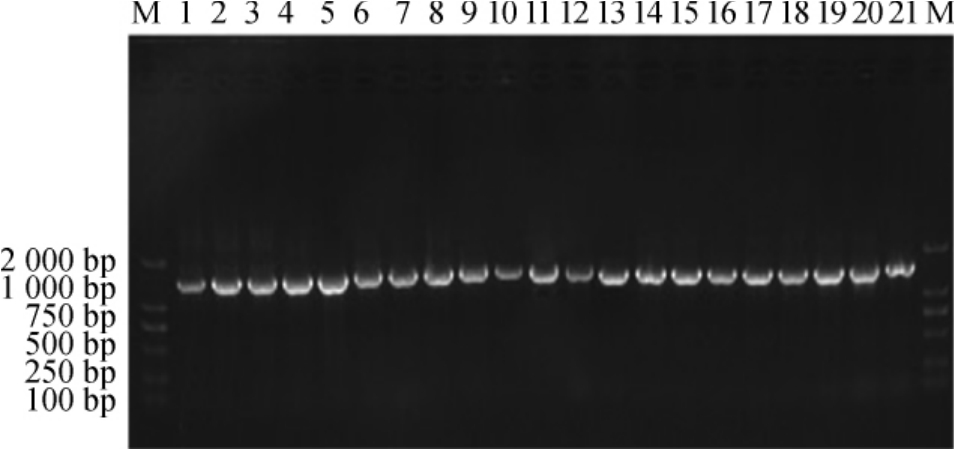
图2 活性菌株16S rRNA PCR扩增产物琼脂糖凝胶电泳图
Fig.2 Agarose gel electrophoresis of 16S rRNA PCR amplification products of active strains
M:DL2000DNAmarker;1~21:部分活性菌株的16SrRNAPCR扩增产物。
将68株活性菌株的16S rRNA PCR扩增产物测序结果提交至NCBI进行Blast同源性搜索,选取同源性较高的模式菌株的16S rRNA基因序列,利用Mega 6.0软件中的NJ法构建系统进化树,结果见图3。由图3可知,菌株FHS2-3为诺卡氏菌属(Nocardiasp.),其他分离菌主要为链霉菌属(Streptomycessp.),这与程少军等[23]的研究结果一致,说明在贵州喀斯特土壤环境中链霉菌属在放线菌中是优势菌属。

图3 基于16S rRNA序列68株活性菌株的系统进化树
Fig.3 Phylogenetic tree of 68 active strains based on 16S rRNA sequences
▲为NCBI模式参考菌株,FHS、DBS、CS分别为凤凰山、大板水、赤水地区土样分离株。
同时Blast比对中,菌株DBS8-4、DBS8-13、CSGY1-11、CSHG2-5、CSDG1-2、CSDG5-5、CSGY1-3、CSGY5-3、CSGY 1-9、FHS4-7与NCBI的GenBank数据库中已有菌株的16SrRNA序列相似性<98%,说明本地区土壤放线菌中可能蕴藏潜在的较新颖菌株,具有开发的潜力。
2.3 次级代谢潜力基因勘探
CHRISTIANSEN G等[24]指出卤化酶基因、聚酮合酶基因、非核糖体肽基因等可以作为指示基因对微生物产次级代谢产物的潜力进行快速评估。利用微生物的基因组信息预测其合成特定天然产物的潜能,进而进行新化合物分离纯化和结构鉴定的基因组挖掘技术。
因此,本研究对具有抗MRSA活性的68株活性菌株的次级代谢产物生物合成过程中的修饰酶基因卤化酶(Hal)及催化主骨架合成的非核糖体肽合成酶(NRPS)、I型聚酮合酶(PKS I)、II型聚酮合酶(PKS II)基因进行PCR扩增,结果见表2。由表2可知,68株活性菌株中,Hal基因阳性的菌株12株,占活性菌株的17.65%,PKS I基因阳性菌株18株,占活性菌株的36.47%,PKS II基因阳性的菌株42株,占活性菌株的61.77%,NRPS基因阳性的菌株22株,占活性菌株的32.35%。说明黔北地区放线菌基因组中存在多种化合物骨架合成酶及后修饰酶相关基因,提示可能含有骨架多样结构新颖的天然产物,应进一步深入挖掘。
表2 68株活性菌株的次级代谢产物基因勘探
Table 2 Secondary metabolic product gene exploration of 68 active strains

菌株 NRPS PKSI PKSII Hal 菌株 NRPS PKSI PKSII Hal DBS10-13 DBS9-1 DBS6-2 DBS11-4 DBS1-2 DBS12-2 DBS12-20 DBS12-5 DBS12-6 DBS16-8 DBS18-4 DBS19-2 DBS21-2 DBS2-2 DBS22-1 DBS22-10 DBS22-6 DBS23-2 DBS5-5 CSDG5-3 CSDG5-5 CSGY1-3 CSGY1-4 CSGY1-9++--+----++----++-+-+--+-+----+-+--++---+----+--+++--++++++++-+-+---++++--+-----++----+----+----CSDG4-1 CSDG4-4 CSGY1-14 FHS10-17 FHS10-21 FHS10-22 FHS4-8 FHS5-10 FHS4-7 FHS10-16 DBS23-4 DBS23-8 DBS5-3 DBS8-13 DBS8-4 CSGY1-17 CSGY1-10 CSGY1-11 CSGY1-5 CSGY1-6 CSGY1-7 CSGY4-13 CSGY4-3 CSGY4-7-----+-+------+-+-+-+-+-----++-+-++--+--+---+---++-+++-+++++++++-+-+-+-+---+-+--+--+-+---+------
续表
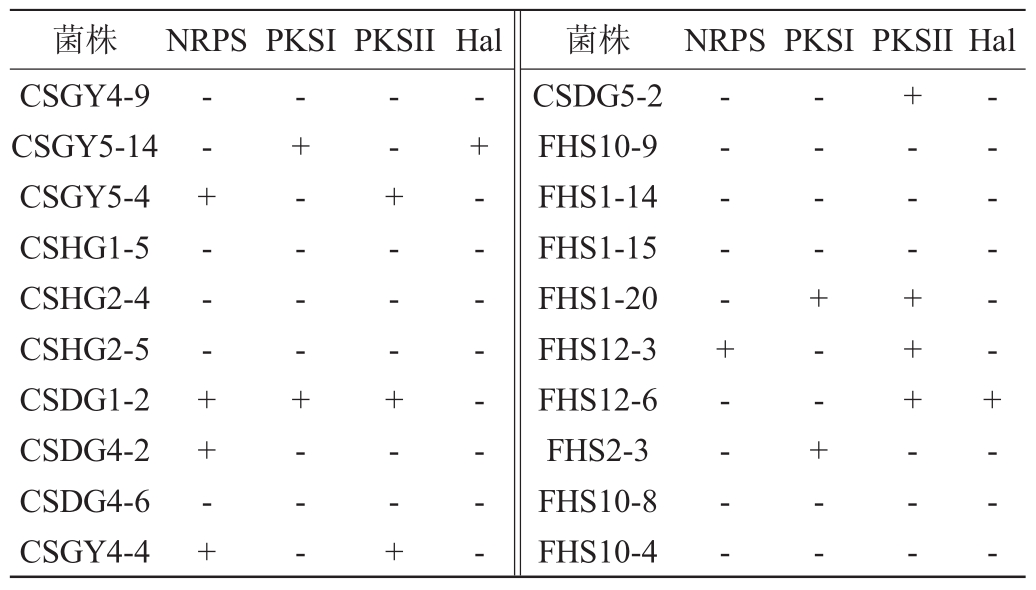
注:“+”表示结果呈阳性,具有该基因;“-”表示结果呈阴性,无该基因。
菌株 NRPS PKSI PKSII Hal 菌株 NRPS PKSI PKSII Hal CSGY4-9 CSGY5-14 CSGY5-4 CSHG1-5 CSHG2-4 CSHG2-5 CSDG1-2 CSDG4-2 CSDG4-6 CSGY4-4--+---++-+-+----+-----+---+--+-+--------CSDG5-2 FHS10-9 FHS1-14 FHS1-15 FHS1-20 FHS12-3 FHS12-6 FHS2-3 FHS10-8 FHS10-4-----+--------+--+--+---+++---------+---
3 结论
本研究从32份土样中共分离纯化到169株放线菌,其中68株菌株具有抗MRSA活性,占总分离菌株的40.24%。通过16S rRNA序列分析可知,68株活性菌株主要为链霉菌属(Streptomycessp.),其中菌株DBS8-4、DBS8-13、CSGY1-11、CSHG2-5、CSDG1-2、CSDG5-5、CSGY1-3、CSGY5-3、CSGY 1-9、FHS4-7与NCBI的Genbank数据库中已有菌株的16S rRNA序列相似性<98%,说明本地区土壤放线菌中可能蕴藏潜在的较新颖菌株,具有开发的潜力。
通过对68株活性菌株的次级代谢产物基因的勘探可知,Hal阳性菌株占17.65%,PKSI阳性菌株占36.47%,PKSII阳性菌株占61.77%,NRPS阳性菌株占32.35%,其中菌株DBS9-1、DBS12-6、DBS8-13、DBS22-6、CSDG1-2、FHS5-10、FHS10-22中同时含有NRPS或Hal、PKSI、PKSII三种基因,本实验可为后续通过激活特定合成的基因组定向挖掘该类化合物等研究提供参考。
[1]MEDINA E,PIEPER D H.Tackling threats and future problems of multidrug-resistant bacteria[J].Curr Top Microbiol,2016,398:3-33.
[2]SHI W,LI J,DONG F,et al.Serotype distribution,antibiotic resistance pattern and multilocus sequence types of invasiveStreptococcus pneumoniaeisolates in two tertiary pediatric hospitals in Beijing prior to PCV13 availability[J].Expert Rev Vaccines,2019,18:89-94.
[3]ZHU H,SWIERSTRA J,WU C,et al.Eliciting antibiotics active against the ESKAPE pathogens in a collection of actinomycetes isolated from mountain soils[J].Microbiology,2014,160(8):1714-1725.
[4]PAYNE K D,DAS A,NDIULOR M,et al.Dosing strategies to optimize currently available anti-MRSA treatment options(Part 2:PO options)[J].Expert Rev Clin Pharmaco,2018,11(2):139-149.
[5]HASSOUN A,LINDEN P K,FRIEDMAN B.Incidence,prevalence,and management of MRSA bacteremia across patient populations-a review of recent developments in MRSA management and treatment[J].Crit Care,2017,21:211.
[6]RAN C,HICKS K,ALEXIEV B,et al.Cervicofacial necrotising fasciitis by clindamycin-resistant and methicillin-resistantStaphylococcus aureus(MRSA)in a young healthy man[J/OL].BMJ Case Rep,2018,http://dx.doi.org/10.1136/bcr-2018-226975.
[7]BERDY J.Thoughts and facts about antibiotics:Where we are now and where we are heading[J].J Antibiot,2012,65(6):385-395.
[8]MINGYAR E,NOVAKOVA R,KNISCHOVA R,et al.Unusual features of the large linear plasmid pSA3239 fromStreptomyces aureofaciensCCM 3239[J].Gene,2018,642:313-323.
[9]AGARWAL V,MILES Z D,WINTER J M,et al.Enzymatic halogenation and dehalogenation reactions:Pervasive and mechanistically diverse[J].Chem Rev,2017,117(8):5619-5674.
[10]LI X G,TANG X M,XIAO J,et al.Harnessing the potential of halogenated natural product biosynthesis by mangrove-derived actinomycetes[J].Mar Drugs,2013,11:3875-3890.
[11]LU Y,YUE C,SHAO M,et al.Molecular genetic characterization of an anthrabenzoxocinones gene cluster inStreptomycessp.FJS31-2 for the biosynthesis of BE-24566B and zunyimycin ale[J].Molecules,2016,21(6):E711.
[12]LU Y,SHAO M,WANG Y,et al.Zunyimycins B and C,new chloroanthrabenzoxocinones antibiotics against methicillin-resistantStaphylococcus aureusandEnterococcifromStreptomycessp.FJS31-2[J].Molecules,2017,22(2):E251.
[13]杨小燕,金晶,周梦洁,等.利用靶向Ⅱ型聚酮合酶的PCR技术从海洋放线菌中发现angucyclinone聚酮类天然产物[J].中国药学,2017(3):173-179.
[14]李园园,保玉心,吕玉红,等.梵净山保护区土壤放线菌分离及其次级代谢产物初步研究[J].长江大学学报,2013(8):60-65.
[15]王苗,李园园,邵美云,等.赤水丹霞来源抗白色念珠菌放线菌分离及进化分析[J].遵义医学院学报,2014,37(4):404-408.
[16]岳昌武,李园园,黄兵,等.赤水丹霞山土壤来源抗菌活性链霉菌的分离及放线菌素产生链霉菌Streptomycessp.CSDX001发酵产物分析[J].中国酿造,2014,33(11):41-46.
[17]王希玮,刘超兰,郭义东,等.湖泊放线菌SIIA-A16124的分类鉴定及基因组挖掘[J].中国抗生素杂志,2018,43(4):424-428.
[18]KUMAR S,STECHER G,LI M,et al.MEGA X:molecular evolutionary genetics analysis across computing platforms[J].Mol Biol Evol,2018,35(6):1547-1549.
[19]SAITOU N,NEI M.The neighbor-joining method:a new method for reconstructing phylogenetic trees[J].Mol Biol Evol,1987,4(4):406-425.
[20]LIAO L,CHEN R,JIANG M,et al.Bioprospecting potential of halogenases from Arctic marine actinomycetes[J].BMC Microbiol,2016,16:34.
[21]HOMUNG A,BERTAZZO M,DZIARNOWSKI A,et al.A genomic screening approach to the structure-guided identification of drug candidates from natural sources[J].Chem Bio Chem,2007,8(7):757-766.
[22]刘发旺,周梦洁,金晶,等.利用靶向非核糖体肽合成酶的PCR技术从链霉菌HMU0027中发现thiazostatin嗜铁素[J].中国药学,2017(10):47-56.
[23]程少军,刘鸿雁,龙云川,等.黔东北喀斯特土壤放线菌多样性研究[J].贵州大学学报,2017(3):35-40.
[24]CHRISTIANSEN G,DITTMANN E,VIA ORDORIKA L,et al.Nonribosomal peptide synthetase genes occur in most cyanobacterial genera as evidenced by their distribution in axenic strains of the PCC[J].Arch Microbiol,2001,176(6):452-458.