竹笋(bamboo shoot)是禾本科多年生植物竹子的嫩茎,一般有春笋、冬笋、夏秋季节毛竹笋等,种类多,适应性强,分布广,除部分鲜食外大部分干制食用。鲜竹笋含有丰富的营养并且味道鲜美,膳食纤维丰富,但因其采收期短、易纤维化及对贮藏条件要求高等问题[1-3],因此我国每年有超过60%的鲜竹笋原料被用来加工制成笋干及即食调味笋等产品[4]。酸笋是以鲜竹笋为原料,配以符合饮用要求的水,不加盐直接浸泡,自然发酵而具有特殊风味的特色食材。鲜竹笋经泡制发酵后不仅风味独特,还可以延长其的保存期[5-6],在我国南方地区及东南亚部分国家都有食用酸笋的习惯。
酸笋在泡制过程中形成特殊的风味和特殊滋味,除了部分来源于竹笋本身营养成分外,与微生物参与代谢有密切关系[7-8],因此研究和分析酸笋泡制中微生物菌群结构对于揭示酸笋风味及酸笋营养物质的转化具有重要作用。SINGHAL P等[9]以印度东北部地区传统发酵竹笋为原料,通过生理生化试验发现发酵大笋中乳酸杆菌属(Lactobacillus)占绝对优势,采用传统的分离培养方法从样品中分离得到13株菌,其中有8株均为乳杆菌属。TAMANG B等[10]对印度东北部地区发酵竹笋制品mesu、soidon、soibum和soijim中的乳酸菌进行分离并结合随机扩增多态性脱氧核糖核酸标记-聚合酶链式反应(randomamplifiedpolymorphic deoxyribonucleicacidpolymerasechainreaction,RAPD-PCR)、16S核糖体核糖核酸(ribosomal ribonucleic acid,rRNA)基因测序和脱氧核糖核酸(deoxyribonucleic acid,DNA)探针杂交技术等方法对乳酸菌进行基因型鉴定,发现其中的主要功能性乳酸菌有短乳杆菌(Lactobacillus brevis)、植物乳杆菌(Lactobacillus plantarum)、弯曲乳杆菌(Lactobacillus curvatus)、戊糖片球菌(Pediococcus pentosaceus)、Leuconostocfallax、乳酸明串珠菌(Leuconostoc lactis)等。THAKURK等[11-12]从非盐渍酸性发酵食品soidon的液体发酵剂“soidon mahi”中共分离得到163株菌,通过扩增核糖体DNA限制性内切酶分析(amplified ribosomal DNA restriction analysis,ARDRA)、16S rDNA测序和随机扩增多态性DNA(RAPD)分析发现,该种酸性发酵剂的pH值为4.50±0.15,其中主要的细菌菌属为芽孢杆菌(Bacillus)和乳杆菌属(Lactobacillus)。目前有关于酸笋的外文文献中多以乳酸菌为研究重点,而国内有一些关于竹笋加盐腌制过程中微生物多样性的研究报道[13-16],低盐发酵过程中乳酸菌相对较多,高盐发酵微生物差异很大。但目前还没有采用泡制不加盐发酵过程中微生物群落变化的相关研究。
本试验以10年老酸水发酵酸笋样品(bamboo shoot full aged fermentation,BSFAF)、泉水发酵6个月块状酸笋样品(massivebambooshootfermentation,MBSF)和丝状酸笋样品(filamentous bamboo shoot fermentation,FBSF)为研究对象,采用Illumina MiSeq高通量测序技术对其细菌多样性进行研究,初步探索酸笋细菌菌群结构,对比不同发酵时长和不同原料形状对样品中菌群结构的影响,为后续探索风味物质的形成与竹笋成分、微生物之间的相关性研究奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
新鲜竹笋(毛竹笋):广西省柳州市桂平县;10年老酸水及泉水:广西省柳州市桂平县;10年老酸水发酵酸笋样品(BSFAF)、利用泉水发酵6个月的块状酸笋样品(massive bamboo shoot fermentation,MBSF)、丝状酸笋(filamentous bamboo shoot fermentation,FBSF)样品:由实验室自制。
1.1.2 化学试剂
Phusion热启动Flex 2X预混液(Pusion Hot Start Flex 2X Master Mix):上海仪涛生物仪器有限公司;DL2000 DNA Marker:日本Takara公司;核酸定量分析试剂盒(QubitdsDNA HSAssayKit):美国英杰生命技术有限公司;BiowestAgarose琼脂糖G-10:法国Biowest公司;50×TAE 缓冲液:上海生工生物工程股份有限公司;AxyPre聚合酶链式反应(poly merase chain reaction,PCR)清洁试剂盒:美国爱思进公司;RStool DNA试剂盒:美国Omega公司。
1.2 仪器与设备
5424型常温离心机:德国Eppendorf公司;Biocen 22 R冷冻离心机:德国WIGGENS公司;WH-861型旋涡振荡仪:太仓华利达实验室设备公司;DK-8D型三温三控恒温水浴锅:上海博迅实业有限公司;A200型PCR仪:杭州朗基科学仪器有限公司;MiSeq测序仪:美国Illumina公司;EPS300型电泳仪、Tanon-2500型凝胶成像仪:上海天能公司;DWHL388型超低温冷冻储存箱:中科美菱低温科技有限责任公司;水系微孔滤膜(Φ50mm,孔径0.22微米):海宁郭店桃园膜分离设备厂。
1.3 试验方法
1.3.1 酸笋制作方法
将新鲜竹笋外壳去除后用清水清洗,切成块状或条状,然后装入容器中填满,并添加泉水将其完全覆盖后,以保鲜膜封口再盖上盖子进行密封发酵。10年老酸水发酵酸笋样品则是以加工成块状的新鲜竹笋泡入10年老酸水中,并按上述步骤进行密封发酵。
1.3.2 酸笋样品总DNA提取
将发酵6个月的块状酸笋样品(MBSF)、丝状酸笋(FBSF)样品和以10年老酸水发酵酸笋样品(BSFAF)搅拌均匀后各取50g,然后用孔径为0.22μm的水系微孔滤膜进行抽滤。将抽滤完成后的滤膜剪碎放入微型离心管中,按照RStool DNA试剂盒说明书提取总DNA。
1.3.3 酸笋16S rDNA高通量测序方法
以酸笋总DNA为模板,进行细菌16S rDNA V3-V4区域扩增。引物为:338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')。
PCR扩增体系:25 μL包含DNA模板25 ng、上下游引物各为2.5 μL、12.5 μL PCR预混合物。
PCR扩增条件:98℃预变性30 s;98℃变性10 s,54℃退火30 s,72℃延伸45 s,循环35次;最后72℃延伸10 min。
取PCR产物10 μL用2%琼脂糖凝胶电泳进行检测,将检测合格样品送至杭州联川生物技术有限公司进行高通量测序。
1.3.4 数据分析
对下机数据首先根据样品的条形码(barcode)信息对数据进行拆分,再根据双端序列的重叠(overlap)关系将序列拼接(merge)成长的tags,并将序列上建库引入的barcode和引物序列去除。为了保证后续结果的可靠性,使用Vsearch(V2.3.4)软件过滤去除长度小于100 bp的序列、含不确定碱基达5%以上的序列及嵌合体序列等低质量序列[17]。再以平均邻近聚类算法通过Verseach(V2.3.4)将具有97%以上相似性的序列划分为同一个操作分类单元(operational taxonomic units,OUT)[18-19],挑选代表性序列使用。在核糖体数据库项目(ribosomal database project,RDP)中进行序列比对,明确序列的分类地位。利用MAFFT(V 7.310)软件对不同类群优势种群的差异进行多序列比对,研究不同OTU间的系统发育关系。同时,可根据每个OTU代表序列与RDP数据库的比对,获得到各样本的物种注释结果[20-21],经过分类统计后可得到各分类水平上物种的相对丰度信息。使用Verseach(V2.3.4)计算样品的各项数据指标,通过Alpha和Beta多样性分析研究单个样本中物种的多样性和多个样本间菌群结构的差异[22-23]。
1.3.5 数据处理
按照生物学样品处理基本要求,所有样品均有3个生物学重复样品。采用SPSS 18.0和Excel 2019进行数理统计分析和相关性分析。
2 结果与分析
2.1 三个不同样品测序数据统计分析
通过IlluminaMiSeq测序后9个样品共产生了148656条有效数据(ValidTags),本次测序有效数据统计结果见表1。
表1 有效数据统计
Table 1 Statistics of effective data
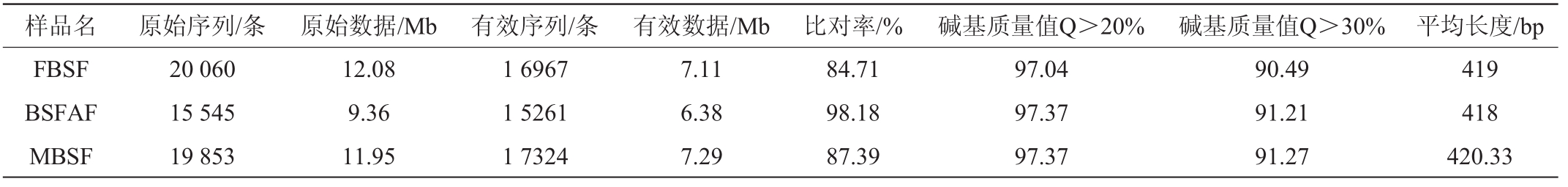
样品名 原始序列/条 原始数据/Mb 有效序列/条 有效数据/Mb 比对率/% 碱基质量值Q>20% 碱基质量值Q>30% 平均长度/bp FBSF BSFAF MBSF 20 060 15 545 19 853 12.08 9.36 11.95 1 6967 1 5261 1 7324 7.11 6.38 7.29 84.71 98.18 87.39 97.04 97.37 97.37 90.49 91.21 91.27 419 418 420.33
由表1可知,碱基质量值>20%(Q>20%)和30%(Q>30%)的数据均在90%以上,平均长度为418.89 bp,表明此次测序质量较好,可进行后续数据分析。
2.2 三个不同样品OTUs统计及分类学分析
对测序获得的148656条有效数据进行聚类分析,OTUs聚类后3组样品中总共得到1116个OTUs,根据3组样品中的OTUs聚类绘制的Venn图见图1。
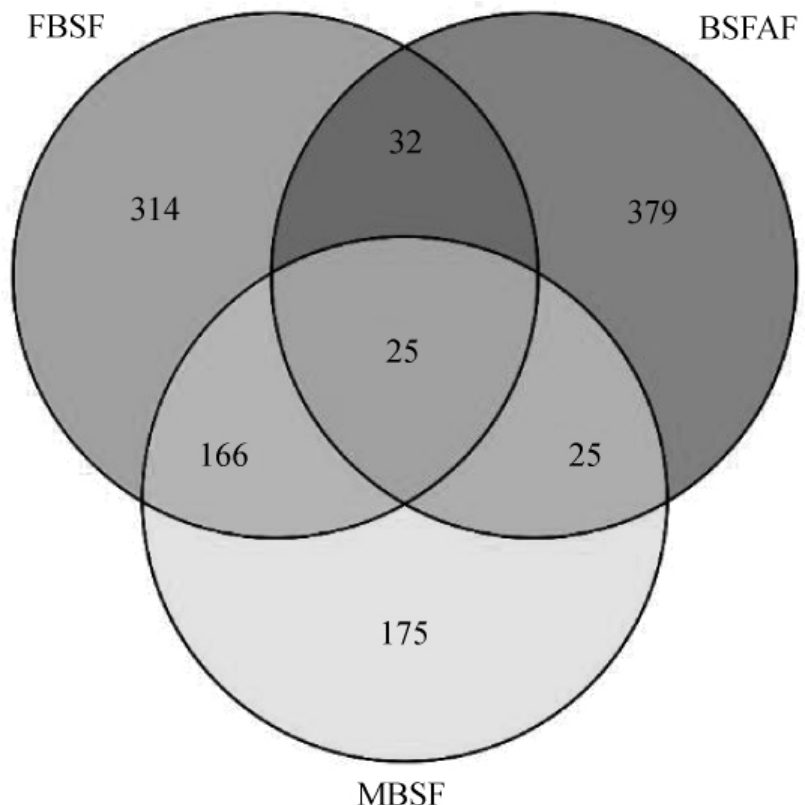
图1 三个样品中OTU分布Venn图
Fig.1 Venn diagram of OTU distribution in three samples
由图1可知,FBSF、BSFAF和MBSF样品中OTUs的数目分别为537、461及391,而3组样品中共有的OTUs仅有25种。FBSF和BSFAF共有57种,FBSF和MBSF共有191种,BSFAF和MBSF共有50种。对FBSF和MBSF样品中OTUs数目进行比较后发现,在相同试验条件下泡制的不同形状的竹笋中细菌菌群数量存在差异,说明原料处理方式如全笋发酵、块状发酵及丝状发酵可能对发酵过程中微生物种类产生影响。另一方面,对比块状酸笋和10年老酸水泡制的酸笋的OTU,可以发现酸水的来源也可能影响微生物数量。
2.3 三个不同样品Alpha多样性分析
Chao1指数曲线是反映样品微生物丰富度的指标,也可以用来说明样本的测序数据量是否合理。当曲线趋向平坦时,说明测序数据量合理,更多的数据量只会产生少量新的OTU,反之则表明继续测序还可能产生较多新的OUT。基于高通量测序技术对酸笋细菌的Chao1指数进行分析,Chao1指数曲线见图2。
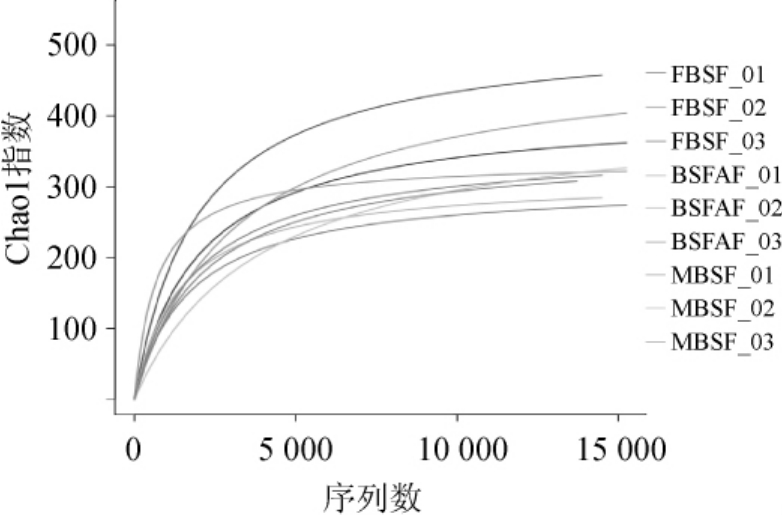
图2 酸笋样品稀释曲线Fig.2 Dilution curves of sour bamboo shoot samples
由图2可知,随着测定序列数目的增加,样本的Chao1指数值均>300,说明3种样品的物种多样性和丰富度都较高。从样品重复性稀释曲线情况来说,曲线均趋于相对平缓,测序达到10000以上基本趋于平缓,可以说明样品中基本代表样品OTU实际情况,能够反应样品中微生物多样性丰富程度,可以评价覆盖所有微生物类群。
3组样品中细菌群落多样性指数的统计结果见表2。由表2可知,coverage指数均>0.99,因此证明本次实验的取样合理。FBSF的Shannon和Simpson指数均显著高于其他酸笋样品(P<0.05),分别为3.60和0.85;而MBSF分别为2.89和0.77,低于FBSF;BSFAF这两个指数均最低,分别为2.26和0.45,且3组样品间差异显著(P<0.05)。
FBSF的ACE指数和检测物种总数最高,显著高于其他两组(P<0.05),而BSFAF和MBSF两组的ACE指数与检测物种总数无显著性差异(P>0.05),表明BSFAF和MBSF两组中微生物物种丰富度几乎相同且与FBSF组显著不同。这些结果表明,原料切分的状态不同,与酸水接触面积差异大,可能导致发酵酸笋中微生物的丰富度与均匀性差异较大。
表2 酸笋样品中细菌群落多样性指数统计
Table 2 Diversity index of fungal community in sour bamboo shoot samples
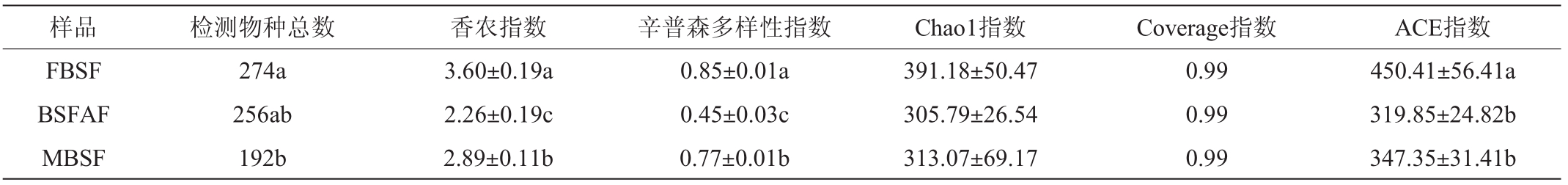
注:同列不同小写字母表示组间差异显著(P<0.05)。
样品 检测物种总数 香农指数 辛普森多样性指数 Chao1指数 Coverage指数 ACE指数FBSF BSFAF MBSF 450.41±56.41a 319.85±24.82b 347.35±31.41b 274a 256ab 192b 3.60±0.19a 2.26±0.19c 2.89±0.11b 0.85±0.01a 0.45±0.03c 0.77±0.01b 391.18±50.47 305.79±26.54 313.07±69.17 0.99 0.99 0.99
此外BSFAF和MBSF两组比较发现,虽然不同来源酸水泡制的样品中物种数量差异不显著(P>0.05),但MBSF中微生物菌群的丰富度和均匀性显著高于10年老酸水泡制的酸笋(P<0.05)。由此说明以泉水泡制发酵酸笋可能更好。
综上,FBSF的Alpha多样性优于其他组,由此说明丝状发酵的酸笋在微生物多样性上有明显优势。
2.4 三种不同样品Beta多样性分析
对测序获得的有效数据进行Beta多样性分析,采用R语言vegan包进行相似性分析,基于weighted unifrac和unweighed unifrac两种距离算法,来检验组间的差异显著性,结果见表3。
表3 相似性分析结果
Table 3 Results of analysis of similarities
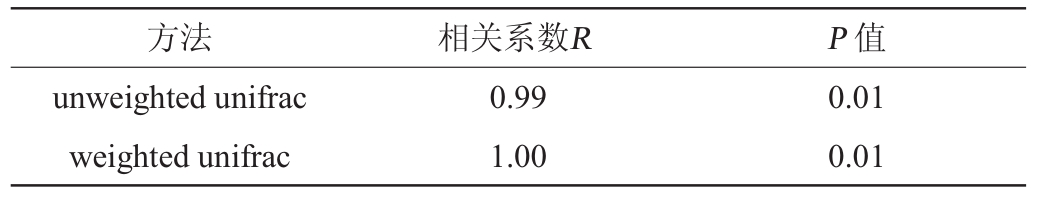
方法 相关系数R P值unweighted unifrac weighted unifrac 0.99 1.00 0.01 0.01
由表3可知,在weighted unifrac和unweighed unifrac两种距离算法下,相关系数R分别为1.00和0.99,且P值均<0.01,表明样本间的Beta多样性差异很大。
3个样品主坐标分析(principalcoordinateanalysis,PCoA)结果见图3。由图3可知,在weighted unifrac算法下,其坐标轴1、2主要贡献度之和为99.52%,表明已经可以较好地反映各样本在图上的种群结构差异,计算3组样本在图3中点的分布距离可以看出,FBSF和MBSF两组距离较近,但与BSFAF组的距离远,表明FBSF和MBSF样品中的细菌菌群结构比较接近,与BSFAF组中细菌菌群结构差异较大,可能与两个6个月发酵样品发酵条件类似有关,但10年老酸水发酵酸笋酸水中积累了较多菌群,因此菌群结构才会出现较大的差异。
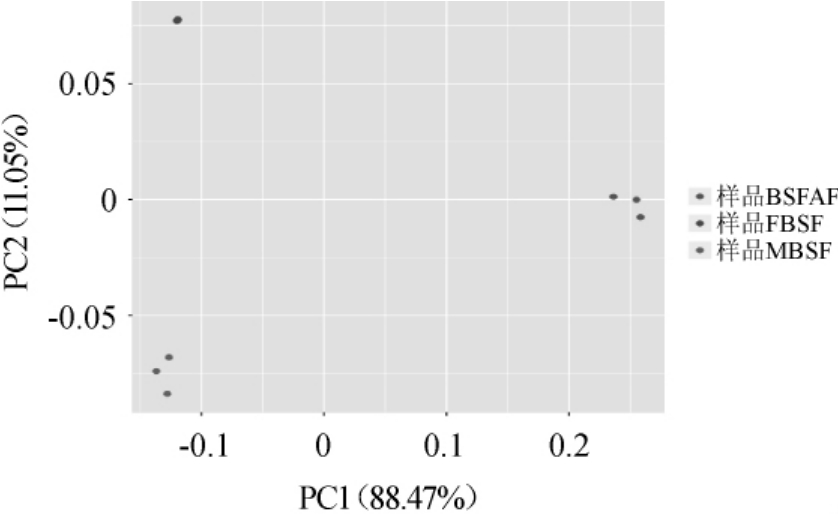
图3 主坐标分析结果
Fig.3 Results of principal coordinate analysis
2.5 三种不同样品中物种注释及分类
将前面分析获得的OTU代表序列与核糖体数据库进行比对,门分类水平上样品菌群差异结果见图4,属分类水平上样品菌群差异结果见图5和图6。
由图4可知,在门分类水平上,3组样品中共注释到12个门类,分别是厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、异常球菌-栖热菌门(Deinococcus-Thermus)、未知细菌类(Bacteria unclassified)、蓝藻门(Cyanobacteria)、Candidatus Saccharibacteria、梭杆菌门(Fusobacteria)、芽单孢菌门(Gemmatimonadetes)、酸杆菌门(Acidobacteria)及绿弯菌门(Chloroflexi)。FBSF、MBSF和BSFAF 3组样品中厚壁菌门(Firmicutes)是占绝对优势的门类,平均相对丰度分别为99.94%、87.02%和92.64%,其次是变形杆菌门(Proteobacteria)和放线菌门(Actinobacteria)。3组样品中BSFAF组中注释到细菌门类最多有11个,而FBSF组和MBSF组样品中注释的门类较少,各有3个,这可能是因为10年老酸水在长期重复发酵中有微生物沉积,所以细菌门类最多。
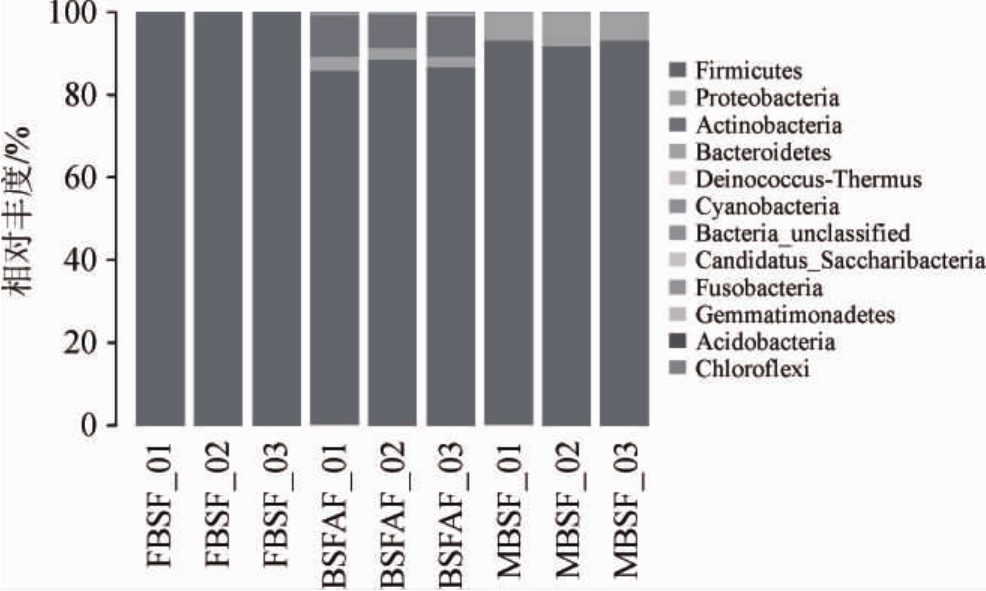
图4 门分类水平上样品相对丰度
Fig.4 Relative abundance of sample at the phylum classification level
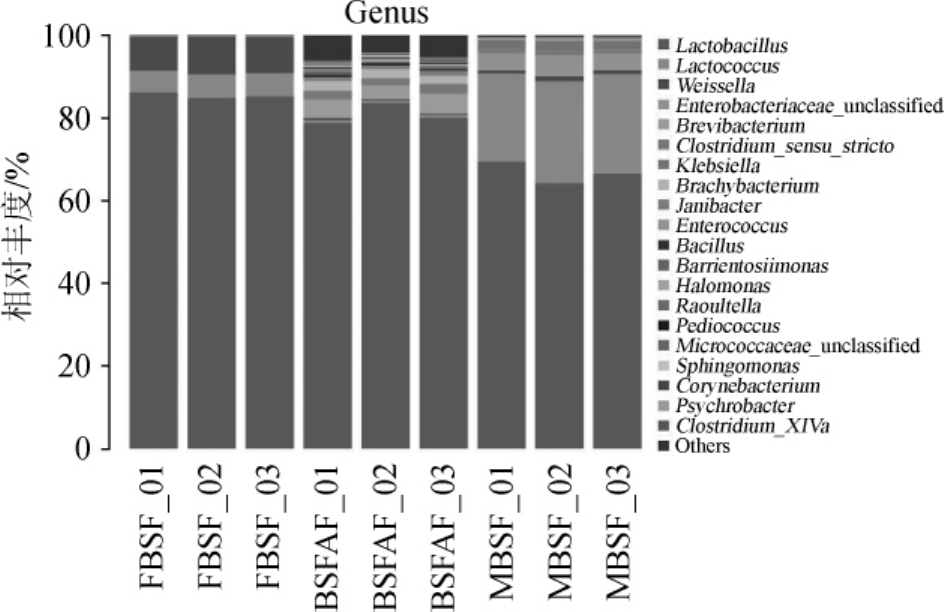
图5 属分类水平上样品相对丰度
Fig.5 Relative abundance of sample at the genus classification level
属分类水平上的物种注释图和热图分别见图5和图6。由图5和图6可知,在FBSF、MBSF和BSFAF3组样品中的优势菌属均为乳杆菌属(Lactobacillus)。其次,从FBSF和MBSF两组中可看出,虽然试验条件相同,但块状和丝状酸笋样品中的细菌菌群结构存在较明显的差异,FBSF组中的乳杆菌属(Lactobacillus)显著高于块状酸笋(MBSF)组,其平均相对丰度分别为85.39%和66.66%。但3组样品中,仅MBSF组中乳球菌属(Lactococcus)的平均相对丰度最高(平均相对丰度为23.39%)。造成这样差异的原因可能是与原料的加工程度有关,当原料被切丝后使竹笋中的内生菌外流并且参与到发酵过程中。最后,对比BSFAF组与发酵6个月的两种酸笋样品组的细菌菌群结构可发现,发酵时间更长的无盐泡制酸笋中细菌种类较其他两组更多,这可能是发酵时间长,老酸水可以作为下一批次发酵“引子”的物质基础。这也说明10年老酸笋水中的微生物菌群一直处于动态变化中,从而在菌群结构及丰度上与发酵时间短的的酸笋混合样品存在较大差异。

图6 属分类水平上热图分析
Fig.6 Heatmap analysis at genus level
此外对3组样品中常见益生菌菌属:乳杆菌属(Lactobacillus)、乳球菌属(Lactococcus)和魏斯氏菌属(Weissella)的平均相对丰度的总和进行统计后发现,FBSF组中平均相对丰度的总和最高,可达99.8%,这3个属的相对丰度含量分别为85.39%、5.52%和8.88%;其次是MBSF组,平均相对丰度的总和为91.17%,3种菌相对丰度分别为66.66%、23.39%和1.07%;而BSFAF组中最低,总和依然能达到81.94%,3种菌相对丰度分别为80.90%、0.52%和0.52%。这些结果与洪秀荣[13]采用干笋糟制过程乳酸菌包括发酵乳杆菌、片球菌、链球菌、肠膜明串珠菌有部分相似。夏秀娟等[14]研究发现,中等盐浓度研制麻竹笋,腌制过程中乳酸乳球菌含量逐渐升高,腌制14 d达到最大值时增加了6个数量级。夏秀娟等[15]研究高盐腌制麻竹笋的细菌群落组成及低盐腌制笋中微生物发酵,发现低浓度盐发酵主要包括绿色气球菌、乳球菌属、食窦魏斯氏菌(Weissella cibaria)、乳酸乳球菌(Lactococcus lactis)、芽孢杆菌属,高盐盐渍以绿色气球菌(Aerococcus viridans)、芽孢杆菌属(Bacillussp.)等[16]为主。本研究采用的新鲜麻竹笋不加盐直接泡制发酵,与高、中、低盐研制酸笋发酵结果相比,乳酸菌相对含量极高,并且Enterobacteriaceaeunclassified、克雷伯氏菌属(Klebsiella)及肠球菌属(Enterococcus)等具有潜在危害的菌属的含量极低,因此,无盐发酵酸笋可能更有利于发酵及生产。3结论
本试验采用高通量测序技术对10年老酸水泡制的酸笋样品(BSFAF)及用泉水泡制发酵6个月块状(MBSF)和丝状酸笋样品(FBSF)中细菌多样性进行研究。在3组样品中共注释到12个门、23个纲、37个目、98个科、190个属。通过在属水平上的物种注释结果可以看出,样品FBSF和MBSF中菌属的结构及相对丰度不同,其中FBSF组中益生菌属的相对丰度高于MBSF组。同时,根据Alpha多样性分析结果可知,丝状酸笋(FBSA)的Shannon、Simpson及ACE指数均显著高于块状发酵酸笋(MBSF)(P<0.05),所以丝状酸笋可能更有利于无盐发酵酸笋的生产。将块状酸笋组(MBSF)与10年老酸水组(BSFAF)对比发现,MBSF中微生物菌群的丰富度和均匀性显著高于10年老酸水泡制的酸笋,由此说明以泉水泡制发酵酸笋可能更好。
此外,本试验发现无盐发酵酸笋体系中存在大量乳酸菌类存在,是一个乳酸菌资源库。之后的研究应对发酵全过程为研究重点,全面剖析发酵过程中微生物群落变化、营养变化、风味变化规律,找到关键微生物及其基因,为竹笋生物技术加工提供强力技术支撑,为具有地域性特色的食材工业化、规模化、可控化提供理论基础。
[1]赵博,胡尚连,刘红.竹笋采后生理及储藏保鲜技术的研究进展[J].竹子学报,2017,36(3):66-71.
[2]杨乐,王洪新.不同可食性涂膜对方竹笋保鲜效果的影响[J].食品工业科技,2011,32(2):305-308.
[3]姚荷,谭兴和.竹笋加工方法研究进展[J].中国酿造,2017,36(11):24-27.
[4]李慧勤,彭见林,赵国华.竹笋加工中的营养成分变化及安全性研究进展[J].食品工业,2012(1):130-134.
[5]朱照华.酸笋的营养成分检测及其主要风味物质的研究[D].南宁:广西大学,2014.
[6]邱坚.傣味酸笋的加工工艺[J].林业科技,2003,28(3):56-57.
[7]解万翠,尹超,宋琳,等.中国传统发酵食品微生物多样性及其代谢研究进展[J].食品与发酵工业,2018,44(10):257-263.
[8]王越男,孙天松.不同发酵方式的益生菌酸奶代谢组学的应用及探究[J].现代食品科技,2017(4):88-94.
[9]SINGHAL P,SHUKLA L,SATYA S,et al.Scientific validation and process mechanism of traditional bamboo shoot fermentation by isolation and characterization of lactic acid[J].Curr Nutr Food Sci,2017,13(3):1-6.
[10]TAMANG B,TAMANG J P,SCHILLINGER U,et al.Phenotypic and genotypic identification of lactic acid bacteria isolated from ethnic fermentedbambootendershootsofNorthEast India[J].Intl J Food Microbiol,2008,121(1):35-40.
[11]THAKUR K,RAJANI C S,TOMAR S K,et al.Fermented bamboo shoots:a rich niche for bioprospecting lactic acid bacteria[J].J Bacteriol Mycol,2016,3(4):1034-1041.
[12]JEYARAM K,ROMI W,SINGH T A,et al.Bacterial species associated with traditional starter cultures used for fermented bamboo shoot production in Manipur state of India[J].Int J Food Microbiol,2010,143(1):1-8.
[13]洪秀荣.竹笋糟制工艺及其不同发酵时期微生物菌群研究[D].福州:福建农林科技大学,2014.
[14]夏雪娟,郑炯,叶秀娟,等.实时荧光定量PCR技术监测腌制麻竹笋中乳酸乳球菌动态变化[J].食品科学,2016,37(4):88-92.
[15]夏雪娟,李冠楠,叶秀娟,等.麻竹笋腌制过程中细菌群落动态变化分析[J].中国食品学报,2015,15(11):11-16.
[16]郑炯,夏雪娟,叶秀娟,等.PCR-DGGE技术分析腌制麻竹笋中微生物多样性[J].食品科学,2014,35(21):170-174.
[17]BLAXTER M,MANN J,CHAPMAN T,et al.Defining operational taxonomic units using DNA barcode data[J].Philosoph Transact Biol Sci,2005,360(1462):1935-1943.
[18]ROGNES T,FLOURI T,NICHOLS B,et al.VSEARCH:a versatile open source tool for metagenomics[J].Peer J,2016,4(10):1-28.
[19]CAPORASO JG,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nature,2010,7(5):335-336.
[20]COLE J R,CHAI B,FARRIS R J,et al.The Ribosomal Database Project(RDP-II):sequences and tools for high-throughput rRNA analysis[J].Nucl Acid Res,2005,33(Database issue):294-296.
[21]佟婷婷,田丰伟,王刚,等.基于宏基因组分析四川泡菜母水作引子的泡菜发酵过程中细菌多样性变化[J].食品工业科技,2015,36(21):173-177.
[22]LOZUPONE C A,HAMADY M,KELLEY S T,et al.Quantitative and qualitative β diversity measures lead to different insights into factors that structure microbial communities[J].Appl Environ Microbiol,2007,73(5):1576-1585.
[23]KÕLJALG U,LARSSON K H,ABARENKOV K,et al.UNITE:a database providing web-based methods for the molecular identification of ectomycorrhizal fungi[J].New Phytologist,2005,166(3):1063-1068.