D-核糖是一种功能性戊糖,是核糖核酸(ribonucleic acid,RNA)、腺嘌呤核苷三磷酸(adenosine triphosphate,ATP)的重要组成成分[1],具有改善心脏缺血、提升心脏功能、增强机体能量、缓解肌肉酸痛等生理功效[2],在食品[3]、医药[4-5]等领域中应用广泛。微生物发酵法是目前公认的最经济、高效的D-核糖生产方式[6-7]。国内外D-核糖工业化生产多采用传统诱变选育的野生型枯草芽孢杆菌(Bacillus subtilis)转酮酶缺陷变异株[8-9]。而传统诱变方法正突变效率低、筛选工作量大、诱变周期长,且多重诱变易造成较多无义突变和负向突变积累,致使菌株活性、生长速度、底物同化能力和环境耐受性降低[10]。因此,亟需新的育种方法构建高效D-核糖高产菌株。
2002年,ZHANG Y X等[11]首次提出了基因组重排(genome shuffling)概念,可将基因重组对象从单个基因扩展到整个基因组,在代谢工程中具有重大的应用潜力[12-13]。相比传统诱变和基因工程育种方法,基因组重排技术操作简单、易推广、见效快,可在微生物基因结构功能以及相关产物代谢表达调控机制并不清楚的情况下,在全基因组不同位置上同时发生多交换、多基因的随机整合重组,迅速将某一理想表型的多个甚至数十个突变基因优化组合,实现目的产物产量的大幅增加[14],并有效剔除负突变,改善菌种性能[15]。目前该技术已经成功应用于乳酸[16]、核黄素[17]等高产菌株的选育研究。
前期,实验室采用多重诱变育种筛选获得一株野生型D-核糖高产菌株枯草芽孢杆菌SFR-4[18],由于负突变积累,菌株SFR-4生长缓慢、抗逆性差,发酵过程易于染菌,显著增加了工业发酵过程控制难度;同时,该菌株难以进行有效基因工程操作,断绝了其工业化定向改造的可能。而以遗传背景清晰的枯草芽孢杆菌模式菌株B.subtilis 168[19]构建的转酮酶突变菌株B.subtilis SFR-3A[20],能够积累较高D-核糖,且生长速度快、环境耐受性强,并具有较成熟的基因工程操作工具,方便实现菌株分子改造,工业应用前景较好,但D-核糖产量略低于SFR-4菌株。经全基因组测序表明,菌株SFR-3A与SFR-4 D-核糖生物合成相关基因序列并无差别,分析其D-核糖产量差异可能源于其他尚未明确的全局代谢调控因子等调控机制,因此,试图筛选单个基因改造来提高B.subtilis SFR-3A D-核糖产量的难度较大。
为此,本研究首先以前期构建B.subtilis SFR-3A时获得的携带博莱霉素抗性基因(zeor)的过程菌株SFR-3A zeor为出发菌株,通过转入木糖诱导型毒性蛋白mazF表达载体,获得具有正反向双重筛选标记菌株,后与菌株SFR-4基因组重排,选育抗逆D-核糖高产工程菌。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与引物
本实验所用的菌株见表1,其中B.subtilis SFR-4[18]为多重诱变筛选获得的转酮酶突变株,B. subtilis SFR-3A zeor[20]为采用SFR-4菌株转酮酶突变序列替换B.subtilis 168菌株相应位点筛选获得的产D-核糖工程菌株,均由本实验室保藏。
表1 本研究所使用的菌株
Table 1 Strains used in the study

菌株 相关特征 来源SFR-4[18]SFR-3A zeor [20]SFR-3A-ZMS SFRCP-100 The wild type D-ribose high-producing strain isolated by mutagenesis B.subtilis 168 replacing the corresponding sites with the mutation site of tkt in SFR-4,harboring zeocin resistance gene B.subtilis SFR-3A zeor,harboring amyE-mazF-cassette The genome shuffling strain本研究本研究
本实验所用引物均由上海派森诺生物科技股份有限公司合成,如表2所示。
表2 本研究中所使用的引物
Table 2 Primers used in the study
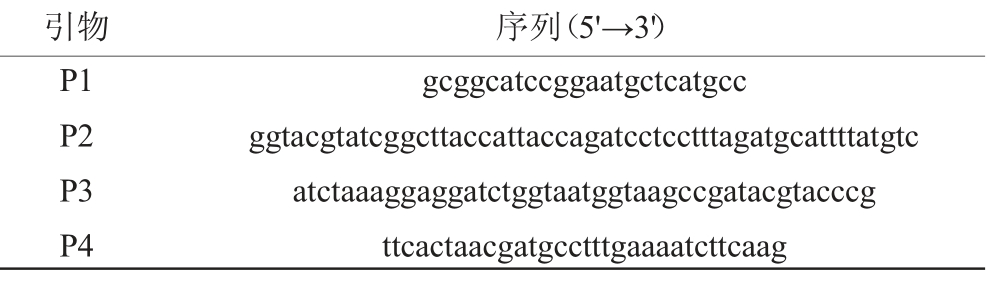
引物 序列(5'→3')P1 P2 P3 P4 gcggcatccggaatgctcatgcc ggtacgtatcggcttaccattaccagatcctcctttagatgcattttatgtc atctaaaggaggatctggtaatggtaagccgatacgtacccg ttcactaacgatgcctttgaaaatcttcaag
1.1.2 化学试剂
酵母浸粉、胰蛋白胨(均为生化试剂):英国Oxoid公司;木糖、牛肉膏(均为生化试剂)、溶菌酶(20 kU/mg)、壮观霉素、博莱霉素:生工生物工程(上海)股份有限公司;顺丁烯二酸、氯化镁、氯化钠、磷酸二氢钾、磷酸氢二钾、硫酸铵、聚乙二醇4000(均为分析纯):国药集团化学试剂(北京)有限公司;玉米浆干粉:西王集团有限公司;Prime STAR Max高保真脱氧核糖核酸聚合酶:大连宝生物工程有限公司。
1.1.3 培养基
LB培养基:酵母浸粉5 g/L,胰蛋白胨10 g/L,NaCl10g/L,121 ℃灭菌20 min。配制LB平板时加20 g/L琼脂。
CMR完全再生培养基:蔗糖171.15 g/L,葡萄糖10 g/L,牛肉膏5 g/L,胰蛋白胨10 g/L,酵母浸粉5 g/L,MgCl·27H2O 4.42 g/L,NaCl 5 g/L,pH 7.2。115 ℃灭菌30 min。
木糖筛选培养基:木糖20 g/L,酵母浸粉5 g/L,玉米浆干粉3 g/L,(NH4)2SO4 3 g/L,pH 6.5。115 ℃灭菌30 min。
种子培养基:葡萄糖20 g/L,酵母浸粉5 g/L,玉米浆干粉3 g/L,(NH4)2SO4 3 g/L,pH 6.5。115 ℃灭菌30 min。
发酵培养基:葡萄糖120 g/L,酵母浸粉10 g/L,玉米浆干粉5 g/L,(NH4)2SO4 5 g/L,pH 6.5。115 ℃灭菌30 min。
SMM缓冲液:蔗糖171.15 g/L,顺丁烯二酸2.32 g/L,MgCl·26H2O 4.06 g/L,pH 7.0。115 ℃灭菌30 min。
SMMP缓冲液:蔗糖342.29 g/L,葡萄糖4 g/L,顺丁烯二酸4.64 g/L,牛肉膏6 g/L,蛋白胨20 g/L,酵母浸粉6 g/L,MgCl·26H2O 8.12 g/L,NaCl 14 g/L,KH2PO4 5.28 g/L,K2HPO4 14.72 g/L,pH 7.0,蔗糖采用0.22 μm无菌过滤器过滤除菌。115 ℃灭菌30 min。
1.2 仪器与设备
PCT-200聚合酶链式反应(polymerase chain reaction,PCR)基因扩增仪:美国BIO-RAD公司;DYY-6C电泳仪:北京六一仪器厂;722型光栅分光光度计:上海精密科学仪器有限公司;5-L全自动机械搅拌发酵罐:上海保兴生物设备工程有限公司;SBA-40D葡萄糖分析仪:山东省科学院生物研究所;Ultimate3000高效液相色谱(high performance liquid chromatography,HPLC)仪:美国Thermo公司。
1.3 方法
1.3.1 反向筛选标记菌株构建方法
参照LIN Z等[21]描述的反向筛选标记构建方法。经化学合成后,采用引物P1/P2 PCR得到连接500 bp淀粉酶基因(amyE)上游DNA序列(amyE-UP)、壮观霉素抗性基因(Spc)r和木糖诱导型启动子PxylA的amyE-UP-Spcr-PxylA基因片段,采用引物P3/P4 PCR得到连接毒素基因mazF和500 bp amyE下游DNA序列(amyE-DN)的mazF-amyE-DN基因片段,后通过融合PCR获得amyE-mazF-cassette(见图1)。采用SPIZIZEN J[22]化学转化法将该片段转入SFR-3A zeor菌株中,并涂布于含有100 μg/mL壮观霉素的LB平板中,经菌落PCR、基因测序验证,筛选获得目的菌株。
图1 amyE-mazF-cassette反向筛选标记
Fig. 1 Schematic representation of the counter selectable marker amyE-mazF-cassette
1.3.2 基因组重排方法
(1)原生质体的制备
将目的菌株稀释涂布于LB固体培养基中,37 ℃培养24 h,挑取单菌落,接种于5 mL LB 液体培养基中,37 ℃、180 r/min摇床培养16~18 h;转接于5 mL LB液体培养基中,接种量2%,37 ℃、180 r/min摇床培养8 h;取5 mL菌液12 000 r/min离心1 min,弃上清。采用5 mL SMMP缓冲液洗涤离心2次,并将菌体悬浮于SMMP缓冲液中;加入溶菌酶(终质量浓度为2 mg/mL),混匀后,37 ℃水浴,每隔30 min进行镜检。将酶解后的细菌悬液经无菌微孔膜(0.45 μm)过滤,4 000 r/min离心10 min,收集细胞,SMMP缓冲液洗涤离心2次,获得相关枯草芽孢杆菌的原生质体。
(2)原生质体的融合
取两亲株的原生质体高渗悬浮液2 mL,混合静置5 min,3 000 r/min离心10 min,弃上清;加入0.2 mL SMM缓冲液,悬浮沉淀;加入1.8 mL 体积分数为40%的聚乙二醇4000溶液,立即混匀,37 ℃水浴融合2 min,3 000 r/min离心10 min,去上清;加入2 mL SMM缓冲液洗涤离心2次;用2 mL SMM缓冲液重新悬浮,37 ℃水浴培养90 min;将悬浮液涂布于CMR完全再生培养基上,37 ℃培养24~48 h;挑取菌落至含有20 μg/mL博莱霉素的木糖筛选培养基中筛选,获得基因组重排目的菌株。
1.3.3 摇瓶发酵培养
将菌株接种至装有50 mL种子培养基的500 mL摇瓶中,37 ℃、180 r/min摇床培养16 h。以2%接种量接种到装有50 mL发酵培养基的500 mL摇瓶中,37 ℃、180 r/min摇床培养96 h。
1.3.4 发酵罐培养
将菌株接种到装有50 mL种子培养基的500 mL摇瓶中,37 ℃、180 r/min摇床培养16 h。以2%接种量接种到装有3.5 L发酵培养基的5 L机械搅拌发酵罐中。发酵罐通风量为1.75 L/min,罐压维持在0.06 MPa,并通过调节搅拌转速维持溶解氧15%,罐温维持37 ℃,发酵过程中维持发酵液pH 6.5。
1.3.5 分析检测
(1)生物量测定
生物量采用比浊法,采用分光光度计于波长600 nm处
测定吸光度值(OD600nm值)。
(2)葡萄糖含量测定
将发酵液离心后,取上清液适当稀释后,采用SBA-40D
葡萄糖分析仪测定。
(3)D-核糖含量的测定
采用高效液相色谱法测定发酵液中D-核糖的含量。HPLC的检测条件为:Hypersil NH2色谱柱(4.6 mm×250 mm),示差折光检测器(differential refraction detector,RID)检测,进样量为10 μL,流动相为80%(V/V)乙腈,流速0.6 mL/min,柱温60 ℃。用流动相对样品进行稀释后,用0.22 μm的滤膜进行过滤,采用外标法对发酵液进行定量。
(4)D-核糖转化率和生产效率的计算
转化率=![]()
×100%生产效率[g/(L·h)]=![]()
式中:CR为D-核糖产量,g/L;CG为底物葡萄糖消耗量,g/L;t为发酵时间,h。
2 结果与分析
2.1 基因组重排选育D-核糖高产菌株
基因组重排育种可以高效地将多种正向优势性状基因集中,并增加菌株稳定性,很大程度上弥补了经典物理、化学诱变对菌株生长、底物利用及耐受性方面造成的不利影响。两株亲株进行基因组重排后,如无有效的筛选标记,可能会挑取大量未融合的无效双亲菌株,显著增加了筛选工作量。由于菌株SFR-4添加筛选标记困难,因此,本研究在菌株SFR-3Azeor中引入木糖诱导型毒性蛋白基因mazF[23]作为反向筛选标记,获得菌株SFR-3A-ZMS。将该菌株与菌株SFR-4原生质体融合后,涂布于CMR完全再生培养基中,并挑取单菌落至含有20 μg/mL博来霉素的木糖筛选培养基中,携带mazF标记的菌株SFR-3A-ZMS和无博来霉素抗性基因(zeo)r的菌株SFR-4都无法生长,大大提高了筛选效率。后对筛选平板上生长菌株摇瓶发酵,发酵结果如图2所示,在挑取100株菌株中有69株菌的D-核糖产量高于菌株SFR-3A。
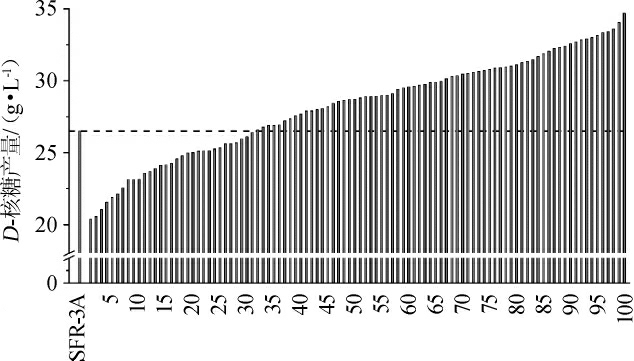
图2 基因组重排菌株摇瓶发酵结果
Fig. 2 Shake flask fermentation results of the recombinant strains by genome shuffling
基因组重排虽然可以对亲株菌株优势基因进行整合,但由于同源重组的随机性,亲本菌株缺陷基因也可能会被整合,其表达效果可能会保留甚至扩增放大。因此,在基因组重排菌株筛选过程中需对融合菌株进行多方面考察。挑取D-核糖产量提高明显的10株转化株,通过摇瓶发酵进一步考察基因组随机重排对菌株生长性能的影响,结果如图3所示,No.94、No.96、No.98菌株生长较慢,No.97、No.99、No.100菌株与菌株SFR-3A生长曲线相似。后经连续7代培养,检测该3株菌均表现出较高的遗传稳定性。选择D-核糖产量、菌株活性、遗传稳定性较高的No.100菌株为目的菌株,并命名为菌株SFRCP-100,经发酵测定,该菌株D-核糖摇瓶产量达到34.69 g/L,较菌株SFR-3A提高了32.15%。
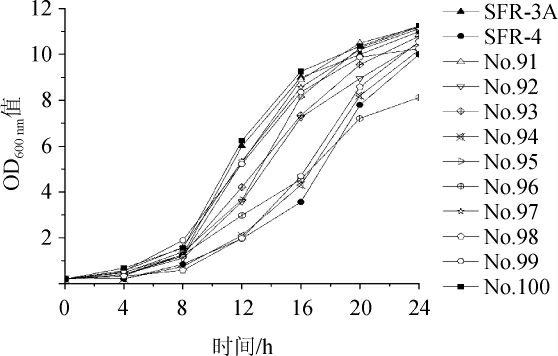
图3 菌株摇床发酵生长曲线
Fig. 3 Growth curves of the strain in shaker flasks of strains
2.2 发酵培养基氮源优化
前期菌株B.subtilis SFR-3A培养基优化研究表明,发酵培养基中氮源含量对菌株D-核糖积累的影响较为显著[20]。为此,本实验在菌株SFR-3A发酵培养基组成的基础上对培养基氮源进行了进一步优化,结果见图4。首先考察酵母浸粉对菌株SFRCP-100 D-核糖发酵影响,结果如图4a所示,对D-核糖产量进行方差分析,表明酵母浸粉添加量对D-核糖产量影响差异显著(P<0.05),在酵母浸粉添加量为5.0~7.5 g/L时,菌株SFRCP-100 D-核糖发酵产量增加明显,当酵母浸粉含量为7.5 g/L时,D-核糖产量达到最大值35.71 g/L,酵母浸粉添加量>7.5 g/L之后,D-核糖产量开始下降。玉米浆干粉添加量对菌株产D-核糖影响结果如图4b所示,对D-核糖产量进行方差分析,表明玉米浆干粉添加量对菌株产D-核糖影响差异显著(P<0.05),玉米浆干粉含量为1~3 g/L时,菌株SFRCP-100 D-核糖产量呈上升趋势,当玉米浆干粉添加量为3 g/L时,D-核糖产量达到最大值36.11 g/L,当玉米浆干粉添加量>3 g/L之后,D-核糖产量开始下降。在有机氮源优化基础上,对无机氮源硫酸铵进行了优化,结果如图4c所示,对D-核糖产量进行方差分析,表明硫酸铵添加量对D-核糖产量影响显著(P<0.05);其中硫酸铵添加量为1~7 g/L时,随着添加量增加,菌株SFRCP-100 D-核糖发酵产量增加,当硫酸铵含量为7 g/L时,D-核糖产量达到最大值37.23 g/L,当硫酸铵添加量>7 g/L之后,D-核糖产量开始下降。因此,酵母浸粉、玉米浆及硫酸铵最适添加量分别为7.5 g/L、3 g/L及7 g/L。氮源优化后菌株SFRCP-100 D-核糖发酵产量较优化前提高了7.32%。优化后的培养基总氮源添加量与菌株SFR-3A相比有所下降,说明菌株SFR-4的一些优良性能在重排菌株中得到显现。

图4 氮源组成对菌株SFRCP-100 D-核糖发酵的影响
Fig. 4 Effects of nitrogen source composition on D-ribose fermentation by strain SFRCP-100
2.3 发酵罐分批培养
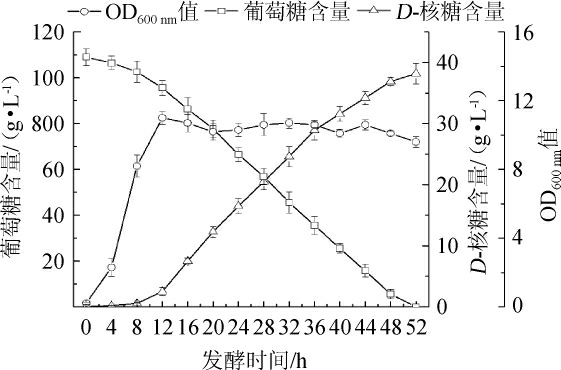
图5 菌株SFRCP-100在5L-发酵罐D-核糖发酵曲线
Fig. 5 D-ribose fermentation curve of strain SFRCP-100 in 5 L-fermentor
为研究菌株SFRCP-100 D-核糖发酵工业化应用潜力,本实验采用5 L机械搅拌发酵罐考察菌株发酵产D-核糖的发酵性能,结果如图5所示。菌株SFRCP-100 在52 h发酵周期内D-核糖产量达38.2 g/L,其转化率和整体生产效率分别达到35%和0.73 g(/L·h)。发酵过程中,工程菌株SFRCP-100在前8 h内,发酵液几乎检测不到D-核糖,8 h后,即对数生长期后期以后,发酵液中的D-核糖浓度明显提高,且直至发酵结束D-核糖仍维持相对稳定的产物生成速率;同时,工程菌株展现出了较强的菌体生长速率和底物消耗速率,在前12 h内,菌体生长较快,12 h后即达到稳定期,菌体量OD600nm值维持在10左右;培养基中葡萄糖在8 h后至发酵结束均能维持相对稳定的消耗速率;说明菌体代谢及产物生产能力随发酵时间的延长衰减较少,这可能是由于相比摇瓶发酵,采用发酵罐分批发酵可以维持稳定的发酵液pH值,且可通过调节搅拌转速实现相对较高的溶解氧条件,这均为菌体生长和产物生成创造了有利条件。发酵罐培养结果进一步表明基因组重排技术能快速提高菌株优势表型、优化代谢途径、增强菌株环境耐受性,在工业化生产菌株选育方面具有广泛的应用前景。
3 结论
基因组重排技术兼具了多种育种技术的优势,且操作简单、易推广、见效快,可在微生物基因结构功能以及相关产物代谢表达调控机制并不清楚的情况下,迅速将某一理想表型的多个甚至数十个突变基因优化组合,并有效剔除负突变,改善菌种性能。本实验通过在D-核糖生产工程菌株SFR-3A中添加正、反向筛选标记,实现基因组重排后目的菌株的高效筛选,选育获得D-核糖高产菌株SFRCP-100抗逆性强,D-核糖产量提高32.15%,经培养基优化后,菌株SFRCP-100 D-核糖发酵产量较优化前提高了7.32%,5 L发酵罐发酵D-核糖产量达到38.2 g/L,其转化率和整体生产效率分别达到35%和0.73 g(/L·h)。本实验成功添加正反向筛选标记,提高了基因组重排技术在枯草芽孢杆菌优良菌株选育中的有效性和高效性,并为结合相关生物技术发展,探索菌株选育中表型改良、耐受性增加、代谢途径调控等机制奠定了一定的理论基础。
[1] SASAJIMA K I, YONEDA M. Carbohydrate metabolism-mutants of a Bacillus species[J].J Agr Chem Soc Jpn,1971,35(4):509-517.
[2]袁保辉,丁琪,刘桂芹,等.D-核糖的生理功能及其应用[J].中国生化药物杂志,2016,36(5):210-212.
[3]DE W P,VANDAMME E J.Production of D-ribose by fermentation[J].Appl Microbiol Biot,1997,48(2):141-148.
[4] PAULY D F, PEPINE C J. D-ribose as a supplement for cardiac energy metabolism[J].J Cardiovascul Pharmacol Therap,2000,5(4):249-258.
[5]MACCARTER D,VIJAY N,WASHAM M,et al.D-ribose aids advanced ischemic heart failure patients[J].Int J Cardiol,2009,137(1):79-80.
[6]PARK Y C,LEE H J,KIM C S,et al.Effects of oxygen supply and mixed sugar concentration on D-ribose production by a transketolase-deficient Bacillus subtilis SPK1[J].J Microbiol Biot,2013,23(4):560-564.
[7]常景玲,李慧,张志宏.D-核糖发酵液预处理研究[J].中国酿造,2008,27(14):60-62.
[8]CHENG J,ZHUANG W,LI N N,et al.Efficient biosynthesis of D-ribose using a novel co-feeding strategy in Bacillus subtilis without acid formation[J].Lett Appl Microbiol,2017,64(1):73-78.
[9] WULF P D, SOETAERT W, SCHWENGERS D, et al. Optimization of D-ribose production with a transketolase-affected Bacillus subtilis mutant strain in glucose and gluconic acid-based media[J]. J Appl Microbiol,1997,83(1):25-30.
[10]王丽宁,赵妍,奚丽萍,等.基因组重排技术及其在真菌育种中的应用[J].食品工业科技,2014,35(18):362-365.
[11]ZHANG Y X,PERRY K,VINCI V A,et al.Genome shuffling leads to rapid phenotypic improvement in bacteria[J].Nature,2002,415(6872):644-646.
[12]MAGOCHA T A,ZABED H,YANG M,et al.Improvement of industrially important microbial strains by genome shuffling:Current status and future prospects[J].Bioresource Technol,2018,258(6):1-14.
[13]WANG Y,ZHANG G,ZHAO X,et al.Genome shuffling improved the nucleosides production in Cordyceps kyushuensis[J].J Biotechnol,2017,260(1):42-47.
[14]蒋承州.后基因组时代基因组重排在微生物菌种选育中的应用及展望[J].微生物学通报,2018,45(11):2494-2502.
[15]BIOTPELLETIER D,MARTIN V J.Evolutionary engineering by genome shuffling[J].Appl Microbiol Biot,2014,98(9):3877-3887.
[16] LUNA FLORES C H, PALFREYMAN R W, KRÖMER J O, et al. Improved production of propionic acid using genome shuffling[J].Biotechnol J,2017,12:1600120.
[17]YING Z,LIU J Z,HUANG J S,et al.Genome shuffling of Propionibacterium shermanii for improving vitamin B12 production and comparative proteome analysis[J].J Biotechnol,2010,148(2-3):139-143.
[18]赵祥颖,刘建军,张家祥,等.D-核糖性质、应用及其生产菌株的选育[J].食品与药品,2005,7(3):23-26.
[19] BELDA E, SEKOWSKA A, LE F F, et al. An updated metabolic view of the Bacillus subtilis 168 genome[J].Microbiology,2013,159(4): 757-770.
[20]ZHAO C,ZHAO X Y,LIU J J,et al.Enhanced D-ribose production by genetic modification and medium optimization in Bacillus subtilis 168[J].Kor J Chem Eng,2018,35(5):1137-1143.
[21]LIN Z,DENG B,JIAO Z,et al.A versatile mini-mazF-cassette for markerfree targeted genetic modification in Bacillus subtilis[J]. J Microbiol Meth,2013,95(2):207-214.
[22]SPIZIZEN J.Transformation of biochemically deficient strains of Bacillus subtilis by deoxyribonucleate[J]. Proceed Natl Acad Sci USA, 1958,44(10):1072-1078.
[23]YU H J,YAN X,SHEN W L,et al.Efficient and precise construction of markerless manipulations in the Bacillus subtilis genome[J]. J Microbiol Biot,2010,20(1):45-53.