梅干菜又称梅菜,是以雪里蕻、芥菜或油菜为主要原料,经挑选、去根、清洗、除水、腌制和干制等传统工艺制作而成的特色发酵蔬菜[1]。采用传统方法生产梅干菜时,制作环境相对开放,使得梅干菜中的微生物丰富且多样。然而,目前对梅干菜的研究多集中于风味物质和微量元素测定[2-3]、抑菌和抗氧化能力分析[4-5]以及工艺优化[6-7]等方面,有关其中微生物群落结构及多样性的研究鲜见报道。
由于某些微生物的特殊生长需求及培养条件的限制,基于纯培养的传统微生物学手段只能将样品中少于1%的微生物分离出来,而非培养技术的出现对于这个问题的解决提供了很好的帮助[8]。近年来,以Illumina MiSeq为代表的高通量测序技术发展迅速,为分析生境菌群多样性提供了有力手段,该技术无需对样品中微生物进行分离纯化,可直接对样本基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)进行分析,能够快速、直接和真实的反映菌群物种丰度及差异[9-10]。
本研究采用Illumina MiSeq技术对采自恩施地区的梅干菜样品中细菌多样性进行解析,同时采用传统微生物学手段对其中所含乳酸菌进行分离鉴定,以期为恩施地区梅干菜中微生物菌种资源的发掘与保护提供一定理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
梅干菜MGC01、MGC02和MGC03:主要原料为雪里蕻。于2017年12月采自湖北省恩施地区土桥坝菜市场。
1.1.2 试剂
氢氧化钠、碳酸钙、过氧化氢、三氯甲烷、氯化钠、三羟甲基氨基甲烷、乙二胺四乙酸二钠、乙酸钙、十二烷基硫酸钠、酚、氯仿、异戊醇、溴化十六烷基三甲铵(cetyltrimethyl ammonium bromide,CTAB)和醋酸钠(均为分析纯):国药集团化学试剂有限公司;MRS培养基:青岛海博生物技术有限公司;Axygen清洁试剂盒:北京科博汇智生物科技发展有限公司;10×PCR Buffer、脱氧核糖核苷三磷酸(deoxyribonucleoside triphosphate,dNTP)mix、DNA聚合酶(5 U/μL)、溶菌酶(400 U/μg)、蛋白酶K(20 U/μg)、pMD18-T vector、Solution I:宝生物工程(大连)有限公司;6×Loading buffer、DL500和DL2000 DNA Marker:宝日医生物技术(北京)有限公司;2×PCRmix:南京诺唯赞生物科技有限公司;QIAGEN DNeasy mericon Food Kit提取试剂盒:德国QIAGEN公司;引物均由武汉天一辉远生物科技有限公司合成,各引物信息如表1所示。
表1 实验所用引物序列信息
Table 1 Sequences information of primers used in the study
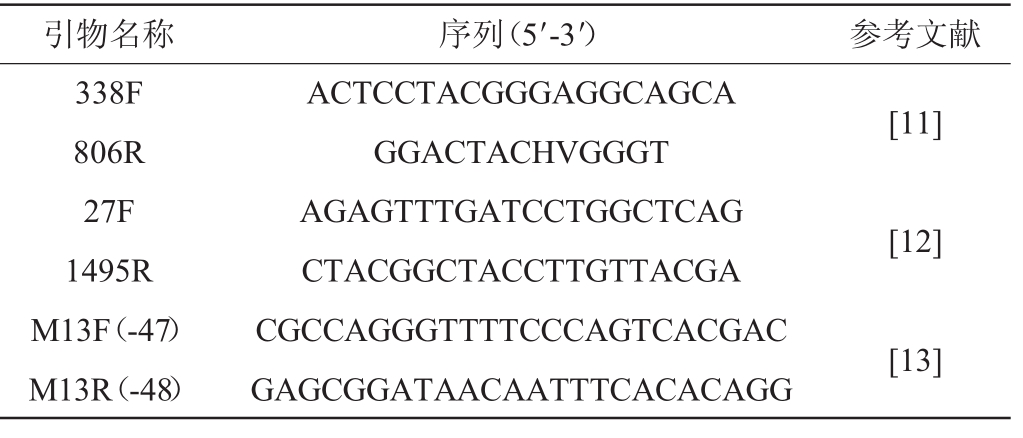
注:“F”表示正向引物;“R”表示反向引物。
引物名称 序列(5′-3′) 参考文献338F 806R 27F 1495R M13F(-47)M13R(-48)ACTCCTACGGGAGGCAGCA GGACTACHVGGGT AGAGTTTGATCCTGGCTCAG CTACGGCTACCTTGTTACGA CGCCAGGGTTTTCCCAGTCACGAC GAGCGGATAACAATTTCACACAGG[11][12][13]
1.2 仪器与设备
HR40-IIB2生物安全柜:青岛海尔特种电器有限公司;VeritiTM 96孔梯度聚合酶链式反应(polymerase chain re action,PCR)扩增仪:美国AB公司;DYY-12水平电泳仪:北京市六一仪器厂;ND-2000C微量紫外分光光度计:美国Nano Drop公司;LRH-70生化培养箱:上海一恒科技有限公司;DG250厌氧工作站:英国DWS公司;5810R台式高速冷冻离心机:德国Eppendorf公司;ECLIPSE Ci生物显微镜:日本Nikon公司;UV PCDS8000凝胶成像分析系统:美国BIORAD公司。
1.3 方法
1.3.1 宏基因组DNA提取
参照QIAGEN DNeasy mericon Food Kit提取试剂盒中的方法提取梅干菜样品宏基因组DNA,并用微量紫外分光光度计检测DNA纯度及浓度。
1.3.2 基于MiSeq高通量测序技术梅干菜细菌多样性的评价
细菌16S rRNA扩增及测序:以微生物宏基因组DNA为模板进行PCR扩增。扩增体系[14]为5×FastPfu Buffer 4μL、2.5 mmol/LdNTPmix 2μL、5μmol/L的正/反引物(338F/806R)各0.8 μL、5 U/μL r Taq酶 0.4 μL、DNA模板 10 ng,双蒸水(ddH2O)补充至20μL。PCR扩增条件为95℃预变性3 min;95℃变性30 s,55℃退火30 s,72℃延伸45 s,共循环30次;72℃再延伸10 min。PCR扩增产物经1.0%的琼脂糖凝胶电泳检测合格后寄至上海美吉生物医药科技有限公司进行Illumina MiSeq高通量测序。
质量控制:对测回的序列进行拼接,拼接时去除重叠区域碱基数>10 bp、最大错配率>0.2和样品特异性条形码(barcode)或引物碱基错配的序列,拼接后依据barcode信息将序列划分至各样品中以备后续分析[15]。
数据分析:使用QIIME数据分析平台,参照CAPORASO J G等[16]的方法对质控后的序列进行分析。首先以97%相似度划分操作分类单元(operational taxonomic units,OTU)[17],然后利用Greengenes[18]和RDP[19]数据库进行同源性比对,统计各分类水平上细菌多样性。
1.3.3 梅干菜中乳酸菌菌株的分离鉴定
乳酸菌分离纯化:采用稀释涂布平板法将样品稀释液涂布于含1.0%~1.2%碳酸钙的MRS琼脂培养基上,37℃厌氧培养48 h后挑选周围有透明圈的单菌落进行纯化,将革兰氏染色阳性和过氧化氢酶阴性的纯种菌株用30%的甘油进行菌种保藏。
乳酸菌DNA提取与鉴定:采用CTAB法[20]提取纯化菌株的DNA,然后以提取的DNA为模板进行PCR扩增,除PCR扩增所需引物为27F和1495R外,PCR扩增体系和条件均与1.3.2相同。PCR扩增产物经琼脂糖凝胶电泳检测、清洁后与PMD18-T载体连接并转化至大肠杆菌(Escherichia coli)Top10,挑取阳性克隆子送至南京金斯瑞生物科技有限公司进行测序。将测序公司返回的序列进行拼接并去除正反引物后置于美国国立生物技术信息中心(national center for biotechnology information,NCBI)上进行同源性比对,选取同源性较高的菌株,使用Mega 7.0中的邻接(Neighbor-Joining,NJ)法构建系统发育树。
1.3.4 数据处理
使用折线图评判本研究测序深度是否合适,使用维恩(Venn)图显示不同样品中共有或特有的OTU及序列数,使用柱状图展示不同样品中各门和属水平细菌种类和含量,使用系统发育树展现不同菌株间进化关系。折线图和柱状图使用Origin2017软件绘制。Venn图由Venny2.1.0在线绘制,系统发育树由Mega 7.0绘制。
2 结果与分析
2.1 基于Illumina MiSeq高通量测序技术梅干菜细菌多样性的评价
高通量测序技术具有通量高、检测速度快等优点,可更加准确、真实的反映样本中微生物群落结构的组成。因此,首先使用Illumina MiSeq高通量测序技术对干梅菜样品中细菌在门、纲、目、科和属各水平上的分类数量进行统计,然后利用超1指数和香农指数对其物种丰度和多样性进行分析,结果如表2所示。
表2 梅干菜样品测序结果及各分类地位数量
Table 2 Sequencing results and numbers at different taxonomical levels of preserved vegetable samples
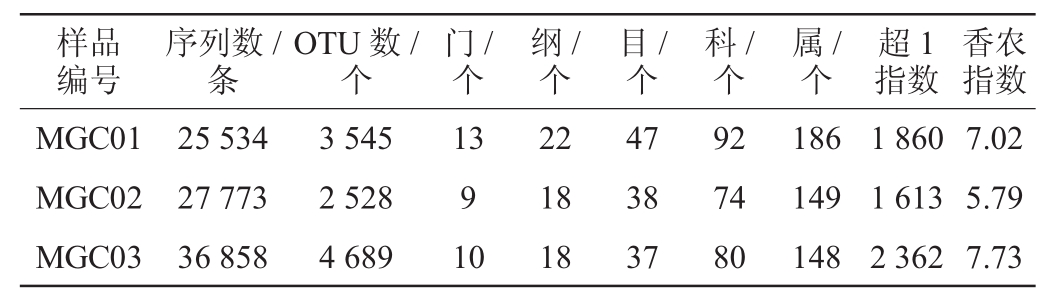
注:超1指数和香农指数均在测序量为23 610条序列时计算所得。
样品编号序列数/条OTU数/个门/个纲/个目/个科/个属/个超1指数香农指数MGC01 MGC02 MGC03 25 534 27 773 36 858 3 545 2 528 4 689 13 9 10 22 18 18 47 38 37 92 74 80 186 149 148 1 860 1 613 2 362 7.02 5.79 7.73
由表2可知,梅干菜样品MGC01、MGC02和MGC03经质控合格后的序列分别为25 534条、27 773条和36 858条,在97%的相似度下划分的OTU数分别为3 545个、2 528个和4 689个。当测序量为23 610条时,梅干菜样品MGC01、MGC02和MGC03的超1指数分别为1 860、1 613和2 362,香农指数分别为7.02、5.79和7.73,其中梅干菜样品MGC03的超1指数和香农指数值均最大,因而梅干菜样品MGC03中细菌丰度和多样性均高于梅干菜样品MGC01和MGC02。进一步对测序深度是否能捕获细菌多样性信息进行了分析,结果如图1所示。
由图1可知,当序列数达到30 000条时,稀疏曲线随序列数的增加而增加,而香农指数曲线在序列数>10 000条时就已经进入平台期,说明随着测序深度的增加可能还会有新的物种被发现但样品中微生物多样性不会再增加。由此可见,本研究的测序深度足以满足后续数据分析要求。本研究进一步对各梅干菜样品特有及样品间共有的OTU数及序列数进行分析,结果如图2所示。
由图2可知,梅干菜样品MGC01、MGC02和MGC03中特有的OTU分别为2 442个、1 464个和3 565个,所包含序列数分别为3 982条、2 305条和9 698条。梅干菜样品MGC01和MGC02共有OTU数为294个,梅干菜样品MGC02和MGC03共有OTU数为315个,梅干菜样品MGC01和MGC03共有OTU数为354个;3个梅干菜样品共有OTU数为455个,其所包含的序列为61 568条,占总序列数的68.28%。由此可见,恩施地区梅干菜样品中含有丰富的细菌物种且各样品间存在大量共有细菌菌群。
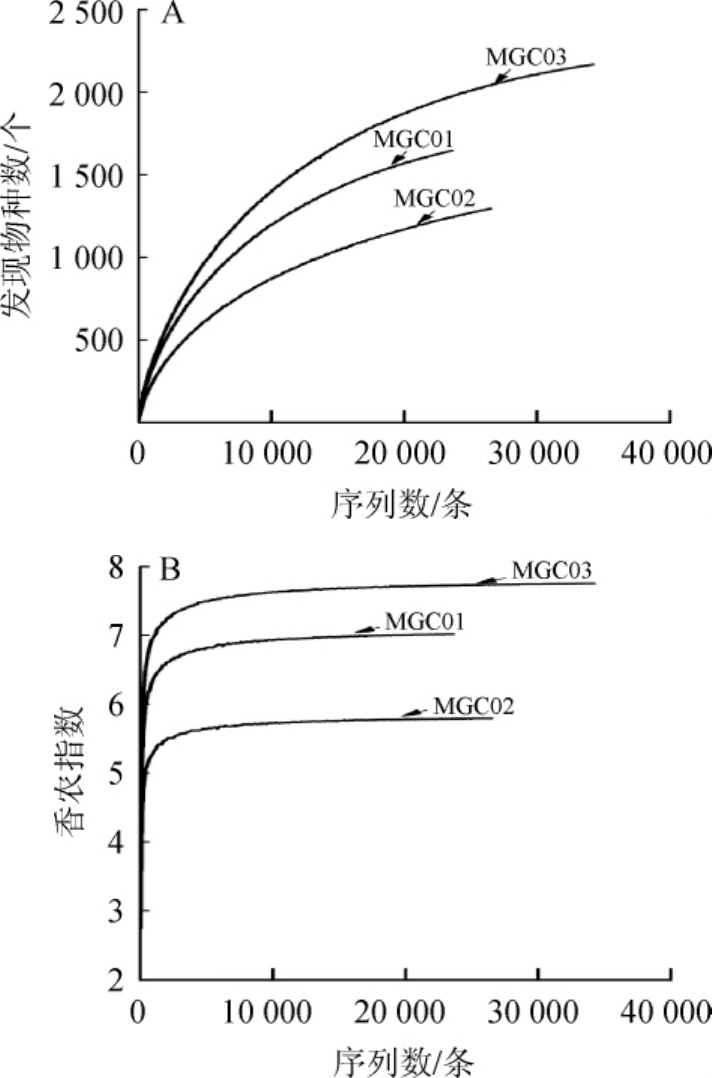
图1 稀疏曲线(A)和香农指数曲线(B)
Fig.1 Rarefaction curves(A)and Shannon index curves(B)
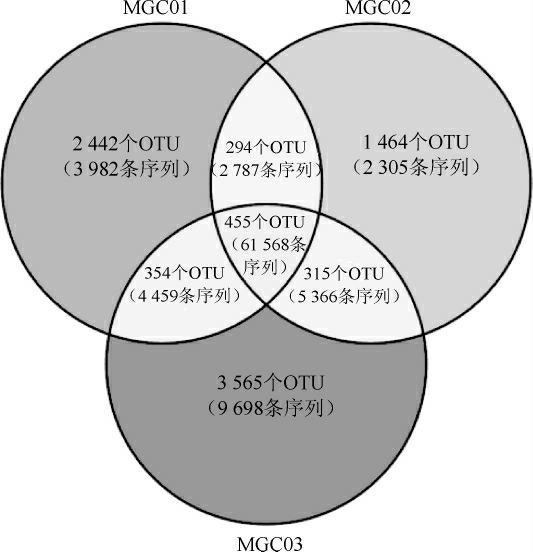
图2 梅干菜样品OTU数及序列数的维恩图
Fig.2 Venn diagram of the OTU and sequence number in preserved vegetable samples
梅干菜样品MGC01、MGC02和MGC03中所含细菌门的数量分别为13个、9个和10个,包含细菌属的数量分别为186个、149个和148个,本研究将相对丰度>1.0%的细菌门和属定义为优势细菌门和属,并对其平均相对含量进行分析,结果如图3所示。
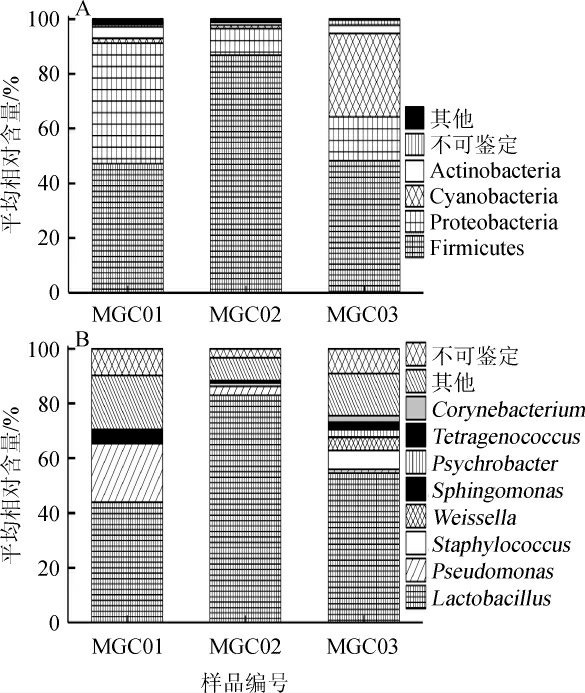
图3 梅干菜样品中优势细菌门(A)和属(B)的相对含量分析
Fig.3 Relative content analysis of dominant bacterial phyla(A)and genus(B)in preserved vegetable samples
由图3可知,恩施地区梅干菜样品中的优势细菌门为硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)、蓝细菌门(Cyanobacteria)和放线菌门(Actinobacteria),其平均相对含量分别为60.70%、23.28%、11.16%、2.76%;优势细菌属主要为乳酸杆菌属(Lactobacillus)、假单胞菌属(Pseudomonas)、葡萄球菌属(Staphylococcus)、魏斯氏菌属(Weissella)、鞘脂单胞菌属(Sphingomonas)、嗜冷杆菌属(Psychrobacter)、四联球菌属(Tetragenococcus)和棒状杆菌属(Corynebacterium),其平均相对含量分别为60.50%、8.69%、2.86%、1.73%、1.28%、1.07%、1.03%和1.00%。值得一提的是,仍有7.40%的序列不能鉴定到属水平,这进一步说明恩施地区梅干菜样品中细菌多样性较高。
2.2 梅干菜中乳酸菌菌株的分离鉴定
在使用Illumina MiSeq高通量测序技术解析发现梅干菜中的细菌主要以乳酸菌为主的基础上,本研究进一步采用传统微生物学手段从3个梅干菜样品中分离得到12株疑似乳酸菌,编号分别为MGC01-1、MGC01-2、MGC01-3、MGC01-4、MGC02-1、MGC02-2、MGC02-3、MGC02-4、MGC03-1、MGC03-2、MGC03-3和MGC03-4,疑似乳酸菌与其近源种模式株的系统发育树如图4所示。
由图4可知,菌株MGC03-3、MGC01-3、MGC01-4、MGC02-1、MGC02-2、MGC02-3和MGC02-4与Lactobacillus plantarum NBRC 15891在同一个进化分枝上,亲缘关系较近,故将其判定为植物乳杆菌(L.plantarum),同理,将菌株MGC03-1鉴定为副干酪乳杆菌副干酪亚种(Lactobacillus paracasei subsp.paracasei),菌株MGC01-1鉴定为融合魏斯氏菌(Weissella confusa),菌株MGC03-2鉴定为食品乳杆菌(Lactobacillus alimentarius),菌株MGC01-2和MGC03-4鉴定为短乳杆菌(Lactobacillus brevis)。
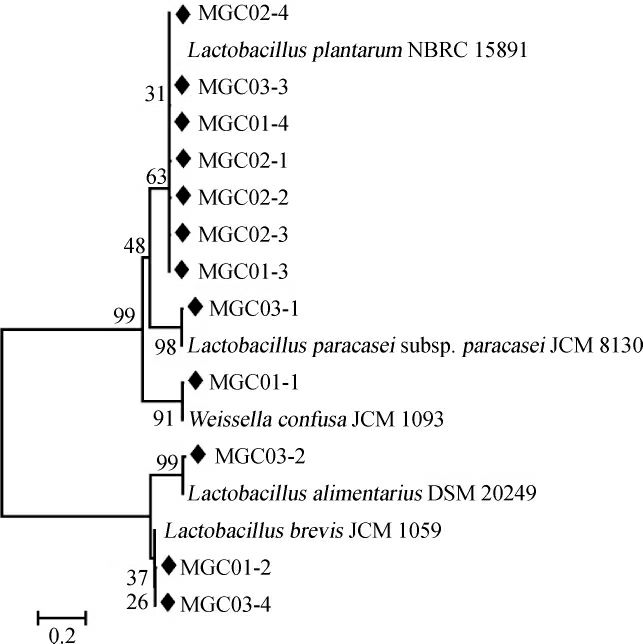
图4 梅干菜中乳酸菌系统发育树
Fig.4 Phylogenetic tree of lactic acid bacteria in preserved vegetable samples
3 结论
本研究通过Illumina MiSeq高通量测序和传统纯培养技术对恩施地区梅干菜中细菌多样性进行了解析,Illumina MiSeq高通量测序结果表明,恩施地区梅干菜样品中的优势细菌门分别为硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)、蓝细菌门(Cyanobacteria)和放线菌门(Actinobacteria),优势细菌属主要为乳酸杆菌属(Lactobacillus)、假单胞菌属(Pseudomonas)、葡萄球菌属(Staphylococcus)、魏斯氏菌属(Weissella)、鞘脂单胞菌属(Sphingomonas)、嗜冷杆菌属(Psychrobacter)、四联球菌属(Tetragenococcus)和棒状杆菌属(Corynebacterium)。经稀释涂布平板法从梅干菜样品中共分离到12株乳酸菌,包括植物乳杆菌(Lactobacillus plantarum)7株、短乳杆菌(Lactobacillus brevis)2株、副干酪乳杆菌副干酪亚种(Lactobacillus paracasei subsp.paracasei)、融合魏斯氏菌(Weissella confusa)和食品乳杆菌(Lactobacillus alimentarius)各1株。
[1]HUANG S,HUANG M,FENG B.Antioxidant activity of extracts produced from pickled and dried mustard(Brassica juncea Coss.var.foliosa Bailey)[J].Int J Food Prop,2012,15(2):374-384.
[2]程玥,徐晓兰,张宁,等.同时蒸馏萃取-气质联用分析三全梅菜扣肉的挥发性风味成分[J].食品科学,2013,34(12):147-150.
[3]卓莉,刘良凤,阮尚全.原子吸收光谱法测定梅干菜中微量元素的含量[J].安徽农业科学,2009,37,30(21):9836.
[4]黄师荣,李豪杰,戴杰辉,等.九头芥梅干菜提取物抗菌活性及其在猪肉保鲜中的应用[J].现代食品科技,2014,30(10):58-62.
[5]沈清,楼乐燕,尹培,等.五种梅干菜的酚类化合物及抗氧化能力比较分析[J].食品科学,2017,38(18):1-11.
[6]黄苇,赵玲华,李远志,等.梅菜漂洗及脱盐工艺参数优选[J].中国调味品,2009,34(6):80-82.
[7]周美琪,周其德,田赛莺,等.低盐腌制对缙云梅干菜加工品质的影响[J].核农学报,2018,32(8):1562-1571.
[8]杨翠,李祖明.分子生物学技术在肠道微生物研究中的应用进展[J].中国微生态学杂志,2017,29(2):229-233.
[9]曹荣,张井,孟辉辉,等.高通量测序与传统纯培养方法在牡蛎微生物群落分析中的应用对比[J].食品科学,2016,37(24):137-141.
[10]沈馨,王艳,代凯文,等.基于Miseq高通量测序技术的辣椒酱核心细菌类群研究[J].食品研究与开发,2018,39(10):151-167.
[11]沈馨,尚雪娇,董蕴,等.基于MiSeq高通量测序技术对3个孝感凤窝酒曲细菌多样性的评价[J].中国微生态学杂志,2018,30(5):525-544.
[12]张会敏,李天婵,孙美青,等.利用非培养技术初步研究古井贡酒窖泥细菌群落结构[J].食品工业科技,2014,35(13):200-206.
[13]OKA K,ASARIM,OMURA T,et al.Genotyping of 38 insertion/deletion polymorphisms for human identification using universal fluorescent PCR[J].Mol Cell Probe,2014,28(1):13-18.
[14]蔡丽云,黄泽彬,须子唯,等.处理垃圾渗滤液的SBR中微生物种群与污泥比阻[J].环境科学,2018,39(2):880-888.
[15]周书楠,席修璞,董蕴,等.琚湾酸浆面浆水细菌多样性评价[J].中国酿造,2018,37(1):49-53.
[16]CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nat Methods,2010,7(4):335-336.
[17]HAAS B J,GEVERS D,EARL A M,et al.Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J].Genome Res,2011,21(3):494-504.
[18]DESANTIS T Z,HUGENHOLTZ P,LARSEN N,et al.Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Appl Environ Microbiol,2006,72(7):5069-5072.
[19]COLE J R,CHAIB,FARRIS R J,et al.The ribosomal database project(RDP-II):introducing myRDP space and quality controlled public data[J].Nucleic Acids Res,2007,35(1):169-172.
[20]SUN X M,CHEN X,DENG Z X,et al.A CTAB-assisted hydrothermal orientation growth of ZnOnanorods[J].Mater Chem Phys,2003,78(1):99-104.