酱豆又称为豆豉,是一种传统特色发酵调味品,其原料为黄豆或黑豆。酱豆不仅风味独特还含有较高的蛋白质、纤维素、维生素等营养元素,且酱豆中的多种营养元素含量均高于大豆[1]。有研究指出,酱豆在预防癌症、延缓衰老、帮助消化和增强免疫等方面具有一定的积极作用[2-4]。传统发酵酱豆制作过程中的环境和工艺手法相对开放,致使其含有较为丰富且复杂的微生物菌群信息[5-6]。发酵过程中微生物在蛋白质、淀粉及脂质的水解代谢中起着重要作用,并影响产品的质量和风味。汤启成等[7]研究发现,毛霉型豆豉与曲霉型豆豉生成的挥发性化合物不一样。聂黔丽等[8]采用高通量测序技术分析遵义干豆豉,发现芽孢杆菌为其主要贡献菌株,其次是乳酸菌,风味成分中酸类含量占比最高。通过对甘肃陇南和庆阳两地传统细菌型豆豉微生物进行研究,ZHANG W B等[9]研究发现,微生物在不同来源样品中存在较大差异,地理因素可能影响豆豉样本微生物组成及丰度。研究人员对不同地区豆豉开展了较为广泛的研究,但对恩施利川豆豉研究较少。
利川市位于湖北西南端,与蜀渝相接,当地酱豆是以黄豆为原料,经选料、浸泡、蒸煮、自然发酵后,密封于陶罐中发酵而成的,制作及食用较为普遍[10]。但目前其微生物组成及风味特征尚不明晰。Illumina MiSeq高通量测序技术有通量高、方便易操作、检测结果准确且成本低等特点[11-12],在食品[13-15]及医学[16]等方面应用比较广泛。电子鼻能够检测并分析样品的整体挥发性成分,其特点是检测的速度快、测定评估的范围广且响应的时间短[17]。目前电子鼻在发酵食品[18]、中药材及农产品[19]和肠道疾病[20]等领域均有应用。这些技术的应用为本研究的开展提供了良好的支撑。
本研究采用MiSeq高通量测序技术对采集自湖北省恩施土家族苗族自治州利川市的自然发酵酱豆样品的细菌微生物群落结构进行分析,同时采用电子鼻系统测定其挥发物质的响应差异,并分析酱豆样品中细菌多样性与其风味品质的相关性,以期为后续改良酱豆风味以及筛选有益工艺生产优良菌株提供数据支持和理论指导。
1 材料与方法
1.1 材料与试剂
利川酱豆:采集自湖北省恩施土家族苗族自治州利川市,属细菌型豆豉且均为自然发酵而成,每份样品采取200g,共采取10 份酱豆样品,分别编号为JD1~JD10。将采集后的样品装入无菌采样管后置于采样箱中快速运回实验室,在-80 ℃冷冻保藏备用;DNeasy mericon Food Kit脱氧核糖核酸(deoxyribonucleic acid,DNA)基因组提取试剂盒:德国QIAGEN公司;DNA聚合酶(5 U/μL)、10×聚合酶链式反应(polymerase chain reaction,PCR)Buffer、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphates,dNTPs)Mix:北京全式金生物技术有限公司;引物338F/806R:天一辉远生物科技有限公司;Axygen清洁试剂盒:康宁生命科学吴江有限公司。
1.2 仪器与设备
5810R台式高速冷冻离心机:德国Eppendorf公司;DYY-12电泳仪:北京六一仪器厂;UVPCDS8000凝胶成像分析系统:美国Protein Simple公司;MiSeq高通量测序平台:美国Illumina公司;R920机架式服务器:美国DELL公司;Vetiri梯度基因扩增仪:美国AB公司;PEN3电子鼻(配置10个金属氧化物半导体型化学传感元件:W1W、W1C、W1S、W5S、W3C、W5S、W6S、W2W、W2S和W3S):德国Airsense公司。
1.3 方法
1.3.1 宏基因组DNA提取
取利川酱豆样品2.0 g,使用DNeasy mericon Food Kit DNA基因组提取试剂盒对酱豆样品的微生物总DNA进行提取后,用1%的琼脂糖凝胶电泳检测DNA条带是否符合试验要求,并将提取的DNA样品置于-20 ℃储存备用。
1.3.2 细菌16S rRNA V3-V4区PCR扩增及MiSeq高通量测序
对酱豆样品的DNA提取并检测后,使用引物338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')对DNA样品进行细菌16S rRNA V3-V4区的PCR扩增,将扩增产物经琼脂糖凝胶电泳检测后寄往上海美吉生物医药科技有限公司用MiSeq高通量测序平台进行测序。扩增体系和扩增条件参照王玉荣等[21]的方法。
1.3.3 序列拼接质控及基因功能预测
高质量序列的获得:对测序的序列进行拼接并质控,同时在拼接过程中剔除重叠区碱基数<10 bp的序列、最大错配比率>0.2的序列、存在barcode碱基错配或引物碱基错配数>2 bp的序列,并切掉barcode和引物后的序列碱基数<50 bp的序列。将得到的高质量序列进行生物信息学分析。
用QIIME(v1.70)平台在建立的脚本程序上对获取的高质量序列进行生物信息学分析[22]。使用PyNAST[23]对序列进行校准排齐后,应用UCLUST进行序列归并,从而在建立16S rRNA V3-V4区序列集的基础上划分操作分类单元(operational taxonomic units,OTU)[24]。最后使用核糖体数据库项目(ribosomal database project,RDP)Release 11.5[25]和Greengenes(Release 13.8)[26]数据库对序列进行同源性比对并确定其分类学地位。用PICRUSt软件对利川酱豆进行基因功能预测[27],并基于蛋白质直系同源簇数据库(clusters of orthologous groups of proteins,COG)进行功能注释[28]。
1.3.4 基于电子鼻技术利川酱豆风味品质的评价
准确称取5.0 g酱豆样品置于电子鼻样品瓶中,在室温下将电子鼻探头插入样品瓶中进行检测。本研究中每个样品测定时间为90 s,测试数据选49 s、50 s和51 s时的平均值,每个样品测3个平行。
1.3.5 多元统计学分析
使用SAS9.0软件进行利川酱豆样品中优势细菌菌属和电子鼻各传感器响应值之间的相关性数据分析,选取相关性系数绝对值>0.6为指标,采用Cytoscape软件(v3.5.1)绘制相关性网络图;使用R软件(v4.0.3)绘制瀑布图和热图;使用Origin 8.5软件绘制柱状堆积图及OTU出现次数统计图。样本细菌序列已上传至MG-RAST数据库,获取编号为mgp100938(https://www.mg-rast.org/mgmain.html?mgpage=metazen2&project=mgp100938)。
2 结果与分析
2.1 基于门和属水平的细菌群落组成分析
采用高通量测序对酱豆样品的细菌菌群多样性进行分析,将样品中相对含量<1.0%的细菌门或属归为其他类,平均相对含量≥1.0%的细菌门或属为优势细菌门或属。首先对酱豆细菌门水平的细菌菌群结构进行分析,结果见图1。
由图1可知,利川酱豆样品中的细菌门有硬壁菌门(Firmicutes)(99.27%)、变形菌门(Proteobacteria)(0.31%)、蓝细菌门(Cyanobacteria)(0.20%)、拟杆菌门(Bacteroidetes)(0.04%)和放线菌门(Actinobacteria)(0.03%),其中有0.15%的门序列未被鉴定到。由此可知利川酱豆样品中的细菌微生物主要隶属于Firmicutes。利川酱豆中细菌属水平分析结果见图2。
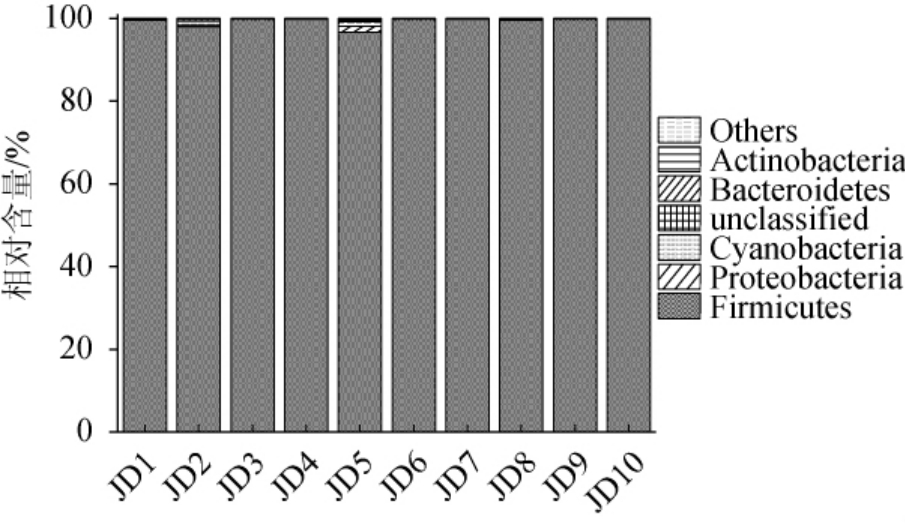
图1 基于门水平利川酱豆样品中细菌菌群结构
Fig. 1 Bacterial community structure of Lichuan Jiangdou samples based on phylum level
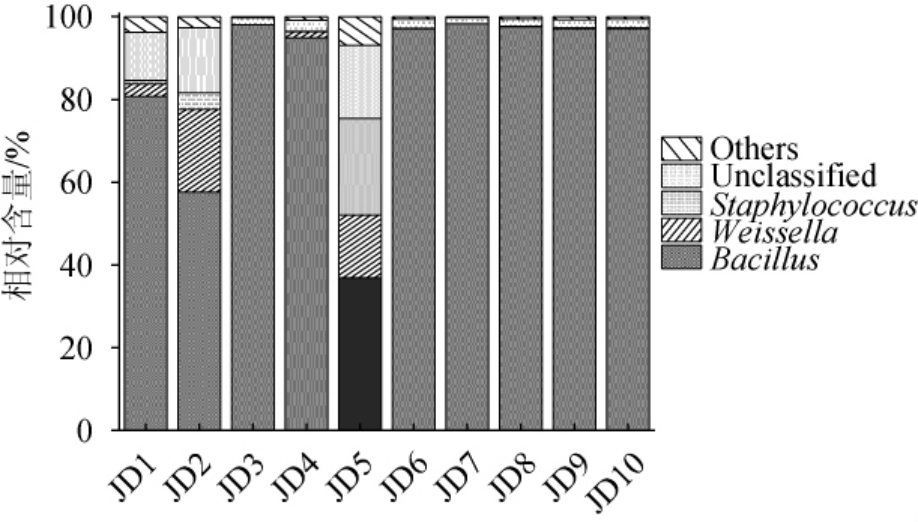
图2 基于属水平利川酱豆样品中细菌菌群结构
Fig. 2 Bacterial community structure of Lichuan Jiangdou samples based on genus level
由图2可知,芽孢杆菌属(Bacillus)、魏斯氏菌属(Weissella)和葡萄球菌属(Staphylococcus)为利川酱豆样品中的优势细菌属,其平均相对含量分别为85.52%、4.03%和2.83%,且这3个菌属均属于Firmicutes。部分样品的优势菌同其他样品存在一定的差异,如样品JD1、JD2和JD5未被鉴定的菌属和魏斯氏菌属相对含量较高,其中样品JD2和JD5的葡萄球菌属的相对含量亦比较高。利川酱豆样品的优势细菌属之间存在差异的原因可能与手工开放式制作外界环境及原辅料比例差异有一定的关系。
有研究发现,Bacillus有耐高温、易培养且在动物消化道内能够分泌多种促进消化吸收的酶,从而维持身体健康,促进生长等特点[29]。隶属于乳杆菌目(Lactobacillales)中的魏斯氏菌属(Weissella)作为乳酸菌能够调节肠道菌群平衡,增强免疫和协助消化等[30]。Staphylococcus为革兰氏阳性球菌,其多数为非致病菌,但是也有少数可以导致疾病的发生[31]。利川酱豆主要是在密封的陶罐中厌氧发酵而成,但是其前期制作环境比较开放,原料多数未经过杀菌,所以参与发酵过程的微生物种类较多,其中既有Bacillus和Weissella等有益菌的存在,同时也有少量Staphylococcus等致病菌或腐败菌。因此,在利川酱豆发酵时可以适当的改善发酵环境,从而减少杂菌污染。
2.2 基于OTU水平细菌群落结构分析
本研究在门和属水平对利川酱豆细菌种类及相对含量进行解析的基础上,进一步在OTU水平上对其细菌群落特征进行分析,结果见图3。
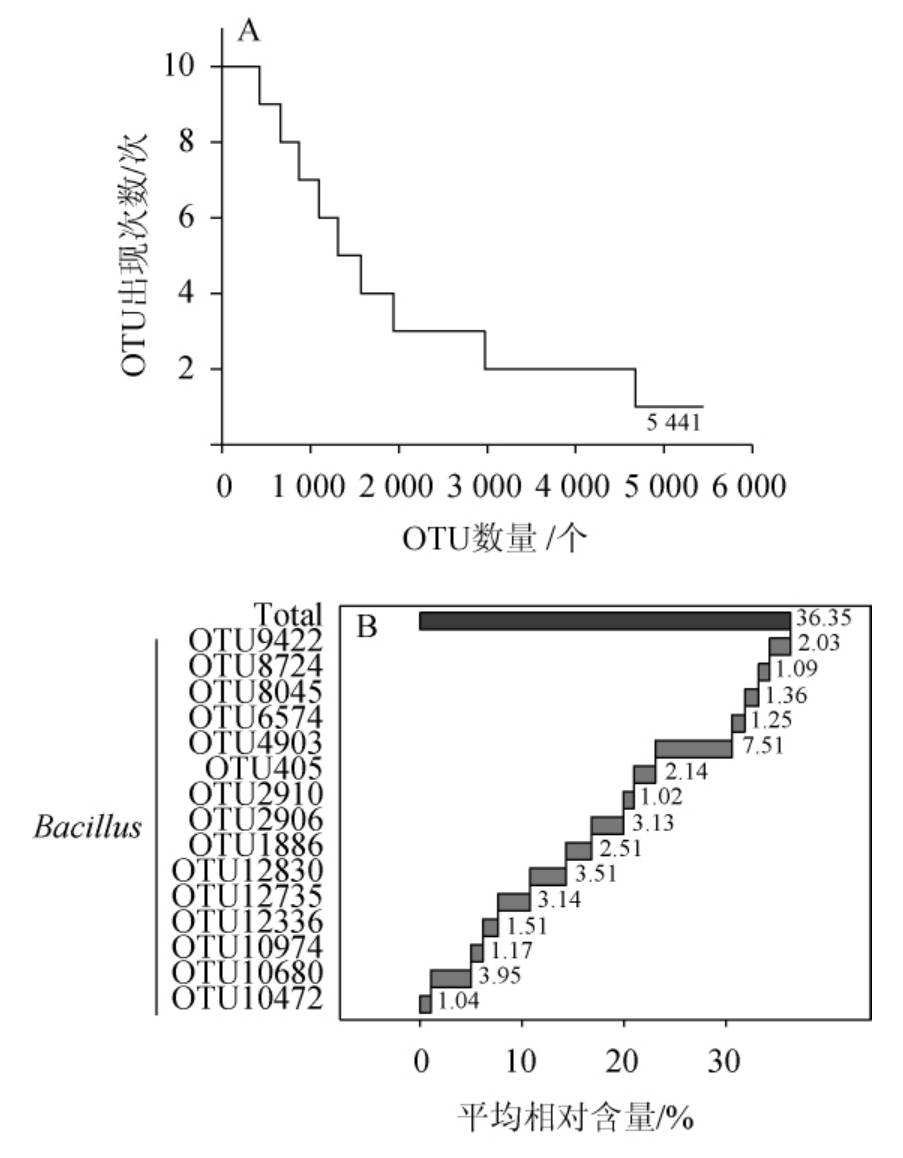
图3 利川酱豆样品中OTU出现次数统计图(A)和核心OTU相对含量瀑布图(B)
Fig. 3 Statistical chart of OTU occurrence times (A) and waterfall chart of core OTU relative content (B) in Lichuan Jiangdou samples
由图3A可知,本研究的利川酱豆样品共包含5 441个OTU,而在10个样品中均出现的OTU有422个,占OTU总数的7.77%,其包含了454 108 条序列,占总序列的84.70%。由图3B可知,平均相对含量>1.0%的OTU有15 个,占OTU总数的0.28%,其包含的序列数占总序列数的36.35%,且这15个OTU均隶属于芽孢杆菌属(Bacillus),由此可进一步说明Bacillus是利川酱豆样品中均存在且相对含量>1%的细菌属,即核心优势细菌。
2.3 基因功能预测
发酵过程中微生物种类及含量对酱豆品质与风味有一定的影响,但目前对利川酱豆细菌潜在功能相关研究较少。本研究进一步对利川酱豆中细菌序列进行了功能预测,结果见图4。
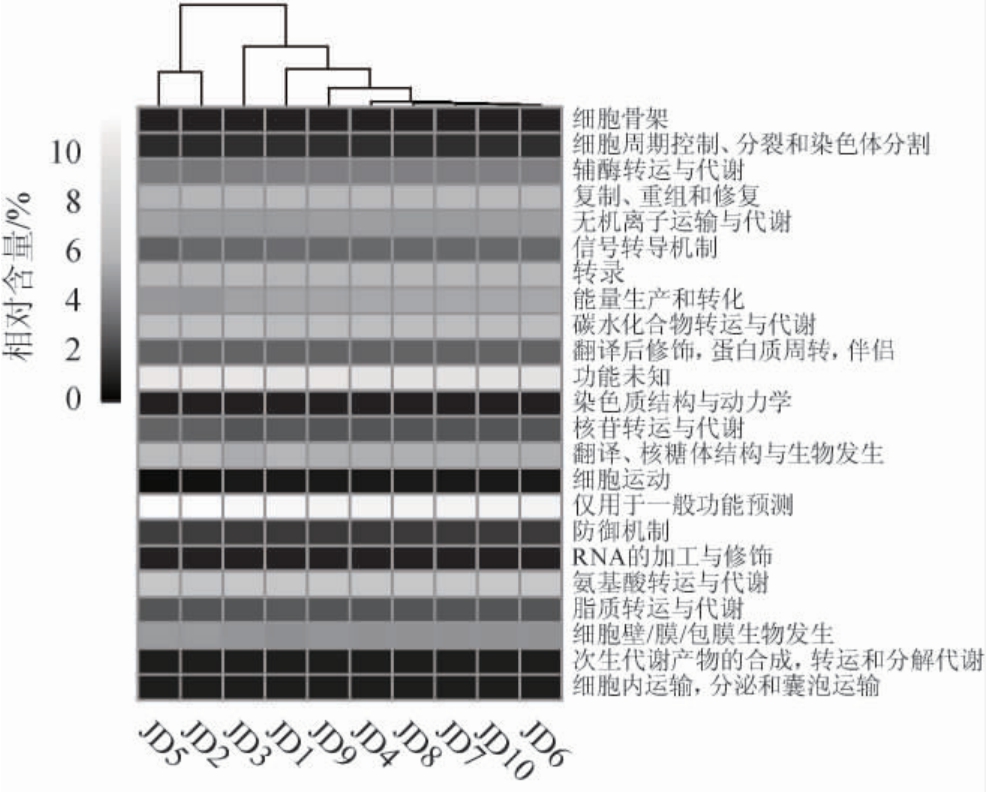
图4 基因功能预测热图
Fig. 4 Heat map of gene function prediction
由图4可知,本研究的10个利川酱豆样品分别注释到了23个功能大类。其中酱豆细菌类群在“氨基酸转运与代谢”、“碳水化合物转运与代谢”、“转录”和“复制、重组和修复”功能上相对含量较高。利川酱豆在自身代谢功能上表达较高可能与其原料中含有较为丰富的氨基酸态氮、总糖和蛋白质等基本营养成分有关[32]。由图4亦可知,酱豆细菌类群序列中具有“核糖核酸(ribonucleic acid,RNA)的加工与修饰”、“染色质结构与动力学”和“细胞骨架”等功能的序列占比则相对低。
2.4 基于电子鼻技术利川酱豆风味品质的评价
采用电子鼻技术对利川酱豆样品的风味品质进行评价,结果见表1。
表1 电子鼻各传感器对利川酱豆样品的响应值
Table 1 Response value of each sensor of electronic nose to Lichuan Jiangdou samples
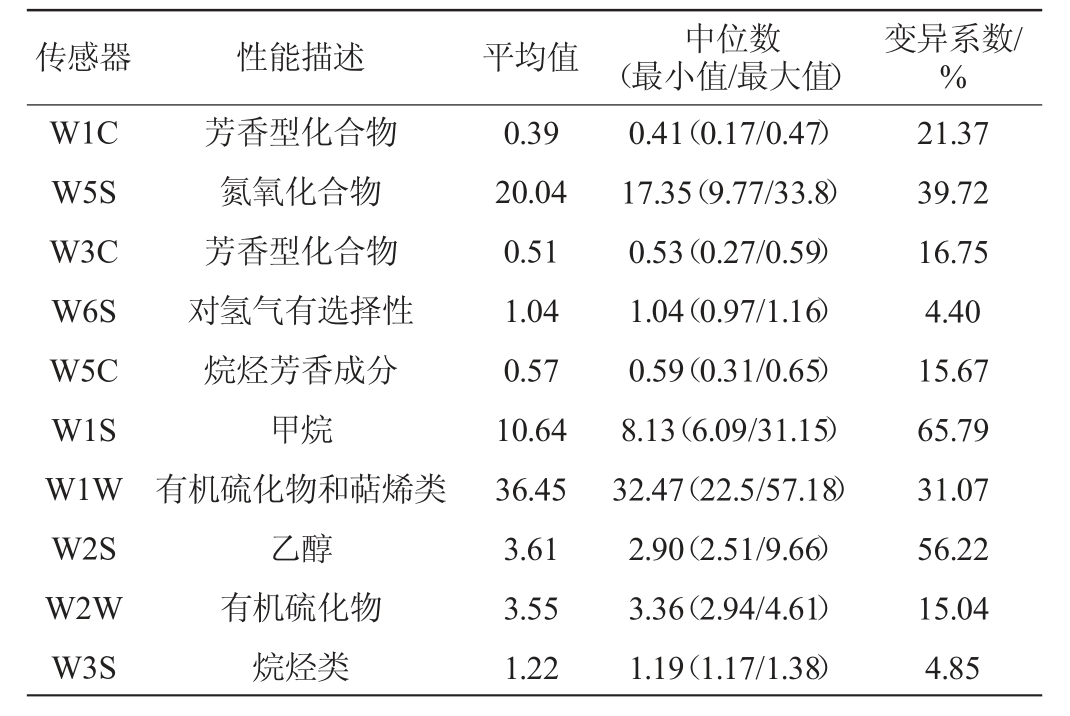
由表1可知,电子鼻传感器W6S和W3S的变异系数分别为4.40%和4.85%,均<10%;而其他8个传感器的变异系数均>10%。即10 个酱豆样品的风味在氢气和烷烃类的差异小,而在芳香成分、甲烷、氮氧化合物、乙醇、有机硫化物和萜烯类的差异大。由此可知,采用传统方法制作的利川酱豆品质不稳定,样品间的风味物质存在较大的差异,可能与样品中不同微生物的影响有关。
2.5 利川酱豆优势细菌属与风味物质的关联性分析
进一步对利川酱豆样品的优势细菌属和风味物质间的相关性进行分析,结果见图5。
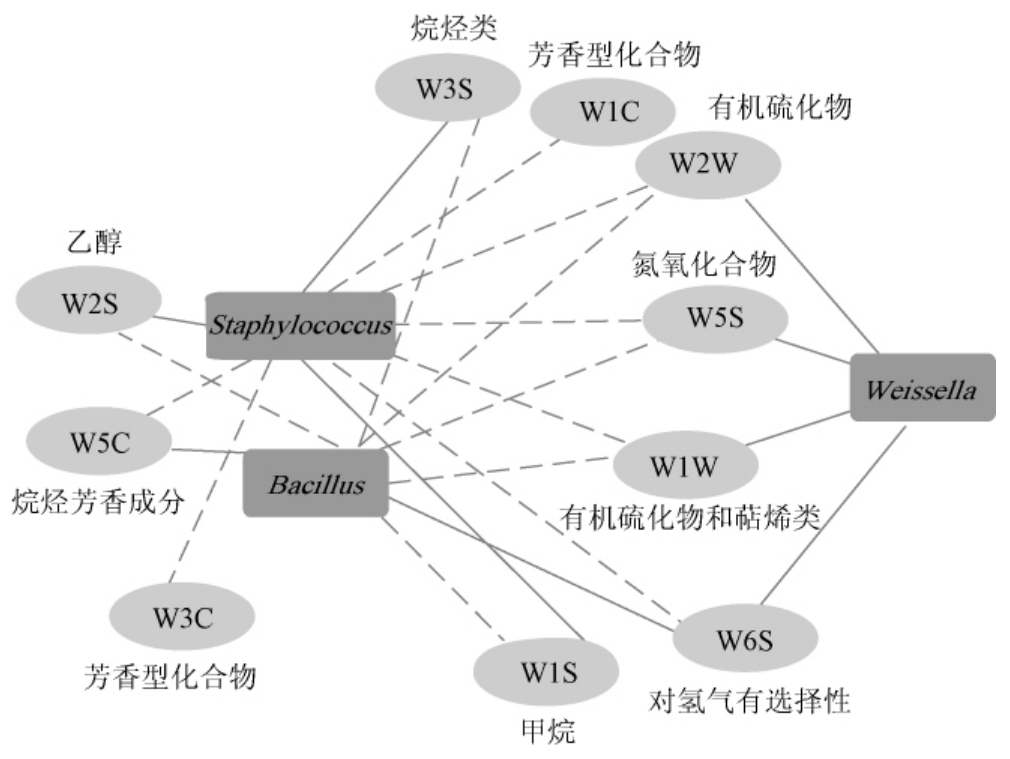
图5 利川酱豆样品中优势细菌属与风味物质相关性网络图
Fig. 5 Correlation network diagram of dominant bacteria genus and flavor substances in Lichuan Jiangdou samples
实线表示正相关,虚线表示负相关。
由图5可知,与有机硫化物等风味物质的生成呈现正相关的菌属为Weissella,Staphylococcus与乙醇、烷烃和甲烷等物质的形成呈现正相关,与芳香型化合物、氮氧化物和氢气等物质的形成呈现负相关;Bacillus与乙醇、烷烃、甲烷、氮氧化物、氢气等物质的形成呈现负相关,而与烷烃芳香成分的形成呈现正相关。由此可知,Bacillus对利川酱豆样品的风味形成可能起到了比较积极的作用。
3 结论
结果显示,利川酱豆样品中的优势细菌门为Firmicutes,优势细菌属为Bacillus、Weissella和Staphylococcus,功能预测结果显示样本中细菌序列潜在功能占比较高的为“氨基酸转运与代谢”、“碳水化合物转运与代谢”、“转录”与“复制、重组和修复”。酱豆样品的风味在氢气和烷烃类的差异小,而在芳香成分、甲烷、氮氧化合物、乙醇、有机硫化物和萜烯类的差异大,且优势细菌属中Bacillus与芳香类风味指标呈明显正相关,对利川酱豆样品的风味形成起到了较为积极的作用。
[1] 胡国军,吴天祥.豆豉营养评价及其粗纤溶酶活性检测[J].中国酿造,2010,29(10):95-99.
[2] XIA X D,LING W H,MA J,et al.An anthocyanin-rich extract from black rice enhances atherosclerotic plaque stabilization in apolipoprotein E-deficient mice[J].J Nutr,2006,136(8):2220-2225.
[3] ORČIĆ D Z,MIMICA-DUKIĆ N M,FRANCIŠKOVIĆ M M,et al.Antioxidant activity relationship of phenolic compounds in Hypericum perforatum L.[J].Chem Cent J,2011,5(1):34.
[4] JAYACHANDRAN M,XU B J.An insight into the health benefits of fermented soy products[J].Food Chem,2019,271(15):362-371.
[5] 陈怡,刘洋,蒋立文,等.基于高通量测序分析不同浏阳豆豉中真菌的多样性[J].中国酿造,2020,39(11):66-70.
[6] 陈怡,刘洋,蒋立文,等.浏阳豆豉发酵中高产酶活菌株的分离鉴定及酶活性分析[J].中国酿造,2020,39(8):37-41.
[7] 汤启成,曾凡玉,汪学荣,等.毛霉型豆豉与曲霉型豆豉挥发性成分的GC-MS分析[J].食品工业,2016,37(10):274-278.
[8] 聂黔丽,王修俊,刘林新,等.遵义细菌型自然发酵干豆豉菌群结构及风味品质分析[J/OL].食品科学:1-14[2022-04-22].http://kns.cnki.net/kcms/detail/11.2206.ts.20210927.2311.038.html
[9] ZHANG W B,LUO Q Q,ZHU Y,et al.Microbial diversity in two traditional bacterial Douchi from Gansu province in northwest China using Illumina sequencing[J].Plos One,2018,13(3):e0194876.
[10] 杨杨,曾剑华,王冰,等.中国传统发酵豆制品风味物质的研究进展[J].大豆科学,2018,37(6):969-974.
[11] 董蕴,许小玲,代凯文,等.基于MiSeq高通量测序技术对细菌型豆豉细菌类群的评价[J].食品工业科技,2018,39(13):150-154,247.
[12] LIANG H P,YIN L G,ZHANG Y H,et al.Dynamics and diversity of a microbial community during the fermentation of industrialized Qingcai paocai,a traditional Chinese fermented vegetable food,as assessed by Illumina MiSeq sequencing,DGGE and qPCR assay[J].Ann Microbiol,2018,68(2):111-122.
[13] 马才仁卓玛,白玛普赤,次仁潘多,等.高通量测序技术对西藏林芝传统发酵酸奶及酪乳细菌多样性研究[J].中国酿造,2020,39(11):116-119.
[14] 宋思家,黄子彧,林莹莹,等.基于Illumina MiSeq高通量测序技术分析不同保质期鸡蛋干的微生物群落多样性[J].食品与发酵工业,2020,46(24):24-30.
[15] CHEN Z J,XU G,DING C Y,et al.Illumina MiSeq sequencing and network analysis the distribution and co-occurrence of bacterioplankton in Danjiangkou Reservoir,China[J].Arch Microbiol,2020,202(4):859-873.
[16] GUO Y J,WANG R L,ZHAO Y,et al.Study on the relationship between microbial composition and living environment in important medical mites based on Illumina miseq sequencing technology[J].J Med Ento-mol,2020,57(4):1049-1056.
[17] 杨芳,杨莉,粟立丹.基于电子鼻和气相-离子迁移谱对美人椒酱的风味分析[J].食品工业科技,2019,40(23):193-198,206.
[18] 任敏,多拉娜,王帅,等.基于电子鼻和电子舌技术评价乳酸乳球菌对发酵乳风味品质的影响[J].中国食品学报,2021,21(1):246-255.
[19] 冯绘敏,侯一哲,黄天赐,等.电子传感技术在中药材及农产品分析领域的应用研究进展[J].分析测试技术与仪器,2020,26(4):239-248.
[20] 郭泽尚,王磊,常志勇.电子鼻在肠道疾病诊断中应用的研究进展[J].吉林大学学报(医学版),2020,46(6):1332-1337.
[21] 王玉荣,折米娜,等.基于MiSeq高通量测序技术内蒙古地区酸粥细菌多样性研究[J].食品工业科技,2018,39(19):124-129.
[22] CAPORASO J G,KUCZYNASKI J,STOMBAUGH J,et al.QIIME allows integration and analysis of high-throughput community sequencing data[J].Nat Methods,2010,7(5):335-336.
[23] CAPORASO J G,BITTINGER K,BUSHMAN F D,et al.PyNAST: a flexible tool for aligning sequences to a template alignment[J].Bioinformatics,2010,26(2):266-267.
[24] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[25] COLE J R,CHAI B,FARRIS R J,et al.The ribosomal database project(RDP-II): introducing myRDP space and quality controlled public data[J].Nucleic Acids Res,2007,35(1):169-172.
[26] DESANTIS T Z,HUGENHOLTZ P,LARSEN N,et al.Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Appl Environ Microbiol,2006,72(7):5069-5072.
[27] WILKINSON T J,HUWS S A,EDWARDS J E,et al.CowPI:A rumen microbiome focussed version of the PICRUSt functional inference software[J].Front Microbiol,2018,9(5):1095.
[28] GALPERIN M Y,KRISTENSEN D M,MAKAROVA K S,et al.Microbial genome analysis:the COG approach[J].Brief Bioinform,2019,20(4):1063-1070.
[29] 谢全喜,侯楠楠,鹿晓慧,等.芽孢杆菌的产酶特性及其对抗生素的耐受性[J].中国酿造,2021,40(5):91-96.
[30] 温永平,沈玲玲,孙志宏,等.海南地区健康青年肠道中乳酸菌和双歧杆菌多样性研究[J].中国食品学报,2019,19(11):14-24.
[31] TONG S Y C,DAVIS J S,EICHENBERGER Emily,et al. Staphylococcus aureus Infections:epidemiology,pathophysiology,clinical manifestations,and management[J]. Clin Microbiol Rev,2015,28(3): 603-661.
[32] 宋朝阳,罗俏俏,杨祥,等.两种传统发酵豆糁的营养成分分析[J].食品与发酵科技,2020,56(3):8-13,30.