食醋是富有营养的酸性调味品,能增进食欲,且具有食物防腐、除腥及预防疾病等功能[1]。在我国,传统食醋酿造是由众多微生物参与并驱动谷物原料发酵,这些微生物因酿造原料[2]、气候特点[3]、制作工艺等不同而具有多样性和复杂性[4-6]。WANG Z M等[7]利用宏基因组方法分析镇江香醋醋酸发酵阶段微生物时共鉴定出151个细菌属和202个真菌属,其中乳酸杆菌属(Lactobacillus)和醋酸杆菌属(Acetobacter)是优势细菌属,链格孢属(Alternaria)和曲霉属(Aspergillus)是优势真菌属。YUN J M等[8]利用高通量测序技术研究发现凉州熏醋酿造过程中出现25个细菌属和18个真菌属,并且优势真菌属为链格孢属(Alternaria)。微生物群落不仅受到外界环境因素的影响[9],群落间的相互作用也塑造了微生物群落结构[10],共现模式可为研究复杂微生物环境的群落结构和功能提供新的视角。共现网络可反映复杂环境下占互作主导地位的优势物种、互作紧密的物种群,这些优势物种以及物种群往往对维持复杂环境的微生物群落结构和功能稳定发挥着独特以及重要的作用[11-14]。目前,在人类肠道[15-16]、土壤[17-19]、植物根际[20]和食品发酵[21-22]等复杂系统中的微生物共现模式研究已得到了学者的广泛关注。
本研究以山西陈醋酿造过程中不同发酵阶段醋醅为研究对象,采用高通量测序技术研究食醋微生物群落的结构和多样性,并应用互作网络数据可视化的软件Cytoscape 3.7.2构建了食醋微生物群落的互作网络,揭示山西陈醋酿造过程中的微生物群落相互作用关系,可为深入认识食醋发酵微生物和食醋发酵过程控制提供重要理论参考。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
醋醅样品:山西清徐县某醋厂食醋淀粉糖化(starch saccharification,Sac)发酵第2天样品(编号Sac2)、酒精发酵(alcohol fermentation,AF)第12天样品(编号AF12)、醋酸发酵(acetic acid fermentation,AAF)第1天、第3天、第7天和第10天样品(编号分别为AAF1、AAF3、AAF7、AAF10)。糖化和酒精发酵阶段样品取自发酵池的顶部、中部和底部,混合后得到测试样;醋酸发酵阶段样品在距离发酵池表面30 cm的深度进行东西南北中五点采集,混合均匀后作为醋酸发酵阶段的测试样。样品用无菌密封袋封装,-20 ℃保存备用。
1.1.2 试剂
酚、氯仿、异戊醇、异丙醇、无水乙醇(均为分析纯):国药集团化学试剂有限公司;脱氧核糖核酸(deoxyribonucleic acid,DNA)Marker、聚合酶链式反应(polymerase chain reaction,PCR)引物、50×TAE缓冲液(Tris acetate 乙二胺四乙酸(ethylene diamine tetraacetic acid,EDTA),TAE)、十六烷基三甲基溴化铵(hexadecyl trimethyl ammonium bromide,CTAB)裂解液、溶菌酶:上海生工生物工程股份有限公司;DNA聚合酶、缓冲液:美国NEB公司;GeneJET胶回收试剂盒:美国Thermo Fisher Scientific公司;TruSeq DNA PCR-Free Library Preparation Kit建库试剂盒:美国Illumina公司。
1.2 仪器与设备
DZKW-D-2电热恒温水浴锅:北京市水光明医疗仪器有限公司;Neofuge 15R高速冷冻离心机:上海力申科学仪器有限公司;DYY-6C电泳仪:北京市六一仪器厂;NanoVue plus超微量紫外分光光度计:英国Cytiva公司;T100梯度PCR仪:美国Bio-Rad公司;Novaseq6000高通量测序平台:美国Illumina公司。
1.3 方法
1.3.1 基因组提取与检测
采用CTAB法[23]对醋醅样本的基因组DNA进行提取,操作如下:①吸取1 000 μL CTAB 裂解液至2.0 mL EP管里,加入20 μL溶菌酶,将适量的样品加入裂解液中,65 ℃水浴(时间为2~3 h),期间颠倒混匀数次,以使样品充分裂解。②离心取950 μL上清,加入与上清等体积的酚(pH 8.0)∶氯仿∶异戊醇(25∶24∶1,V/V),颠倒混匀,12 000 r/min离心10 min。③取上清,加入等体积的氯仿∶异戊醇(24∶1,V/V),颠倒混匀,12 000 r/min离心10 min。④吸取上清至1.5 mL离心管里,加入上清液3/4体积的异丙醇,上下摇晃,-20 ℃沉淀。⑤12 000 r/min离心10 min,倒出液体。用1 mL体积分数为75%乙醇洗涤2次,剩余的少量液体可再次离心收集,然后用枪头吸出。⑥超净工作台吹干或者室温晾干。⑦加入51 μL双蒸水(ddH2O)溶解DNA样品。⑧加RNase A 1μL消化核糖核酸(ribonucleic acid,RNA),37 ℃放置15 min。之后利用琼脂糖凝胶电泳检测DNA完整性并用超微量分光光度计测定DNA的浓度与纯度。
1.3.2 PCR扩增与高通量测序
用引物515F(5'-GTGCCAGCMGCCGCGGTAA-3')、806R(5'-GGACTACHVGGGTWTCTAAT-3')和引物ITS3-2024F(5'-GCATCGATGAAGAACGCAGC-3')、ITS4-2409R(5'-TCCTCCGCTTATTGATATGC-3')分别扩增细菌16S rRNA基因V4区和真菌ITS2区。PCR扩增体系:Phusion Master Mix(2×)15 μL,引物各1.5 μL,基因组DNA(genomic DNA,gDNA)10 μL,ddH2O 2 μL。PCR扩增条件:98 ℃预变性1 min;98 ℃变性10 s,50 ℃退火30 s,72 ℃延伸30 s,共30个循环;72 ℃再延伸5 min。PCR产物使用2%琼脂糖凝胶电泳检测,并进行回收和纯化。高通量测序委托北京诺禾致源科技股份有限公司。
1.3.3 高通量测序数据分析
测序原始序列通过序列拼接、过滤、质量控制、去除嵌合体,得到最终有效序列[24-25]。利用Uparse算法(Uparse v7.0.1001)以97%的一致性将所有样本的有效序列聚类成为操作分类单元(operational taxonomic units,OTUs)[26]。用Mothur软件与SILVA(v138.1)数据库对细菌OTUs序列进行物种注释分析,用基本局部比对搜索工具(basic local alignment search tool,BLAST)与Unite(v8.2)数据库对真菌OTUs序列进行物种注释分析,并分别在门、纲、目、科、属水平上统计各样本的群落组成。最后对各样本数据进行均一化处理用于后续Alpha多样性分析。
使用Qiime软件(Version 1.9.1)计算Observed-OTUs、Shannon、Simpson、Chao1、ACE及Coverage指数。利用Origin 8.5软件绘制稀释曲线和门水平、属水平物种相对丰度柱状图。通过对所有样本进行斯皮尔曼相关系数(Spearman's correlation coefficient,SCC)计算,得到物种相关系数矩阵后,设置过滤条件如下:①显著性阈值设置为0.05;②去除相关系数绝对值<0.6的连接;③过滤掉节点自连接;④最大物种数目设置为50。利用Cytoscape 3.7.2软件绘图,以门水平为着色单位可视化山西陈醋酿造过程中细菌和真菌的共现网络,其中显示标签的节点为平均相对丰度>1%的菌属。
2 结果与分析
2.1 醋醅样品测序数据分析
基于高通量测序技术对醋醅样品中细菌和真菌的有效序列和OTUs进行研究,结果见表1。
表1 醋醅样品中细菌和真菌的序列信息
Table 1 Sequence information of bacteria and fungi in Cupei samples
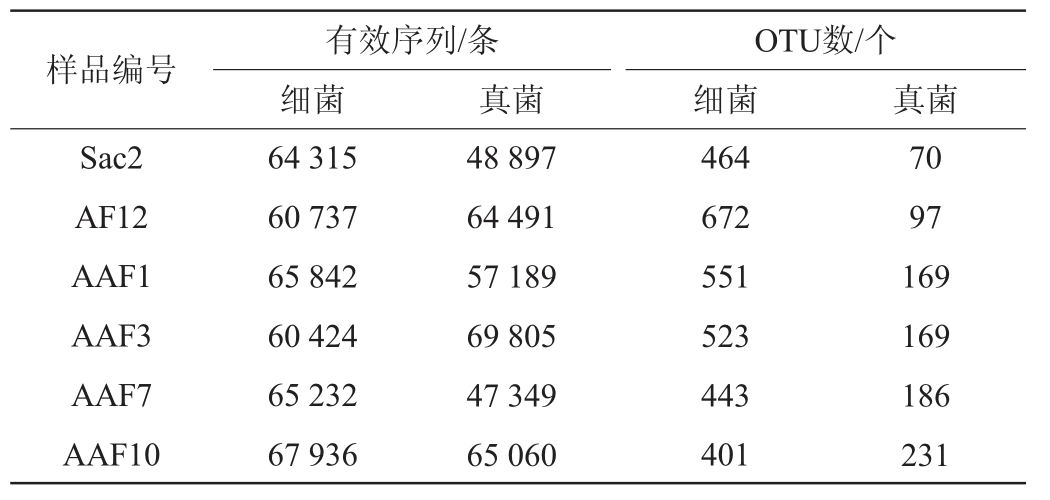
6个醋醅样品共产生384 486条细菌有效序列,352 791条真菌有效序列。将有效序列以97%的一致性聚类成为OTUs,共得到1 197个细菌OTUs和432个真菌OTUs,并且在各个发酵阶段细菌OTUs均大于真菌OTUs,说明醋醅中细菌群落多样性远高于真菌群落。用Mothur软件与SSUrRNA数据库进行物种注释分析,将所有的细菌序列鉴定为36个门、67个纲、153个目、238个科、429个属。用BLAST与Unite(v8.2)数据库进行物种注释分析,将所有的真菌序列鉴定为8个门、27个纲、54个目、108个科、168个属。
2.2 醋醅样品Alpha多样性分析
稀释曲线直接反映测序数据量的合理性,山西陈醋酿造过程中醋醅样品的微生物测序稀释曲线见图1。山西陈醋酿造过程中醋醅样品中微生物的Alpha多样性指数结果见表2。

图1 醋醅样品中细菌(a)和真菌(b)的稀释曲线
Fig.1 Dilution curves of bacteria (a) and fungi (b) in Cupei samples
表2 醋醅样品中细菌和真菌群落Alpha多样性指数
Table 2 Alpha diversity indexes of bacterial and fungal community in Cupei samples
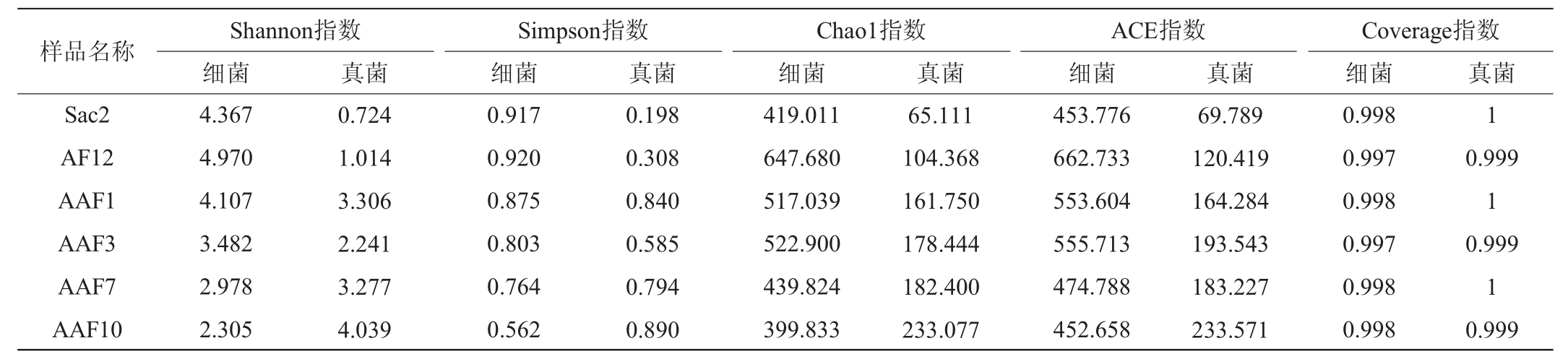
由图1可知,所有醋醅样品的稀释曲线渐近平坦并且Coverage指数均>0.99,表明测序可以反映醋醅样本中绝大多数微生物群落特征和多样性信息。
多样性指数包括Shannon指数和Simpson指数,用于表征微生物群落内物种分布的多样性和均匀度。微生物群落多样性越高,物种分布越均匀,Shannon指数和Simpson指数越大。丰富度指数包括Chao1指数和ACE指数,用于估计微生物群落中包含的物种总数。微生物群落丰富度越高,Chao1指数和ACE指数越大。由表2可知,细菌群落的多样性和丰富度变化趋势一致,在酒精发酵第12天(AF12)有最高的丰富度和多样性,随着醋酸发酵的进行,多样性和丰富度逐渐下降。真菌群落的多样性和丰富度整体呈现逐渐增加的趋势。除醋酸发酵第7天和第10天外(AAF7、AAF10),细菌群落的Shannon指数和Simpson指数高于真菌群落,而在整个发酵过程中细菌群落的Chao1指数和ACE指数均高于真菌群落,表明食醋酿造过程中细菌群落比真菌群落更具复杂性。
2.3 醋醅微生物群落结构分析
基于门水平醋醅中的细菌和真菌群落结构分析,结果见图2。

图2 基于门水平醋醅样品中细菌(a)和真菌(b)群落结构分析
Fig.2 Community structure analysis of bacterial (a) and fungal (b) in Cupei samples based on phylum level
其中Other表示平均相对丰度<1%的菌门。
由图2a可知,山西陈醋酿造过程中硬壁菌门(Firmicutes)、变形菌门(Proteobacteria)和蓝藻菌门(Cyanobacte ria)是主要的细菌门(平均相对丰度>1%),平均相对丰度分别为60.68%、33.78%和3.18%。由图2b可知,子囊菌门(Ascomycota)和担子菌门(Basidiomycota)是主要的真菌门,平均相对丰度分别为97.46%和1.58%。
基于属水平醋醅中的细菌和真菌群落结构分析,结果见图3。由图3可知,山西陈醋酿造过程中有6个细菌属和8个真菌属的平均相对丰度>1%,其中8个真菌属均来自子囊菌门(Ascomycota)。与自然发酵的谷物醋类似[5,27-29],乳酸杆菌属(Lactobacillus)和醋酸杆菌属(Acetobacter)是优势细菌属,其平均相对丰度分别为36.06%和23.98%。其中乳酸杆菌属(Lactobacillus)随着发酵的进行相对丰度逐渐降低,在醋酸发酵第10天(AAF10)降低至5.33%。相反,醋酸杆菌属(Acetobacter)的相对丰度随着发酵的进行不断增加,在醋酸发酵第10天(AAF10)增加至64.56%。此外,魏斯氏菌属(Weissella)、假单胞菌属(Pseudomonas)、unidentified_Mitochondria和unidentified_Chloroplast也是食醋酿造过程的主要菌属。unidentified_Mitochondria和unidentified_Chloroplast仅在醋酸发酵第1天(AAF1)具有较高的相对丰度,分别为23.62%和17.41%,表明其是在酒精发酵结束后,酒醅与辅料、老醅混合后引入的,并且不适应醋酸发酵环境导致相对丰度很快降低。
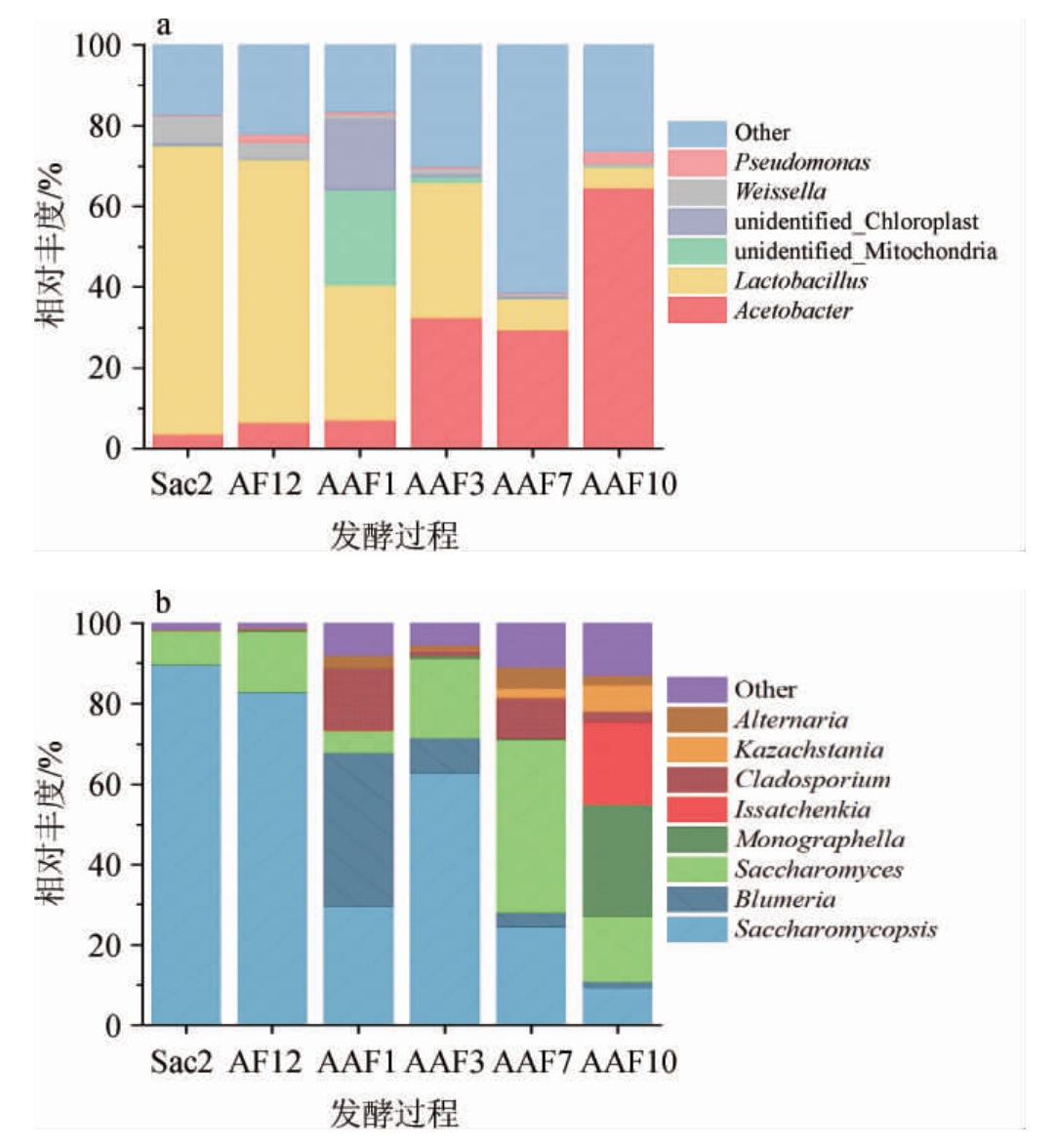
图3 基于属水平醋醅样品中细菌(a)和真菌(b)群落结构分析
Fig.3 Community structure analysis of bacterial (a) and fungal (b) in Cupei samples based on genus level
其中Other表示平均相对丰度<1%的菌属。
复膜孢酵母属(Saccharomycopsis)是醋醅中的优势真菌属,其平均相对丰度为49.75%。已有研究发现,镇江香醋和山西老陈醋酿造过程中的优势真菌属是酵母菌属(Saccharomyces)[27,29-30],凉州熏醋酿造过程中的优势真菌属是链格孢属(Alternaria)[8],这些差异可能是由地理气候、酿造原料和工艺等因素造成。此外,布氏白粉菌属(Blumeria)、枝孢属(Cladosporium)、明梭孢属(Monographella)、伊萨酵母属(Issatchenkia)、链格孢属(Alternaria)和哈萨克斯坦酵母属(Kazachstania)也是食醋酿造过程中的主要真菌属,平均相对丰度分别为8.74%、4.98%、4.84%、3.48%、1.98%和1.55%。
2.4 醋醅中微生物群落互作网络分析
醋醅样品中细菌、真菌群落的相关性网络分析分别见图4和图5。
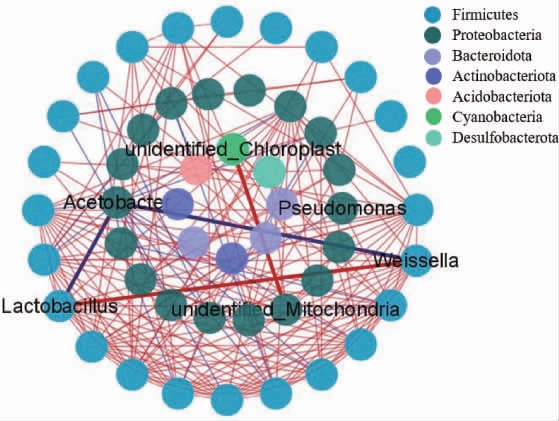
图4 醋醅样品中细菌群落的相关性网络分析
Fig.4 Correlation network analysis of bacterial community in Cupei samples
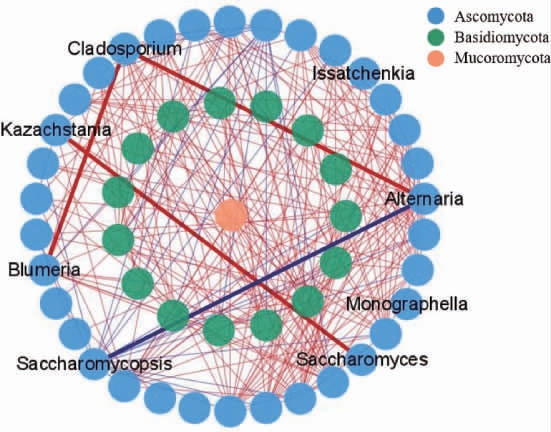
图5 醋醅样品中真菌群落的相关性网络分析
Fig.5 Correlation network analysis of fungal community in Cupei samples
由图4可知,细菌互作网络包括50个节点,182条边,网络密度为0.074,聚集系数为0.304,平均相邻节点数为7.280,正相关关系占87.36%。细菌互作网络中,48%的节点来自硬壁菌门(Firmicutes),36%的节点来自变形菌门(Proteobacteria),43.48%的拮抗作用来自醋酸杆菌属(Acetobacter)。隶属于变形菌门(Proteobacteria)的醋酸杆菌属(Acetobacter)与硬壁菌门(Firmicutes)的乳酸杆菌属(Lactobacillus)、魏斯氏菌属(Weissella)具有显著的拮抗性,这可能是由于醋酸杆菌属(Acetobacter)代谢的乙酸逐渐积累抑制其他菌属的生长。乳酸杆菌属(Lactobacillus)和魏斯氏菌属(Weissella)存在协同作用,unidentified_Mitochondria和unidentified_Chloroplast呈正相关。微生物之间的协同作用表明这些微生物在食醋酿造环境中有共同的营养和环境偏好,或通过代谢物交换形成相互依赖的模式。
由图5可知,真菌互作网络包括50个节点,246条边,网络密度为0.1,聚集系数为0.304,平均相邻节点数9.84,以正相关关系占主导,占比达88.62%。根据以上结果推论,在食醋酿造过程中,微生物群落的互作以协同作用为主;真菌互作网络具有更复杂的连接。真菌互作网络中68%的节点来自子囊菌门(Ascomycota),30%的节点来自担子菌门(Basidiomycota),57.14%的拮抗作用来自复膜孢酵母属(Saccharomycopsis)。复膜孢酵母属(Saccharomycopsis)与链格孢属(Alternaria)存在显著负相关关系,枝孢属(Cladosporium)与布氏白粉菌属(Blumeria)、链格孢属(Alternaria)存在显著正相关关系,酵母菌属(Saccharomyces)和哈萨克斯坦酵母属(Kazachstania)存在协同作用。
3 结论
本研究基于高通量测序技术研究了山西陈醋酿造过程中醋醅微生物群落的结构以及互作网络。结果表明,醋醅中细菌群落多样性远高于真菌群落。醋醅中优势细菌属为乳酸杆菌属(Lactobacillus)和醋酸杆菌属(Acetobacter),并且乳酸杆菌属(Lactobacillus)随着发酵的进行丰度逐渐降低,而醋酸杆菌属(Acetobacter)则相反。细菌互作网络分析表明,醋酸杆菌属(Acetobacter)贡献了43.48%的拮抗作用,其中醋酸杆菌属(Acetobacter)与乳酸杆菌属(Lactobacillus)、魏斯氏菌属(Weissella)具有显著的拮抗性;乳酸杆菌属(Lactobacillus)和魏斯氏菌属(Weissella)存在协同作用。醋醅中的优势真菌属为复膜孢酵母属(Saccharomycopsis)。真菌互作网络分析表明,57.14%的拮抗作用来自复膜孢酵母属(Saccharomycopsis),并且复膜孢酵母属(Saccharomycopsis)与链格孢属(Alternaria)存在显著负相关关系。本研究从共现网络视角探究了山西陈醋酿造过程中的复杂微生物群落结构演替特征,并提出了可能的互作模式,既丰富了酿造微生物信息数据,也可为食醋酿造过程控制和开发复配醋曲提供重要的参考数据。
[1]李江.浅谈食醋检验标准中存在的疏漏[J].监督与选择,2006(4):57.
[2]TR CˇEK J,MAHNI CˇA,RUPNIK M.Diversity of the microbiota involved in wine and organic apple cider submerged vinegar production as revealed by DHPLC analysis and next-generation sequencing[J].Int J Food Microbiol,2016,223:57-62.
[3]VILACRESPO J,NOGALES J M R,FERNÁNDEZ-FERNÁNDEZ E,et al.Strategies for the enhancement of malolactic fermentation in the new climate conditions[M]//Current research,technology and education topics in applied microbiology and microbial biotechnology.Seville:University of Seville publisher,2010:920-929.
[4]GAN X,TANG H L,YE D D,et al.Diversity and dynamics stability of bacterial community in traditional solid-state fermentation of Qishan vinegar[J].Ann Microbiol,2017,67(10):703-713.
[5]ZHU Y P,ZHANG F F,ZHANG C N,et al.Dynamic microbial succession of Shanxi aged vinegar and its correlation with flavor metabolites during different stages of acetic acid fermentation[J].Sci Rep,2018,8(1):8612.
[6]CHAI L J,QIU T,LU Z M,et al.Modulating microbiota metabolism via bioaugmentation with Lactobacillus casei and Acetobacter pasteurianus to enhance acetoin accumulation during cereal vinegar fermentation[J].Food Res Int,2020,138:109737.
[7]WANG Z M,LU Z M,SHI J S,et al.Exploring flavour-producing core microbiota in multispecies solid-state fermentation of traditional Chinese vinegar[J].Sci Rep,2016,6:26818.
[8]YUN J M,ZHAO F Q,ZHANG W W,et al.Monitoring the microbial community succession and diversity of Liangzhou fumigated vinegar during solid-state fermentation with next-generation sequencing[J].Ann Microbiol,2019,69(3):279-289.
[9]ZHANG Q,ZHAO C M,WANG X B,et al.Bioaugmentation by Pediococcus acidilactici AAF1-5 improves the bacterial activity and diversity of cereal vinegar under solid-state fermentation[J].Front Microbiol,2021,11:603721.
[10]BARBERÁN A,BATES S T,CASAMAYOR E O,et al.Using network analysis to explore co-occurrence patterns in soil microbial communities[J].ISME J,2012,6(2):343-351.
[11]JIAO S,LIU Z S,LIN Y B,et al.Bacterial communities in oil contaminated soils:Biogeography and co-occurrence patterns[J].Soil Biol Biochem,2016,98:64-73.
[12]QIN J J,LI Y R,CAI Z M,et al.A metagenome-wide association study of gut microbiota in type 2 diabetes[J].Nature,2012,490:55-60.
[13]关琼,马占山.人类母乳微生物菌群的生态学分析[J].科学通报,2014,59(22):2205-2212.
[14]SHEN C C,WANG J,JING Z W,et al.Plant diversity enhances soil fungal network stability indirectly through the increase of soil carbon and fungal keystone taxa richness[J].Sci Total Environ,2022,818:151737.
[15]RAKOFF-NAHOUM S,FOSTER K R,COMSTOCK L E.The evolution of cooperation within the gut microbiota[J].Nature,2016,533:255-259.
[16]魏晓.乙肝肝硬化的肠道微环境及菌群代谢网络分析[D].北京:军事科学院,2018.
[17]MA B,WANG H Z,DSOUZA M,et al.Geographic patterns of co-occurrence network topological features for soil microbiota at continental scale in eastern China[J].ISME J,2016,10(8):1891-1901.
[18]周恒.不同海拔高寒草原土壤微生物特征及其适应机制[D].兰州:甘肃农业大学,2019.
[19]马骢毓.民勤退耕区次生草地土壤微生物多样性研究及优势植物根际促生菌资源筛选[D].兰州:甘肃农业大学,2017.
[20]DURAN P,THIERGART T,GARRIDO-OTER R,et al.Microbial in-terkingdom interactions in roots promote arabidopsis survival[J].Cell,2018,175(4):973-983.
[21]FANG G Y,CHAI L J,ZHONG X Z,et al.Deciphering the succession patterns of bacterial community and their correlations with environmental factors and flavor compounds during the fermentation of Zhejiang rosy vinegar[J].Int J Food Microbiol,2021,341:109070.
[22]戴奕杰.酱香型白酒酿造过程中微生物群落组成及其与酒品质的关系[D].长沙:湖南农业大学,2019.
[23]ARSENEAU J R,STEEVES R,LAFLAMME M.Modified low-salt CTAB extraction of high-quality DNA from contaminant-rich tissues[J].Mol Ecol Resour,2016,17(4):686-693.
[24]MARTIN M.Cutadapt removes adapter sequences from high-throughput sequencing reads[J].EMBnet J,2011,17(1):10-12.
[25]SCHLOSS P D,GEVERS D,WESTCOTT S L.Reducing the effects of PCR amplification and sequencing artifacts on 16S rRNA-based studies[J].Plos One,2011,6(12):e27310.
[26]EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nat Methods,2013,10(10):996-998.
[27]NIE Z Q,ZHENG Y,XIE S K,et al.Unraveling the correlation between microbiota succession and metabolite changes in traditional Shanxi aged vinegar[J].Sci Rep,2017,7:9240.
[28]LI S,LI P,LIU X,et al.Bacterial dynamics and metabolite changes in solid-state acetic acid fermentation of Shanxi aged vinegar[J].Appl Microbiol Biot,2016,100(10):4395-4411.
[29]NIE Z Q,ZHENG Y,DU H F,et al.Dynamics and diversity of microbial community succession in traditional fermentation of Shanxi aged vinegar[J].Food Microbiol,2015,47:62-68.
[30]XU W,HUANG Z Y,ZHANG X J,et al.Monitoring the microbial community during solid-state acetic acid fermentation of Zhenjiang aromatic vinegar[J].Food Microbiol,2011,28(6):1175-1181.