蔗糖是一种可再生的低碳资源,可以从植物中获得,如热带地区的甘蔗和温带地区的甜菜等[1]。由于蔗糖是一种低廉的碳源,且大部分的微生物都可以利用蔗糖作为碳源进行生长,因此,目前常利用微生物发酵蔗糖合成一些大众化工品以及高附加值的化合物[2-3],如乙醇、乙酸、丙酮酸、氨基酸以及一些萜烯类化合物等[4-6]。
目前,可利用蔗糖的微生物主要有大肠杆菌(Escherichia coli)、酵母菌(Saccharomyces)和枯草芽孢杆菌(Bacillus subtilis)等,大肠杆菌主要发酵蔗糖产酸、产气[7];酵母菌在工业上主要利用蔗糖生产乙醇[8],枯草芽孢杆菌可发酵蔗糖产氨基酸等[9],其中,能够利用蔗糖快速生长的最主要的微生物是酵母菌[8]。酵母菌可通过蔗糖水解酶的作用,使蔗糖分解成葡萄糖和果糖,从而被利用,有报道证实酵母菌利用蔗糖的速度较其他微生物更快[8,10]。在酵母菌中,酿酒酵母(Sacchromyces cerevisiae)具有遗传操作简单、操作技术与方法更成熟、基因组小、研究方便等优点[11],其常被作为模式菌株来进行蔗糖消耗途径的研究[12]。酿酒酵母以两种形式来代谢利用蔗糖:一种是通过由Suc基因家族编码的细胞外蔗糖转化酶,其属于β-半乳糖苷酶类蔗糖水解酶,被分泌到细胞外并将蔗糖水解成葡萄糖和果糖[8,13-14];另一种形式是蔗糖可以通过质子同向转运蛋白的作用进入细胞,并在胞质溶胶中被酿酒酵母代谢麦芽糖的酶麦芽糖酶及胞内的蔗糖转化酶水解[8,15]。其中蔗糖转化酶基因Suc2[16-18]、麦芽糖渗透酶基因MALx1、麦芽糖酶基因MALx2以及一些调节基因MALx3是酿酒酵母中蔗糖消耗途径中的关键基因[8]。有研究表明,除蔗糖转化酶基因Suc2外,酿酒酵母中的MAL12、MAL22以及MAL32基因主要参与蔗糖的水解,而MAL11、MAL21及MAL31等基因负责蔗糖的转运,这些MAL基因在不同酿酒酵母菌株中的位置以及拷贝数不同[8,19]。有研究表明,同时敲除蔗糖的一些转运蛋白基因及蔗糖水解酶类基因之后,酵母无法在以蔗糖为唯一碳源的培养基上进行生长,而单纯敲除Suc2基因,酿酒酵母可以继续代谢蔗糖[8,16]。因此,在不敲除转运蛋白基因的情况下,阻断酿酒酵母中一些重要的蔗糖水解酶基因,其对酿酒酵母代谢蔗糖方面的影响还需进一步研究。同时,由于酿酒酵母中的重要水解酶基因MAL12、MAL22以及MAL32具有较高的同源性,其位于不同的染色体上,如何利用Crispr-Cas9的技术对其进行精确的敲除具有一定的研究价值及理论意义[20-21]。
因此,本研究以酿酒酵母IMX581为出发菌株,利用Crispr-Cas9基因编辑[22-23]的方法敲除其蔗糖消耗途径中的蔗糖转化酶基因Suc2和麦芽糖酶基因MAL32、MAL12、MAL22,并对其生长特性及蔗糖消耗情况进行分析,为后续在酿酒酵母中研究蔗糖代谢及调控提供一定理论参考。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株及引物
本研究所用菌株见表1,引物均由江苏金唯智生物科技有限公司合成,见表2。
表1 本研究所用的菌株
Table 1 Strains used in the study

表2 本研究所用的引物
Table 2 Primers used in the study

1.1.2 试剂
蛋白胨、酵母粉、葡萄糖、氯化钠(均为分析纯或生化试剂):上海生工生物工程有限公司;琼脂糖(分析纯):北京全式金生物科技有限公司;无氨基酵母氮源(yeast nitrogen base without amino acids,YNB)、尿嘧啶省却培养物:西格玛奥德里奇(上海)贸易有限公司;尿嘧啶、乙酸锂、聚乙二醇3350(均为分析纯):北京索莱宝生物科技有限公司。
1.1.3 培养基
酵母浸出粉胨葡萄糖(yeast extract peptone dextrose,YPD)培养基[24]:酵母粉10 g/L、蛋白胨20 g/L、葡萄糖20 g/L,pH 7.0。121 ℃高压蒸汽灭菌20 min。固体培养基中加入20 g/L琼脂粉。
SC-Ura营养缺陷培养基[24]:YNB 6.7 g/L、尿嘧啶省却培养物1.4 g/L、葡萄糖20 g/L、亮氨酸(10 g/L)10 mL/L、组氨酸(10 g/L)2 mL/L、色氨酸(10 g/L)2 mL/L,pH 7.0。121 ℃高压蒸汽灭菌20 min。固体培养基中加入20 g/L琼脂粉。
5-氟乳清酸(5-fluoroorotic acid,5-FOA)固体培养基[25]:YNB 6.9 g/L、尿嘧啶省却培养物0.77 g/L、葡萄糖20 g/L、琼脂粉20 g/L、尿吡啶2 g/L、5-FOA 1 g/L,pH 7.0,121 ℃高压蒸汽灭菌15 min。
最小液体培养基[25]:(NH4)2SO47.5 g/L、KH2PO414.4 g/L、MgSO4·7H2O 0.5 g/L、金属溶液(乙二胺四乙酸(ethylene diaminetetraaceticacid,EDTA)15.0 g/L、ZnSO4·7H2O 0.45 g/L、MnCl2·2H2O 1 g/L、CoCl2·6H2O 0.3 g/L、CuSO4·5H2O 0.3 g/L、Na2MoO4·2H2O 0.4 g/L、CaCl2·2H2O 0.45 g/L、FeSO4·7H2O 0.3 g/L、H3BO3 0.1 g/L、KI 0.1 g/L,pH 4.0)2 mL/L、维生素溶液(生物素0.05 g/L、对氨基苯甲酸0.2 g/L、烟酸1 g/L、泛酸钙1 g/L、吡哆醇1 g/L、硫胺素1 g/L、肌醇25 g/L)1 mL/L。pH 6.5。121 ℃高压蒸汽灭菌15 min。固体培养基中加入20 g/L琼脂粉。
1.2 仪器与设备
Agilent1290高效液相色谱(high performance liquid chromatography,HPLC)仪:美国安捷伦公司;DH-400型恒温培养箱:上海鸿都电子科技有限公司;LX-C50立式自动电热压力蒸汽灭菌锅:合肥华泰医疗设备有限公司;C1000梯度聚合酶链式反应(polymerase chain reaction,PCR)仪:美国Bio-Rad公司;NanoDrop 2000C超微量分光光度计:德国艾本德公司;SW-CJ-2D双人超净工作台:苏州净化设备有限公司;TGL-16G离心机:上海安亭科学仪器厂;DYCZ-24KF四板垂直电泳仪:北京六一生物科技有限公司;UV Bluestar A紫外可见分光光度计:北京莱伯泰科仪器有限公司。
1.3 实验方法
1.3.1 酿酒酵母IMX581中蔗糖水解酶基因MAL32、MAL12、MAL22的分析
有文献报道,酿酒酵母IMX581与酿酒酵母CEN.PK 113-7D的亲缘关系最近[7,26]。因此,首先,在美国国立生物技术信息中心(national center for biotechnology information,NCBI)中检索酿酒酵母CEN.PK 113-7D的基因组序列。然后,结合文献[25]报道的蔗糖水解酶基因MAL32、MAL12及MAL22在酿酒酵母CEN.PK 113-7D中的染色体序号,对酿酒酵母CEN.PK 113-7D的染色体基因序列分别检索,确定MAL32、MAL12以及MAL22所在的位置及基因序列信息,并确定其左(起始密码子前100 bp)右(终止密码子后100 bp)边界基因序列信息。最后,利用DNAMAN软件对MAL32、MAL12以及MAL22基因序列以及其左右边界基因序列进行比对,确定MAL32、MAL12以及MAL22基因序列以及其边界基因序列的同源性。
1.3.2 基因敲除质粒及repair片段的构建
Suc2基因的敲除引导核糖核酸(ribose nucleic acid,RNA)序列(guideRNA,gRNA)及重组修复(repair)片段的设计主要根据参考文献[22]进行。MAL32、MAL12及MAL22基因敲除guideRNA的设计则根据比对结果选取同源部分序列进行:采用网站(http://www.e-crisp.org/E-CRISP/)设计gRNA序列,repair片段选取MAL32、MAL12及MAL22基因左右边界同源部分进行设计,设计方法见图1。
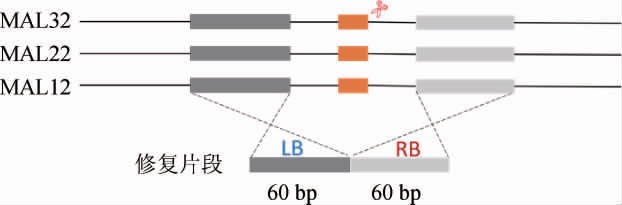
图1 酿酒酵母IMX581中基因MAL12、MAL22及MAL32的guideRNA和repair片段的设计
Fig.1 Design of guideRNA and repair fragments of gene MAL12,MAL22 and MAL32 of Saccharomyces cerevisiae IMX581
以实验室保存的用于酿酒酵母基因敲除的质粒ΔPex31-gRNA为模板,采用引物对gRNA-Suc2-R和pgRNAf1-F以及gRNA-Suc2-F和pgRNA-f1-R,通过PCR扩增得到Suc2基因敲除组件gRNA-Suc2-F1和gRNA-Suc2-F2,PCR产物通过胶回收试剂盒纯化片段。通过Gibson组装连接构建质粒gRNA-Suc2。
以ΔPex31-gRNA为模板,采用引物对gRNA-MAL-R和pgRNA-f1-F以及gRNA-MAL-F和pgRNA-f1-R,通过PCR扩增得到MAL基因敲除组件gRNA-MAL-F1和gRNA-MAL-F2,PCR产物通过胶回收试剂盒纯化片段。通过Gibson组装连接构建质粒gRNA-MAL。
通过NEBuilder HiFi DNA Assembly方法,将两条互补的单链suc2-repair-FW和suc2-repair-RV以1∶1摩尔比混合,加热到95 ℃,然后冷却到室温,合成suc2-repair片段。同理,将两条互补的单链MAL-repair-FW和MAL-repair-RV以1∶1摩尔比混合,加热到95 ℃,然后冷却到室温,合成MAL-repair片段。
1.3.3 Suc2、MAL32、MAL12和MAL22基因的敲除
通过醋酸锂化学转化法[27],将基因敲除质粒gRNA-Suc2和Suc2-repair片段转入酿酒酵母IMX581中,涂布于SC-Ura营养缺陷型固体培养基中,30 ℃条件下培养2 d。从中随机挑选转化子,提取基因组脱氧核糖核酸(deoxyribonucleic acid,DNA),采用引物Suc2-dg-FW和Suc2-dg-RV进行PCR验证。将验证正确的菌株划线于5-FOA固体培养基,30 ℃条件下培养2~3 d,待培养基上出现单菌落,挑选单菌落于5 mL YPD液体培养基中,30 ℃、180 r/min条件下培养12~16 h,提取基因组DNA进行阳性检测。将鉴定正确的菌株IMX581ΔSuc2划线于SC-Ura营养缺陷型固体培养基中,30 ℃条件下培养2 d没有单菌落出现,说明质粒gRNA-Suc2丢失成功。
将基因敲除质粒gRNA-MAL和MAL-repair片段转入酿酒酵母IMX581ΔSuc2中,涂布于SC-Ura营养缺陷型固体培养基中,30 ℃条件下培养2 d。从中随机挑选转化子,提取基因组DNA,采用引物对MAL-dg1-FW、MAL-dg1-RV和MAL-dg2-FW、MAL-dg2-RV进行PCR验证。将验证正确的菌株划线于5-FOA固体培养基,30 ℃条件下培养2~3 d,待培养基上出现单菌落,挑选单菌落于5 mL YPD液体培养基中,30 ℃、180 r/min培养12~16 h,提基因组DNA进行阳性检测。将鉴定正确的菌株IMX581ΔSuc2ΔMAL32ΔMAL12 ΔMAL22划线于SC-Ura营养缺陷型固体培养基中,30 ℃条件下培养2 d没有单菌落出现,说明质粒gRNA-MAL丢失成功。
1.3.4 酿酒酵母生长特性的测定
将酿酒酵母IMX581、IMX581ΔSuc2和IMX581ΔSuc2 ΔMAL32ΔMAL12ΔMAL22划线于YPD培养基,30 ℃条件下恒温培养2~3 d,挑单菌落接种到YPD液体培养基中,30 ℃、180 r/min条件下摇床培养24 h。按初始OD600nm值为0.1的接种量接种到含20 g/L蔗糖(葡萄糖)、2 g/L尿嘧啶的最小液体培养基中,30 ℃、180 r/min条件下培养,每隔4 h取样,采用紫外可见分光光度计在波长600 nm处测定OD600nm值。以发酵时间(x)为横坐标,OD600nm值(y)为纵坐标绘制生长曲线。
分别挑取酿酒酵母IMX581、IMX581ΔSuc2和IMX581 ΔSuc2ΔMAL32ΔMAL12ΔMAL22的单菌落接种于含20 g/L葡萄糖、2 g/L尿嘧啶的最小液体培养基中,30 ℃、180 r/min条件下摇床培养24 h,得到3个菌株的种子液。将种子液按照10-1、10-2和10-3三个梯度进行稀释,分别取1.5 μL点接到含20 g/L蔗糖(葡萄糖)、2 g/L尿嘧啶的最小固体培养基上,30 ℃条件下恒温培养48 h,观察3个菌株在平板中的生长情况。
1.3.5 酿酒酵母中蔗糖消耗的测定
分别挑取酿酒酵母IMX581、IMX581ΔSuc2和IMX581 ΔSuc2ΔMAL32ΔMAL12ΔMAL22的单菌落接种于YPD液体培养基中,30 ℃、180 r/min条件下培养12~16 h,得到种子液。按照初始OD600nm值为0.1的接种量接种到含20 g/L葡萄糖、2 g/L尿嘧啶的最小液体培养基中,30 ℃、180 r/min条件下培养24 h,再分别按照初始OD600nm值为0.1、5.0、10.0接种量接种到含20 g/L蔗糖、2 g/L尿嘧啶的最小液体培养基中发酵培养,分别在发酵24 h、48 h、72 h和96 h取1 mL发酵液,8 000 r/min离心2 min,收集上清并过膜制备样品。利用高效液相色谱分析样品中蔗糖的含量[16]。
2 结果与分析
2.1 酿酒酵母IMX581中蔗糖水解酶基因MAL32、MAL12、MAL22的分析
酿酒酵母IMX581中蔗糖水解酶基因MAL32、MAL12、MAL22的同源性比对结果见图2。由图2可知,MAL12、MAL22以及MAL32基因的同源性高达99%,3个基因的左右边界基因序列同源性分别达99%、99.3%,因此,根据这3个基因的特点,按照图1设计得到共同的gRNA引导序列(5'-TATAAGGAACTTATGATTTA-3')以及repair片段序列(5'-TTAAGCAGTTTTTTTGATAATCTCAAATGTACATCAGTCAAGCGTAACTAAAATACATAAAATTAGTGCCGACTTTTTTTTAGCGCGTACTTTAACGAAATAACAGATGATTTTTCACAT-3')。

图2 酿酒酵母IMX581中基因MAL12、MAL22及MAL32的序列比对分析结果
Fig.2 Sequence alignment analysis results of gene MAL12, MAL22 and MAL32 in Saccharomyces cerevisiae IMX581
MAL12、MAL22以及MAL32基因边界各取100 bp的比对结果,LB为左边界,RB为右边界。
2.2 Suc2、MAL32、MAL12及MAL22基因敲除菌株的构建及其鉴定
Suc2及Suc2、MAL32、MAL12、MAL22基因敲除菌株IMX581ΔSuc2和IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22的鉴定结果见图3。
由图3可知,未敲除Suc2基因的对照菌株IMX581的PCR扩增产物碱基长度为3 000 bp左右,而敲除Suc2基因的菌株IMX581ΔSuc2的PCR扩增产物碱基长度为900 bp左右;未敲除MAL32、MAL12及MAL22基因的对照菌株IMX581的PCR扩增产物碱基长度为3 000 bp左右,而敲除MAL32、MAL12及MAL22基因的工程菌株IMX581ΔSuc2 ΔMAL32ΔMAL12ΔMAL22的PCR扩增产物碱基长度为750 bp左右,结果表明,Suc2、MAL32、MAL22以及MAL12基因被敲除成功。

图3 菌株IMX581ΔSuc2、IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22的PCR验证结果
Fig.3 PCR identification results of strains IMX581ΔSuc2 and IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22
a中M为DL 2 000 DNA Marker;1、2、3均为Suc2基因敲除后的验证结果;4、5、6、7、8、9均为MAL32、MAL12及MAL22基因敲除后的验证结果。b中M为DL 10 000 DNA Marker;1、2、3均为Suc2基因敲除前的验证结果;4、5、6、7、8、9均为MAL32、MAL12和MAL22基因敲除前的验证结果。
2.3 菌株IMX581、IMX581ΔSuc2及IMX581ΔSuc2ΔMAL32 ΔMAL12ΔMAL22的生长情况分析
菌株IMX581、IMX581ΔSuc2和IMX581ΔSuc2ΔMAL32 ΔMAL12ΔMAL22在以葡萄糖或蔗糖为唯一碳源的培养基中的生长情况见图4。

图4 菌株IMX581、IMX581ΔSuc2和IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22生长能力的比较
Fig.4 Comparison of growth ability of strains IMX581,IMX581ΔSuc2 and IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22
a、b为各菌株在以葡萄糖、蔗糖为唯一碳源的培养基中的生长曲线;c、d为不同稀释浓度的各菌株在以葡萄糖、蔗糖为唯一碳源的培养基上的生长情况。
由图4可知,以葡萄糖为唯一碳源进行培养时,菌株IMX581、IMX581ΔSuc2和IMX581ΔSuc2ΔMAL32ΔMAL12 ΔMAL22均可以生长,但是敲除Suc2基因后,酿酒酵母的生长明显受到限制,进一步敲除MAL32、MAL12以及MAL22基因后,酿酒酵母的生长速率明显降低,且平稳期OD600nm值只有4.4。此外,当以蔗糖为唯一碳源对各菌株进行培养时,菌株IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22几乎不生长,而菌株IMX581ΔSuc2在前12 h生长缓慢,之后进入指数生长期,平稳期的OD600nm值可达4.3,但其与对照菌株IMX581相比,生长受到较明显的影响。为了进一步观察3个菌株在以葡萄糖或蔗糖为唯一碳源的培养基上的生长情况,选择在最小固体培养基中进行培养,结果表明,在以葡萄糖为唯一碳源的最小固体培养基中,菌株IMX581 ΔSuc2ΔMAL32ΔMAL12ΔMAL22的生长明显受到影响,而单独敲除Suc2基因的菌株IMX581ΔSuc2的生长影响较小;在以蔗糖为唯一碳源的最小固体培养基中,菌株IMX581 ΔSuc2ΔMAL32ΔMAL12ΔMAL22几乎不生长,而敲除Suc2基因的菌株IMX581ΔSuc2相比之下受到的影响较小。结果表明,敲除Suc2、MAL32、MAL12以及MAL22基因后,酿酒酵母IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22消耗蔗糖的途径大部分被阻断,而酿酒酵母IMX581ΔSuc2利用蔗糖的能力受到一定影响,但仍然可以利用蔗糖。
为进一步验证敲除不同蔗糖消耗途径中的基因对于酵母利用蔗糖的影响,考察不同初始OD600nm值条件下各酿酒酵母菌株对20 g/L蔗糖的消耗情况,结果见图5。由图5可知,当初始OD600nm值为0.1时,培养72 h后,菌株IMX581 ΔSuc2、IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22的蔗糖消耗率分别为28.5%、7.0%,而对照菌株IMX581消耗蔗糖的速度较快,24 h已经全部消耗完。而当初始OD600nm值提高到5.0时,菌株IMX581ΔSuc2和IMX581ΔSuc2ΔMAL32 ΔMAL12ΔMAL22消耗蔗糖的能力都比之前有所增加,培养72h后,蔗糖消耗率分别为35.5%、18.5%,对照菌株IMX581培养12 h就可以完全消耗蔗糖。继续提高初始OD600nm值到10.0时,其结果与初始OD600nm值为5.0时各菌株消耗蔗糖的情况相似,菌株IMX581ΔSuc2和IMX581ΔSuc2ΔMAL32 ΔMAL12ΔMAL22在培养72 h后,蔗糖消耗率分别为38.5%、23.0%。结果表明,酿酒酵母IMX581只敲除Suc2基因仍可进行部分蔗糖的利用,而敲除Suc2、MAL32、MAL12、MAL22基因的酵母在低浓度培养时几乎不消耗蔗糖,而高浓度培养时可以进行部分蔗糖的消耗,这可能是高浓度培养条件下,酵母体内基因的表达量增加,个别受蔗糖调控的基因表达之后而参与了部分蔗糖的分解,这与于平等[9]对MAL11和MAL12基因的驯化实验结果相一致。研究不同初始OD600nm值条件下各酿酒酵母菌株对20 g/L蔗糖的消耗情况,有助于为以酿酒酵母为宿主细胞进行生物转化蔗糖从而合成蔗糖基化合物提供理论基础。
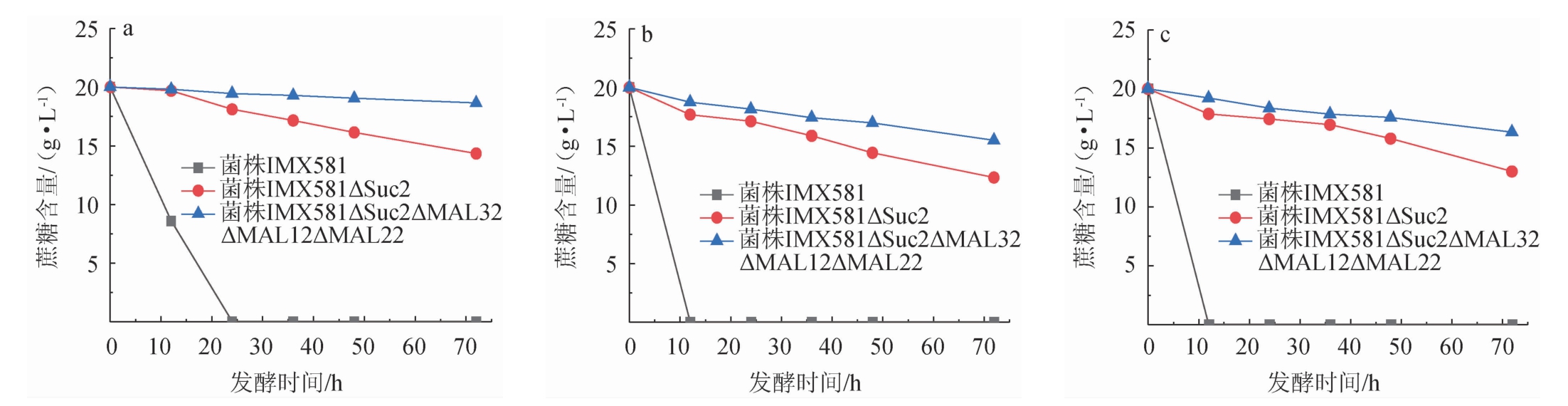
图5 不同初始OD600nm值条件下菌株IMX581、IMX581ΔSuc2和IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22对蔗糖的消耗情况
Fig.5 Sucrose consumption by strains IMX581,IMX581ΔSuc2 and IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22 under different initial OD600nm value
a、b、c分别为初始OD600nm值为0.1、5.0、10.0条件下各菌株对蔗糖的消耗情况。
3 结论
本研究通过Crispr-Cas9基因编辑的方法,对酿酒酵母IMX581的蔗糖代谢途径进行改造,敲除了与蔗糖水解相关的4个基因,成功构建了菌株IMX581ΔSuc2和IMX581ΔSuc2 ΔMAL32ΔMAL12ΔMAL22。当初始OD600nm值为0.1时,菌株IMX581ΔSuc2在以蔗糖为唯一碳源的培养基上仍能够生长,但培养56 h时,OD600nm值是出发菌株IMX581的52%;而菌株IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22几乎不能生长。同时,进一步考察不同初始OD600nm值(0.1、5.0、10.0)条件下菌株IMX581ΔSuc2和IMX581ΔSuc2ΔMAL32ΔMAL12 ΔMAL22对蔗糖的消耗情况,发现菌株IMX581ΔSuc2和IMX581ΔSuc2ΔMAL32ΔMAL12ΔMAL22的蔗糖消耗率较出发菌株IMX581均显著降低,培养72 h后,蔗糖消耗率分别为28.5%、35.5%、38.5%和7.0%、18.4%、22.5%。可以得出菌株IMX581只敲除Suc2基因后,在低浓度接种条件下,仍可消耗部分蔗糖;敲除Suc2、MAL32、MAL12和MAL22四个基因后,在低浓度接种条件下,几乎不消耗蔗糖。而高浓度接种条件下,酵母体内基因的表达量增加,个别受蔗糖调控的基因表达之后而参与了部分蔗糖的分解。总体而言,敲除Suc2、MAL32、MAL12和MAL22四个基因,可使酿酒酵母几乎不能利用蔗糖,这为后续酿酒酵母利用蔗糖转化合成一些新的化合物奠定基础。
[1]MARQUES W L,RAGHAVENDRAN V,STAMBUK B U,et al.Sucrose and Saccharomyces cerevisiae:a relationship most sweet[J].FEMS YeastRes,2016,16(1):107-123.
[2]GOMBERT A K,MARIS A J A V.Improving conversion yield of fermentable sugars into fuel ethanol in 1st generation yeast-based production processes[J].Curr Opin Biotechnol,2015,33:81-86.
[3]DELLA-BIANCA B E,BASSO T O,STAMBUK B U,et al.What do we know about the yeast strains from the Brazilian fuel ethanol industry?[J].Appl Microbiol Biot,2013,97(3):979-991.
[4]陈娜,林卢奇,毛银,等.代谢改造酿酒酵母生产葡萄糖二酸[J].食品与发酵工业,2018,44(12):19-25.
[5]黄晓斌,仲蕾蕾.遗传改造酿酒酵母生产植物萜类药物的策略[J].微生物学通报,2015,42(10):2019-2025.
[6]邹春悦.酵母菌在食品中的作用研究进展[J].化工中间体,2018(3):96-97.
[7]殷泽禄,万虎.大肠杆菌的研究综述[J].甘肃畜牧兽医,2019,49(5):33-35.
[8]MARQUES W L,MANS R,MARELLA E R,et al.Elimination of sucrose transport and hydrolysis in Saccharomyces cerevisiae:a platform strain for engineering sucrose metabolism[J].FEMS Yeast Res,2017,17(1):1-11.
[9]于平,黄星星,张一舒.枯草芽孢杆菌ZJS18发酵生产γ-聚谷氨酸培养条件的优化[J].食品科学,2018,39(22):87-92.
[10]BADOTTI F,D魣RIO M G,ALVES S L,et al.Switching the mode of sucrose utilization by Saccharomyces cerevisiae[J].Microb Cell Factories,2008,7(1):1-11.
[11] ZHOU Y J,BUIJS N A,ZHU Z W,et al.Production of fatty acid-derived oleochemicals and biofuels by synthetic yeast cell factories[J].Nat Commun,2016,7(1):11709.
[12]MEADOWS A L,HAWKINS K M,TSEGAYE Y,et al.Rewriting yeast central carbon metabolism for industrial isoprenoid production[J].Nature,2016,537(7622):694-697.
[13]V魣SQUEZ-BAHENA J M,VEGA-ESTRADA J,SANTIAGO-HERN魣-NDEZ J A,et al.Expression and improved production of the soluble extracellular invertase from Zymomonas mobilis in Escherichia coli[J].Enzyme Microb Technol,2006,40(1):61-66.
[14]MAIORELLA B L,BLANCH H W,WILKE C R,et al.Economic evaluation of alternative ethanol fermentation processes[J].Biotechnol Bioeng,1984,26(9):1003-1025.
[15] KORSHUNOVA I V,NAUMOVA E S,NAUMOV G I.Comparativemolecular-genetic analysis of the beta-fructosidases in yeast Saccharomyces[J].Mol Biol,2005,39(3):413-419.
[16]张志凯,汤宏赤,樊少林,等.酿酒酵母蔗糖关键代谢途径suc2基因的敲除及其蔗糖代谢特性的变化分析[J].基因组学与应用生物学,2016,35(3):622-628.
[17] WANG S A,LI F L.Invertase SUC2 is the key hydrolase for inulin degradation in Saccharomyces cerevisiae[J].Appl Environ Microbiol,2013,79(1):403-406.
[18] KOK S D,YILMAZ D,SUIR E,et al.Increasing free-energy (ATP)conservation in maltose-grown Saccharomyces cerevisiae by expression of a heterologous maltose phosphorylase[J].Metab Eng,2011,13(5):518-526.
[19] DENG X,PETITJEAN M,TESTE M A,et al.Similarities and differences in the biochemical and enzymological properties of the four isomaltases from Saccharomyces cerevisiae[J].FEBS Open Bio,2014,4(1):200-212.
[20]张驹.CRISPR/Cas9系统介导羊MSTN基因敲除和定点整合fat-1基因的研究[D].呼和浩特:内蒙古大学,2016.
[21] SCHAAFF I,HEINISCH J,ZIMMERMANN F K.Overproduction of glycolytic enzymes in yeast[J].Yeast,1989,5(4):285-290.
[22] MANS R,ROSSUM H M V,WIJSMAN M,et al.CRISPR/Cas9:a molecular Swiss army knife for simultaneous introduction of multiple genetic modifications in Saccharomyces cerevisiae[J].FEMS yeast Res,2015,15(2):1-15.
[23]钟成,黄龙辉,刘其敬,等.CRISPR/dCas9在细菌转录调控中的应用及展望[J].天津科技大学学报,2019,34(2):1-5.
[24]郝彦玲,王颖,朱本忠,等.卷枝毛霉Δ6-脂肪酸脱氢酶基因的克隆及在酿酒酵母中的高效表达[J].遗传学报:英文版,2005,32(3):303-308.
[25]安伯格.酵母遗传学方法实验指南:2005版[M].北京:科学出版社,2009:123-130.
[26]NIJKAMP J F,BROEK M V D,DATEMA E,et al.De novo sequencing,assembly and analysis of the genome of the laboratory strain Saccharomyces cerevisiae CEN.PK113-7D,a model for modern industrial biotechnology[J].Microb Cell Fact,2012,11(1):36.
[27]GIETZ R D,SCHIESTL R H.High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method[J].Nat Protoc,2007,2(1):31-41.