3-甲硫基丙醇(俗称菠萝醇)是一种重要的含硫高级醇,常温下为易流动的淡黄色液体,低浓度时具有强烈的肉类芬芳香气,天然存在于苹果、番茄、酱油、奶酪、白酒、葡萄酒、啤酒、威士忌、白兰地等食品或饮料中,能赋予众多食品其特有的风味[1]。3-甲硫基丙醇香气阈值低,在1~3 mg/L之间即可表现出一种令人愉悦的、肉类的或者烤制奶酪的香气,是调配肉味香精的重要香料之一,是国家允许使用的食品用香料,广泛应用于香精香料调配和食品增香,是一类应用非常广泛的食用香料化合物,市场需求量大[2-3]。
目前,3-甲硫基丙醇的生产方法包括化学合成法和微生物发酵法,其中化学合成法是目前3-甲硫基丙醇生产的主要方法。化学合成法虽然成本低,但却存在合成过程污染较大等问题,不能满足消费者日益对环保、绿色和纯天然香精香料青睐的需求。由于生物转化制备天然3-甲硫基丙醇具有构型单一、绿色环保,成为近年来生产3-甲硫基丙醇的重要发展方向[4]。相关研究表明,微生物,尤其是酵母能代谢L-蛋氨酸发酵合成3-甲硫基丙醇,是生物转化生产天然3-甲硫基丙醇的有效途径。目前,有关微生物发酵合成3-甲硫基丙醇的研究相对较少,需要对其进行深入研究。本文正是基于微生物合成3-甲硫基丙醇的重要性,通过对微生物合成3-甲硫基丙醇的主要微生物及其代谢途径中的关键酶进行简述,加强对微生物产天然3-甲硫基丙醇研究现状的了解,为相关研究提供参考。
1 3-甲硫基丙醇在食品中的作用
3-甲硫基丙醇具有强烈的甜香、汤或肉样香气,早在1973年美国食用香料与提取物制造者协会(Flavour and Extract Manufactuere's Association,FEMA)将其确定为一般认为安全的香料物质,在食品中建议用量为0.1~50 mg/kg[5],1986年,我国相关食品国家标准也将其列为暂时允许使用的食品香料。3-甲硫基丙醇特有的肉香味及其低阈值特性,对众多食品呈现独特的风味发挥了重要的作用,如在切达干酪、蓝奶酪、卡门培尔干酪等奶酪中呈现发酵卷心菜的独特气味,能很容易在奶酪中被辨识出,是奶酪整体风味的重要组成部分[6-7];尽管3-甲硫基丙醇是否是芝麻香型白酒中的特征风味物质存有争论,但其在部分品牌芝麻香型白酒的风格形成中发挥着重要作用,并且在豉香型白酒及果酒的呈味中也具有重要的贡献,表现出花椰菜和煮熟蔬菜的香气,对风味品质的贡献显著[1,8];3-甲硫基丙醇是黄酒中的重要含硫化合物,在清爽型黄酒中香气强度较高,主要赋予黄酒青香和煮熟蔬菜的气味,丰富黄酒的香气[9];3-甲硫基丙醇也是葡萄酒中重要的风味物质,能赋予葡萄酒煮土豆味,丰富其风味[10]。除此之外,因其具有甜的肉及肉汤气味,3-甲硫基丙醇是调配肉味香精的重要原料,还可用于调配诸如水果、蔬菜、酱油、酒等众多食用香精,用途广泛[11]。
2 3-甲硫基丙醇的生产方法
2.1 化学合成法
化学合成方法因具有成本低、合成快、产物浓度高的优势,一直是3-甲硫基丙醇主流的生产方法。目前,有关3-甲硫基丙醇的化学合成方法主要有四种:①采用甲硫醇和烯丙醇的加成反应。②利用3-氯-1-丙醇和甲硫醇钠在醚中进行取代反应。③采用烯丙基甲基硫醚的分解反应。④3-巯基-1-丙醇和硫酸二甲酯的取代反应(图1)[11]。其中,前两种反应是目前实际生产中所采用的方法,相关研究也较多。刘玉平等[11]以3-氯-1-丙醇和甲硫醇钠为原料,在相转移催化剂四甲基溴化铵的作用下发生取代反应,制得了纯度较高的3-甲硫基丙醇,产率达到81%。虽然,化学合成成本低廉,但存在的一些诸如原料毒性高、生产过程中的毒副产物难以去除及污染较大等问题引起关注,在一定程度上限制了其在工业化生产中的长期使用,必须拓展新的生产方法[12]。
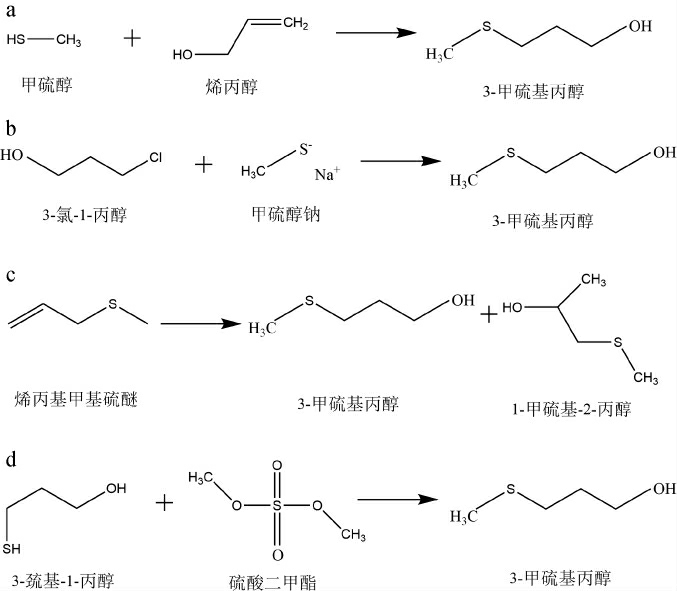
图1 3-甲硫基丙醇的4种化学合成方法
Fig.1 Four chemical synthesis methods for 3-methithiopropanol
2.2 微生物合成法
有关研究表明,微生物可以将甲硫氨酸转化为3-甲硫基丙醇[7,13]。如,BUZZINI P等[13]筛选获得10株能够转化氨基酸生成3-甲硫基丙醇的酵母菌株,产量在0.04~0.40 g/L。虽然,当前微生物转化合成3-甲硫基丙醇的速率不及化学方法,但由于其具有反应条件温和、产物构型单一、绿色环保、所用原料安全无污染等特点,成为近年来制备“绿色”和“天然”的3-甲硫基丙醇的重要发展方向。
2.2.1 生产3-甲硫基丙醇的天然菌株
当前,国内外有关微生物转化生产天然3-甲硫基丙醇的研究相对较少,能够产生3-甲硫基丙醇的微生物种类不多,主要集中于酵母菌株(表1)。已报道能够产3-甲硫基丙醇的菌株包括酿酒酵母(Saccharomyces cerevisiae)、乳酸克鲁维酵母(Kluyveromyces lactis)、汉逊德巴利酵母(Debaryomyces hansenii)、酒类酒球菌(Oenococcus oeni)、担子菌酵母(Basidiomycetous yeasts)、解脂耶氏酵母(Yarrowia lipolytica)、鲁氏接合酵母(Zygosaccharomyces rouxii)、拟威尔酵母(Williopsis)等[4,13-21]。目前,天然微生物转化生产3-甲硫基丙醇的能力相对较弱,CASTILLO-LOZANO M L D等[7]通过摇瓶发酵培养5株酵母菌株,其中乳酸克鲁维酵母KL640和酿酒酵母SC45能转化生成3-甲硫基丙醇,产量分别为14.51 mg/L和25.70 mg/L;BUZZINI P等[13]从37株担子菌酵母中获得10株可以生成3-甲硫基丙醇的酵母,其产量在0.04~0.40 g/L;侯晟等[4]优化了酿酒酵母SC408发酵合成3-甲硫基丙醇的条件,在最优条件下,该菌株所产3-甲硫基丙醇的量达到1.6 g/L;为进一步提高酿酒酵母SC408发酵合成3-甲硫基丙醇的产量,王成涛等[22]采用发酵-分离耦合技术降低3-甲硫基丙醇对细胞的毒害作用,最终产量可达2.26 g/L,为已报道天然菌株中所产3-甲硫基丙醇最高产量;另外,WILLIAMS A G等[23]从切达干酪中分离获得29株能产3-甲硫基丙醇的乳酸菌,产量也较低,在0.04~0.30 g/L之间。天然菌株所产3-甲硫基丙醇普遍低的现象(几乎都低于0.1 g/L),成为采用天然菌株进行工业化生产3-甲硫基丙醇最主要的限制因素[24]。
表1 产3-甲硫基丙醇的天然菌株
Table 1 Natural strains that produce 3-methylthiopropanol
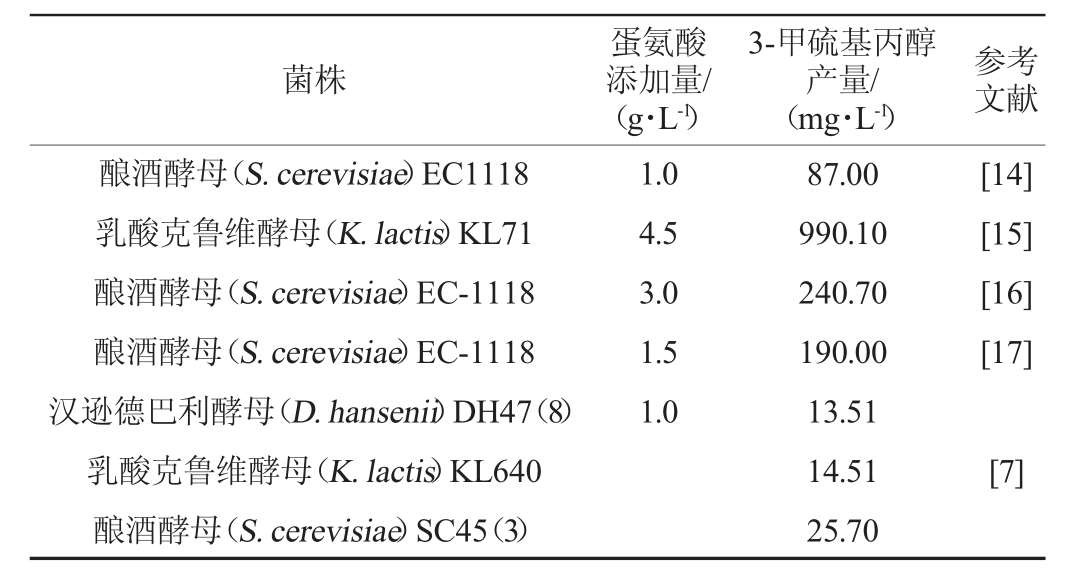
续表

2.2.2 生产3-甲硫基丙醇的基因工程菌
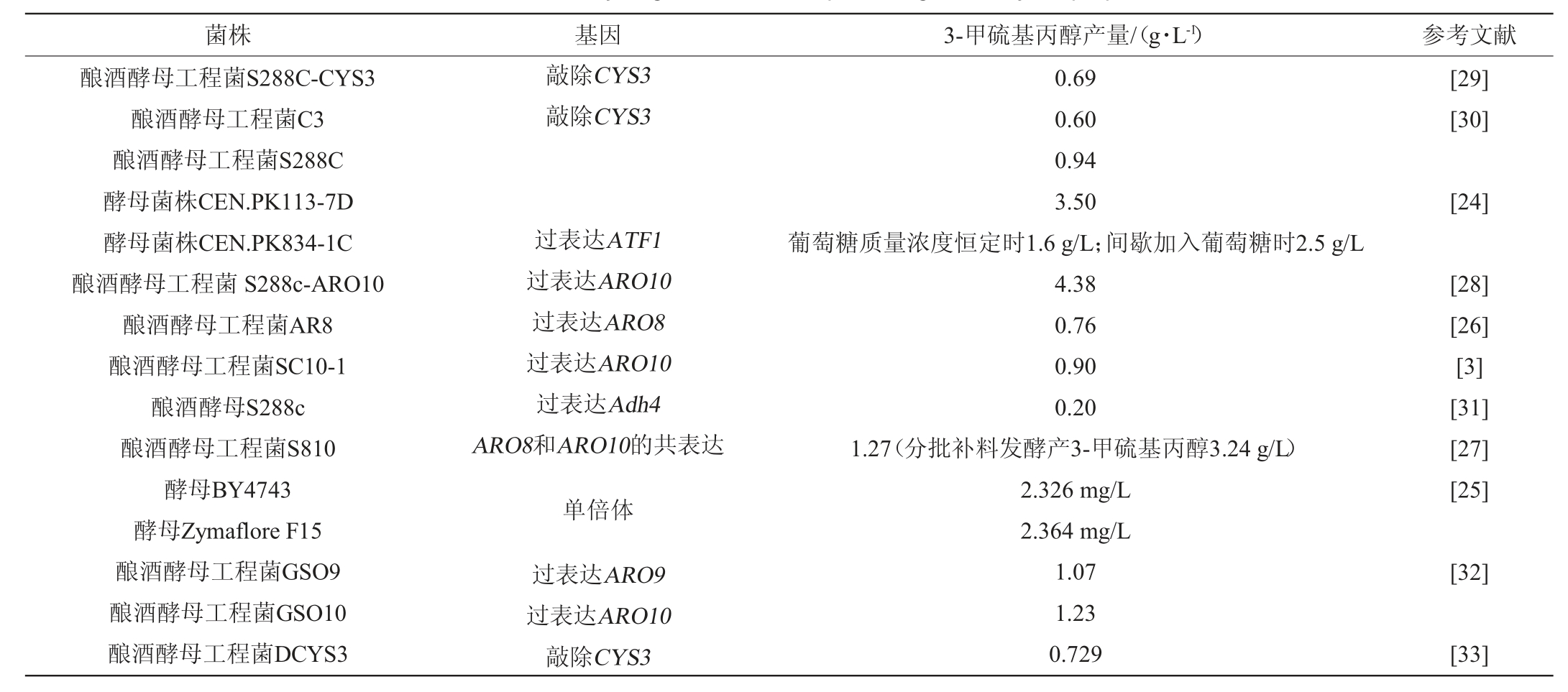
随着人们对天然生产的3-甲硫基丙醇的需求增加以及基因工程技术的不断发展,越来越多的学者开始采用分子生物学技术对菌株进行改造,以此实现高产3-甲硫基丙醇的工程菌株。刘丽等[26]通过过表达酿酒酵母S288C中的氨基转移酶ARO8基因构建工程菌ARO8,工程菌所产3-甲硫基丙醇达到0.76 g/L,相比野生型S288C提高了28.8%;温明显等[3]通过过表达酿酒酵母INVSc1中的脱羧酶基因ARO10构建了酿酒酵母转化子SC10-1,其产3-甲硫基丙醇的量相比天然菌株提高了55.2%,达到0.90 g/L;YIN S等[27]通过共表达氨基转移酶基因ARO8和ARO10构建了酿酒酵母转化子S810,在摇瓶水平上,该工程菌所产3-甲硫基丙醇达到1.27 g/L,通过分批补料发酵罐培养,产量达到3.24 g/L;ETSCHMANN M M W等[24]通过分批补料发酵方法实现了工程菌酿酒酵母CEN.PK113-7D高产3-甲硫基丙醇的目的,产量可达3.5 g/L;杨雪莲等[28]优化了过表达脱羧酶基因ARO10的酿酒酵母工程菌合成3-甲硫基丙醇的发酵条件,在最优条件下,工程菌株所产3-甲硫基丙醇可达4.38 g/L,为已报道中的最高水平。部分产3-甲硫基丙醇的基因工程菌株见表2,由表2可知,通过基因改造可以实现3-甲硫基丙醇产量的大幅度提高,为工业化应用微生物转化生产3-甲硫基丙醇打下基础。
表2 产3-甲硫基丙醇的基因工程菌株
Table 2 Genetically engineered strains producing 3-methylthiopropanol
3 微生物生物转化生产3-甲硫基丙醇的关键基因和酶
在酵母细胞中,Ehrlich途径是生物转化生成高级醇的最重要的途径[2]。3-甲硫基丙醇是高级醇的一种,可以由蛋氨酸经过Ehrlich途径代谢产生[34]。在3-甲硫基丙醇的生物合成过程中,如果没有蛋氨酸,3-甲硫基丙醇几乎不产生,这足以表明Ehrlich途径在合成3-甲硫基丙醇中的重要性[24]。在Ehrlich途径中,首先,蛋氨酸在氨基转移酶(aminotransferase)作用下发生转氨基作用生成α-酮-γ-甲硫基丁酸(α-keto-γ-(methylthio)butyrate,KMBA),然后在脱羧酶(decarboxylase)作用下生成3-甲硫基丙醛,最后在乙醇脱氢酶(alcohol dehydrogenase,ADH)作用下发生还原反应生成3-甲硫基丙醇[24,35]。
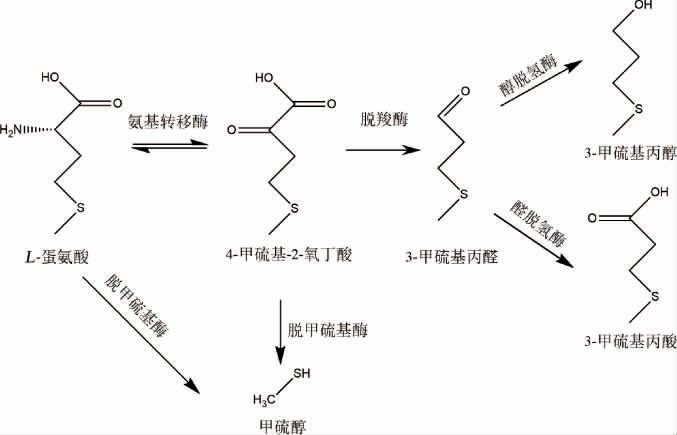
图2 微生物转化L-蛋氨酸产3-甲硫基丙醇的Erich途径
Fig.2 Erich pathway of microbial transformation of L-methionine to produce 3-methylthiopropanol
3.1 氨基转移酶
在Ehrich途径中具有转氨作用的基因有4种,分别为BAT1、BAT2、ARO8和ARO9[2]。CHOLET O等[36]对汉斯德巴氏酵母菌(Debaryomyces hansenii)、马克斯克鲁维酵母(Kluyveromyces marxianus)和解脂耶氏酵母(Yarrowia lipolytica)中的86个基因进行了脱氧核糖核酸(deoxyribonucleic acid,DNA)微阵列分析,发现能高效同化L-蛋氨酸的解脂耶氏酵母中,与酿酒酵母同源的基因BAT2和ARO8表达量较高,表明这两种转氨酶在酵母通过Ehrich途径转化蛋氨酸生成3-甲硫基丙醇中起到一定的作用。研究表明,基因BAT1编码线粒体氨基转移酶BAT1p,优先参与支链氨基酸(BCAAs)的生物合成,BAT2则编码细胞质氨基转移酶BAT2p,参与BCAAs的分解代谢,两者在3-甲硫基丙醇的合成中起到的作用较小,而在合成其他高级醇中的作用显著[37-40]。基因ARO8和ARO9则分别编码Aro8p和Aro9p,两者作用的底物是芳香族氨基酸,分别被称为芳香族氨基酸转移酶Ⅰ和Ⅱ[41-42]。Aro8p属于组成型表达蛋白,是主要的芳香族氨基酸转移酶,它的表达易受到氨基酸合成代谢的影响;Aro9p则是一种诱导表达蛋白,其表达受到氮源调控以及ARO8基因缺失等因素的影响。在已有研究中发现,虽然L-蛋氨酸不是芳香族氨基酸,但其代谢过程与芳香族氨基酸相似,基因ARO8和ARO9在利用L-蛋氨酸合成3-甲硫基丙醇中起到重要的作用[42-43]。Aro8p和Aro9p都具有广泛的底物特异性,其表达水平在以L-蛋氨酸为唯一氮源、葡萄糖受限的条件下较高,Aro8p表现出较强的催化蛋氨酸转氨基生物活性[2,43-44]。URRESTARAZU A等[43]研究表明,单独缺失ARO8或ARO9基因的酿酒酵母都能在含有蛋氨酸存在的基础培养基上生长,而同时缺失ARO8和ARO9基因后则无法生长,通过体外实验发现,ARO8基因编码的芳香族氨基酸转移酶I不仅对芳香族氨基酸具有转氨作用,而且还对蛋氨酸、α-氨基乙二酸和亮氨酸具有转氨活性,ARO9基因编码的芳香族氨基酸转移酶II同样具有较宽广的底物特异性。YIN S等[27]研究发现,过表达ARO8基因提高的芳香族氨基酸转移酶活性明显高于过表达ARO9基因所获得的活性,表明相比芳香族氨基酸转移酶II,芳香族氨基酸转移酶I在转化L-蛋氨酸合成3-甲硫基丙醇中效果更为明显。DEED R C等[25]研究表明,单独缺失基因ARO8可使酿酒酵母BY4743合成3-甲硫基丙醇的能力下降44%,使商用葡萄酒酵母F15合成3-甲硫基丙醇的能力降低92%;单独缺失ARO9基因对酿酒酵母BY4743合成3-甲硫基丙醇没有影响,然而却使菌株F15的3-甲硫基丙醇含量相比野生型菌株提高了46%。另外,相关研究还表明,转氨酶的转氨作用具有可逆性,Aro8p和Aro9p可以催化α-KMBA发生转氨作用产生L-蛋氨酸[45-46]。综上可见,酵母通过Ehrlich途径合成3-甲硫基丙醇中转氨酶Aro8p所发挥的转氨作用最为重要。
3.2 脱羧酶
在转氨酶的作用下,L-蛋氨酸转化为α-KM BA,α-KMBA则在脱羧酶作用下生成3-甲硫基丙醛,这种醛也具有独特的肉香味,对番茄、奶酪和酱油呈味也发挥重要的作用[47-48]。脱羧酶与转氨酶不同,在Ehrlich途径中是不可逆的[2]。VURALHAN Z等[48-49]研究了酿酒酵母CEN.PK113-7D中的脱羧酶,结果表明其拥有5个候选基因可以编码:ARO10(YDR380w)、THI3(YDL080c)、PDC1(YLR044c)、PDC5(YLR134w)和PDC6(YGR087c),表达的脱羧酶属于硫胺二磷酸依赖性脱羧酶[27]。ARO10基因编码的脱羧酶具有广泛的底物特异性,尤其对芳香族氨基酸具有很强的转氨活性,是产生支链和含硫杂醇类的关键贡献者,尚未发现THI3编码具有活性的脱羧酶,PDC1基因和PDC5基因编码为丙酮酸脱羧酶,与ARO10基因编码的脱羧酶类似,对所有支链和含硫的2-oxo酸均表现出活性,两者中一种的缺失都会激发另一种的大量表达,而PDC6仅能在含低硫的条件下表达,并且PDC5基因编码的脱羧酶脱羧效率与PDC1基因编码的相当,而PDC6基因编码的脱羧酶的脱羧效率最低[50-52]。
VURALHAN Z等[48]研究发现,在以苯丙氨酸、亮氨酸或蛋氨酸为唯一氮源时,5种脱羧酶基因中只有ARO10基因转录水平上调,其中使用L-Met作为唯一氮源时,ARO10基因的转录水平上调15倍,当其他四个基因失活时,ARO10能编码足够量的苯丙酮酸脱羧酶活性[49]。PERPETE P等[35]研究发现,KMBA在Ydr380wp作用下可脱羧生成3-甲硫基丙醛,若Ydr380wp失活,酿酒酵母不能将L-蛋氨酸转化为3-甲硫基丙醇,而缺失其他四个脱羧酶仍能转化3-甲硫基丙醇,由此推测KMBA的脱羧作用受Ydr380wp特异性影响。LIU B等[21]研究发现,盐度可以提高鲁氏接合酵母中ARO10基因的表达,从而增加其合成3-甲硫基丙醇的含量。YIN S等[27]通过基因过表达研究同样发现,ARO10基因的过表达可显著提高细胞脱羧酶活性,从而提高了3-甲硫基丙醇的产量。由此可见,5种脱羧酶中,ARO10基因所编码的脱羧酶对酵母通过Ehrlich途径合成3-甲硫基丙醇更为重要。
3.3 醇脱氢酶
在Ehrlich途径中,3-甲硫基丙醇合成的最后一步是氧化还原反应,其由3-甲硫基丙醛在醇脱氢酶的作用下发生还原反应而生成的,这一步是非限制性速度,不限制3-甲硫基丙醇的转化速度,因为酵母中存在许多乙酸脱氢酶,可被其中的任何一种催化完成[2]。
BOER V M等[53]研究发现,醛的氧化或还原受培养条件的影响,在葡萄糖限制型的恒化培养基中,当进行有氧发酵时大部分醛被氧化为酸,进行无氧发酵时大部分醛被还原为醇。VALLET A等[54]研究表明,仅有的能合成3-甲硫基丙醇细菌-酒类酒球菌IOEB 8406中存在乙醇脱氢酶(ADH)活性,在3-甲硫基丙醇的合成中起到重要的作用。DICKINSON J R等[55]采用13C核磁共振(nuclear magnetic resonance,NMR)和气相色谱-质谱(gas chromatographymass spectrometry,GC-MS)分析发现,醇脱氢酶Adh1p、Adh2p、Adh3p、Adh4p、Adh5p和Sfa1p中的任何一种都能将醛转化成3-甲硫基丙醇。然而,CHE Y X等[31]分析了在添加3-甲硫基丙醛的条件下酵母中7个ADH基因的表达情况,结果发现,只有ADH4表达量明显增加,在酿酒酵母S288c中过表达ADH4后,3-甲硫基丙醇产量明显增加,这表明不同醇脱氢酶在合成3-甲硫基丙醇中的重要性不同,有待进一步研究。
3.4 脱甲硫基酶
在Ehrlich途径中,L-蛋氨酸或其衍生物α-KMBA可以通过脱甲基化转化,从而产生甲硫醇(methanthiol,MTL),从而对3-甲硫基丙醇的合成产生影响,具有脱甲硫基功能的酶有胱硫醚-γ-裂解酶(cystathionine γ-lyase,CGL)和胱硫醚-β-裂解酶(cystathionine β-lyase,CBL)等[35]。KNOLL C等[56]研究发现酒类酒球菌中胱硫醚-γ/β-裂解酶似乎参与了蛋氨酸代谢途径生成甲硫醇,在过表达胱硫醚-γ-裂解酶基因的菌株中,显著增加了蛋氨酸的分解,产生了更多的甲硫醇。JIA K Z等[57]研究发现胱氨酸-β-裂解酶(CBL)可以将蛋氨酸和KMBA降解为甲硫醇,是合成甲硫醇的限速酶。然而,刘丽等[30]敲除胱硫醚-γ-裂解酶基因后,酿酒酵母S288c所产3-甲硫基丙醇产量降低,推测可能除了胱硫醚-γ-裂解酶外,还有其他酶参与了L-蛋氨酸的去甲基化。ZHANG Q等[58]在粉红螺旋聚孢霉和酿酒酵母中过表达E3-泛素-蛋白连接酶HUWE1后显著提高了去甲基硫醇途径中甲硫醇及其衍生物的生物合成,而抑制了Ehrlich途径中的3-甲硫基丙醛和3-甲硫基丙醇的生物合成。由此可见,影响Ehrlich途径的支路上的酶对于3-甲硫基丙醇合成也具有重要影响。
4 展望
3-甲硫基丙醇是许多食品中的重要风味物质,随着人们对天然添加剂3-甲硫基丙醇需求的增加,微生物转化法生产3-甲硫基丙醇成为满足消费者需求的重要方法。但已报道的天然菌株中具有高产3-甲硫基丙醇的菌株较少,需要进一步筛选高产产3-甲硫基丙醇的天然菌株,另外,可以利用基因工程对天然菌株进行改造,如可以通过过表达Ehrlich途径中的关键酶,也可以敲除胱硫醚-γ-裂解酶基因,减少蛋氨酸的支路代谢,从而提高3-甲硫基丙醇的产量,再次,也可以通过在发酵过程中分离出发酵产物3-甲硫基丙醇以减少其对微生物的毒性,从而连续不断的转化生成3-甲硫基丙醇。随着对微生物研究的深入,也可利用微生物群落法对不同的微生物进行最佳组合从而获得3-甲硫基丙醇的最大产量。由此可见,通过传统筛选方法、分子生物技术及发酵技术等组合可以实现生物法高效转化合成3-甲硫基丙醇,为生物法合成天然的3-甲硫基丙醇提供广阔的应用空间。
[1]赵东瑞,柳金龙,张锋国,等.GC-MS/SIM内标法定量分析两种类型酒中的3-甲硫基丙醇[J].中国食品学报,2016,16(5):196-204.
[2]HAZELWOOD L A,DARAN J,VAN MARIS A J A,et al.The Ehrlich pathway for fusel alcohol production:A century of research on Saccharomyces cerevisiae metabolism[J].Appl Environ Microb,2008,74(8):2259-2266.
[3]温明显,王成涛,杨雪莲,等.ARO10基因在Saccharomyces cerevisiae中的克隆表达及其对3-甲硫基丙醇合成代谢的影响[J].食品与发酵工业,2012,38(4):1-5.
[4]侯晟,赵磊,杨雪莲,等.酿酒酵母SC408转化3-甲硫基丙醇条件的研究[J].北京工商大学学报(自然科学版),2011,29(2):18-22.
[5]NEWBERNE P,SMITH R L,DOULL J,et al.GRAS flavoring substances 18[J].Food Technol,1998,52(9):58-76.
[6]FRANK D C,OWEN C M,PATTERSON J.Solid phase microextraction(SPME)combined with gas-chromatography and olfactometry-mass spectrometry for characterization of cheese aroma compounds[J]. Food Sci Technol,2004,37(2):139-154.
[7]CASTILLO-LOZANO M L D,DELILE A,SPINNLER H E,et al.Comparison of volatile sulphur compound production by cheese-ripening yeasts from methionine and methionine-cysteine mixtures[J]. Appl Microbiol Biot,2007,75(6):1447-1454.
[8] DU R B,LIU J,JIANG J,et al.Construction of a synthetic microbial community for the biosynthesis of volatile sulfur compound by multimodule division of labor[J].Food Chem,2021,347:129036.
[9]罗涛.清爽型黄酒香气特征及麦曲对其香气的影响[D].无锡:江南大学,2008.
[10]席艳茹,唐柯,徐岩,等.应用定量描述分析和气相色谱-闻香/质谱法研究黄土高原赤霞珠干红葡萄酒香气特征[J].食品与发酵工业,2016,42(5):192-197.
[11]刘玉平,孙宝国,郑福平,等.3-甲硫基-1-丙醇的合成研究[J].香料香精化妆品,2003(6):1-2.
[12]杨尚彤,陈洪,黄友和,等.生物转化法生产天然香料3-甲硫基-1-丙醇的研究进展[C].上海:第三届全国香料香精化妆品专题学术论坛,2013.
[13]BUZZINI P,ROMANO S,TURCHETTI B,et al.Production of volatile organic sulfur compounds(VOSCs)byBasidiomycetousyeasts[J].FEMS Yeast Res,2005,5(4-5):379-385.
[14] SEOW Y,ONG P K C,LIU S.Production of flavour-active methionol from methionine metabolism by yeasts in coconut cream[J].Int J Food Microbiol,2010,143(3):235-240.
[15]MATTHEW K P,SUN J C,LIU S Q,et al.Optimization of L-methionine bioconversion to aroma-active methionol by Kluyveromyces lactis using the taguchi method[J].J Food Res,2013,2(4):90-100.
[16]LWA H,SUN J C,LIU S Q.Optimization of methionol bioproduction by Saccharomyces cerevisiae using response surface methodology[J]. Ann Microbiol,2015,65(1):197-205.
[17] QUEK J M B,SEOW Y X,ONG P K,et al.Formation of volatile sulfur-containing compounds by Saccharomyces cerevisiae in soymilk supplemented with L-methionine[J]. Food Biotechnol,2011,25(4):292-304.
[18]VALLET A,LUCAS P,LONVAUD-FUNEL A,et al.Pathways that produce volatile sulphur compounds from methionine in Oenococcus oeni[J].J Appl Microbiol,2008,104(6):1833-1840.
[19]TAN A W J,LEE P,SEOW Y,et al.Volatile sulphur compounds and pathways of L-methionine catabolism in Williopsis yeasts[J].Appl Microbiol Biot,2012,95(4):1011-1020.
[20]LIU S Q,CROW V L.Production of dairy-based,natural sulphur flavor concentrate by yeast fermentation[J]. Food Biotechnol,2010,24(1):62-77.
[21]LIU B,WANG X F,ZHAO J H,et al.Effects of salinity on the synthesis of 3-methylthiopropanol,2-phenylethanol,and isoamyl acetate in Zygosaccharomyces rouxii and Z.rouxii 3-2[J].Bioproc Biosyst Eng,2020,43(5):831-838.
[22]王成涛,侯晟,赵磊,等.发酵-分离耦合技术转化3-甲硫基丙醇的研究[C].Bali Island:Proceedings of 2011 International Conference on Biomedicine and Engineering,2011.
[23]WILLIAMS A G,NOBLE J,BANKS J M.Catabolism of amino acids by lactic acid bacteria isolated from Cheddar cheese[J].Int Dairy J,2001,11(4):203-215.
[24]ETSCHMANN M M W,KOETTER P,HAUF J,et al.Production of the aroma chemicals 3-(methylthio)-1-propanol and 3-(methylthio)-propylacetate with yeasts[J].Appl Microbiol Biot,2008,80(4):579-587.
[25]DEED R C,HOU R,KINZURIK M I,et al.The role of yeast ARO8,ARO9 and ARO10 genes in the biosynthesis of 3-(methylthio)-1-propanol from L-methionine during fermentation in synthetic grape medium[J].FEMS Yeast Res,2019,19(2):doi:10.1093/femsyr/foy109.
[26]刘丽,尹胜,梁婧如,等. Saccharomyces cerevisiae氨基转移酶基因ARO8克隆及对3甲硫基丙醇合成的影响[J].食品工业科技,2014,35(9):132-135.
[27] YIN S,LANG T D,XIAO X,et al.Significant enhancement of methionol production by co-expression of the aminotransferase gene ARO8 and the decarboxylase gene ARO10 in Saccharomyces cerevisiae[J].FEMS Microbiol Lett,2015,362(5):https://doi.org/10.1093/femsle/fnu043.
[28]杨雪莲,张凯丽,李金玉,等.酿酒酵母工程菌产3-甲硫基丙醇的发酵条件优化[J].河北农业大学学报,2014,37(1):94-101.
[29]杨雪莲,张凯丽,李金玉,等.响应面法优化酿酒酵母产3-甲硫基丙醇发酵条件[J].河北大学学报(自然科学版),2014,34(4):398-404.
[30]刘丽,张婵,梁婧如,等.CYS3基因的敲除及其对Saccharomy cescerevisiae 3-甲硫基丙醇合成代谢的影响[J].食品科学,2014,35(5):139-143.
[31]CHE Y X,YIN S,WANG H,et al.Production of methionol from 3-methylthiopropionaldehyde by catalysis of the yeast alcohol dehydrogenase Adh4p[J].J Agr Food Chem,2020,68(16):4650-4656.
[32]温明显. ARO9、ARO10基因过量表达对Saccharomyces cerevisiae产3-甲硫基丙醇代谢调控的影响[D].新疆:石河子大学,2012.
[33]张小溪.CYS3基因敲除对Saccharomyces cerevisiae产3-甲硫基丙醇代谢调控的影响[D].北京:北京工商大学,2013.
[34] BUWALDA R J,ELSTON P D,JOHNSTON K,et al.Dairy product and process,EP1482780 A1[P].2004.
[35]PERPETE P,DUTHOIT O,De MAEYER S,et al.Methionine catabolism in Saccharomyces cerevisiae[J].FEMS Yeast Res,2006,6(1):48-56.
[36] CHOLET O,HENAUT A,CASAREGOLA S,et al.Gene expression and biochemical analysis of cheese-rinening yeasts:Focus on catabolism of L-methionine,lactate,and lactose[J]. Appl Environ Microb,2007,73(8):2561-2570.
[37]刘芳志,张翠英,李维,等.BAT基因改造对酿酒酵母高级醇生成量的影响[J].现代食品科技,2016,32(6):142-147.
[38]徐佳,黄雪芹,杨建飞,等.酿酒酵母中BAT2基因敲除对桑葚酒中高级醇产生的影响[J].食品与发酵工业,2021:47(19):133-139.
[39] ZHANG C Y,QI Y N,MA H X,et al.Decreased production of higher alcohols by Saccharomyces cerevisiae for Chinese rice wine fermentation by deletion of bat aminotransferases[J].J Ind Microbiol Biot,2015,42(4):617-625.
[40]COLON M,HERNANDEZ F,LOPEZ K,et al.Saccharomyces cerevisiae Bat1 and Bat2 aminotransferases have functionally diverged from the ancestral-like Kluyveromyces lactis orthologous enzyme[J].PLoS One,2011,6(1).
[41] IRAQUI I,VISSERS S,CARTIAUX M,et al.Characterisation of Saccharomyces cerevisiae ARO8 and ARO9 genes encoding aromatic aminotransferases I and II reveals a new aminotransferase subfamily[J].Molecul General Genet,1998,257(2):238-248.
[42] IRAQUI I,VISSERS S,ANDRE B,et al.Transcriptional induction by aromatic amino acids in Saccharomyces cerevisiae[J]. Mol Cell Biol,1999,19(5):3360-3371.
[43] URRESTARAZU A,VISSERS S,IRAQUI I,et al.Phenylalanine-and tyrosine-auxotrophic mutants of Saccharomyces cerevisiae impaired in transamination[J].Mol General Genet,1998,257(2):230-237.
[44] GHAEMMAGHAMI S,HUH W,BOWER K,et al.Global analysis of protein expression in yeast[J].Nature,2003,425(6959):737-741.
[45]PIRKOV I,NORBECK J,GUSTAFSSON L,et al.A complete inventory of all enzymes in the eukaryotic methionine salvage pathway[J].FEBS J,2008,275(16):4111-4120.
[46] WU Y P,ZHA M S,YIN S,et al.Novel method for l-methionine production catalyzed by the aminotransferase ARO8 from Saccharomyces cerevisiae[J].J Agr Food Chem,2018,66(24):6116-6122.
[47] TODA Y,NAKAGITA T,HIROKAWA T,et al.Positive/Negative allosteric modulation switching in an umami taste receptor (T1R1/T1R3)by a natural flavor compound,methional[J]. Sci Rep-Uk,2018,8(1):https://doi.org/10.1038/s41598-018-30315-x.
[48]VURALHAN Z,LUTTIK M,TAI S L,et al.Physiological characterization of the ARO10-dependent,broad-substrate-specificity 2-oxo acid decarboxylase activity of Saccharomyces cerevisiae[J]. Appl Environ Microb,2005,71(6):3276-3284.
[49]VURALHAN Z,MORAIS M A,TAI S L,et al.Identification and characterization of phenylpyruvate decarboxylase genes in Saccharomyces cerevisiae[J].Appl Environ Microb,2003,69(8):4534-4541.
[50]ROMAGNOLI G,LUTTIK M A H,KOETTER P,et al.Substrate specificity of thiamine pyrophosphate-dependent 2-oxo-acid decarboxylases in Saccharomyces cerevisiae[J].Appl Environ Microb,2012,78(21):7538-7548.
[51]TAI S L,BOER V M,DARAN-LAPUJADE P,et al.Two-dimensional transcriptome analysis in chemostat cultures-Combinatorial effects of oxygen availability and macronutrient limitation in Saccharomyces cerevisiae[J].J Biol Chem,2005,280(1):437-447.
[52]AGARWAL P K,UPPADA V,NORONHA S B.Comparison of pyruvate decarboxylases from Saccharomyces cerevisiae and Komagataella pastoris(Pichia pastoris)[J].Appl Microbiol Biot,2013,97(21):9439-9449.
[53] BOER V M,TAI S L,VURALHAN Z,et al.Transcriptional responses of Saccharomyces cerevisiae to preferred and nonpreferred nitrogen sources in glucose-limited chemostat cultures[J]. FEMS Yeast Res,2007,7(4):604-620.
[54]VALLET A,SANTARELLI X,LONVAUD-FUNEL A,et al.Purification of an alcohol dehydrogenase involved in the conversion of methional to methionol in Oenococcus oeni IOEB 8406[J]. Appl Microbiol Biot,2009,82(1):87-94.
[55] DICKINSON J R,ESHANTHA L,SALGADO J,et al.The catabolism of amino acids to long chain and complex alcohols in Saccharomyces cerevisiae[J].J Biol Chem,2003,278(10):8028-8034.
[56]KNOLL C,DU TOIT M,SCHNELL S,et al.Cloning and characterisation of a cystathionine beta/gamma-lyase from two Oenococcus oeni oenological strains[J].Appl Microbiol Biot,2011,89(4):1051-1060.
[57]JIA K Z,ZHANG Q,SUN L Y,et al. Clonostachys rosea demethiolase STR3 controls the conversion of methionine into methanethiol[J]. Sci Rep,2016,6(1):21920.
[58]ZHANG Q,JIA K Z,XIA S T,et al.Regulating Ehrlich and demethiolation pathways for alcohols production by the expression of ubiquitinprotein ligase gene HUWE1[J].Sci Rep,2016,6(1):20828.