酸粥作为我国内蒙地区一种传统的发酵淀粉制品,已有数千年历史,因其特殊的发酵工艺和营养价值,以及特有的酸滑口感而深得消费者的喜爱[1]。近年来,越来越多的研究者针对酸粥微生物多样性进行了研究。王玉荣等[2]对内蒙古地区3份酸粥样本中的细菌多样性进行解析发现,优势菌属为乳酸杆菌(Lactobacillus)和醋酸杆菌(Ace tobacter),通过传统微生物学手段共分离到9株乳酸菌,分别为发酵乳杆菌(Lactobacilus fermentum)、植物乳杆菌(Lactobacillus plantarum)和卷曲乳杆菌(Lactobacillus crispatus);薛建岗等[3]对内蒙古西部地区自然发酵酸粥中微生物组成进行分析发现,其含有较高的乳酸菌属。前人的研究为解析微生物的多样性提供一定的参考依据,而这些研究大多采用传统的生物学手段,并不能准确揭示酸粥中的物种构成和多样性,因此,采用分子生物学技术手段对酸粥中的细菌多样性进行研究显得尤为必要。
随着测序技术的快速发展,以Illumina MiSeq为代表的第二代测序技术已经广泛应用于传统发酵食品的微生物多样性解析中[4],其不仅准确性高,还可以多样品平行测序,提升测序速度。贾丽艳等[5]采用Illumina MiSeq测序技术解析了传统清香型白酒发酵过程中细菌群落的结构和动态演替,为改善白酒风味提供了依据;王玉荣等[6]采用此技术解析了当阳鲊广椒中的细菌多样性,发现乳酸杆菌能改善鲊广椒的风味品质。Illumina MiSeq测序技术的广泛应用也为解析酸粥中的细菌多样性提供了有效的技术手段。
本研究采用Illumina MiSeq高通量测序技术对采集自内蒙古巴彦淖尔地区的10份酸粥样品中的细菌多样性进行解析,同时结合多元统计学和基因预测等手段对酸粥中细菌菌群结构和细菌功能及表型进行比较研究,为开发优质的乳酸菌资源提供一定的参考依据。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
酸粥:采集自内蒙古自治区巴彦淖尔市的农户家中,分别从10户农户家中共采集10个样本,分别编号为BM1~BM10。
1.1.2 试剂
食品基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒:德国QIAGEN公司;脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs)Mix、FastPfu Fly DNA Polymerase、5×TransStartTM FastPfu Buffer:北京全式金生物技术有限公司。
1.2 仪器与设备
R930机架式服务器:美国Dell公司;IlluminaMiSeqPE250高通量测序平台:美国Illumina公司;CR21N高速离心机:日本日立公司;vetiri梯度基因扩增仪:美国AB公司;ND-2000C微量紫外分光光度计:美国Nano Drop公司。
1.3 试验方法
1.3.1 酸粥样品微生物宏基因组DNA提取
称取3.0 g酸粥样品,使用食品基因组DNA提取试剂盒对酸粥样品中的微生物宏基因组DNA进行提取。
1.3.2 细菌16S rRNA V3-V4区PCR扩增及测序
以提取的宏基因组为模板,使用包含不同核苷酸标签(barcode)的引物338F/806R,对提取出的宏基因组DNA的16S rRNA V3-V4区序列进行聚合酶链式反应(polymerase chain reaction,PCR)扩增[2]。
使用琼脂糖凝胶电泳和紫外分光光度计对PCR扩增产物的质量进行检验,并将检验合格的扩增产物送至上海美吉生物医药科技有限公司,使用Illumina MiSeq PE250高通量测序平台进行测序。
1.3.3 生物信息学分析
参照郭壮等[7]对窖泥微生物多样性研究中序列质控的参数对下机数据进行质控,并依照barcode信息进行归类,将序列分发到不同的样本中。
利用QIIME v1.70平台对酸粥中的细菌结构和多样性进行解析[8]。具体的生物信息分析过程:(1)使用PyNAST软件[9]对高质量序列进行校准和比对;(2)通过100%相似度建立非冗余的序列集后,按97%的相似度构建操作分类单元(operational taxonomic units,OTU);(3)使用ChimeraSlayer软件[10]对OTU中的嵌合体序列进行识别和删除;(4)基于Greengenes v13.8[11]、RDP v11.5[12]和SILVA v128[13]数据库对每个OTU的代表性序列进行注释;(5)使用FastTree软件构建基于代表性序列的系统发育树;(6)计算α多样性指数(超1(Chao1)指数和辛普森指数)和β多样性,以评估每个酸粥样品中微生物物种的丰度和多样性。
基于Greengenes数据库对序列进行划分和注释,使用PICRUSt软件[14]和BugBase在线网站(https://bugbase.cs.umn.edu/)[15]对酸粥中微生物的基因功能和表型进行预测,同时参照蛋白质直系同源簇数据库进行基因功能预测。
1.3.4 多元统计学分析
基于加权和非加权的主成分分析(principal component analysis,PCA)对酸粥的菌群结构进行分析;使用围绕中心点的划分(partitioning around medoid,PAM)对酸粥的属水平结构进行分析;使用Wilcoxon test对不同分组酸粥中细菌的基因功能和表型进行显著性分析;使用R软件进行矩阵数据的分析和绘图;使用Origin 2019b软件进行数据的可视化。
2 结果与分析
2.1 酸粥中细菌菌群丰富度和多样性分析
采用高通量测序技术对酸粥样品中细菌菌群的丰富度和多样性进行分析,结果见表1。
表1 酸粥样品细菌菌群丰富度及多样性分析
Table 1 Analysis of richness and diversity of bacterial community in sour porridge samples
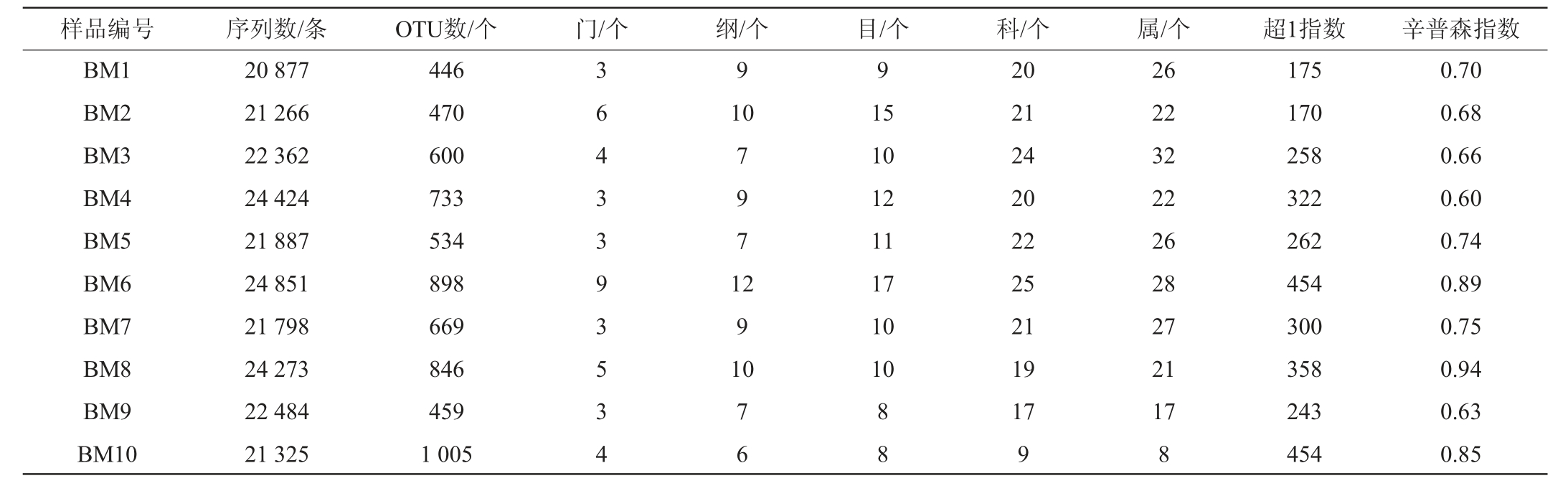
由表1可知,所有样本共产生225 547条高质量序列,平均每个样本22 555条。按97%的相似度进行OTU的划分后共得到6 681个,删除21个嵌合体OTU后平均每个样本666个OTU。由表1亦可知,10份酸粥样品间的OTU数和α多样性指数存在较大差异,其中样品BM10的OTU数和超1指数最大,而样品BM8的辛普森指数最大,辛普森指数和超1指数常用来评价环境样本中微生物的多样性和丰度[16]。由此可见,样品BM8中细菌的多样性最高,样品BM10中微生物的丰度最高,且同一地区酸粥样本中的细菌微生物的多样性和丰度之间存在着明显的差异。
2.2 酸粥中细菌菌群结构分析
从10份酸粥样本中共检测出9个细菌门和55个细菌属,其中优势细菌门和优势细菌属相对含量的比较见图1。
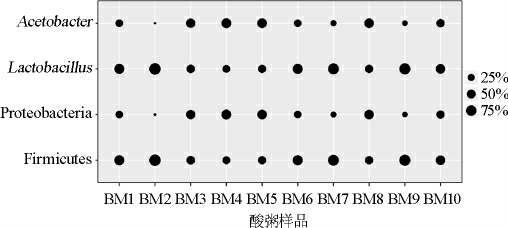
图1 酸粥样品中优势菌门和优势菌属相对含量的比较分析
Fig.1 Comparison and analysis of the relative contents of dominant phylum and genus in sour porridge samples
由图1可知,10份酸粥样本中有2个优势菌门(平均相对含量>1%),分别为硬壁菌门(Firmicutes)和变形菌门(Proteobacteria),平均相对含量分别为61.91%和37.33%;优势菌属(平均相对含量>1%)亦有2个,分别为乳杆菌属(Lactobacillus)和醋杆菌属(Acetobacter),平均相对含量分别为61.37%和37.22%,两者累计平均相对含量为98.59%。由此说明,酸粥中的细菌几乎均为产酸菌。王玉荣等[6]对内蒙古乌拉特前旗、杭锦后旗和准格尔旗地区酸粥的细菌多样性进行研究发现,其优势菌门和优势菌属与本研究一致,但其平均相对含量与本研究的结果存在一定的差异。酸粥中主要以乳酸杆菌和醋酸杆菌为主,但其不同农户家制作的酸粥中细菌菌群之间也存在着一定的差异。由此说明,两者在酸粥的发酵过程中有着至关重要的作用,而不同的制作方法和制作环境也可能直接或间接影响着酸粥中的菌群构成[17-18]。
乳酸菌作为现代食品加工产业中常用的发酵菌株之一,在发酵蔬菜制品和发酵乳制品等方面应用广泛,其具有应用范围广和经济价值高等特点。研究人员围绕乳酸菌开展了许多卓有成效的研究,HUANG L等[19]研究发现一株分离于中国传统食品豆腐中的植物乳杆菌(Lactobacillus plantarum)C88能抑制黄曲霉毒素B1,而KHAN I等[20]研究表明,植物乳杆菌DGK-17能够明显改善韩国泡菜的营养品质,说明乳杆菌属具有改善发酵制品品质的潜力。醋杆菌属亦广泛存在发酵食品中[21],其产生的乙酸和葡萄糖酸等产物能显著改善产品的品质。目前,产业化中应用的乳酸菌菌株大多来自于发酵乳制品和发酵蔬菜制品,通过对酸粥中的乳酸菌进行相关研究,进一步对乳酸菌进行收集,以期对后续乳酸菌遗传多样性研究和产业化推动具有积极的意义。
进一步对酸粥样本中OTU出现的频率和平均相对含量进行分析,以便于对酸粥样品中的核心细菌类群进行研究,结果见图2。
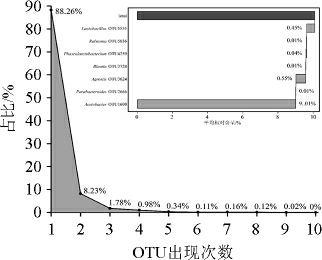
图2 酸粥样本中OTU的出现次数和共有OTU
Fig.2 Occurrence times and common OTU in sour porridge samples
由图2可知,独有OTU数(仅在一个样品中出现)占总数的88.26%,拥有序列数3 900条,占全部序列数的1.81%。而在所有样品中出现的OTU数为0。由图2亦可知,酸粥样品中共有OTU(在80%的样品中出现)数有6个,其所包含的序列数占全部序列数的10.08%,其中醋杆菌属、农杆菌属(Agrostis)和乳杆菌属的平均相对含量分别为9.01%、0.55%和0.45%。由此说明,虽然酸粥样本中存在大量的核心菌群,但每份样本中均存在许多独特的细菌类型。酸粥中乳杆菌属平均相对含量>60%,但其共有OTU的比例<0.50%,说明酸粥中隶属于乳杆菌属的细菌在种或株水平上存较大的差异。
2.3 酸粥中细菌菌群结构的比较
为进一步研究酸粥中细菌菌群结构在整体上的差异,基于非加权和加权的UniFrac距离对酸粥中的菌群结构进行主成分分析,结果见图3。
由图3可知,酸粥在两份坐标轴的空间排布并不相同,图3a坐标轴中的样本在空间排布上较为分散,而图3b坐标轴中的样本在空间排布上呈现明显的聚类趋势。由此说明,酸粥中存在着许多低丰度物种,且不同样本中低丰度物种之间存在着较大差异,而不同酸粥样本中优势菌的差异却相对较小。
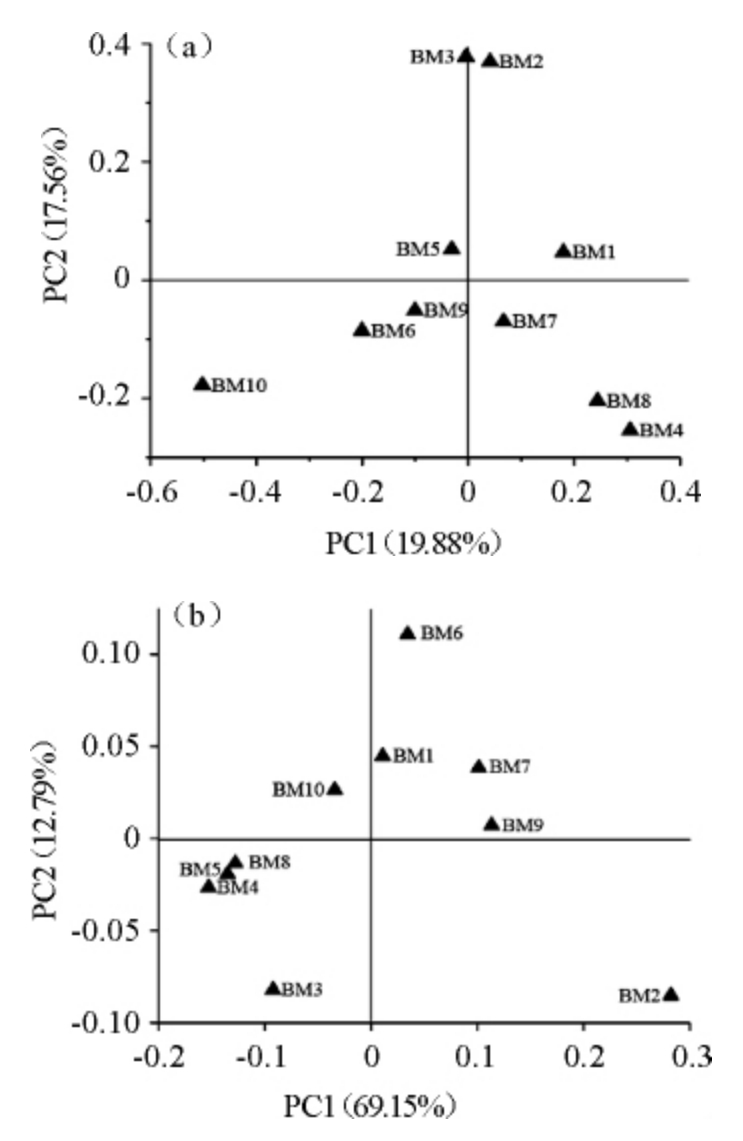
图3 基于非加权(a)和加权(b)的UniFrac距离酸粥样品中细菌菌群结构主成分分析
Fig.3 Principal component analysis of bacterial community structure in sour porridge samples based on unweighted (a) and weighted(b) UniFrac distance
为进一步探究不同酸粥样本在属水平上的差异,本研究基于属水平对10份酸粥样品进行PAM分析,结果见图4。
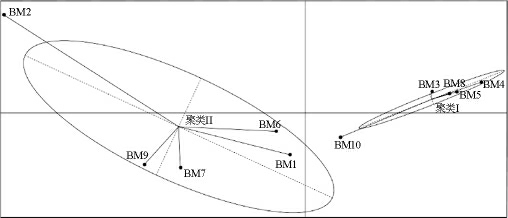
图4 基于细菌属水平酸粥样品的围绕中心点的划分分析
Fig.4 Partitioning around medoid analysis of sour porridge samples based on bacterial genera level
由图4可知,10份酸粥样品被分为2个聚类,其中样品BM3、BM4、BM5、BM8和BM10隶属于聚类I,而样品BM1、BM2、BM6、BM7和BM9隶属于聚类II。对比不同聚类中细菌属的相对含量发现,聚类I以醋酸菌为主(Acetobacter与Lactobacillus的平均比值为1.31),而聚类II以乳杆菌为主(Lactobacillus与Acetobacter的平均比值为4.39)。由此说明,不同聚类中醋杆菌属和乳杆菌属的相对含量差异较大,对酸粥中菌群的整体结构具有较大的影响。
2.4 酸粥中细菌的功能预测和表型预测
以PAM分析的结果为分组依据,进一步对酸粥中细菌的基因功能和表型进行预测和差异化分析。酸粥中细菌一级功能层的比较分析见图5。
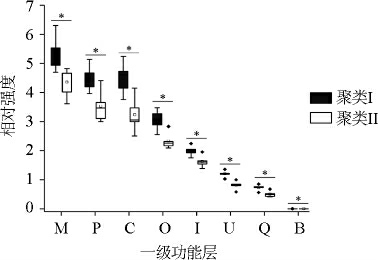
图5 酸粥样品中细菌一级功能层相对丰度的箱型图
Fig.5 Box diagram of the relative abundance of first-level functional layer of bacteria in sour porridge samples
B为染色质结构与动力学;C为能量生产和转换;I为脂质转运与代谢;M为细胞壁/膜/包膜生物发生;O为翻译后修饰,蛋白质周转,伴侣;P为无机离子运输与代谢;Q为次生代谢产物的合成.转运和分解代谢;U为细胞内运输,分泌和囊泡运输。菱形表示每组数据中的特异值大小;正方形表示每组数据的平均值;“*”表示差异显著(P<0.05)。
由图5可知,所有的一级功能层中,聚类I与聚类II的8个一级功能层具有显著性差异(P<0.05),且均在聚类I中显著富集。聚类I的样品中有更多的序列与物质的转运代谢和能量的生产转换相关。由此说明,聚类I样品中细菌菌群对于碳水化合物的利用更加频繁,细菌的生长繁殖更加快速,这可能有利于酸粥的发酵。酸粥中细菌的表型预测结果见图6。
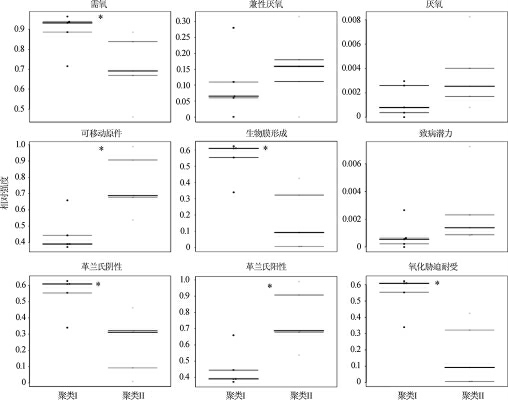
图6 酸粥样品中细菌表型的比较分析
Fig.6 Comparison and analysis of bacterial phenotypes in sour porridge samples
“*”表示差异显著(P<0.05)。
由图6可知,在9种细菌表型中,聚类I与聚类II有6种表型存在显著性差异(P<0.05),分别为需氧、可移动原件、生物膜的形成、革兰氏阴性、革兰氏阳性和氧化胁迫耐受,其中需氧、生物膜的形成、革兰氏阴性和氧化胁迫耐受在聚类I中的强度显著较高(P<0.05),而其他显著表型呈现相反趋势。由此说明,与聚类II相比,聚类I酸粥样品中革兰氏阴性菌的繁殖更加强烈,即醋杆菌属(革兰氏阴性菌、专性好氧菌)在聚类I中具有更强的繁殖能力。
3 结论
内蒙古自治区巴彦淖尔盟酸粥样品中的细菌隶属于9个细菌门的55个属,样品间细菌的多样性和丰度存在明显差异,但优势细菌门均为硬壁菌门(Firmicutes)和变形菌门(Proteobacteria),两者累计占比为99%;优势细菌属均为乳杆菌属(Lactobacillus)和醋杆菌属(Acetobacter),两者累计占比为98.59%。所有酸粥样品可分为两个聚类,聚类I以醋杆菌属为主,聚类II以乳杆菌属为主。基因预测结果表明,聚类I样品中细菌菌群对于碳水化合物的利用更加频繁,细菌的生长繁殖更加快速,而表型预测结果亦表明聚类I中样品中革兰氏阴性菌的繁殖更加强烈,即醋杆菌属在聚类I中具有更强的繁殖能力。
[1]秦慧彬,黄志伟,张志强,等.传统食品酸粥的发酵工艺[J].食品与发酵工业,2019,45(17):111-116.
[2]王玉荣,折米娜,刘康玲,等.基于MiSeq 高通量测序技术内蒙古地区酸粥细菌多样性研究[J].食品工业科技,2018,39(19):124-129.
[3]薛建岗,陈永福,于海静,等.内蒙古西部地区自然发酵酸粥化学成分及微生物组成分析[J].食品科技,2013,38(7):10-14.
[4]LIANG H,YIN L,ZHANG Y,et al.Dynamics and diversity of a microbial community during the fermentation of industrialized Qingcaipaocai,a traditional Chinese fermented vegetable food,as assessed by Illumina MiSeq sequencing,DGGE and qPCR assay[J].Ann Microbiol,2018,68(2):111-122.
[5]贾丽艳,荆旭,田宇敏,等.传统清香型白酒发酵过程中细菌群落结构及其动态演替[J].中国食品学报,2020,20(2):196-204.
[6]王玉荣,沈馨,董蕴,等.鲊广椒细菌多样性评价及其对风味的影响[J].食品与机械,2018,34(4):25-30.
[7]郭壮,葛东颖,尚雪娇,等.退化和正常窖泥微生物多样性的比较分析[J].食品工业科技,2018,39(22):93-98.
[8] YANG C,ZHAO F,HOU Q,et al.PacBio sequencing reveals bacterial community diversity in cheeses collected from different regions[J].J Dairy Sci,2020,103(2):1238-1249.
[9] CAPORASO J G,BITTINGER K,BUSHMAN F D,et al.PyNAST:a flexible tool for aligning sequences to a template alignment[J].Bioinformatics,2010,26(2):266-267.
[10] EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[11] EDGAR R C,HAAS B J,CLEMENTE J C,et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194-2200.
[12] DESANTIS T Z,HUGENHOLTZ P,LARSEN N,et al.Greengenes,a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Appl Environ Microbiol,2006,72(7):5069-5072.
[13] MAIDAK B L,COLE J R,LILBURN T G,et al.The RDP (ribosomal database project)continues[J].Nucl Acid Res,2000,28(1):173-174.
[14] WILKINSON T J,HUWS S A,EDWARDS J E,et al.CowPI:a rumen microbiome focussed version of the PICRUSt functional inference software[J].Front Microbiol,2018,9(5):1095.
[15]WARD T,LARSON J,MEULEMANS J,et al.BugBase predicts organism level microbiome phenotypes[J].Bio Rxiv,2017,5(2):133462.
[16]姚国强.传统发酵乳中细菌多样性及其功能基因研究[D].呼和浩特:内蒙古农业大学,2017.
[17] ASHANG U J,ANTAI S P,TIKU D R.Distribution of antibiotic resistant bacteria flora in ready to-eat food samples sold in Calabar Metropolis[J].World J Pharmaceut Med Res,2017,3(11):198-125.
[18]王鑫毅,谢骁,金珊,等.基于高通量测序的缢蛏及其养殖池塘菌群结构的季节变化[J].应用生态学报,2019,30(12):4267-4276.
[19]HUANG L,DUAN C,ZHAO Y,et al.Reduction of aflatoxin B1 toxicity by Lactobacillus plantarum C88:a potential probiotic strain isolated from Chinese traditional fermented food "tofu"[J].PloS One,2017,12(1):e0170109.
[20]KHAN I,KANG S C.Probiotic potential of nutritionally improved Lactobacillus plantarum DGK-17 isolated from Kimchi-A traditional Korean fermented food[J].Food Control,2016,60(2):88-94.
[21] MOUNIR M,SHAFIEI R,ZARMEHRKHORSHID R,et al.Simultaneous production of acetic and gluconic acids by a thermotolerant Acetobacter strain during acetous fermentation in a bioreactor[J].J Biosci Bioeng,2016,121(2):166-171.