刺梨(Rosa roxburghii)是一种广泛分布于我国西南地区的蔷薇科、蔷薇属植物,具有丰富的营养价值和药用价值[1-2]。但刺梨果实中酚类物质含量较高,鲜果生食口感酸涩,比较适宜进行精深加工,如发酵生产刺梨果酒、果醋等饮品[3-4]。不同种类微生物的代谢活动,均影响着酿造酒的质量[5],这些微生物包括酵母菌、霉菌、细菌等[6-8]。研究表明,在果实的自然发酵过程中,除了酵母菌,醋酸杆菌属(Acetobacter)、乳杆菌属(Lactobacillus)、葡糖杆菌属(Gluconobacter)等细菌也会对发酵过程产生重要影响。如乳酸菌的异常代谢,可产生多种挥发酸;醋酸菌的甘油代谢、糖代谢和酒精的醋化,都会引起葡萄酒的病害[9-12];葡糖杆菌属细菌可产生乙酸,影响葡萄酒的感官品质[13]。谢丹等[14]采用高通量测序技术分析了刺梨果渣自然发酵过程中细菌群落结构及多样性。张源等[15]基于高通量测序分析了草莓酒发酵过程中的细菌群落特征,发现欧文氏菌属(Erwinia)、柠檬酸菌属(Citrobacter)、乳酸球菌属(Lactococcus)、泛菌属(Pantoea)以及未知属细菌在草莓酒的发酵过程中动态变化,且与酒体特征相关。本研究采用高通量测序技术,解析刺梨自然发酵过程中细菌菌群多样性及其动态变化,对优质刺梨酿造菌种的选育及控制发酵酒的品质具有重要的指导意义。
1 材料与方法
1.1 材料与试剂
1.1.1 原料
刺梨(Rosa roxburghii),品种为贵龙5号,采购于2019年9月份贵州龙里地区,平均单果质量(22.26±0.82)g,糖度(9.89±0.23)°Bx,pH值为3.58±0.11。
1.1.2 化学试剂
脱氧核糖核酸(deoxyribonucleic acid,DNA)抽提试剂盒:美国MP Biomedicals公司;Pfu高保真DNA聚合酶(100 U/管):北京TransGen Biotech有限公司;琼脂糖凝胶:西班牙Biowest公司;DNA Marker:生工生物工程(上海)股份有限公司;引物338F(5'-ACTCCTACGGGAGGCAGCAG-3')、806R(5'-GGACTACHVGGGTWTCTAAT-3'):上海美吉生物科技有限公司合成。
1.2 仪器与设备
N13462C移液器、5430 R小型离心机:德国Eppendorf公司;ABSON MiFly-6微型离心机:合肥艾本森科学仪器有限公司;NanoDrop2000超微量分光光度计:美国Thermo Fisher Scientific公司;GeneAmpR 9700聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;Illumina Miseq测序仪:美国Illumina公司。
1.3 方法
1.3.1 刺梨自然发酵
取新鲜、成熟、无霉烂的贵农5号刺梨,用无菌水冲洗果实表面泥沙,切碎,置于无菌1 L三角瓶中。28 ℃进行静置自然发酵,在自然发酵的1 d、3 d、5 d、15 d分别取样1 mL,命名为F1、F3、F5、F15,每个样本3个平行重复,保存于-80 ℃冰箱用于高通量测序。
1.3.2 高通量测序与生物信息学分析[16-18]
按照试剂盒说明书提取刺梨自然发酵液中细胞基因组脱氧核糖核酸(DNA),利用NanoDrop2000进行DNA纯度与浓度检测,琼脂糖凝胶电泳进行DNA完整性检测。取适量的DNA模板,以338F和806R为引物进行PCR扩增,扩增细菌16S rRNA基因,PCR扩增体系为5×FastPfu缓冲液4 μL,2.5 mmol/L脱氧核苷三磷酸(deoxyribonucleoside triphos phate,dNTPs)2 μL,5 μmol/L 338F引物0.8 μL,5 μmol/L 806R引物0.8 μL,FastPfu聚合酶0.4 μL,DNA模板10 ng,补水至20 μL。PCR扩增条件为:95 ℃、3 min;95 ℃、30 s,55 ℃、30 s,72 ℃、45 s,共30个循环;72 ℃再延伸10 min。PCR产物经琼脂糖凝胶检测后,进行回收和纯化。文库的构建与Miseq测序由深圳微生太科技有限公司完成。
Miseq测序得到的序列,在深圳微生太平台上进行数据的生物信息学分析。首先,根据取样的时间和方式,把测序数据的样本分为自然发酵1 d样本(F1)、自然发酵3 d样本(F3)、自然发酵5 d样本(F5)和自然发酵15 d样本(F15)共4组,每组3个平行重复。然后,按照相似度为97%,按照最小样本序列数抽评样本序列,运用QIIME2 dada2插件对测序原始数据进行质量控制,修剪,去噪,拼接及去除嵌合体。运用QIIME2 feature-classifier插件将代表序列与GREENGENES数据库进行比对,获得物种分类信息表。采用QIIME2 core-diversity插件计算Alpha多样性指数。采用PCoA和NMDS图展示Beta多样性指数。基于样本中主要微生物物种相对丰度分析,采用共现网络分析(co-occurrence analysis)计算斯皮尔曼等级相关系数,从而了解物种之间的关联。PICRUSt软件预测微生物群体可能的功能组成[19-20]。
2 结果与分析
2.1 琼脂糖凝胶电泳鉴定
刺梨自然发酵不同阶段的样品中细菌16S rRNA PCR扩增产物琼脂糖凝胶电泳结果见图1。由图1可知,刺梨自然发酵4个阶段(F1、F3、F5和F15)12个样本的PCR扩增产物条带特异性和亮度均较好,可满足下一步的测序需求。
图1 刺梨自然发酵液样品中细菌群落16S rDNA PCR扩增产物琼脂糖凝胶电泳图
Fig. 1 Agarose gel electrophoresis of 16S rDNA PCR amplified product of bacterial community in nature fermentation liquid samples of Rosa roxburghii
2.2 测序样本数据分析
刺梨自然发酵液4个阶段的样本,经过测序样本数据分析,结果见表1。由表1可知,共得到230 692条读数,经质量控制(过滤、去噪、去嵌合体)后共得到150 385条有效序列,序列的平均碱基长度约500 bp。
表1 刺梨自然发酵液样品测序样本读数结果
Table 1 Results of sequencing sample reads of nature fermentation liquid samples of Rosa roxburghii
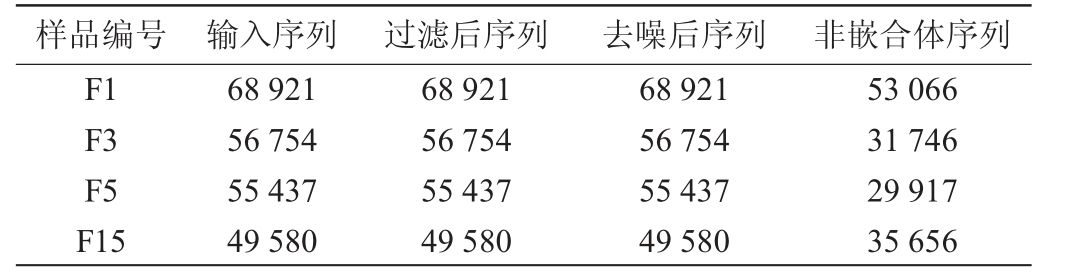
样品编号 输入序列 过滤后序列 去噪后序列 非嵌合体序列F1 F3 F5 F15 68 921 56 754 55 437 49 580 68 921 56 754 55 437 49 580 68 921 56 754 55 437 49 580 53 066 31 746 29 917 35 656
2.3 Alpha多样性分析
稀释曲线可用于验证测序数据量是否满足反映样品中物种多样性的需求,刺梨自然发酵液样品的稀释曲线见图2。由图2可知,刺梨自然发酵液4个样品细菌操作分类单元(operational taxonomic units,OTU)图平缓性较好,能够反映样本中绝大部分细菌菌落特征和多样性信息。稀释曲线还可以间接反映样品中物种的丰富度,结果表明,F1样品中细菌多样性最大,4个样品中细菌丰富度程度大小为F1>F15>F3>F5。
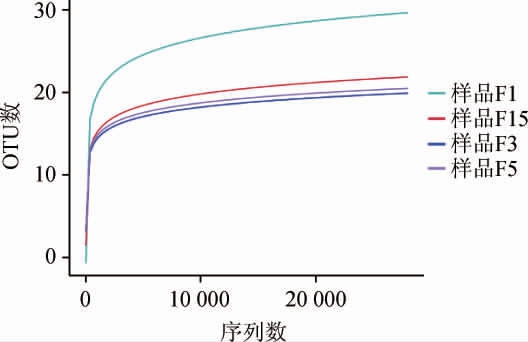
图2 刺梨自然发酵液样品稀释曲线
Fig. 2 Dilution curve of nature fermentation liquid samples of Rosa roxburghii
Shannon指数可综合衡量样本中的物种多样性,通常Shannon指数数值越大,表示样本中物种的多样性越高,反之,则样本中物种多样性越低。刺梨自然发酵样品Alpha多样性指数指数结果见表2。
表2 刺梨自然发酵液样品Alpha多样性指数
Table 2 Alpha diversity indexes of nature fermentation liquid samples of Rosa roxburghii
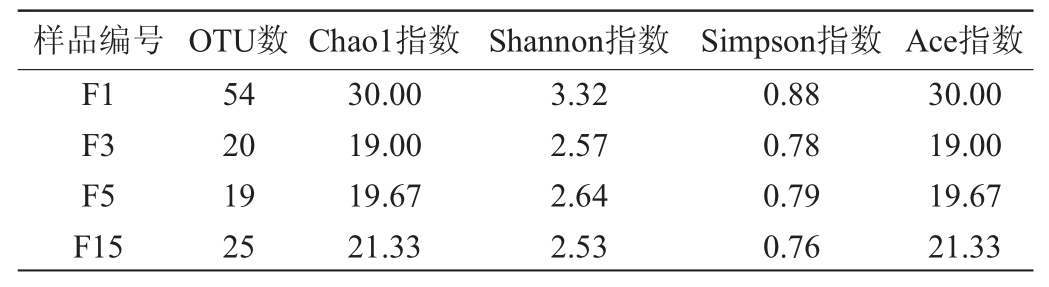
样品编号 OTU数Chao1指数 Shannon指数 Simpson指数 Ace指数F1 F3 F5 F15 54 20 19 25 30.00 19.00 19.67 21.33 3.32 2.57 2.64 2.53 0.88 0.78 0.79 0.76 30.00 19.00 19.67 21.33
由表2可知,样品F1的Shannon指数和OTU数均最大,说明样本F1中物种多样性最丰富。比较4个样本的Shannon指数和OTU数可以发现,随着刺梨自然发酵的进行,样品中细菌多样性表现出先逐渐降低,再增加的趋势。Ace指数和Chao1指数也可用来衡量样本中的物种的种类。Ace指数和Chao1指数越大,则样本中物种种类越多。由表2可知,在刺梨自然发酵样本中,样本F1的Ace指数和Chao指数均最大。
2.4 物种组成
基于OTU的绝对丰度及注释信息,对每个样品在界、门、纲、目、科、属、种7个分类水平上的序列数目占总序列数的比例进行统计,可以有效的评估样本的物种注释分辨率。每个样本中OTU在各分类水平注释的相对程度结果见图3。刺梨发酵液各样本细菌菌群属水平分布Venn图见图4,属水平细菌菌群相对丰度见图5。
由图3可知,在门水平上,刺梨自然发酵过程中共检测到变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、绿弯菌门(Chloroflexi)、蓝细菌门(Cyanobacteria)等8个门的细菌类,优势细菌门为Proteobacteria,占98.66%;在刺梨自然发酵的前期,包括样品F1、F3和F5中基本上未检测到厚壁菌门(Firmicutes)菌群,但在发酵后期的样品F15中,厚壁菌门(Firmicutes)的比例达到5.26%,暗示其在刺梨自然发酵的后期发挥作用。
图3 刺梨自然发酵液样品序列注释程度
Fig. 3 Sequence annotation degree of nature fermentation liquid samples of Rosa roxburghii
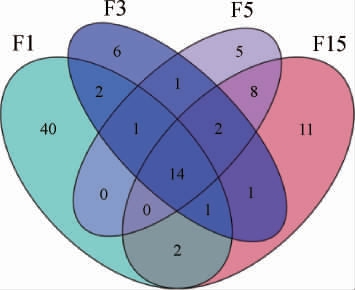
图4 刺梨自然发酵液各样本细菌菌群属水平分布Venn图
Fig. 4 Venn diagram of bacteria community distribution at the genus level of nature fermentation liquid samples of Rosa roxburghii
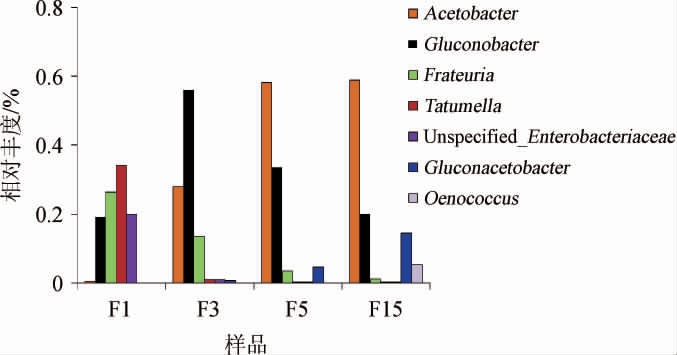
图5 刺梨自然发酵液各样本属水平细菌菌群相对丰度
Fig. 5 Relative abundance of bacteria community of nature fermentation liquid samples of Rosa roxburghii at the genus level
由图4可知,在属水平,样品F1特有属40种,样品F3特有属6种、样品F5特有属5种,样品F15特有属11种;4个样本共有属14种。
由图5可知,随着刺梨发酵的不断进行,葡糖杆菌属细菌(Gluconobacter)表现出先增加后降低的趋势;弗拉托氏菌属(Frateuria)、肠杆菌属(Enterobacteriaceae)、塔特姆菌属(Tatumella)在刺梨发酵过程中含量逐渐降低;而醋酸杆菌属(Acetobacter)和葡糖醋杆菌属(Gluconacetobacter)、酒酒球菌属(Oenococcus)细菌含量在发酵过程中含量不断增加。表明随着刺梨自然发酵的不断进行,参与糖代谢相关菌群量逐渐降低,而参与酒精发酵、乳酸发酵以及醋酸发酵相关菌群量逐渐增加。
2.5 样本比较分析
LEfSe法是非参数检验和线性判别分析(linear discriminant analysis,LDA)的结合,适合菌群丰度差异检验。由图6可知,在刺梨自然发酵的样品F1中,菌群总体丰度较高,且甲基杆菌属(Methylobacterium)、弗拉托氏菌属(Frateuria)、痰塔特姆氏菌(Tatumella)、拉恩氏菌属(Rahnella)、黄单胞菌属(Xanthomonadaceae)、肠杆菌属(Enterobacter)等菌属在样品F1中分布较为特异性;在发酵终点样品F15中,醋酸杆菌属(Acetobacter)菌群丰度较高,较为特异性。
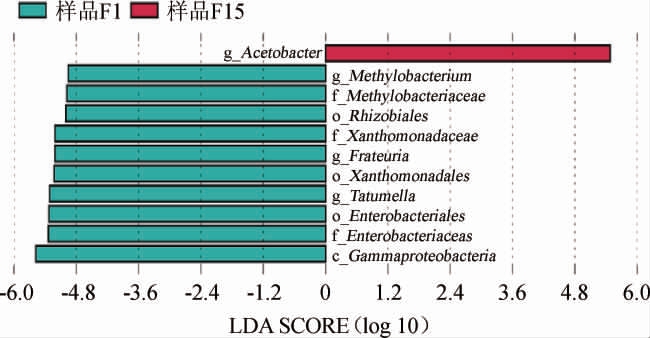
图6 刺梨自然发酵液样品细菌菌群LEfSe法线性判别分析
Fig. 6 Linear discriminant analysis of bacteria community of nature fermentation liquid samples of Rosa roxburghii by LEfSe method
2.6 功能分析
基于KEGG功能注释的方法分析刺梨自然发酵过程中各菌群的功能特性,结果见图7。由图7可知,在发酵过程中,菌群中与细胞处理(Cellular Processes)相关过程和环境信息处理(Environmental Information Processing)过程逐渐降低;而与代谢(Metabolism)相关、遗传信息处理(Genetic Information Processing)过程逐渐增加。
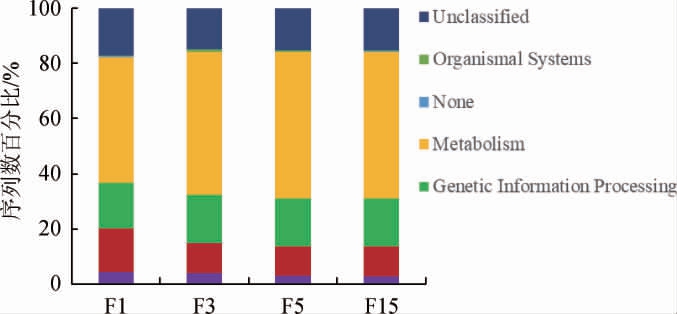
图7 刺梨自然发酵液样本菌群功能分析
Fig. 7 Functional analysis of bacteria community of nature fermentation liquid samples of Rosa roxburghii
3 结论
本研究基于高通量测序技术的研究结果表明,在刺梨发酵的起始阶段微生物多样性最高,随着刺梨果实发酵的不断进行,葡糖杆菌属细菌(Gluconobacter)表现出先增加后降低的趋势;弗拉托氏菌属(Frateuria)、肠杆菌属(Enterobacteriaceae)、塔特姆菌属(Tatumella)细菌在刺梨发酵过程中含量逐渐降低;而醋酸杆菌属(Acetobacter)和葡糖醋杆菌属(Gluconacetobacter)、酒酒球菌属(Oenococcus)细菌含量在发酵过程中含量不断增加。通过高通量技术较为全面的掌握了刺梨自然发酵过程中细菌多样性及其菌群结构变化,为后续进行优质刺梨酿造菌种的选育奠定了基础。
[1] LIU M H, ZHANG Q, ZHANG Y H, et al. Chemical analysis of dietary constituents in Rosa roxburghii and Rosa sterilis fruits[J].Molecules,2016,21(9):1204.
[2]CHEN G,KAN J.Characterization of a novel polysaccharide isolated from Rosa roxburghii Tratt fruit and assessment of its antioxidant in vitro and in vivo[J].Int J Biol Macromol,2018,107:166-174.
[3]姚敏.刺梨果酒技术研究[D].贵阳:贵州大学,2015.
[4]徐廷.刺梨果醋饮料的研制及其发酵过程中的品质变化[D].恩施:湖北民族学院,2018.
[5]TAMANG J P,WATANABE K,HOLZAPFEL W H.Diversity of microorganisms in global fermented foods and beverages[J]. Front Microbiol,2016,7:377.
[6]FLEET G H.Yeast interactions and wine flavour[J].Int J Food Microbiol,2003,86(1-2):11-22.
[7]LIU Y,ROUSSEAUX S,TOURDOT-MARÉCHAL R,et al.Wine microbiome: a dynamic world of microbial interactions[J]. Crit Rev Food Sci Nutr,2017,57(4):856-873.
[8]TEMPÈRE S,MARCHAL A,BARBE J C,et al.The complexity of wine:clarifying the role of microorganisms[J].Appl Microbiol Biot,2018,102(9):3995-4007.
[9]CAPPELLO M S,ZAPPAROLI G,LOGRIECO A,et al.Linking wine lactic acid bacteria diversity with wine aroma and flavour[J].Int J Food Microbiol,2017,243:16-27.
[10]TRISTEZZA M,DI FEO L,TUFARIELLO M,et al.Simultaneous inoculation of yeasts and lactic acid bacteria:Effects on fermentation dynamics and chemical composition of Negroamaro wine[J].LWT-Food Sci Techn,2016,66:406-412.
[11]CAGGIANIELLO G,KLEEREBEZEM M,SPANO G.Exopolysaccharides produced by lactic acid bacteria:from health-promoting benefits to stress tolerance mechanisms[J].Appl Microbiol Biot,2016,100(9):3877-3886.
[12]BARTOWSKY E J,XIA D,GIBSON R L,et al.Spoilage of bottled red wine by acetic acid bacteria[J].Lett Appl Microbiol,2003,36(5):307-314.
[13] JOYEUX A, LAFON-LAFOURCADE S, RIBÉREAU-GAYON P. Evolution of acetic acid bacteria during fermentation and storage of wine[J].Appl Environ Microbiol,1984,48(1):153-156.
[14]谢丹,刘晓燕,毕远林,等.基于高通量测序分析刺梨果渣自然发酵过程中细菌群落结构及多样性[J].食品工业科技,2019,40(22):110-114.
[15]张源,兰伟,王明跃,等.基于高通量测序的草莓酒发酵过程中的细菌群落分析[J].宿州学院学报,2018,33(8):98-102,106.
[16]王春艳,宋建阳,吕慧鑫,等.基于高通量测序的宋河浓香型白酒不同窖龄窖泥细菌群落结构分析[J].中国酿造,2019,38(9):163-166.
[17]周恩民,李文均.未培养微生物研究:方法、机遇与挑战[J].微生物学报,2018,58(4):706-723.
[18]BROWN C M,STALEY C,WANG P,et al.A high-throughput DNA-sequencing approach for determining sources of fecal bacteria in a Lake Superior estuary[J].Environ Sci Technol,2017,51(15):8263-8271.
[19] MOTATO K E, MILANI C, VENTURA M, et al. Bacterial diversity of the Colombian fermented milk "Suero Costeño" assessed by culturing and high-throughput sequencing and DGGE analysis of 16S rRNA gene amplicons[J].Food Microbiol,2017,68:129-136.
[20]李妍,徐兴祥.高通量测序技术的研究进展[J].中国医学工程,2019,27(3):32-37.