化石燃料是一种不可再生能源,随着化石燃料的日益枯竭,可再生能源受到了越来越多的关注,而生物丁醇具有可再生性、热值高、蒸气压低、腐蚀性小等[1]优点,可以替代部分的化石燃料,受到了广泛的关注。丁醇的生产方式主要有化学合成法与生物发酵法,化学合成法主要有丙烯羰基合成法与乙醛醇醛缩合法[2],原料的来源依然是化石燃料,而生物发酵法利用葡萄糖、纤维素、淀粉等可再生的物质作为原料,通过微生物发酵生产丁醇,具有可再生的优势。粮食安全一直是国家关注的重点,随着发酵工业的发展,粮食类原料价格不断在提高,越来越多的非粮原料被投入到丁醇的生产中,如稻草[3],甚至有一些是工业生产的废弃物,如生产乙醇的废糟[4]。这些原料存在成分复杂,营养成分及比例不适宜,淀粉与还原糖含量低,抑制物多等问题;加上菌株对原料的利用能力差,对溶剂的耐受能力差,严格厌氧等问题。另外,没有合适的发酵工艺相匹配,导致在丁醇发酵过程中存在菌株无法正常生长、副产物过多、丁醇产量低等问题[5]。该文将从菌种、培养基配方、培养工艺三个方面总结一些已有的生产丁醇的优化策略,为改善发酵法生产丁醇与提高丁醇生产效益提供参考。
1 优化菌种
1.1 高产菌株的筛选
近些年来,分离筛选丁醇生产菌的工作一直在进行,新发现菌株尽管在丁醇的产量上很难有提高,但是菌株拥有的新性状有利于在丁醇发酵工业中应用,提高生产效益,一些丁醇生产菌的报道情况如表1所示。
表1 一些丁醇生产菌的报道
Table 1 Partial report of butanol producing strains

诱变育种是在已经获得了能够生产丁醇的菌的基础上,通过添加诱变剂的方式来提高菌的遗传物质在复制过程中的错配机率,并将这些发生变异的遗传信息遗传给后代,通过一定的分离筛选手段得到具有优良性状的菌的育种方式,一些丁醇生产菌诱变育种的报道见表2。诱变育种操作简单,被广泛的运用在菌种的改良中,通过这种方式有一定的机率可以得到性状优良的菌株,但是突变结果具有随机性,无意义的突变占绝大部分,试验的盲目性大,工作量也庞大。
表2 一些丁醇生产菌诱变育种的报道
Table 2 Partial report on mutation breeding of butanol producing strains

1.2 代谢途径的分子修饰或改造
野生型丁醇生产菌最常被报道的代谢产物有乙醇、乙酸、丙酮、丁酸、丁醇这些物质,这些物质的产生途径如图1所示,每种代谢产物都需要利用前体物质丙酮酸去合成,因此,减少副产物的生成与增加丁醇的生成是提高丁醇产量的一个重要方向。另外,菌株专性厌氧,对丁醇的耐受能力不高也是丁醇生产工业的一个障碍[16]。目前,主要从两个方面修饰或改造菌种的代谢途径:一种是从碳源的流向入手,改造糖酵解途径(embden-meyerhof pathway,EMP)代谢途径,使用于丙酮-丁醇-乙醇(acetone-butanol-ethanol,ABE)合成的前体物质过表达,阻断丙酮、乙酸及丁酸这些副产物的代谢途径,减少碳源流入副产物,一定程度上可以将更多的碳源流入到生成丁醇的代谢中;另一种是提高菌株对发酵环境的适应能力,解除其他基因对菌体生长与产物合成的抑制作用,如提高菌株对丁醇的耐受性与对氧气的耐受能力,使菌株更加适应发酵环境,从而达到改善发酵的作用。一些通过分子手段修饰或改造代谢途径的报道见表3。
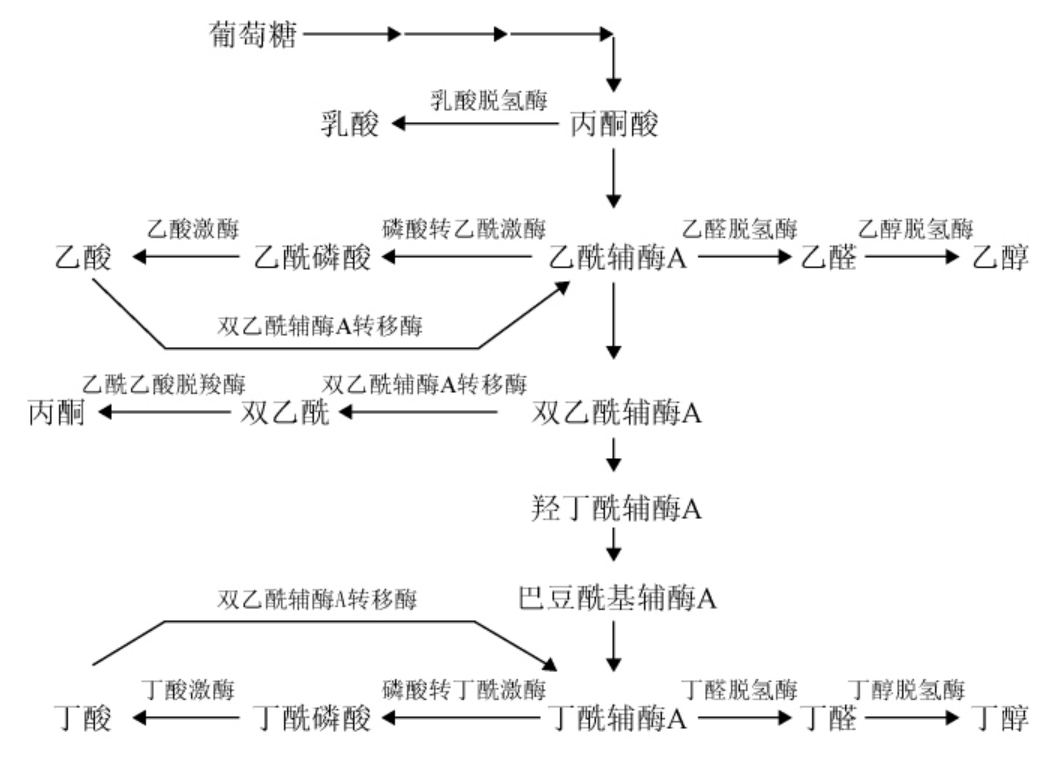
图1 丁醇生产菌的代谢途径[24]
Fig.1 Metabolic map of butanol-producing bacteria
表3 部分关于代谢途径通过分子手段修饰或改造的报道
Table 3 Partial report on molecular means of modification or modification of metabolic pathway

相比于传统的诱变育种,利用分子改造或修饰菌株的方式目的性强,可以促进有利于产物生成的基因的表达,阻断或抑制非必需的副产物基因的表达,提高菌株适应性。然而丁醇梭菌的代谢途径复杂,需要考虑通过基因的过表达、敲除、重组等方式进行修饰改造菌株对其生长代谢带来的影响。各个代谢途径存在着相互联系,丁醇梭菌本身专性厌氧,会增加分子试验操作的难度。
1.3 构建工程菌株
基因工程手段通常也被用于菌种的改良,该方法能够借助工程菌的优势。该方法通常是将与丁醇代谢相关的基因片段导入到特性优良的宿主细胞体内,并将导入的基因在宿主细胞体内表达。通常生物发酵法生产丁醇有两种途径[25],一种是辅酶A(coenzyme A,CoA)依赖途径,类似于脂肪酸在生物体内的合成方式,梭菌先将乙酰CoA合成丁酰CoA,然后将丁酰CoA还原成正丁醇,这是野生菌最常见的丁醇代谢方式,例如丙酮丁醇梭菌、拜氏梭菌、糖丁酸梭菌与糖乙酸多丁醇菌。该方式也被用于构造丁醇生产的工程菌株,如LAN E I等[26]将丁醇的CoA依赖途径相关基因导入蓝细菌中表达,构造了一株能够利用光能合成丁醇的菌株,其中丁醇产量0.014 5 g/L。ZHANG J等[27]将丁醇的CoA依赖途径部分基因导入酪丁酸梭菌,构造了一株对丁醇耐受能力强的菌株,通过分批发酵丁醇产量达26.2 g/L;YANG X R等[28]将醛/醇脱氢酶基因(edhE2)引入纤维素分解菌嗜纤维杆菌(Clostridium cellulovorans),其能够将丁酰-CoA转化为正丁醇和乙酰-CoA转化为乙醇,构造的菌株可以直接利用纤维素生产正丁醇,产量为1.42 g/L。另一种是2-酮酸途径,生物体内的苏氨酸被用于中间产物2-酮丁酸的生产,然后中间产物被脱羧还原产生正丙醇与正丁醇,一般这种途径用于异源表达,代谢副产物少,如利用大肠杆菌重组菌株BW25113就是利用这一途径,丁醇产量达0.8 g/L[29]。
将涉及丁醇代谢的关键基因转移到特性优良的宿主体内进行异源表达,其他方面加以调整,具有生产丁醇的潜力与可行性。这种生产方式能够借助宿主细胞的优良特性,使得能够用于丁醇发酵的原料种类增多,同时对发酵环境具有强的适应能力,然而丁醇代谢相关的基因在宿主体内的兼容性也是不可忽略的,增加了改造的难度。
2 优化培养基配方
微生物生产丁醇的代谢网络复杂,通常提高或降低某些物质的含量就可以影响某些代谢途径的表达量,从而影响丁醇的产量。随着研究深入,已经有报道表明,适量地添加氨基酸[30]、醋酸铵[31]、丁酸根[32]、乙酸根[33]、锌离子、钙离子、铁离子[34]等对丁醇产量有提高的作用,各物质存在相互影响,适宜的物质配比能够得到更高的丁醇产量。借助数理统计工具就可以通过少量的试验数据模拟生物发酵过程,建立发酵的数学模型。再借助分析工具分析模型就能得到培养基中影响显著的成分及其适宜的添加量,从而达到提升丁醇产量的目的。
2.1 回归分析模拟优化
响应面是回归分析模型之一,是一个基于二次二阶的数学模型。将试验所得的数据用于拟合一个二次多项式模型方程式,通过显着性检验与方差分析(analysis of variance,ANOVA),分析参数及其相互作用对输出结果的影响[35],用相关系数R2值评估多项式模型的可靠性。通过拟合的多项式方程通过绘制三维表面图,可以观察到两两因素之间的相互作用关系,通过回归方程可以精确得到最优的培养基配方及相应的预测结果。NAJEEB K N S等[36]采用中心组合设计来提高丁醇的产量,以木薯为原料,按照中心组合设计添加葡萄糖与丁酸以及调整C/N比例,将试验结果用于拟合模型,按照模型得出添加葡萄糖与丁酸含量以及调整C/N比例分别是50 g/L、10 g/L、12.8,预测的丁醇产量13.82 g/L,发酵验证的丁醇产量是13.87 g/L,高于优化前的最高产量9.48 g/L。
2.2 神经网络模拟与遗传算法优化
人工神经网络是一种人工模拟大脑运行用于处理某种信息的系统,被用于广泛运用于微生物的培养基优化上[37-38]。遗传算法则是模拟自然进化,在合理范围内随机产生培养基配方,并将这些配方带入训练有素的神经网络模型,多次筛选变异之后,就能得到一个相对较优的优化结果以及相应的配方。相比于响应面来说,采用神经网络建模预测结合遗传算法优化具有更高的准确性,SALIM N等[39]使用人工神经网络对菌株Trichoderma harzianum建立发酵模型,采用遗传算法从模型中找到最优培养的条件,蛋氨酸酶预测值是33.76 U/mg,预测的配方是乳糖、L-蛋氨酸、KH2PO4、K2HPO4、ZnCl2分别加13.9 g/L、11.37 g/L、1.58 g/L、3.98 g/L、0.01 g/L。某些物质对于菌的影响来说并非符合二次连续函数模型,阈值函数的加入适用于处理这类非连续的数学模型,其拟合的模型能够更加吻合生物发酵的特征。
3 优化发酵工艺
除了优化培养基配比外,丁醇梭菌的培养工艺同样重要。菌株对丁醇的耐受性没有很大提高[40],限制了丁醇产量的进一步提高;越来越多的非粮原料用于丁醇的生产,存在菌株对原料的利用能力差的问题,各种处理会释放原料中的抑制物,都会影响丁醇的生产[41],采用合适的培养工艺可以缓解这些问题。
3.1 产物原位提取
丁醇梭菌的代谢产物有许多,如乙醇、乙酸、丙酮、丁酸、丁醇等,其中丁醇对菌株的抑制性最大,正丁醇影响膜的脂质组成,流动性与电位,并且能够中断细胞的磷脂双分子层[42]。原位提取是一个避免高浓度产物对菌体有毒害作用的方式,将发酵产物丁醇脱离发酵体系,这样能够实现在营养物质足够的情况下,菌株能够源源不断地生产丁醇。一种方式是利用一些物质对于水与丁醇的溶剂度不同构建双相系统,如利用丁醇易溶于有机溶剂可以液-液萃取[43]、活性炭对丁醇吸附丁醇能力强,可以用吸附[44]、丁醇比水易溶于空气,通过通气的方式将丁醇携带出发酵体系,可以用气提[45]等。另一种是利用一些材料对水和丁醇的选择透过性不同可以将丁醇从发酵体系中分离,如膜蒸发[46]、渗透[47]与反向渗透[48]。原位提取能够很好回避丁醇对菌的毒害作用,然而最大的问题在于寻找合适的提取分离体系,要求溶剂收集相能够对发酵产生的溶剂溶解度足够大,并与发酵液互不相溶,对菌体无毒无害,易于分离,或者材料对丁醇的选择透过性高,其营养物质不能透过膜。
3.2 共培养发酵
目前报道的用于生产丁醇的菌绝大多数是厌氧菌,以木质纤维素为原料进行发酵生产丁醇的方式受到了越来越多的关注。木质纤维素难以被丁醇梭菌所利用,水解过程会产生大量的抑制物[41],通过与其他微生物共培养,可以为丁醇梭菌制造厌氧环境[49],更重要的是让其他微生物先行分解这类难以利用的大分子物质为一些容易吸收利用营养物质,中间代谢产物,生长因子等[50],协助丁醇梭菌对原料中营养物质的吸收利用,同时,协助分解一些抑制物,从而达到提高丁醇产量的目的。ALEJANDRA G O R等[51]使用龙舌兰水解液作为原料,丙酮丁醇梭菌与枯草芽孢杆菌共培养丁醇产量达8.2 g/L,而丙酮丁醇梭菌单独发酵产量是6 g/L,提高了36.7%。LI L等[52]使用甘蔗糖蜜作为原料,将拜氏梭菌与酪丁酸梭菌共发酵生产丁醇产量达12.1 g/L,而单独使用拜氏梭菌发酵产量是11.2 g/L,提高了8%。另外,研究还发现,通过丁醇梭菌与其他菌共培养的方式生产丁醇的产量,高于直接在发酵培养基中添加这些物质的方式与直接发酵的方式,一方面原因是人为添加这些物质的时机没有共培养微生物代谢产生的好;另一方面是受技术水平的限制,共培养微生物产生的一些物质对丁醇梭菌产丁醇有提高作用,但由于其含量极低,在其以往的研究过程中并没有被发现,这类物质在培养基上的添加往往会被忽略掉。
一些研究发现,在食物匮乏或者营养物质不全的条件下,丁醇梭菌能够与其他菌互利共生,两者不仅能够在体外进行营养物质代谢产物的交换,而且还能够进行细胞质中的物质交换。SAIDA B等[53]研究发现,丙酮丁醇梭菌与脱硫弧菌能够互换细胞质中的物质,脱硫弧菌的存在使得丙酮丁醇梭菌生长的更好。
3.3 两阶段发酵
大多数的丁醇梭菌在生产丁醇的过程中存在着产酸与产醇两个阶段,产醇阶段可以利用产酸阶段产生的部分有机酸转化为醇[54]。也有研究表明,将有机酸在发酵前加入培养基中,可以提高丁醇梭菌对丁醇的耐受性,提高丁醇的产量[55]。分阶段发酵就是利用产酸菌先将部分难以分解利用的碳源转化为丁醇梭菌容易吸收利用的有机酸,然后丁醇梭菌将这些酸转化为丁醇;另外,产酸菌还可以降解原料中存在的一些抑制物,促进丁醇梭菌的生长代谢。LI J Z等[56]使用稻草作为生产丁醇的原料,先利用产酸菌发酵产生乙酸与丁酸,然后利用拜氏梭菌发酵生产丁醇,丁醇产量高达13.8 g/L,高于直接使用拜氏梭菌对原料进行发酵的方式,还高于在原料上添加乙酸、丁酸再使用拜氏梭菌发酵的方式。相比共培养来说,可以避免其他菌对丁醇梭菌产生抑制作用,但是分阶段发酵的操作工艺相对复杂,同时也会带来发酵周期的延长。
4 结论与展望
生物发酵法生产丁醇一直是一个研究的热点,真正用于工业生产还存在着菌株对产物耐受性差,对原料利用能力差,对发酵环境适应能力差,副产物多等问题。这些问题可以从三个方面找到缓解的策略:
性状优良的菌种用于发酵是高产目标产物的前提,自然界筛选与诱变育种的操作简单,是得到性状优良生产菌的重要途径,今后的探索依然会继续。随着丁醇梭菌代谢方面的研究深入,分子生物学的快速发展,代谢途径修饰改造或者丁醇代谢基因异源表达的技术越来越成熟,生产菌株可以通过这些方式获得优良的特性,在提高菌株对产物的耐受性,对原料的利用,对发酵环境的适应,减少副产物的产量,增加目标产物的产量等方面具有广泛的应用前景,菌种改良的目标比较明确,依旧是今后研究的一个热点方向。
随着研究的深入,越来越多的非粮原料用于丁醇生产,成分的多样性,原料的更新,需要不断优化配比,今后,借助各种处理工具优化培养基配比的方式将继续。神经网络与遗传算法适合解决一类优化的问题,在其他生物的培养基配比优化方面优于回归分析,推动该方法在丁醇生产中在培养基配比优化上的运用。
面对复杂的发酵环境,发酵工艺依然是一个研究的热点,尤其是菌株对发酵产物丁醇的耐受性难以提高的情况下,原位提取工艺结合其他的发酵工艺是一个值得借鉴考虑的工艺优化方式,将会有更多的研究。
[1]PETER D.Biobutanol:An attractive biofuel[J]. Food Fuel,2007,2(12):1525-1534.
[2]王洪,罗惠波,廖玉琴,等.发酵法产丁醇的研究进展[J].中国酿造,2017,36(4):10-14.
[3]NARISETTY V,LAKSHMI M N,BINOOP M,et al.Biobutanol production from rice straw-recent trends,possibilities,and challenges[J]. Bioresource Technol Rep,2019,7:100224.
[4]OCEGUERA-CONTRERAS E,AGUILAR-JUAREZ O,OSEGUERA-GALINDO D,et al.Biohydrogen production by vermihumus-associated microorganisms using agro industrial wastes as substrate[J].Int J Hydr Energ,2019,44(20):9856-9865.
[5]WANG Y,GUO W Q,CHEN B Y,et al.Exploring the inhibitory characteristics of acid hydrolysates upon butanol fermentation:A toxicological assessment[J].Bioresource Technol,2015,198:571-576.
[6]NG C Y,TAKAHASHI K,LIU Z.Isolation,characterization,and optimization of an aerobic butanol-producing bacterium from Singapore[J].Biotechnol Appl Biochem,2016,63(1):86-91.
[7]JYOTSANA D,MRIGANKO D,SAM J,et al.Efficient isopropanol-butanol(IB)fermentation of rice straw hydrolysate by a newly isolated Clostridium beijerinckii strain C-01[J].Biomass Bioenergy,2019,127:105292.
[8]NAJEEB K N A S,MOHD S K,WAN M W Y,et al.Improvement of the butanol production selectivity and butanol to acetone ratio(B:A)by addition of electron carriers in the batch culture of a new local isolate of Clostridium acetobutylicum YM1[J].Anaerobe,2015,36:65-72.
[9]LI H G,FRED K O,LI K T,et al.Acetone,butanol,and ethanol production from gelatinized cassava flour by a new isolates with high butanol tolerance[J].Bioresource Technol,2014,172:276-282.
[10]NUTPAPHAT P,KHANITTHA F,JIRAWAN A.Isolation of Cellulolytic clostridia and their performance for one-step butanol production from sugarcane bagasse[J].Energy Procedia,2017,138:163-168.
[11]冯鸿燕,李东,闫志英,等.Clostridium acetobutylicum 的亚硝基胍和钴-60 复合诱变[J].太阳能学报,2016,37(12):3240-3245.
[12]金超楠,刘莉,史吉平,等.离子束诱变与连续驯化选育耐毒性产丁醇菌株[J].中国酿造,2015,34(6):25-28.
[13]LI H G,LUO W,WANG Q,et al.Direct fermentation of gelatinized cassava starch to aceton,butanol,and ethanol using Clostridium acetobutylicum mutant obtained by atmospheric and room temperature plasma[J]. Appl Biochem Biotechnol,2014,172(7):3330-3341.
[14]JANG Y S,MALAVIYA A,LEE S Y.Acetone-butanol-ethanol production with high productivity using Clostridium acetobutylicum BKM19[J].Biotechnol Bioeng,2013,110(6):1646-1653.
[15]毛绍名,章怀云.丙酮丁醇梭菌高耐丁醇突变株的选育及其生理特性的研究[J].中南林业科技大学学报,2012,32(8):103-107.
[16]HOLGER J,CHRISTINA G,ARMIN E,et al.A transcriptional study of acidogenic chemostat cells of Clostridium acetobutylicum-solvent stress caused by a transient n-butanol pulse[J]. J Biotechnol,2012,161(31):354-365.
[17]VENTURA J S,HU H,JAHNG D.Enhanced butanol production in Clostridium acetobutylicum ATCC 824 by double overexpression of 6-phosphofructokinase and pyruvate kinase genes[J].Appl Microbiol Biotechnol,2013,97(16):7505-7516.
[18]TUMMALA S B,WELKER N E,PAPOUTSAKIS E T.Design of antisense RNA constructs for downregulation of the acetone formation pathway of Clostridium acetobutylicum[J].J Bacteriol,2003,185(6):1923-1934.
[19]KUIT W,MINTON N P,LOPEZ C A M,et al.Disruption of the acetate kinase(ack)gene of Clostridium acetobutylicum results in delayed acetate production[J].Appl Microbiol Biotechnol,2012,94(3):729-741.
[20]WANG Y,LI X,MILNE C B,et al.Development of a gene knockout system using mobile group II introns(targetron)and genetic disruption of acid production pathways in Clostridium beijerinckii[J]. Appl Environ Microbiol,2013,799(19):5853-5863.
[21]XU M,ZHAO J,YU L,et al.Engineering Clostridium acetobutylicum with a histidine kinase knockout for enhanced n-butanol tolerance and production[J].Appl Microbiol Biotechnol,2015,99(2):1011-1022.
[22]SCOTCHER M C,BENNETT G N.SpoIIE regulates sporulation but does not directly affect solventogenesis in Clostridium acetobutylicum ATCC 824[J].J Bacteriol,2005,187(6):1930-1936.
[23]裴建新,庞浩,左文朴,等.perR 基因的敲除对丙酮丁醇梭菌摇瓶发酵的影响[J].食品与发酵工业,2014,40(2):94-98.
[24]曹长海,关浩,高慧鹏,等.产丁醇梭菌优化改造的研究进展[J].当代化工,2015,44(8):2014-2017.
[25]YE W H,LI J,HAN R Z,et al.Engineering coenzyme A-dependent pathway from Clostridium saccharobutylicum in Escherichia coli for butanol production[J].Bioresource Technol,2017,235:140-148.
[26]LAN E I,LIAO J C.Metabolic engineering of cyanobacteria for 1-butanol production from carbon dioxide[J].Metab Eng,2011,13:353-363.
[27]ZHANG J,ZONG W M,HONG W,et al.Exploiting endogenous CRISPR-Cas system for multiplex genome editing in Clostridium tyrobutyricum and engineer the strain for high-level butanol production[J].Metab Eng,2018,47:49-59.
[28]YANG X R,XU M M,YANG S T.Metabolic and process engineering of Clostridium cellulovorans for biofuel production from cellulose[J]. Metab Eng,2015,32:39-48.
[29]SHEN C R,LIAO J C.Metabolic engineering of Escherichia coli for 1-butanol and 1-propanol production via the keto-acid pathways[J].Metab Eng,2008,10(6):312-320.
[30]PRANHITA R N,MANISHA A K,RAHUL K K,et al.Strategic intensification in butanol production by exogenous amino acid supplementation:Fermentation kinetics and thermodynamic studies[J].Bioresource Technol,2019,288:121521.
[31]LI S B,ZHOU Y,LUO Z T,et al.Dual function of ammonium acetate in acetone-butanol-ethanol fermentation by Clostridium acetobutylicum[J].Bioresource Technol,2018,267:319-325.
[32]LEHMANN D,TINA L E.Switching Clostridium acetobutylicum to an ethanol producer by disruption of the butyrate/butanol fermentative pathway[J].Metab Eng,2011,13(5):464-473.
[33]W KUIT,NIGEL P M,ANAM L C,et al.Disruption of the acetate kinase(ack)gene of Clostridium acetobutylicum results in delayed acetate production[J].Appl Microbiol Biotechnol,2012,94(3):729-741.
[34]MAYURKETAN M,PAYEL S,GARGI G,et al.Regulation of butanol biosynthesis in Clostridium acetobutylicum ATCC 824 under the influence of zinc supplementation and magnesium starvation[J].Enzyme Microbial Technol,2019,129:109352.
[35]SHARON M J P,SATHYANARAYANA N G.Artificial neural network and regression coupled genetic algorithm to optimize parameters for enhanced xylitol production by Debaryomycesnepalensis in bioreactor[J].Biochem Eng J,2017,120:136-145.
[36]NAJEEB K N S,HAFIZA S,PEYMAN A,et al.Enhanced butanol production by optimization of medium parameters using Clostridium acetobutylicum YM1[J].Saudi J Biol Sci,2018,25(7):1308-1321.
[37]ABDOLLAHI H,NOAPARAST M,SHAFAEI S,et al.Prediction and optimization studies for bioleaching of molybdenite concentrate using artificial neural networks and genetic algorithm[J]. Mineral Eng,2019,130:24-35.
[38]郭慧慧.应用BP 神经网络和遗传算法优化苏云金芽孢杆菌BRCLLP29、BRC-LLB19 及BRC-ZQL3 菌株的发酵培养基[D].福州:福建农林大学,2013.
[39]SALIM N,SANTHIAGU A,KJOJ I.Process modeling and optimization of high yielding L-methioninase from a newly isolated Trichoderma harzianum using response surface methodology and artificial neural network coupled genetic algorithm[J]. Biocatal Agr Biotechnol,2019,17:299-308.
[40]RASTEGAR S O,MOUSAVI S M,REZAEI M,et al.Statistical evaluation and optimization of effective parameters in bioleaching of metals from molybdenite concentrate using Acidianus brierleyi[J].J Ind Eng Chem,2014,20(5):3096-3101.
[41]周智友,李志,张庆华,等.利用木质纤维素生产丁醇的研究进展[J].微生物学通报,2019,46(12):3461-3468.
[42]HOLGER J,CHRISTINA G,ARMIN E,et al.A transcriptional study of acidogenic chemostat cells of Clostridium acetobutylicum-Solvent stress caused by a transient n-butanol pulse[J]. J Biotechnol,2012,161(31):354-365.
[43]ANCA M D,IONUT B,GHEORGH B.Process modeling and simulation for butanol removing from fermentation broth by extraction with biodiesel[J].Renew Energ,2019,131:137-143.
[44]CHUANG X,LIU F F,XU M M,et al.Butanol production in acetonebutanol-ethanol fermentation with in situ product recovery by adsorption[J].Bioresource Technol,2016,219:158-168.
[45]ROCHON E,FERRARI M D,LAREO C.Integrated ABE fermentationgas stripping process for enhanced butanol production from sugarcane-sweet sorghum juices[J].Biomass Bioenerg,2017,98:153-160.
[46]HODA A,HANDAN T,JULES T.Optimisation of the in-situ recovery of butanol from ABE fermentation broth via membrane pervaporation[J/OL].Chem Eng Res Design,2019,https://doi.org/10.1016/j.cherd.2019.07.012.
[47]QURESHI N,MADDOX I S.Reduction in butanol inhibition by perstraction[J].Food Bioprod Process,2005,83(1):43-52.
[48]AGENSON K O.Urase change in membrane performance due to organic fouling in nanofiltration(NF)/reverse osmosis(RO)applications Sep[J].Purif Technol,2007,55:147-156.
[49]HANH T M,BENJAMAS C,BRIAN H,et al.Potential use of Bacillussubtilis in a co-culture with Clostridium butylicum for acetone-butanolethanol production from cassava starch[J].Biochem Eng J,2010,48:260-267.
[50]WU J W,DONG L L,ZHOU C S,et al.Developing a coculture for enhanced butanol production by Clostridium beijerinckii and Saccharomyces cerevisiae[J]. Bioresource Technol Rep,2019,6:223-228.
[51]ALEJANDRA G O R,JULIAN Q,MIGUEL A M M,et al. Clostridium strain selection for co-culture with Bacillus subtilis for butanol production from agave hydrolysates[J].Bioresource Technol,2019,275:410-415.
[52]LI L,AI H X,ZHANG S X,et al.Enhanced butanol production by coculture of Clostridium beijerinckii and Clostridium tyrobutyricum[J].Bioresource Technol,2013,143:397-404.
[53]SAIDA B,DAVUD R,MARIA L C,et al.Nutritional stress induces exchange of cell material and energetic coupling between bacterial species[J].Nat Commun,2015,6283(6).http://doi.org/10.1038/ncomms7283.
[54]LI X,SHI Z,LI Z.Increasing butanol/acetone ratio and solvent productivity in ABE fermentation by consecutively feeding butyrate to weaken metabolic strength of butyrate loop[J].Bioprocess Biosyst Eng,2014,37:1609-1616.
[55]SHENG Y C,WU J W,ZHAO L,et al.Optimization of culture conditions for enhanced butanol production by a high butanol tolerant Clostridium beijerinckii F-6[J].Energy Procedia,2019,158:471-476.
[56]LI J Z,CHI X,ZHANG Y F,et al.Enhanced coproduction of hydrogen and butanol from rice straw by a novel two-stage fermentation process[J].Int Biodeterior Biodegradat,2018,127:62-68.