放线菌是原核生物中经济和生物学上最具有价值的细菌,可以产生各种生物活性物质,如抗生素、抗肿瘤药物以及生物酶等[1]。目前,放线菌已成为新型抗生素生产的主要来源之一,包括最重要的抗菌药物,如β-内酰胺、四环素、利福霉素、氨基糖苷、大环内酯和糖肽[2]。因此,放线菌在天然化合物开发方面具有至关重要的作用和潜力[3]。据统计,已发现超过一半以上的天然活性物质来自于放线菌[4]。然而,随着抗生素的大规模使用,病原菌耐药性逐渐增加,使得现有的抗生素越来越不足以应对,因此迫切需要产生新的抗生素。现代基因组研究成果表明,特殊生境可能蕴藏着新物种,而新物种可能具有合成新化合物的基因,因此从特殊生境中发现放线菌资源是寻找新化合物的有效方法[5]。
由于温泉的持续高温环境条件及其独特的物理化学特征,促进了特殊微生物群的形成,因此在温泉环境中,聚集了大量适合极端环境条件生存的微生物如嗜热菌[6]。近年来,由于高通量测序技术的发展和宏基因组学方法的出现,国内外越来越多的学者开始关注温泉微生物群体的多样性以及功能基因分析[7]。关于温泉微生物多样性以及筛选抗生素合成相关基因的研究具有良好的医学应用前景。RUCKMANI A等[8-9]分别研究印度高止山脉西部温泉和哥伦比亚安迪斯山脉的El Coquito酸性温泉中的微生物多样性。肖凯等[10]研究广东金山温泉微生物的多样性,发现菌株S-X2与嗜热蓝细菌聚球藻BP-1(47118315)具有高度的相似性。近年来,对于温泉中放线菌的多样性和抗生素合成基因筛选的研究很少。1991年,徐丽华等[11]研究发现,在云南安宁温泉次生林中共分离到放线菌8个种属。王勇等[12]研究发现,长白山的不同环境以及温泉中采集的土样中分离出来的BOS-009菌株具有一定的抗植物病原菌作用。
山西省是我国的内陆省份,也是地热资源丰富的地区,温泉遍布全省,其中大多数温泉集中分布在某一区域[13]。山西太子滩温泉属于地热井水,水温达46.5℃。本研究以太子滩温泉地热井周围的土壤样品为研究对象,通过对土壤中的放线菌进行分离鉴定并对其菌株进行功能基因筛选,旨在探索放线菌新物种及其潜在活性。
1 材料与方法
1.1 材料与试剂
1.1.1 土壤样品
2018年2月采集于太子滩温泉泉眼周围土壤样品4份(编号:W1、W2、W3、W4),样品采集信息如表1。
表1 采集的土壤样品信息
Table 1 Information of collected soil sample

样品 采集地点W1 W2 W3 W4 N35°40'28",E111°28'22"N35°40'28",E111°28'22"N35°40'28",E111°28'22"N35°40'18",E111°28'31"
1.1.2 培养基
(1)分离培养基
高氏1号培养基(M1):可溶性淀粉 20.0 g,KNO31 g,NaCl 0.5 g,K2HPO40.5 g,MgSO40.5 g,FeSO40.01 g,琼脂20.0 g,加双蒸水定容至1 L。
自来水酵母粉琼脂培养基(M2):酵母浸汁0.25 g,K2HPO40.5 g,琼脂18.0 g,加双蒸水定容至1 L。
葡萄糖酵母麦芽(glucose yeast malt extract,GYM)培养基(M3):葡萄糖4.0 g,CaCO32.0 g,麦芽抽提物10.0 g,琼脂粉15.0 g,酵母抽提物共4.0 g,加双蒸水定容至1 L。
牛肉膏蛋白胨培养基(M4):牛肉膏5.0 g,氯化钠5.0 g,蛋白胨10 g,琼脂15.0 g,加双蒸水定容至1 L。
LB培养基(M5):蛋白胨10.0g,酵母粉5.0g,NaCl0.5g,琼脂15.0 g,加双蒸水定容至1 L。
大豆酪蛋白琼脂(tryptic soy agar,TSA)培养基(M6):TSA30.0g,琼脂20.0g,加双蒸水定容至1L。
(2)纯化培养基
链霉菌培养基二号ISP2培养基:葡萄糖4.0 g,酵母粉4.0 g,麦芽浸粉10.0 g,琼脂20.0 g,加双蒸水定容至1 L。
1.1.3 主要试剂
2×EasyTaq SuperMix、pEASY-T1 Cloning Kit、EasyPure Quick Gel Extraction Kit、Trans-T1感受态细胞、DNAMarker、X-gal、聚合酶链式反应(polymerase chain reaction,PCR)引物27F和1492R,A3F和A7R,K1F和M6R,KSα和KSβ:上海生物工程股份有限公司;High Fidelity PCR Super Mix:北京全式金生物技术有限公司;Chelex-100树脂,美国BioRad公司;琼脂糖、TAE缓冲液:购自北京索莱宝公司;萘啶酮酸(20 mg/L)、重铬酸钾(60 mg/L)[14]:上海麦克林生化科技有限公司。
1.2 仪器与设备
BSC-1600IIA2生物安全柜:苏州安泰空气技术有限公司;ZXSD-B1270生化培养箱:上海智城分析仪器制造有限公司;SHKE481HP型落地式恒温制冷摇床:美国Thermo公司;SX-500高压蒸汽灭菌锅:日本Tomy公司;Microfuge系列台式微量离心机:美国Beckman公司;聚合酶链式反应(polymeras chain reaction,PCR)T100扩增仪、Gel DocTMXR+凝胶成像仪:美国BioRad公司;DYY-6C型电泳仪:北京市六一仪器厂。
1.3 方法
1.3.1 样品处理与菌株的稀释平板分离
菌种分离前2周,取等量土壤样品置于培养皿中,自然风干。将风干后的土壤研磨过筛后,每份样品取2 g放入装有18 mL无菌水的离心管中,28℃、180 r/min振荡过夜。然后取样品进行10倍梯度稀释,使其终浓度为10-2和10-3。取终浓度为10-3样品200μL涂布于上述分离培养基上,倒置放于28℃培养箱中培养4~8周。
1.3.2 菌株的纯化及保藏
观察分离培养基上的菌落形态和生长状况,并用肉眼判断将放线菌菌落进行编号。挑取单菌落于ISP2固体平板上,采用四区划线法进行纯化,重复这一过程直至得到纯培养物。同时,将生长好的菌落置于20%甘油中-80℃保存。
1.3.3 基于16S rRNA基因序列的系统发育分析
(1)16S rRNA基因测序和分析
按Chelex-100法进行菌株DNA的提取[15]。PCR扩增引物为通用引物27F(5'-AGAGTTTGATCCTGGCTCAG-3)'和1492R(5'-GGTTACCTTGTTACGACTT-3)'。PCR反应体系:2×EasyTaqSuperMix 25 μL,27F引物2 μL,1492R引物2 μL,模板2 μL,无菌水19 μL。PCR反应条件:95 ℃、5 min;94 ℃、1 min,55 ℃、1 min,72 ℃、2 min,35个循环;72 ℃、10 min。PCR扩增产物经琼脂糖凝胶电泳检测,将电泳检测结果显示为阳性的PCR扩增产物送交生工生物工程(上海)股份有限公司进行序列测定。
(2)序列分析和系统发育树构建
测序获得的16S rRNA基因序列利用EzBioCloud(http://www.ezbiocloud.net)[16]数据库中的EzTaxon在线比对服务进行相关标准菌株的相似性比对搜索,确定菌株的分类学地位,并从中调出相关的标准菌株的序列作为参比对象,用ClustalX[17]软件进行多序列比对,采用MEGA 5.0[18]软件以邻接法(neighbor-joining,NJ)[19]进行聚类分析并构建系统发育树,系统进化矩阵根据Kimura-2-parameter模型估算,重复取样1000次进行自展值分析以评估进化树拓扑结构的稳定性。
1.3.4 抗生素生物合成基因的探测
放线菌基因组DNA的提取同1.3.3(1),以基因组DNA作为模板PCR扩增非核糖体多肽合成酶(nonribosomal peptide synthetases,NRPS)基因A结构域(引物A3F:5'-GCSTACSYSATSTACACSTCSGG-3';A7F:5'-SASGTCVCCSGTSCGGTAS-3)'[20]。PCR扩增反应条件:95℃、5min;95℃、0.5 min,59 ℃、2 min,72 ℃、4 min,35个循环;72℃、10min。扩增I型聚酮合酶(I type polyketide synthase,PKSI)基因KS结构域(引物K1F:5'-TSAAGTCSAACATCGGBCA-3';M6R:5'-CGCAGGTTSCSGTACCAGTA-3)'[20]。PCR扩增反应条件:95℃、5 min;95℃、0.5min,59℃、2min,72℃、4min,35个循环;72℃、10min。扩增II型聚酮合酶(IItypepolyketide synthase,PKSII)基因KS结构域(引物KSα:5'-TSGCSTGCTTGGAYGCSATC-3';KSβ:5'-TGGAANCCGCCGAABCCTCT-3)'[15],扩增反应条件:96℃、2min;96℃、1 min,60℃、2 min,73 ℃、1.5 min,35个循环;72 ℃、8.5 min。PCR反应体系都为50 μL,其中模板DNA 2 μL,A3F/A7R(10 μmol/L)、K1F/M6R(10 μmol/L)、KSα/KSβ(10 μmol/L)分别为2 μL,2TaqPCR Master Mix为25 μL,用ddH2O补至50 μL。PCR扩增产物用1%琼脂糖凝胶电泳进行检测,凝胶成像仪观察结果。
2 结果与分析
2.1菌种分离结果
使用6种分离培养基分离从曲沃太子滩温泉周围收集的4份土壤样品中的放线菌。根据放线菌菌落形态初步筛选,选择111株菌进行16S rRNA基因序列扩增和序列分析。结果表明,104株菌的16S rRNA基因序列比对结果是放线菌。4个样品中,1号土壤样品中分离到的放线菌数量最多,为34株,其次是3号土壤样品,分离到放线菌27株。从不同培养基中分离的放线菌数量如表2所示。
表2 6种培养基分离放线菌的结果
Table 2 Result of actinomycetes isolated by 6 media

培养基 属 属数目菌株数目M1 M2 M3 M4 M5 M6?Streptomyces Streptomyces,Plantibacter,Arthrobacter,Paenarthrobacter,Rhodococcus,Arthrobacter Streptomyces,Nocardioides,Cellulosimicrobium,Curtobacterium,Promicromonospora Streptomyces,Agromyces,Microbacterium Streptomyces,Microbacterium,Cellulosimicrobium Streptomyces,Microbacterium,Agromyces,Arthrobacter,Pseudomonas 1 6 5 3 3 5 11 24 13 22 12 22
由表2可知,在6种分离培养基中,从M2培养基中分离的放线菌数量最多,为24株,M1培养基分离的菌数最少。不同培养基中分离的菌株具有特殊性,植物杆菌属(Plantibacter),红球菌属(Rhodococcas)为M2培养基所分离,原小单胞菌属(Promicromonospora)为M3培养基所分离,壤霉菌属(Agromyces)为M4培养基所分离,纤维菌属(Cellulosimicrobium)为M5培养基所分离,(Pseudomonas)为M6培养基所分离。根据分离到的菌株数量及种属数目,M2培养基和M6培养基的菌数量和种属数目较多,可能较适合培养温泉来源的放线菌。
2.2 土壤放线菌多样性分析
104株菌的分布情况如表3所示,部分菌株的系统发育树构建结果见图1。由表3可知,分离到的104株放线菌分布于3目、5科、12属,分别是链霉菌科的链霉菌属(Streptomyces);微杆菌科的壤霉菌属(Agromyces)、微杆菌属(Microbacterium)、植物杆菌属(Plantibacter)和短小杆菌属(Curtobacterium)。微球菌科的Pseudarthrobacter属、节杆菌属(Arthrobacter)和Paenarthrobacter属;原小单胞菌科的原小单胞菌属(Promicromonospora)和纤维菌属(Cellulosimicrobium);Nocardio科的类诺卡氏菌属(Nocardioides)和红球菌属(Rhodococcus)。其中,链霉菌76株,占所分离菌株的73.07%,属优势种;微球菌科最具多样化,包括6株微杆菌属、5株壤霉菌属、2株植物杆菌属和1株短小杆菌属。就放线菌的多样性而言,从太子滩温泉附近的土壤中所获得的稀有放线菌和种属数量相对较高,多样性较好。
表3 104株放线菌的多样性分布情况
Table 3 Diversity and distribution of 104 actinomycetes

属 菌株数量链霉菌目(Streptomycetales)目科链霉菌科(Sterptomycetaceae)76微杆菌科(Microbacteriaceae)微球菌目(Micrococcales)微球菌科(Micrococcaceae)原小单胞菌科(Promicromonosporaceae)棒杆菌目(Propionibacteriales)类诺卡氏菌科(Nocardiaceae)链霉菌属(Streptomyces)微杆菌属(Microbacterium)壤霉菌属(Agromyces)植物杆菌属(Plantibacter)短小杆菌属(Curtobacterium)节杆菌属(Arthrobacter)(Paenarthrobacter)(Pseudarthrobacter)原小单孢菌属(Promicromonospora)纤维菌属(Cellulosimicrobium)诺卡氏菌属(Nocardioides)红球菌属(Rhodococcus)6 5 2 1 5 1 1 2 2 2 1
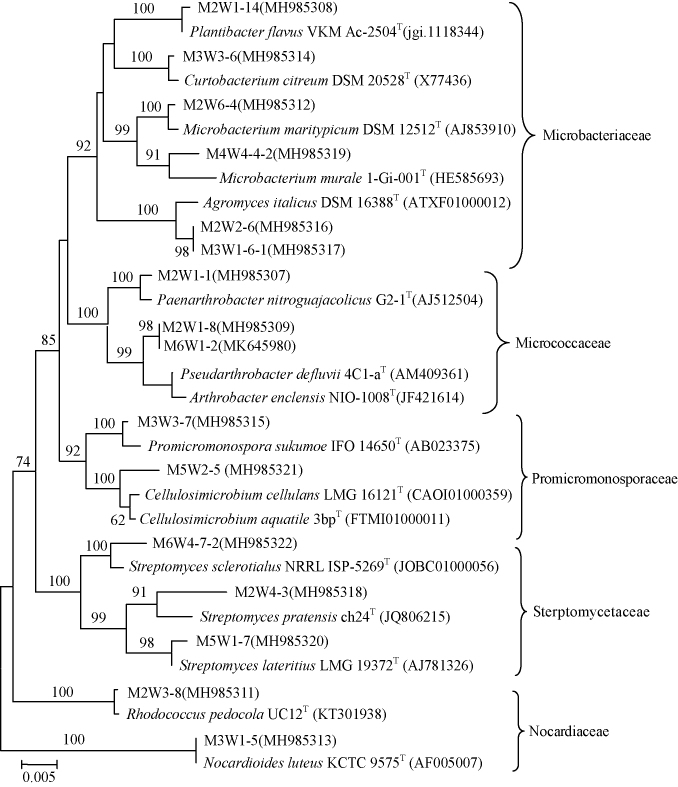
图1 基于16S rRNA序列构建的部分菌株的系统进化树
Fig.1 Phylogenetic tree of partial strains based on 16S rRNA gene sequences
2.3 土壤放线菌新颖性分析
以98.65%作为区分两个原核物种的“金标准”[21],即菌株16S rRNA基因序列最大相似率低于98.65%的视为潜在新物种,对山西太子滩温泉放线菌进行新颖性分析,结果见图2。
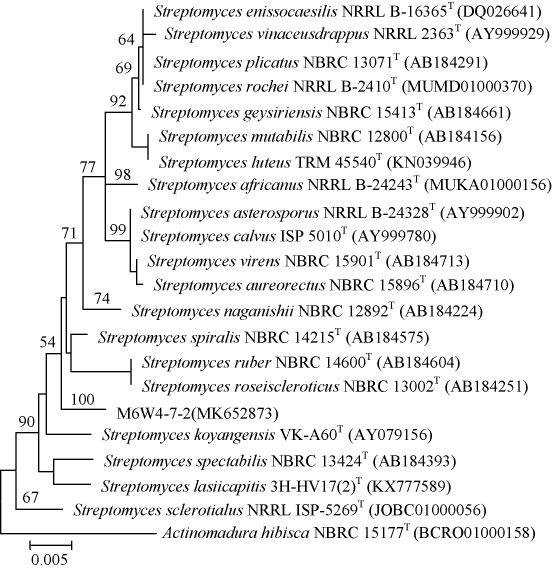
图2 基于16S rRNA基因序列构建的菌株M6W4-7-2的系统进化树
Fig.2 Phylogenetic tree of strain M6W4-7-2 based on 16S rRNA gene sequences
由图2可知,有一株菌的16SrRNA基因序列相似率低于98.65%,菌株编号为M6W4-7-2,其与最近发表菌株Streptomyces ruberNRRC 14600T(GenBank 登录号:AB184604)的相似率为98.58%,在基于16S rRNA基因序列构建的NJ法系统发育树中,菌株M6W4-7-2在Streptomyces属中形成单独的一个分支。基于相似率和进化树分析,推测菌株M6W4-7-2可能为链霉菌科链霉菌属的潜在新种,但是其确切的分类地位需要进行形态特征分析、生理生化特性分析等分类工作来确定。
2.4 NRPS、PKS I和PKS II基因的筛选结果
放线菌的抗菌活性与其产生的次级代谢产物密切相关,而微生物次级代谢产物中的主要结构是通过非核糖体多肽合成酶和聚酮合酶合成[22]。微生物次级代谢产物是重要的药物来源,但是传统方法获得活性次生代谢产物存在代谢量低,很难检测及提纯等诸多因素的限制[23]。近年来,随着生物技术和生物信息学的快速发展,许多微生物活性次生代谢产物的生物合成机制已得到解释。它为通过筛选功能基因预测微生物活性次生代谢产物的结构和类型提供了理论基础[24]。从分离的放线菌菌株中随机筛选出69株菌进行NRPS、PKS I和PKS II生物合成基因的PCR筛选,结果如图3所示,筛选电泳图结果见图4。
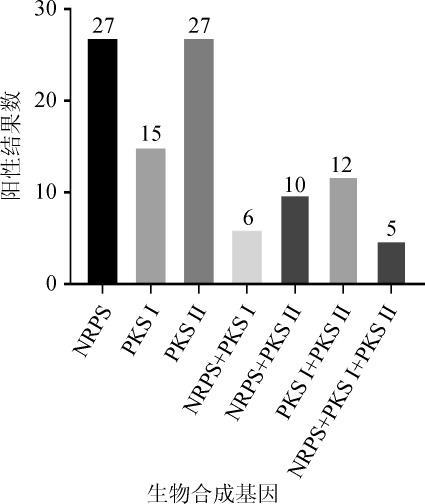
图3 NRPS、PKS I和PKS II基因的PCR筛选结果
Fig.3 Screening results of genes from NRPS,PKS I and PKS II by PCR
其中,NRPS、PKS I以及PKS II基因PCR扩增片段长度分别是在700~800 bp、1 200~1 400 bp和600 bp左右。48株放线菌含有至少一种生物合成基因,总阳性率为69.5%。其中,含有PKS II基因的阳性菌株数目与NRPS基因的阳性菌株数均为27株,含有PKS I基因的放线菌有15株,5株同时含有三种生物合成基因。同时还发现,潜在新种菌株M6W4-7-2同时具有NRPS、PKS II两种抗生素合成基因。因此,对该区域土壤样品的采集以及对菌株M6W4-7-2活性次生代谢产物的分离与鉴定将是下一步研究工作的重点,以期发现该区域土壤中存在的潜在放线菌新种以及从菌株M6W4-7-2中获得新颖的生物活性物质,为放线菌次生代谢产物的进一步开发应用提供理论依据。
图4 NRPS基因(A)、PKS I基因(B)、PKS II基因(C)筛选电泳图
Fig.4 Electrophoregram of genes screening from NRPS(A)、PKS I(B)、PKS II(C)
3 结论
以山西太子滩温泉周围土壤样品为研究对象,选用6种分离培养基对土壤中的放线菌进行分离鉴定与抗生素合成基因的筛选。结果表明,4份土壤样品中共分离得到104株放线菌,分布于放线菌域的3目、5科和12属。随机选取69株放线菌进行功能基因筛选,有69.5%的放线菌具有至少一个生物合成基因簇,阳性率较高。这说明山西太子滩温泉周围土壤中放线菌具有丰富的多样性和抗生素产生潜力,是后期放线菌分离方法研究的高质量材料,能为寻找新活性新结构化合物提供可能存在的潜力菌株。
[1]POOMTHONGDEE N,DUANGMAL K,PATHOM-AREE W.Acidophilic actinomycetes from rhizosphere soil:diversity and proprieties beneficial to plants[J].J Antibiot,2014,68(2):106-114.
[2]GENILLOUD O.Actinomycetes:still a source of novel antibiotics[J].Nat Prod Rep,2017,349(10):1203-1232.
[3]KUNOVA A,BONALDI M,SARACCHI M,et al.Selection of Streptomyces against soil borne fungal pathogens by a standardized dual culture assay and evaluation of their effects on seed germination and plant growth[J].BMC Microbiol,2016,16:272.
[4]BERDY J.Thoughts and facts about antibiotics:where we are now and where we are heading[J].J Antibiot,2012,65(8):385-395.
[5]MOHAMED H,MILOUD B,ZOHRA F,et al.Isolation and characterization of actinobacteria from Algerian Sahara soils with antimicrobial activities[J].Int J Mol Med,2017,6(2):109-120.
[6]于新娟,王莉莉,贾盛佼,等.温泉微生物多样性与酶类分析[J].微生物学通报,2014,41(1):130-135.
[7]JIMENEZ D J,ANDREOTE F D,CHAVES D,et al.Structural and functional insights from the metagenome of an acidic hot spring microbial planktonic community in the colombianandes[J].PLoS One,2012,7(12):e52069.
[8]RUCKMANI A,CHAKRABARTI T.Analysis of bacterial community composition of a spring water from the Western Ghats,India using culture dependent and molecular approaches[J].Curr Protocol Microbiol,2011,62(1):7-15.
[9]BOHORQUEZ L C,DELGADO-SERRANO L,LOPEZ G,et al.In-depth characterization via complementing culture-independent approaches of the microbial community in an acidic hot spring of the Colombian Andes[J].Microb Ecol,2012,63(1):103-115.
[10]肖凯,曹理想,陆勇军,等.广东金山温泉沉积物中原核与真核微生物多样性初步分析[J].微生物学报,2008,48(6):717-724.
[11]徐丽华,杨宇容,郭光远,等.南若干地区土壤放线菌区系及资源考察Ⅷ.滇中高原的放线菌资源[J].微生物学通报,1991,18(6):336-339.
[12]王勇,纪明山,张文革,等.放线菌BOS-009的分离、拮抗活性及鉴定[J].农药,2009,48(2):150-152.
[13]郝召剑.关于山西地热资源开发利用的探究[J].山西建筑,2015,41(5):172-174.
[14]姜怡,段淑蓉,唐蜀昆,等.稀有放线菌的分离方法[J].微生物学通报,2006,33(1):181-183.
[15]ZHOU S Q,HUANG X L,HUANG D Y,et al.A rapid method for extracting DNA from actinomycetes by Chelex-100[J].Biotechnol Bullet,2010,20(2):123-125.
[16]YOONSH,HASM,KWONS,etal.Introducing EzBioCloud:A taxonomically united database of 16S rRNA gene sequences and whole-genome assemblies[J].Int J Syst Evol Microbiol,2017,67(5):1613-1617.
[17]THOMPSON J D,GIBSON T J,PLEWNIAK F,et al.The clustal_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucl Acid Res,1997,25(24):4876-4882.
[18]TAMURA K,STECHER G,PETERSON D,et al.MEGA6:Molecular evolutionary genetics analysis version6.0[J].Mol Biol Evol,2013,30(12):2725-2729.
[19]SAITOU N,NEI M.The neighbor-joining method:a new method for reconstructing phylogenetic trees[J].Mol Biol Evol,1987,4(4):406-425.
[20]AVUSO-SACIDO A,GENILLOUD O.New PCR primers for the screening of NRPS and PKS-I systems in actinomycetes:detection and distribution of these biosynthetic gene sequences in major taxonomic groups[J].Microb Ecol,2005,49(1):10-24.
[21]METSA-KETELA M,SALO V,HALO L,et al.An efficient approach for screening minimal PKS genes from Streptomyces[J].FEMS Microb Lett,1999,180(1):1-6.
[22]冯玲玲,杨芹,张露,等.海南三亚红树林放线菌多样性及抗菌活性[J].中国抗生素杂志,2018,43(11):1355-1363.
[23]LI J W,VEDERAS J C.Drug discovery and natural products:end of an era or an endless frontier?[J]Science,2009,325(5937):161-165.
[24]叶美金,郭海霞,杨财容,等.渐绿木霉基因组中PKS和NRPS基因的多样性分析[J].分子植物育种,2018,16(14):4607-4613.