褐藻胶又称藻酸、海藻酸钠,是一些大型藻类的细胞壁、细胞间质的主要组成成分[1]。褐藻胶是由β-D-甘露糖醛酸(β-D-mannuronate,M)和α-L-古洛糖醛酸(α-L-guluronate,G)两种糖醛酸残基通过β-1,4糖苷键相互连接而成的线性高分子聚合物[2]。依据海藻种类、藻体组织部位、生态环境、季节等的不同,M和G单体的含量比也具有差异[3]。褐藻胶及其衍生物的独特理化性质,如良好的凝胶特性和稳定性,使其被广泛应用于食品、纺织和化工等领域[4]。但天然褐藻胶为分子质量较大的高聚物,黏度高、难溶于水,限制了褐藻胶的应用。褐藻胶寡糖作为一种功能性寡糖,不仅解决了天然褐藻胶应用上的瓶颈问题,且其具有多种生物活性,如免疫调节[5]、抗凝血、抗高血脂[6]、抗氧化[7-9]等,也可以作为一种植物生长调节剂,应用于绿色农业[10-11]。因此,如何有效获得褐藻胶的降解产物——褐藻寡糖势在必行。
目前,褐藻寡糖的制备方法有物理法、化学法和生物酶法[2]。常用的物理法包括辐射、热降解和超声等,但此类方法不能将天然褐藻胶完全降解,且操作繁琐、价格昂贵,容易造成资源浪费。化学法以酸降解为主,但该方法降解条件难以控制,耗时长且产物品质较差,可能对褐藻胶还原端造成一定程度的破坏,进而影响降解产物即褐藻寡糖的生物活性;同时大量化学试剂的应用造成环境污染。生物酶法是采用褐藻胶降解酶通过β-消去反应断裂褐藻胶的糖苷键,产生的新还原端为饱和糖醛酸盐,该方法专一性强、效率高、反应温和、可控性强,不会对环境造成污染,且能为后续研究寡糖活性化学结构提供信息,并满足不同用途的需要[12],故酶法降解逐步成为优先选择的方法。
褐藻胶降解酶来源广泛[13],在细菌、真菌中均有发现[14],其中,海洋来源的微生物是褐藻胶裂解酶的重要来源。由于现阶段报道的微生物来源的褐藻胶降解酶中,仍然存在酶活力低、作用位点单一等缺点[15-16]。因此,筛选能够降解褐藻胶的新菌株,对发掘具有潜在产业化价值的褐藻胶裂解酶具有重要意义。本研究从大连长兴岛采集的腐烂海带样品中,筛选能够降解褐藻胶的菌株,并通过形态观察、生理生化试验及分子生物学技术对其进行鉴定,丰富褐藻胶降解酶新菌种资源,为褐藻胶降解的深入研究及酶法制备褐藻胶寡糖产品奠定了基础。
1 材料与方法
1.1 材料与试剂
1.1.1 样品
腐烂海带:采自辽宁省大连市长兴岛海域。
1.1.2 培养基
无机盐溶液:NaCl 5 g/L,KH2PO4 1 g/L,MgSO4·7H2O 0.5 g/L,CaCl2 0.2 g/L,FeSO4·7H2O 0.02 g/L,去离子水配制。
分离培养基:在1 L无机盐溶液中加入褐藻酸钠7 g,(NH4)SO4 5 g,琼脂20 g。
产酶发酵培养基:在1 L无机盐溶液中加入褐藻酸钠10 g,蛋白胨5 g,酵母粉1 g。
以上培养基均在121℃条件下高压蒸汽灭菌20 min。
1.1.3 试剂
细菌基因组抽提试剂盒:上海碧云天生物技术有限公司;BM5000脱氧核糖核酸(deoxyribonucleic acid,DNA)Marker:北京博迈德科技发展有限公司;聚合酶链式反应(polymerase chain reaction,PCR)所用试剂:宝生物工程(大连)有限公司;褐藻胶(分析纯):上海阿拉丁生化科技股份有限公司;HBI细菌生理生化鉴定条:青岛海博生物技术有限公司;革兰氏染色试剂盒:北京索莱宝生物科技有限公司;其他所有试剂均为国产分析纯。
1.2 仪器与设备
ML104/02电子天平:梅特勒-托利多国际贸易(上海)有限公司;YXQ-LS-50sII立式压力蒸汽灭菌锅、SPX-250B恒温培养箱:上海博迅实业有限公司;MS-S磁力搅拌器:大连元博科学器材有限公司;QHZ-98A全温振荡培养箱:太仓市华美生化仪器厂;Hybaid型PCR仪:美国Thermo Fisher Scientific公司;CR-22E高速冷冻离心机:日本HITACHI公司;DYY-6C型电泳仪:北京市六一仪器厂;Multiskan Ascent酶标仪:芬兰Labsystems公司;IX73型显微镜、FV1000MPE双光子共聚焦显微镜:日本Olympus公司;GelDoc-It型凝胶成像仪:美国UVP公司。
1.3 实验方法
1.3.1 菌株的分离纯化
称取样品10g于装有90 mL无菌水及转子的三角瓶中,于磁力搅拌器上600 r/min条件下搅拌10 min,待样品充分分散后,静置10 min,转移至超净台,取上清液制备10倍系列稀释样品匀液,稀释度为10-1~10-9。分别取各梯度稀释液0.2 mL涂布于以褐藻酸钠为唯一碳源的分离培养基平板,于30℃恒温培养箱中倒置培养2~4 d,挑取颜色及菌落形态不一的菌株进行多次划线,分离纯化。
1.3.2 产褐藻胶降解酶菌株的筛选
挑取单菌落分别接种于装有7.5 mL产酶发酵培养基的50 mL离心管中,于37℃、150 r/min条件下培养12 h,作为种子液备用。以2%(V/V)的接种量将种子液接种于装有50 mL产酶发酵培养基的250 mL的三角瓶中,于37℃、150 r/min条件下培养24 h,测定褐藻胶降解酶活力,筛选褐藻胶降解酶活力高的菌株。
1.3.3 褐藻胶降解酶活力测定
采用3,5-二硝基水杨酸(3,5-dinitrosalicylic acid,DNS)法[17]测定褐藻胶降解酶活力。以OD540nm值(y)为纵坐标,葡萄糖含量(x)为横纵标,绘制葡萄糖标准曲线。葡萄糖标准曲线回归方程为y=5.194x-0.026 7,相关系数R2=0.999 3,线性关系良好,可用于褐藻胶降解酶活力的测定。
发酵液经12 000 r/min离心5 min,上清液即为粗酶液。取500 μL粗酶液与500 μL 0.3%褐藻胶于37℃水浴锅中反应1 h,然后于沸水浴中加热10 min终止反应,12 000 r/min离心2 min,取400 μL上清液加入300 μL DNS试剂,沸水浴加热10 min后立即冷却,加无菌水定容至1.5 mL,取200 μL于波长540 nm处测定吸光度值。以沸水浴10 min高温进行灭活的酶作为对照组,每组实验设定3个平行。根据葡萄糖标准曲线回归方程计算出还原糖含量,从而计算出酶活力大小。
酶活力单位定义:每分钟催化褐藻胶产生1 μmol还原糖(以葡萄糖计)所需要的酶量为1个酶活力单位(U/L)。
1.3.4 菌株的鉴定
形态观察:将筛选菌株划线于分离培养基平板上,30℃倒置培养24 h,肉眼观察单菌落形态。然后挑取单菌落于油镜下观察其显微形态,并进行革兰氏染色观察。
生理生化试验:采用HBI细菌生理生化鉴定条对菌株的生理生化特征进行测定,观察并记录实验结果。
分子生物学鉴定:利用细菌基因组抽提试剂盒提取筛选菌株的基因组DNA,以其为模板,利用16S rDNA通用引物(27F:5′-AGAGTTTGATCCTGGCTCAG-3′;1492R:5′-GGTTACCTTGTTACGACTT-3′)[18]进行PCR扩增。PCR扩增体系:10×PCR 缓冲液10 μL,基因组1.5 μL,10 μmoL/L通用引物各4 μL,脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)Mixture8μL,r Taq酶1μL,双蒸水(ddH2O)补充至100 μL,混匀。PCR扩增条件:94℃预变性2 min;94℃变性30 s,55℃退火30 s,72℃延伸1 min 30 s,共进行30个循环;72℃再延伸5 min。PCR扩增产物经琼脂糖凝胶电泳检测正确后,切胶回收,送至北京六合华大基因科技有限公司进行测序。将测序结果与EZBioCloud数据库中模式菌株的核酸序列进行比对分析,选取亲缘关系相近的模式菌株,通过MEGA5.0软件[19]中的邻接(neighbor-joining,NJ)法构建系统发育树后进行分析。
2 结果与分析
2.1 产褐藻胶降解酶菌株的筛选
以褐藻胶为唯一碳源,共筛选到8株能够稳定生长的菌株,编号为E6-1、E6-2、E5、E8、F3、F1、E10、F4。将其分别转接于发酵培养基,采用DNS法对菌株降解褐藻胶的能力进行测定,结果见图1。
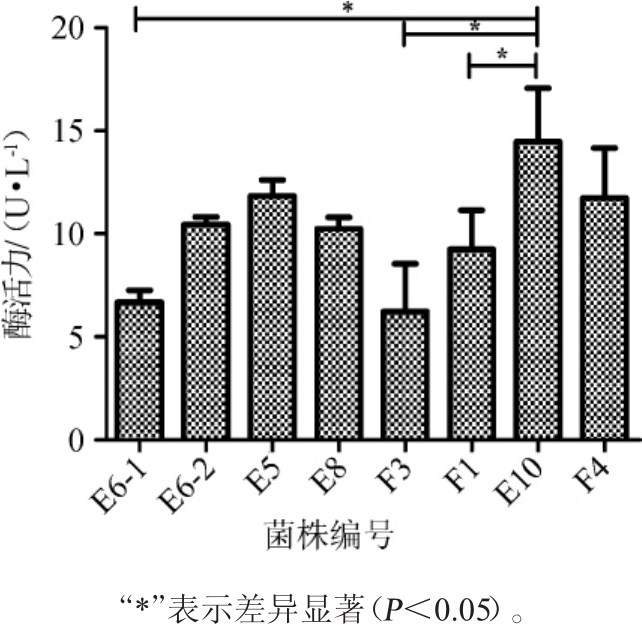
图1 不同菌株降解褐藻胶能力的比较
Fig.1 Comparison of alginate degradation ability between different strains
由图1可知,筛选得到的8株菌株均具有降解褐藻胶的能力。其中,菌株E-10所产褐藻胶降解酶活力最高,为14U/L,其褐藻胶降解酶活力显著高于菌株E6-1(6 U/L)、F1(9 U/L)和菌株F3(6 U/L)(P<0.05);其次为菌株E5(11 U/L);菌株F3的褐藻胶降解酶活力最低,为6 U/L。LANGE B等[20]研究表明,克雷伯氏菌来源的褐藻胶降解酶作用底物为多聚葡萄糖醛酸组成的褐藻胶,特异性断裂G-G之间的糖苷键,本文中所使用的底物为G和M交替连接组成的褐藻胶,故此可能为E6-1和F3酶活力较低的原因。现在,通过基因重组或酶的定向进化技术已经实现对酶的改造和酶活力的提升[21-22],本研究所筛选获得的菌株E-10虽活性较低,但是作为一种新的产酶菌株,可通过上述生物技术对其进一步研究以拓宽褐藻胶降解酶酶源。因此,筛选菌株E-10进行深入研究。
2.2 菌株鉴定
2.2.1 形态学鉴定
菌株E-10在分离培养基上的菌落形态见图2A、细胞形态见图2B,细胞经革兰氏染色后的细胞形态见图2C。

图2 菌株E-10的菌落(A)和细胞(B和C)形态
Fig.2 Colonial(A)and cell(B and C)morphology of strain E-10
由图2A可知,菌株E-10的菌落呈圆形,边缘整齐,初始生长表面光滑透明、湿润,后期菌落表面呈乳白色半透明,且有干粉状物质位于菌落表面;由图2B可知,菌株E-10的细胞为短小棒状;由图2C可知,菌株E-10经革兰氏染色后为紫色,说明菌株E-10为革兰氏阳性菌。
2.2.2 生理生化试验结果
根据《细菌系统鉴定手册》[23],对菌株E-10的生理生化特征进行测定,结果见表1。
表1 菌株E-10的生理生化特征
Table 1 Physiological and biochemical characteristics of strain E-10

注:“+”表示结果呈阳性;“-”表示结果呈阴性。
项目 结果 项目 结果苯丙氨酸酶西蒙氏枸橼酸盐吲哚鸟氨酸脱羧酶肉汤氨基酸脱羧酶对照D-甘露醇水杨苷- - - + - + +明胶尿素酶木糖麦芽糖脂酶(玉米油)七叶苷水解+-++-+
由表1可知,明胶试验呈阳性,说明菌株E-10具有运动性;菌株E-10产鸟氨酸脱羧酶;能利用D-甘露醇、水杨苷、木糖、麦芽糖以及七叶苷;不产苯丙氨酸酶、尿素酶和脂酶;西蒙氏枸橼酸盐、吲哚试验呈阴性。结合形态观察结果,初步判定菌株E-10为莱茵海默氏菌属(Rheinheimera)。
2.2.3 分子生物学鉴定
菌株E-10的16S rDNA PCR扩增产物经琼脂糖凝胶电泳检测,结果见图3。
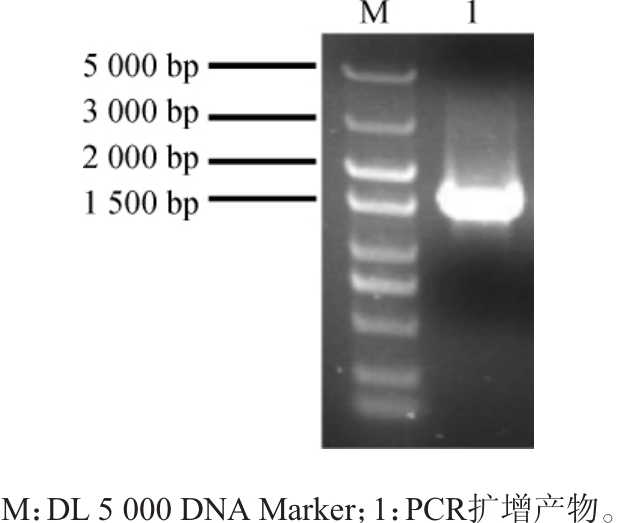
图3 菌株E-10 16S rDNA PCR扩增产物琼脂糖凝胶电泳结果
Fig.3 Agarose gel electrophoresis result of 16S rDNA PCR amplification product of strain E-10
由图3可知,PCR扩增产物的碱基长度约为1 500 bp,与目的基因的碱基长度一致,且条带较为完整,说明PCR扩增成功。
菌株E-10 16S rDNA序列与EZBioCloud数据库中模式菌株的核苷酸序列进行比对发现,菌株E-10的16S rDNA基因序列与莱茵海默氏菌属(Rheinheimera)的相似度较高,与水莱茵海默氏菌(Rheinheimeraaquimaris)SW-353(EF076757)、日本粳稻莱茵海默氏菌(Rheinheimerajaponica)KMM 9513(LC004727)、太平洋莱茵海默氏菌(Rheinheimerapacifica)KMM1406(AB073132)、明岑贝格莱茵海默氏菌(Rheinheimera muenzenbergensis)E49(JQ922424)的相似度分别为99.93%、98.51%、98.50%、98.43%。为了进一步确定菌株E-10的分类地位,选取相似度较高的模式菌株,采用MEGA 5.0软件中的NJ法构建系统发育树,结果见图4。

图4 基于16S rDNA序列菌株E-10的系统发育树
Fig.4 Phylogenetic tree of strain E-10 based on 16S rDNA sequences
由图4可知,菌株E-10与水莱茵海默氏菌(Rheinheimera aquimaris)(EF076757)聚于同一分支,亲缘关系最近,因此,将菌株E-10鉴定为一株水莱茵海默氏菌(Rheinheimera aquimaris)。
3 结论
本研究以褐藻胶为唯一碳源,从长兴岛采集的腐烂海带中筛选得到能够高效降解褐藻胶的菌株E-10,褐藻胶降解酶活力为14 U/L。通过形态观察、生理生化试验及分子生物学鉴定菌株E-10为水莱茵海默氏菌(Rheinheimera aquimaris)。首次报道了莱茵海默氏菌具有降解褐藻胶的能力,此菌株的筛选和鉴定丰富了褐藻胶降解酶新微生物酶源,对发掘具有潜在产业化价值的褐藻胶降解酶具有重要意义。
[1]GACESA P.Enzymic degradation of alginates[J].Int J Biochem Cell Biol,1992,24(4):545-552.
[2]ZHU B W,CHEN M,YIN H,et al.Enzymatic hydrolysis of alginate to produce oligosaccharides by a new purified endo-type alginate lyase[J].Mar drug,2016,14(6):108-119.
[3]AARSTAD O A,TONDERVIK A,SLETTA H,et al.Alginate sequencing:an analysis of block distribution in alginates using specific alginate degrading enzymes[J].Biomacromolecules,2012,13(1):106-116.
[4]YONG T J,MUNUSAMY Y,CHEE S Y.The effect of acrylic acid and acrylamide graft-copolymerization on the properties of sodium alginatebased superabsorbent polymer[J].Int J Gen Eng Technol,2015,4(1):1-14.
[5]王庭欣,赵文,蒋东升,等.海带多糖对小鼠免疫功能的调节作用[J].卫生毒理学杂志,2000,14(2):75.
[6]王庭欣,王庭祥,庞佳宏.海带多糖降血糖、血脂作用的研究[J].营养学报,2007,29(1):99-100.
[7]胡博旸,聂莹,孙路,等.酸降解法制备褐藻寡糖抗氧化性的研究[J].食品工业科技,2016,37(10):130-134.
[8]S,EN M,ATIK H.The antioxidant properties of oligo sodium alginates prepared by radiation-induced degradation in aqueous and hydrogen peroxide solutions[J].Radiat Phys Chem,2012,81(7):816-822.
[9]孙丽萍,薛长湖,许家超,等.褐藻胶寡糖体外清除自由基活性的研究[J].中国海洋大学学报,2005,35(5):811-814.
[10]ZHANG Y,YIN H,ZHAO X,et al.The promoting effects of alginate oligosaccharides on root development in Oryza sativa L.mediated by auxin signaling[J].Carbohyd Polym,2014,113(26):446-454.
[11]TRAN C V,NGUYEN T T,KIM D.Marine alginate oligosaccharides-a promising biomaterial:current use and future perspectives in food industry and pharmaceutical applications[J].Vietnam J Sci Technol,2018,56(2):133-147.
[12]RAVANAL M C,SHARMA S,GIMPEL J,et al.The role of alginate lyases in the enzymatic saccharification of brown macroalgae,Macrocystis pyrifera and Saccharina latissima[J].Algal Res,2017,26:287-293.
[13]MARTIN M,PORTETELLE D,MICHEL G,et al.Microorganisms living on macroalgae:diversity,interactions,and biotechnological applications[J].Appl Microbiol Biot,2014,98(7):2917-2935.
[14]李丽妍,管华诗,江晓路,等.海藻工具酶—褐藻胶裂解酶的研究进展[J].生物工程学报,2011,27(6):838-845.
[15]罗丹丹,薛永常.褐藻胶裂解酶的研究进展[J].生物学杂志,2016,33(6):95-98.
[16]黄李淑馨,刘钢,岳敏,等.产褐藻胶裂解酶菌株的分离与鉴定[J].食品工业科技,2013,34(23):147-151.
[17]MILLER G L.Use of dinitrosalicylic acid reagent for determination of reducing sugar[J].Anal Chem,1959,31(3):426-428.
[18]HOGG J C,LEHANE M J.Identification of bacterial species associated with the sheep scab mite(Psoroptes ovis)by using amplified genes coding for 16S rRNA[J].Appl Environ Microbiol,1999,65(9):4227-4229.
[19]TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Mol Biol Evol,2011,28(10):2731-2739.
[20]LANGE B,WINGENDER J,WINKLER U K.Isolation and characterization of an alginate lyase from Klebsiella aerogenes[J].Arch Microbiol,1989,152(3):302-308.
[21]李树,张伟,赵春梅.易错PCR技术定向进化褐藻胶裂解酶Alg-2的研究[J].食品科学,2019,40(4):146-151.
[22]KIM S H.Enhancing the alginate degrading activity of Streptomyces sp.strain M3 alginate lyase by mutation[J].J Life Sci,2012,22(1):7-15.
[23]东秀珠,蔡妙英.细菌系统鉴定手册[M].北京:科学出版社,2001:242.