在白酒酿造领域,一般认为甲烷菌利用氢气(H2)还原二氧化碳(CO2)产生甲烷(CH4),使菌群代谢平衡向促进有机酸、酒精生成的方向转化,提高了酒醅中的酸和酯的含量,也提高了产酒率[1-2]。根据“种间氢转移”原理,甲烷菌属利用己酸菌发酵产生的H2用于产生甲烷,使己酸菌消除了氢的抑制而促进产酸,继而提高己酸乙酯含量。但是上述过程只能在窖池的有限的位置发生,因为只有一定深度条件下的窖池底部密封发酵一段时间以后才可能满足甲烷菌属的厌氧生长条件。甲烷菌促进白酒发酵质量的途径肯定是多方面的,理论上不同的甲烷菌属在窖泥-黄水-酒醅界面的厌氧发酵过程中发挥的作用与方式会有所不同。但是这些甲烷菌属到底通过哪些生物学途径发挥作用、甲烷菌属之间存在哪些重要的相互作用等问题还需要大量的研究[1,3-4]才能逐步搞清楚,需要检测各个甲烷菌属在发酵动力过程中的含量变化。所以设计一批针对各个甲烷菌属的属或种的特异性引物很有必要。
张会敏[5]的高通量测序结果显示,古井贡酒窖泥包含的甲烷菌属至少有甲烷八叠球菌属(Methanosarcina)(1-458),未知甲烷菌-1(unkown genus-1)(1-2),未知甲烷菌-2(unkown genus-2)(2-8),产甲烷袋菌属(Methanofollis)(3-896),甲烷囊菌属(Methanoculleus)(16-14155),甲烷粒菌(Methanocorpusculum)(6-2180),甲烷球菌属(Methanoc-occus)(2-10),甲烷短杆菌属(Methanobrevibacter)(3-903)以及甲烷杆菌属(Methanobacterium)(5-1514),括号里面的两个数字分别是各自操作分类单元(operational taxonomy unit,OTU)的个数及其序列总个数,显示古井贡酒窖泥中甲烷囊菌属(Methanoculleus)这个属的序列复杂度及菌属含量在全部甲烷菌中相对较大。在窖泥质量评估的方法体系中,甲烷菌属的属特异性检测方法属于重要的建设内容。
甲烷菌属在浓香型白酒窖泥中的含量分布情况比较类似,既有的检测报道一般是3~6个属。如五粮液酒的窖泥中报道了4个优势甲烷菌属,分别为甲烷细菌属(Methanobacterium)、甲烷短杆菌属(Methanobrevibacter)、甲烷囊菌属(Methanoculleus)和甲烷八叠球菌属(Methanosarcina),其中Methanosarcina可同时利用乙酸和氢气[6]。四川绵阳丰谷酒业的窖泥中至少含有5个甲烷菌属,分别是Methanoculleus、Methanosarcina、Methanobacterium、甲烷粒菌属(Methanocorpusculum)和甲烷微菌属(Methanomicrobium)[7-8]。四川泸州某公司白酒生产窖池中发现至少有6个甲烷菌属,分别是Methanobrevibacter、甲烷鬃毛菌属(Methanosata)、Methanoculleus、Methanobacterium、Methanocorpusculum和甲烷丝菌属(Methanothri)[9],而泸州老窖的百年窖泥中至少含有4类不同的甲烷菌属,分别是甲烷短杆菌属Methanobrevibacter、甲烷杆菌属(Methanobacterium)、甲烷鬃菌属(Methanosaeta)和Methanoculleus[10]。显然,不同的浓香型白酒在日常质控过程中需要对不同组合的甲烷菌属设计特异性(快速)检测技术。
甲烷菌的特异性检测已经有不少报道,韦艳霞等[11]研究根管感染中产甲烷古细菌的多样性,利用甲基辅酶M还原酶(methylcoenzymeMreductase,MCR)基因α亚基(mcrA)的特异性引物,发现所选样本中产甲烷古细菌的多样性局限于类口腔甲烷短杆菌序列型。华金玲等[12]也是利用MCR基因特异性基因引物发现黄淮白山羊瘤胃中的甲烷菌主要是瘤胃甲酸甲烷杆菌。兰阿峰等[13]利用甲烷菌16S rDNA特异性引物Met86F和Met1340R测定了野生秦岭羚牛瘤胃中甲烷菌种类,发现至少包括3个科,若干个属,其多样性比较丰富。
利用MCR等基因设计的甲烷菌特异性引物,以及利用甲烷菌16SrDNA特异性引物的检测方法,理论上适合于大多数产甲烷菌,但是难以把不同的甲烷菌种属区分开来。在白酒研究领域也有若干甲烷菌特异性检测技术的报道,比如罗青春等[14]运用已知特异性引物[15-17]和荧光定量聚合酶链式反应(polymerase chain reaction,PCR)测定了不同年份川酒窖泥中几种主要产甲烷菌的含量,发现随着窖泥年份的增加,3种产甲烷菌属甲烷鬃毛菌属(Methanosaeta)、甲烷八叠球菌属(Methanosarcina)及甲烷杆菌属(Methanobacter)的含量均呈不同幅度的增加趋势。不过对这些已知特异性引物的原始文献进行勘察时发现它们是目(order)分类层面上的特异性引物,而且并未交待引物特异性的基本验证数据。
本研究以甲烷囊菌属为例提供了一个基于16S rDNA的属特异性单核苷酸多态性(single nucleic polymorphism,SNP)和半随机引物(semi-arbitrarily primer,SAP)技术[18-20]设计属特异性PCR引物的方法。利用本研究的方法原则上可以设计任意其他菌属在特定复杂菌群背景下的特异性引物,一般应该都可以找到比较满意的实用引物,这对于白酒酿造过程中的质量控制有明显的潜在应用价值。
1 材料与方法
1.1 材料与试剂
1.1.1 窖泥样本
共使用了18个古井窖泥样本,包括A厂区6个(40年优质窖池,样本1~6),B厂区3个(百年优质窖池,样本7~9),C厂区9个(5年普通窖池,样本10~18)。
1.1.2 试剂
NPK02和NPK62 PCR试剂盒:威海晓东生物技术有限公司;Ezup柱式基因组提取试剂盒、Marker D:上海生工生物有限公司;特异性引物:上海生工生物有限公司合成;其他生化试剂均购自上海生工生物有限公司。
1.2 仪器与设备
TDL-60B台式离心机:上海安亨科学仪器厂;TP600 PCR仪:大连TaKaRa公司;StepOne实时定量PCR(real-time quantitative PCR,RT-QPCR)仪:美国ABI公司;JY300C核酸电泳仪:北京君意东方电泳设备有限公司;WD-9413C凝胶成像仪:北京市六一仪器厂;HC-3108R(-80℃)低温冰箱:海尔集团公司;HC-3018R高速冷冻离心机:安徽中科中佳科学仪器有限公司。
1.3 方法
1.3.1 窖泥宏基因组的提取
利用Ezup柱式基因组提取试剂盒提取窖泥的宏基因组脱氧核糖核酸(deoxyribonucleicacid,DNA),来自200mg的窖泥的基因组DNA溶于100 μL TE缓冲液,再进一步稀释至500 μL作为PCR或RT-QPCR的模板,RT-QPCR测定的是500 μL该溶液中甲烷囊菌属16S rDNA的拷贝数浓度。
1.3.2 16S rRNA序列比对分析及甲烷囊菌属SNP位点确认
古井窖泥微生物菌群中含量最高的30个属及甲烷囊菌属的16S rRNA全长序列源自于高通量测序实验[21];傅里叶转换多重序列比对(multiple alignment using fast Fourier transform,MAFFT)的一个重要优点是其比对结果中序列越相近则排列距离越接近,这样可以对几百条到几千条高度相似的序列快速进行分组,非常有利于大量序列中SNP位点的寻找。使用MAFFT对上述总共5 000多条序列和从Genbank数据库搜索得到的210条甲烷囊菌属(Methanoculleus)的16S rRNA(有些是全长)基因序列进行比对,选出各自的代表性序列,以进行各属之间的SNP识别分析。SNP标记作为第三代分子标记,在分子遗传学、药物遗传学、法医学以及疾病的诊断和治疗等方面发挥着重要作用[22-26]。借助MAFFT多序列比对工具对序列进行比对寻找SNP位点,对列出的所有SNP位点,返回甲烷囊菌属内与自身其余序列进行SNP查证,要求该位点碱基与其余序列该位置碱基绝大部分相同;再与古井菌群各属其余序列比对查证,要求与该位置碱基绝大部分不同,从而选出理论上效果最好的SNP位点。
1.3.3 甲烷囊菌属特异性引物设计
PCR引物的碱基长度需在20~25 bp之间,退火温度需在60℃左右,碱基GC含量应在40%~60%,以此保持引物的稳定性。根据SAP理论的一般设计方法,引物的3′端的末端应该是SNP位点,在SNP位点的前一位点(引物的倒数第二位)人为的引入错配,以致于引物与靶序列(甲烷囊菌属)只有在倒数第二位有错配,而与非靶序列在最后两个碱基位点都是错配。因此,可以通过合适的扩增条件使得非靶序列不能得到有效的扩增。本研究基于该理论在错配的位置做了稍许调整,SNP位点只需要放置在引物3′端的末位,不一定是最后一位,可以是倒数第二位;错配位置也放在末位,不一定是倒数第二位也可以是倒数第三位附近,宗旨是让引物与靶序列在引物的3′端末端几个碱基的配对强度大于非靶序列,在这方面实验室已经做了若干摸索实验[18]。引物的特异性经过PCR扩增及产物测序验证,确定特异性无误的引物可以进一步用于RT-QPCR再验证。
1.3.4 引物特异性检测
利用PCR对两对引物的特异性进行验证。18个窖泥基因组DNA等量混合,以其为模板进行PCR扩增,PCR扩增体系:NPK02 2×buffer 6 μL,物种特异性引物1.5 μL,模板1 μL,Taq酶0.3 μL,双蒸水(ddH2O)3.2 μL。PCR扩增程序:94℃预变性4 min;94℃变性30 s,60℃退火30 s,72℃延伸45 s,37个循环;72 ℃再延伸2 min。取8 μL反应液进行1%琼脂糖凝胶电泳。
1.3.5 RT-QPCR定量
要求RT-QPCR的熔解曲线为单峰且没有明显的二聚体扩增。以设计好的两对甲烷囊菌属特异性引物PCR扩增得到的产物作为RT-QPCR定量的标准物质,其母液(质量浓度为ng/μL)经10倍系列稀释(10-1~10-)7,后,选择10-2~10-7的稀释液制作标准曲线。摸索到的最佳RT-QPCR条件为:第1对引物RT-QPCR扩增程序:95℃、4min;95℃、30s,60℃、30s,72℃、5s,42个循环;72℃、2min。第2对引物QPCR扩增程序:95℃、4 min;95 ℃、30 s,65 ℃、30 s,72 ℃、55 s,42个循环;72℃、2 min。2对引物RT-QPCR扩增体系:NPK62 2×Buffer 6 μL,上下游引物(2 μmol/L)各1.75 μL,窖泥宏基因组DNA模板4 μL,Taq酶0.25 μL。每对引物在最佳条件下重复扩增3次,选择其中一个最具代表性的结果绘图和计算。
DNA拷贝量计算公式如下:
式中:C为测定得到的各个样本中甲烷囊菌属DNA片段的浓度,ng/μL;L为PCR扩增产物碱基长度,bp。
2 结果与分析
2.1 SNP位点选取及引物设计
通过使用傅里叶转换多序列比对在线多序列比对工具,分析了甲烷囊菌属在古井窖泥中含量前30属中的SNP情况,通过各属自身的序列比对,挑选出各属的代表序列,如表1所示。
表1 属代表序列选取结果
Table1 Selection results of genus-representative sequence

注:“*”表示该菌属未能确定到属(k_Bacteria、p_Firmicutes、c_Clostridia、o_Clostridiales、f_Lachnospiraceae)。
?
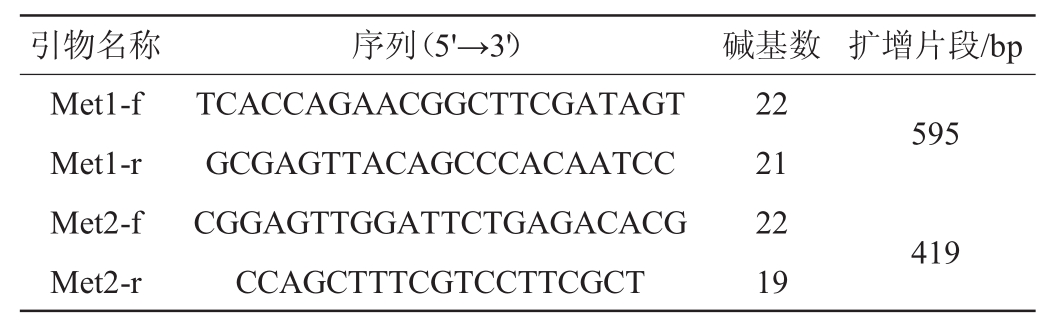
由表1可知,古井菌群30个属,每个属2条代表序列,共60条;甲烷囊菌属代表序列1条;对61条序列进行比对,初步找出了甲烷囊菌的114个SNP位点。首先对SNP位点进行甲烷囊菌属内可行性验证,保留了41个SNP位点;然后对41个SNP位点进行属外可行性验证,与古井菌群30个属所有序列(共计5 499条)比对验证后,找出了3个效果最好的SNP位点,SNP位点5′-TCCAGGCCC-3′的同属代表性是94.3%,他属代表性是0.4%;位点5′-AAAACTTT-3′的同属代表性是94.8%,他属代表性是0.8%;位点5′-TTCGACAGT-3′的同属代表性是94.8%,他属代表性是0.4%。随机选择SNP位点5′-TTCGACAGT-3′设计两对引物,其信息见表2。
表2 两对引物信息
Table2 Information of two pairs of primers
?
2.2 引物特异性的验证
以18个窖泥样本的混合宏基因组DNA[19]为模板,采用设计好的两对引物进行PCR扩增,PCR扩增产物经琼脂糖凝胶电泳检测,结果如图1所示。由表2可知,两对引物的预计扩增片段大小分别为595 bp和419 bp;而图1显示的电泳条带大小与预测值相一致,表明这两对引物能够扩增出预计的核酸序列,满足特异性的基本要求。扩增产物需要测序来进一步验证扩增特异性。将PCR扩增产物纯化后送至上海生工进行测序。分别使用引物Met1-f和Met2-f进行单向测序,测序结果见表3。测序所得的碱基长度分别为545bp和357 bp。考虑到引物测序一般从引物位置后面30~50个碱基处才能开始得到可读序列,实际的PCR产物大小需要在545bp和357bp基础上再加上30~50个碱基,这样就完全符合595 bp和419 bp的预期大小。将表3序列在美国国家生物技术信息中心(national center of biotechnology information,NCBI)中进行BLAST验证,确认其均为甲烷囊菌目标序列。
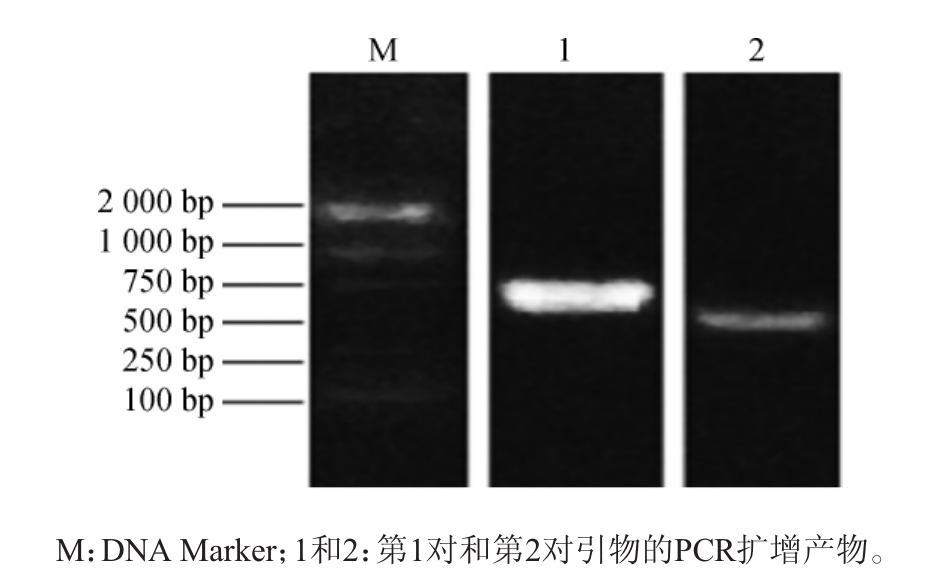
图1 两对引物特异性验证结果
Fig.1 Verification results of specificity of two pairs of primers
表3 PCR产物单向测序结果
Table3 PCR product sequencing results using forward primers

?
2.3 实时荧光定量PCR
RT-QPCR的熔解曲线是衡量引物特异性的一个重要指标。本研究预期的熔解曲线是一个主峰下有很多熔点非常接近的次峰。这是因为扩增出来的是一组大小和熔点都非常接近的DNA片段。两对引物的熔解曲线见图2,RT-QPCR定量计算结果见图3。由图2可知,Met1为引物的熔解曲线中含有少量(Tm)值有明显偏差的小峰;虽然对定量结果影响不大,但是值得在应用过程中加以关注和继续评估。由图3可知,两对引物的定量结果的基本趋势也是一致的,就是甲烷囊菌含量在样本中含量的明显趋势为老窖池>新窖池。即使在老厂区的窖泥样本中也有一些甲烷囊菌含量偏低(如3、5、6号样本),其中可能的具体原因有待于确认,可能来自样本取样的不均匀性。每个窖池的窖泥虽然都是9点取样并混合[21],但是窖泥粘性很大,很难混合均匀;加上本次研究取样时每个混合样本中只取出200 mg,很可能带来一定的样本均匀性问题。
从窖泥宏基因组的高通量测序结果来看[17],甲烷囊菌在样本中的丰度排名为第7位,属于高丰度菌属。但是其他种类的甲烷菌属丰度排名都在45名以后,比如Methanocorpusculum排第45名,Methanobacterium排第62名,Methanobrevibacter排第86名,Methanofollis排第88名。这个丰度排名数据提示Methanoculleus应该是浓香型白酒酿造过程中“种间氢转移”的主要参与菌属。本研究对18个古井窖泥样本的初步检测结果表明,老窖池窖泥对应的基因组DNA溶液中甲烷囊菌DNA的拷贝数大概在2000×108个/L(图3),最终换算的结果是每毫克窖泥中甲烷囊菌DNA目标片段的个数大概为1.5×106个。这个含量的准确度还可以在后续的研究中进一步验证。不过,未来确实需要检测发酵过程中不同甲烷菌属的动态变化过程,同时要逐步研究还需要哪些其他的厌氧菌协同完成上述“种间氢转移”过程。
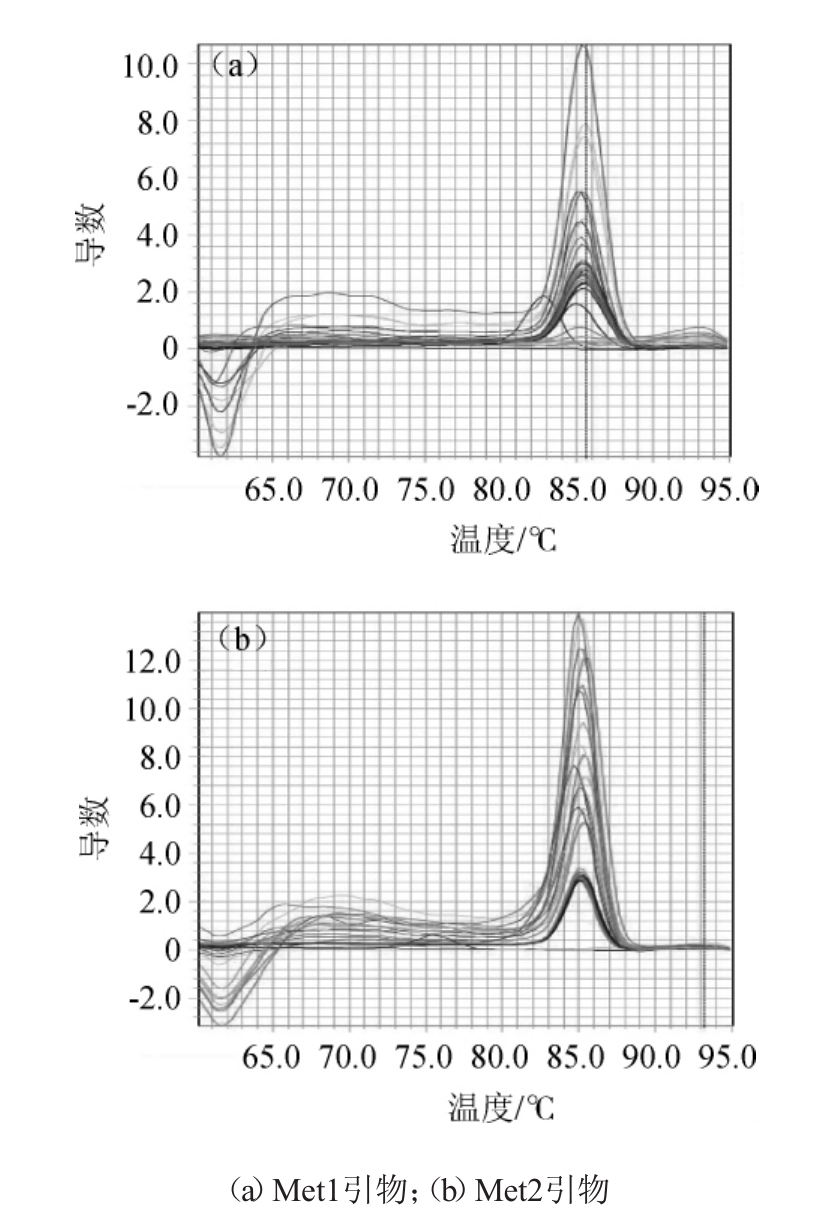
图2 两对引物实时定量聚合酶链反应的熔解曲线
Fig.2 Melting curves of real-time RT-QPCR of two pairs of primers
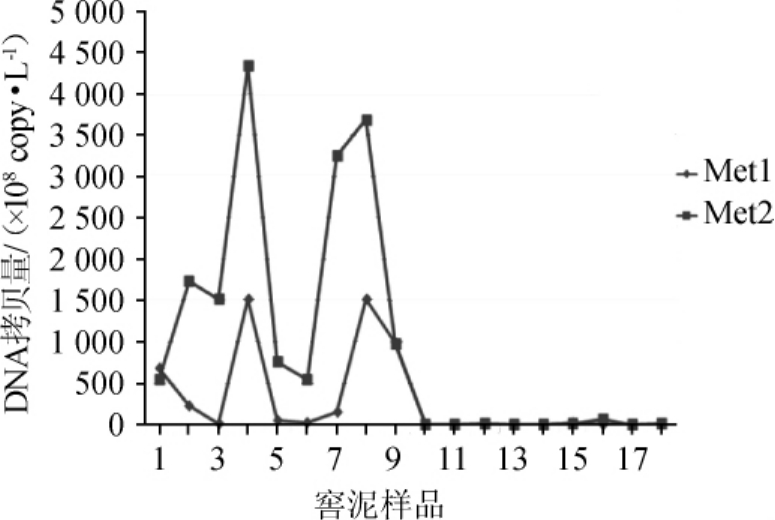
图3 两对引物的实时定量聚合酶链反应结果
Fig.3 Results of RT-QPCR of two pairs of primers
已知甲烷囊菌的特异性引物非常少[12]。本研究的两对引物为研究人员提供了更多选择。鉴于甲烷菌属在浓香型白酒发酵过程中的作用非常独特,后续需要对不同的甲烷菌属都要设计更多更好的PCR引物。本研究并没有比较已知甲烷囊菌引物与2对引物在测定18个样本中的特异性差异和QPCR定量结果差异。主要原因在于,一是菌群中既有的基因组序列背景信息很有限,这决定了引物的特异性还达不到100%;另外,不同白酒的不同发酵样本中菌群的组成差异非常大,而几对引物面向不同菌群基因组背景时各自表现出来的特异性程度肯定是不同的。所以到底哪一对引物的特异性最好取决于使用的具体的样本,研发人员需要在实际的测试过程中找到适合所用样本的最佳引物。
3 结论
本研究以古井贡酒窖泥中的甲烷囊菌为研究对象,通过使用傅里叶转换多序列比对(MAFFT)工具,分析了甲烷囊菌属在古井贡酒窖泥中含量前30属中的SNP情况,选出了3个效果最好的SNP位点,并据此设计出两对甲烷囊菌属特异性引物。特异性检测表明,两对引物从窖泥混合宏基因组样本中扩增出来的两段序列皆属于甲烷囊菌属,可以用于RT-QPCR测定古井贡酒窖泥样本中该菌属的含量,发现老窖池优质窖泥中的甲烷囊菌含量最多,是新窖池普通窖泥4 000多倍。考虑到甲烷囊菌在浓香型白酒窖泥中比较广泛存在,本研究所报道的两对引物应该可以在其他不同种类的白酒窖泥中所测试和应用。另外,本研究提供的甲烷囊菌属的特异性测定方法与浓香型白酒窖泥质量评价有直接的相关性。窖泥质量一般从窖泥感官、理化指标、微生物组成及基础酒产质量等方面综合评判。单独任何一种因素往往只能作为辅助评判的工具。研究组前期多项研究已经发现古井贡酒老窖池的优质窖泥其甲烷囊菌平均含量一直比新窖池的普通窖泥明显要高很多,所以通过窖泥中甲烷囊菌DNA目标片段的定量测定可以辅助评估窖泥的质量。
[1]李俊辉,刘英杰,隋丽娜,等.浓香型白酒增加己酸乙酯降低乳酸乙酯的研究进展[J].中国酿造,2019,38(1):1-4.
[2]李芳莉.浓香型白酒窖泥微生物群落结构分析及其对原酒风味物质的影响[D].郑州:郑州轻工业学院,2017.
[3]李付丽,吴鑫颖,王晓丹,等.微生物技术在浓香型白酒增香方面的应用[J].中国酿造,2014,33(1):9-13.
[4]姚万春,唐玉明,任道群,等.功能性人工窖泥培养配方筛选研究[J].中国酿造,2014,33(8):63-66.
[5]张会敏.古井贡酒微生物群落结构及其与主要风味物质的关联研究[D].哈尔滨:哈尔滨工业大学,2016.
[6]赵东,郑佳,彭志云,等.高通量测序技术解析五粮液窖泥原核微生物群落结构[J].食品与发酵工业,2017,43(9):1-8.
[7]刘茂柯,唐玉明,邹兰,等.浓香型白酒窖泥古菌群落结构及其多样性[J].食品与发酵工业,2013,39(10):22-28.
[8]邓杰,卫春会,罗惠波,等.基于高通量测序的浓香型白酒窖池古菌群落结构分析[J].现代食品科技,2015,31(8):205-210.
[9]邓杰,卫春会,边名鸿,等.浓香型白酒不同窖龄窖池窖泥中古菌群落结构分析[J].食品科学,2017,38(8):37-42.
[10]刘茂柯,唐玉明,邹兰,等.浓香型白酒窖泥古菌群落结构及其多样性[J].食品与发酵工业,2013,39(10):22-28.
[11]韦艳霞,郑纪伟,杨锋,等.根管感染中产甲烷古细菌基于mcrA序列的系统发育分析[J].口腔生物医学,2014,5(4):181-184.
[12]华金玲,王立克,杨芝,等.PCR技术在黄淮白山羊瘤胃产甲烷菌检测中的应用[J].东北林业大学学报,2013,41(7):134-136.
[13]兰阿峰,杨曼,郭素芬,等.野生秦岭羚牛瘤胃产甲烷古菌多样性研究[J].安徽农业科学,2018,46(8):88-91.
[14]罗青春,刘超兰,吴正云,等.不同年份窖泥中主要产甲烷菌的荧光定量 PCR 研究[J].酿酒科技,2013(12):17-20.
[15]YU Y,LEE C,KIM J,et al.Group-specific primer and probe sets to detect methanogenic communities using quantitative real-time polymerase chain reaction[J].Biotechnol Bioeng,2005,89(6):670-679.
[16]YU Y,KIM J,HWANG S.Use of real-time PCR for group-specific quantification of aceticlastic methanogens in anaerobic processes:population dynamics and community structures[J].Biotechnol Bioeng,2006,93(3):424-433.
[17]胡贝,陈瑶,张文学,等.几种主要窖泥微生物丰度与窖龄的关系[J].应用与环境生物学报,2014,20(3):389-394.
[18]罗锡梅.基于单碱基识别的qPCR微生物群落定量技术的研究[D].哈尔滨:哈尔滨工业大学,2015.
[19]高志磊.古井窖泥微生物群落纲层级定量识别的引物设计和应用[D].哈尔滨:哈尔滨工业大学,2015.
[20]BUI M,LIU Z.Simple allele-discriminating PCR for cost-effective and rapid genotyping and mapping[J].Plant Meth,2009,5(1):1.
[21]LIU Z L,HAN Y,LI J W,et al.Analysis of microbial community structure of pit mud for Chinese strong-flavor liquor fermentation using next generation DNA sequencing of full-length 16S rRNA[J].Bio Rxiv,2018,doi:https://doi.org/10.1101/380949.
[22]唐立群,肖层林,王伟平,等.SNP分子标记的研究及其应用进展[J].中国农学通报,2012,28(12):154-158.
[23]PEARSON T,FALKOW S.Phylogenetic discovery bias inBacillus anthracisusing single-nucleotide polymorphisms from whole-genome sequencing[J].Proceed Natl Acad Sci USA,2004,101(37):13536-13541.
[24]WANG H T,SUN H,KWON W S,et al.A PCR-based SNP marker for specific authentication of Korean ginseng(Panax ginseng)cultivar "Chunpoong "[J].Molecul Biol Report,2010,37(2):1053-1057.
[25]FERNÁNDEZ-NO I C,BOHME K,CAAMANO-ANTELO S,et al.Identification of single nucleotide polymorphisms(SNPs)in the 16S rRNA gene of foodborneBacillusspp[J].Food Microbiol,2015,46(4):239-245.
[26]TAKAJO I,YAMADA A,UMEKI K,et al.Development of a simple and practical method of discrimination betweenVibrio furnissiiandV.fluvialisbased on single-nucleotide polymorphisms of 16S rRNA genes observed inV.furnissiibut not inV.fluvialis[J].J Microbiol Meth,2018,144(1):22-28.