高温大曲是以小麦和水为原料,稻草为辅料,经自然培菌、高温发酵等传统工序制成,富含多菌群多酶系,可将原料中的淀粉糖化、发酵转化成乙醇及微量香气成分,具有产酒和生香功能,是酱香型白酒生产的糖化发酵剂[1]。高温大曲同时是酱香型白酒酿造过程中生物催化剂和微生物的来源之一,在制曲的过程中微生物的代谢产物和原料的分解产物直接或间接地影响了风味物质的形成,使白酒具有不同的风格特征[2-3]。
稻草是水稻将大米打出后剩的稻杆,我国将水稻分为籼稻和粳稻两个亚种,对应稻草为籼稻草和粳稻草[4-5]。 籼稻草多缩戊糖含量高,草秆比例小,草叶、草穗所占比例大,木素、果胶、淀粉等非纤维含量成份较高,纤维组织松软。粳稻草茎杆较矮,叶子较窄,分蘖直立,叶色较深无毛,质地较硬,叶片较短与茎间角度较小。大曲中富集培养的微生物主要来源于原辅料、空气、生产场地等,经研究发现,小麦、稻草和制曲用水是曲块发酵过程中微生物的重要来源[6-7]。其中,稻草作为高温大曲生产中的重要辅料,在大曲培菌过程中起着保温、保湿、排潮、接种微生物的作用,能够提升大曲发酵微生物的多样性[8-9]。因此,探索稻草中微生物群落结构,对大曲微生物富集、培养具有重要意义。研究发现,稻草的微生物多样性受到品种、生长环境、化学成分等多种因素的影响,不同水稻土壤的微生物群落和多样性存在显著差异[10]。目前,关于稻草的研究多聚焦于新稻草与陈稻草的差异分析,黄瑜[6]研究发现陈稻草因微生物富集,其群落多样性与新稻草存在差异并影响大曲风味代谢。 然而,有关不同品种稻草的微生物群落特征差异的研究鲜见报道,有必要对其进行研究。
本研究通过高通量测序技术对籼稻草和粳稻草的微生物群落进行分析,探究不同品种的稻草在微生物群落与代谢特征的差异,以期为高温大曲发酵过程中不同稻草品种的选择提供理论依据。
1 材料与方法
1.1 材料与试剂
籼稻草、粳稻草:采购自泸州某公司。
脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒:美国Omega公司;DL2000 Marker:宝日医生物技术(北京)有限公司;Q5R超保真DNA聚合酶:美国纽英伦生物技术公司;琼脂糖、双链DNA(double-stranded DNA,dsDNA)定量试剂盒:赛默飞世尔科技(中国)有限公司;High Sensitivity DNA Kit试剂盒:美国安捷伦公司。
1.2 仪器与设备
DYY-6C电泳仪:北京六一生物科技有限公司;BGgdsAUTO(130)凝胶成像系统:北京百晶生物科技有限公司;NC2000-超微量紫外分光光度计、2720聚合酶链式反应(polymerase chain reaction,PCR)扩增仪、FLX800T酶标仪:美国赛默飞科技有限公司;NovaSeqPE250测序仪:美国Illumina公司。
1.3 方法
1.3.1 样品前处理
分别取放置于泸州某酒厂稻草房3 d的样品(编号籼稻草_1、粳稻草_1)以及放置0 d的籼稻草和粳稻草样品(编号籼稻草_2、粳稻草_2),在每个稻草房的3个位置采集样品,充分粉碎后混合为一个样品,重复取样3次。 将样品分别装入无菌密封取样袋中,贮存在-80 ℃用于后续高通量测序分析。
1.3.2 DNA的提取与PCR扩增
采用DNA提取试剂盒提取不同稻草样品中微生物宏基因组DNA,利用0.8%琼脂糖凝胶电泳与超微量分光光度计检测样品DNA纯度与浓度。将样品中DNA稀释,以稀释后的基因组DNA为模板,使用引物338F(5'-ACTCCTACGGGAGGCAGCA-3')、806R(5'-GGACTACHVGGGTWTCTAAT-3')对细菌的16S rDNA V3-V4区域进行扩增;使用引物ITS1F(5'-GCTGCGTTCTTCATCGA-TGC-3')、ITS2(5'-GCTGCGTTCTTCATCGA-TGC-3')对真菌的ITS1区域进行PCR扩增;PCR扩增条件:98 ℃预变性2 min;98 ℃变性15 s,54 ℃退火15 s,72 ℃延伸50 s,共35个循环;72 ℃再次延伸5 min,4 ℃保存;PCR扩增体系:5×TransStart FastPfu缓冲液4 μL,2.5 mmol/L脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphates,dNTPs)2 μL,上游引物(5 μmol/L)1 μL,下游引物(5 μmol/L)1 μL,Trans Start FastPfu DNA聚合酶0.5 μL,模板DNA 10 ng,用双蒸水(ddH2O)补足至20 μL。
1.3.3 高通量测序
使用fastp(https://github.com/OpenGene/fastp,version 0.19.6)软件对双端原始测序序列进行质控,使用FLASH(http://www.cbcb.umd.edu/software/flash,version 1.2.11)软件进行拼接,使用UPARSE v7.1软件(http://drive5.com/uparse/),根据97%的相似度对质控拼接后的序列进行操作分类单元(operational taxonomic unit,OTU)聚类并剔除嵌合体。
1.3.4 统计分析
所有的数据分析均在派森诺基因云平台(https://www.genescloud.cn/home)上进行,具体如下:采用mothur[11]软件(http://www.mothur.org/wiki/Calculators)计算Alpha多样性Chao1指数、Shannon指数、Simpson指数等;使用Venn图来描述群落之间的相似性和差异性;使用分类层次树状图显示了不同样品在不同分类水平上的物种组成树图;利用Mantel test分析基于Spearman进行微生物相关性分析[12];使用PICRUSt2[13](version 2.2.0)软件进行功能预测分析;以上数据均进行3次平行重复分析。
2 结果与分析
2.1 不同稻草样品高通量测序数据分析
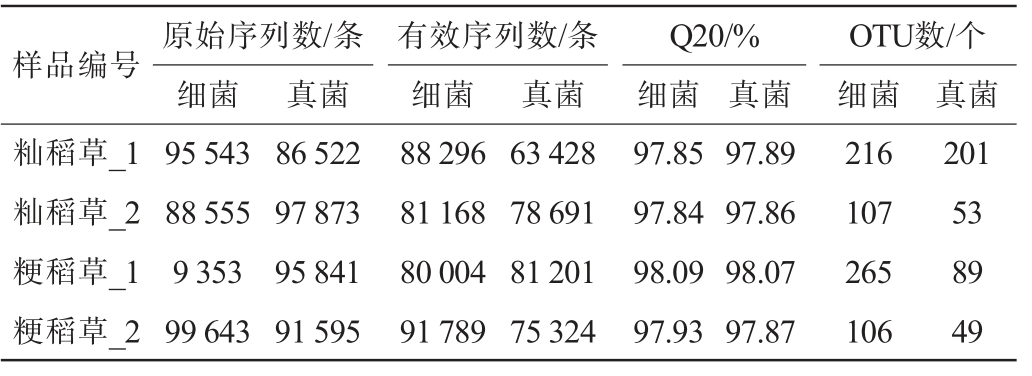
不同稻草样品测序数据结果见表1。 由表1可知,在对4个样品进行质量控制后,共获得了371 831条真菌ITS原始序列和373 094条细菌16S rRNA原始序列,过滤后最终获得298 644条真菌有效序列和341 257条细菌有效序列。 通过聚类获得了392个真菌OTUs和694个细菌OTUs,同源性为98%,Q20值(碱基质量值≥20的碱基所占百分比)均大于97%,表明数据可信度较高,能够进一步分析。
表1 稻草样品高通量测序数据处理结果
Table 1 High-throughput sequencing data processing results of straw samples
?
2.2 不同稻草样品中微生物群落Alpha多样性分析
Chao1指数反映了物种的丰富度,数值越高表示丰富度越高;而Shannon指数和Simpson指数反映了物种的多样性,数值越高表示多样性越大[14]。 对不同种类稻草样品进行微生物Alpha多样性分析,结果见图1。由图1a可知,粳稻草中细菌Chao1指数、Shannon指数与Simpson指数略高于籼稻草,说明粳稻草整体细菌菌群丰富度与多样性略高于籼稻草。而由图1b可知,籼稻草中真菌Chao1指数、Shannon指数与Simpson指数明显高于粳稻草,说明籼稻草真菌菌群丰富度与多样性远远高于粳稻草。 经放置3 d的稻草中细菌及真菌Chao1指数、Shannon指数与Simpson指数高于放置0 d的,说明放置3 d后的籼稻草和粳稻草细菌、真菌菌群的丰富度与多样性均高于放置0 d。另外,经放置3 d后的籼稻草微生物丰富度与多样性高于放置3 d后的粳稻草,说明籼稻草富集、培养微生物的能力较强,能够更好的应用于后续大曲培菌糖化发酵阶段。
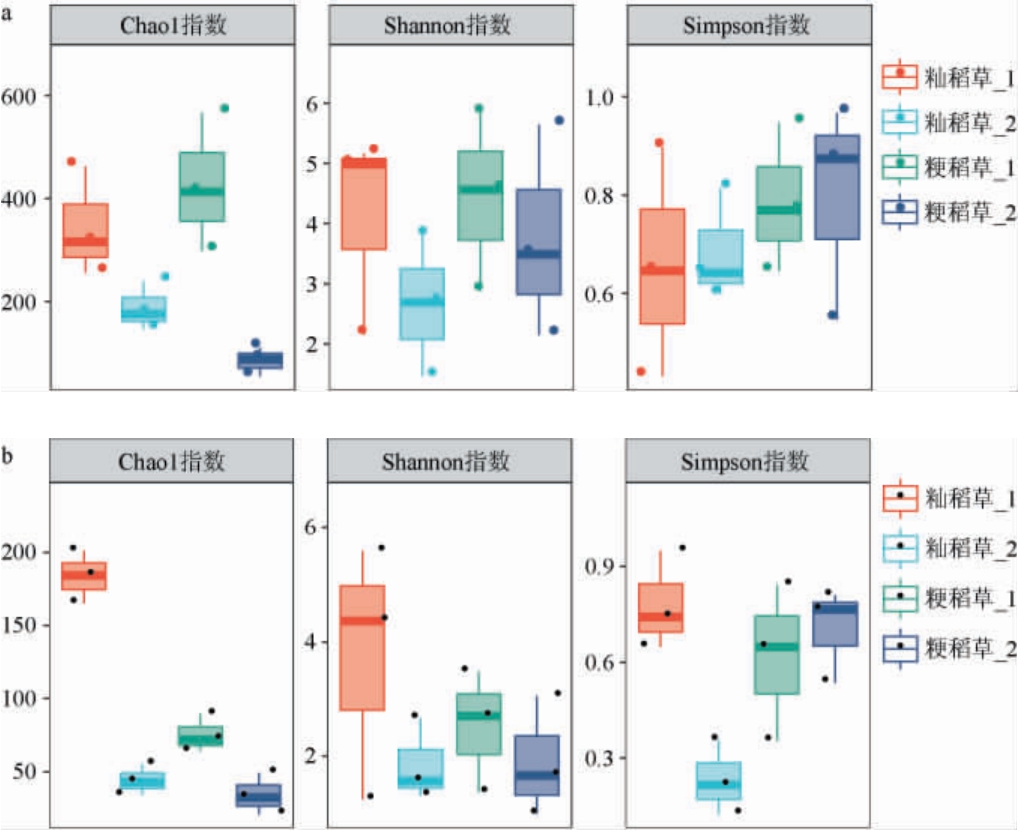
图1 不同稻草细菌(a)及真菌(b)菌群的Alpha多样性分析结果
Fig.1 Alpha diversity analysis results of bacteria (a) and fungi (b)communities of different straws
对4种样品进行微生物群落结构分类并绘制韦恩图,结果见图2。由图2可知,4种样品间共有的细菌OTU数为3个,真菌OTU数为7个,说明不同稻草微生物群落具有特异性。籼稻草_1、籼稻草_2、粳稻草_1、粳稻草_2特有的细菌OTU数分别有207个、85个、222个、74个; 特有的真菌OTU数分别有166个、30个、46个、15个。 结合特有OTU数分析可知,经放置3 d后的籼稻草、粳稻草微生物多样性较放置前更为丰富,其中放置3 d后的籼稻草特有真菌OTU数达到166个,籼稻草的真菌种类远远大于粳稻草的真菌种类,说明其真菌种类更为复杂。
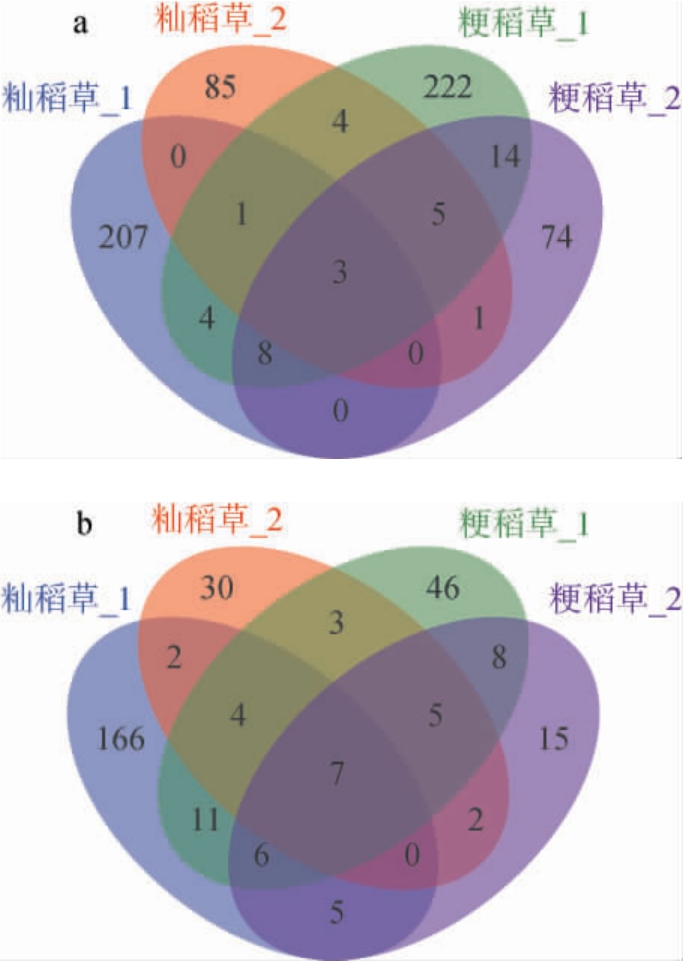
图2 不同稻草细菌(a)及真菌(b)操作分类单元的Venn图
Fig.2 Venn diagram of operational taxonomic unit of bacteria (a)and fungi (b) in different straw
2.3 不同稻草样品中微生物群落组成结构
基于门水平与属水平,籼稻草和粳稻草微生物群落组成结构见图3。由图3a可知,在门水平上,从4种样品中共注释到3个优势细菌门(平均相对丰度≥1%),分别是变形菌门(Proteobacteria)(53.82%)、放线菌门(Actinobacteria)(41.37%)、厚壁菌门(Firmicutes)(3.24%)。其中,经放置3 d后的籼稻草放线菌门相对丰度达到97.24%,是绝对优势细菌门;放置0 d的籼稻草变形菌门相对丰度达到97.02%,是绝对优势细菌门;放置前后的粳稻草相对丰度最高的是变形菌门和放线菌门,相对丰度之和均>90%,说明这两种细菌门是粳稻草的优势细菌门。 由图3b可知,在真菌门水平上4种样品结构组成相似,共注释到2个优势真菌门(平均相对丰度≥1%),分别是子囊菌门(Ascomycota)(81.75%)、担子菌门(Basidiomycota)(2.12%);放置3 d的粳稻草子囊菌门相对丰度达到96.12%,占据了绝对优势。由图3c可知,放置前后籼稻草细菌组成结构有着显著的差异,放置前主要优势细菌属为泛菌属(Pantoea)(73.07%),放置3 d后主要优势细菌属为拟诺卡氏菌属(Nocardiopsis)(55.30%)、糖多孢菌属(Saccharopolyspora)(35.82%),该结论与DU H等[15]的研究结果相似,泛菌属仅在大曲发酵的早期阶段大量存在,据报道它在大曲发酵过程中参与脂质合成,产生低分子量脂多糖[16-17],促进含硫化合物转化为挥发性较低的含硫氨基酸[18]。 放置前后粳稻草细菌组成结构相似,主要细菌属为甲烷细菌属(Methylobacterium)、动球属(Kineococcus)、假单胞菌属(Pseudomonas),通过对比发现,放置3 d后的粳稻草主要细菌属相对丰度高于放置前。 此外,据研究表明[19],高温大曲中优势细菌属为泛菌属、糖多孢菌属等,因此可以证明籼稻草能为大曲发酵提供优势微生物。由图3d可知,经放置3 d后籼稻草真菌组成结构发生变化,放置前共注释到1个优势真菌属(平均相对丰度≥1%),是帚枝霉属(Sarocladium)(80.86%);而放置3 d后优势菌属为曲霉属(Aspergillus),而在粳稻草在真菌属水平上结构组成相似,共注释到1个优势真菌属(平均相对丰度≥1%),是曲霉属(Aspergillus)(56.41%)。 据研究表明曲霉属为大曲中的优势真菌[20]。在白酒酿造过程中,曲霉属能够将大量的葡萄糖淀粉酶分泌到环境中,使淀粉糖化,进一步促进酵母的生长和新陈代谢[21-22]。
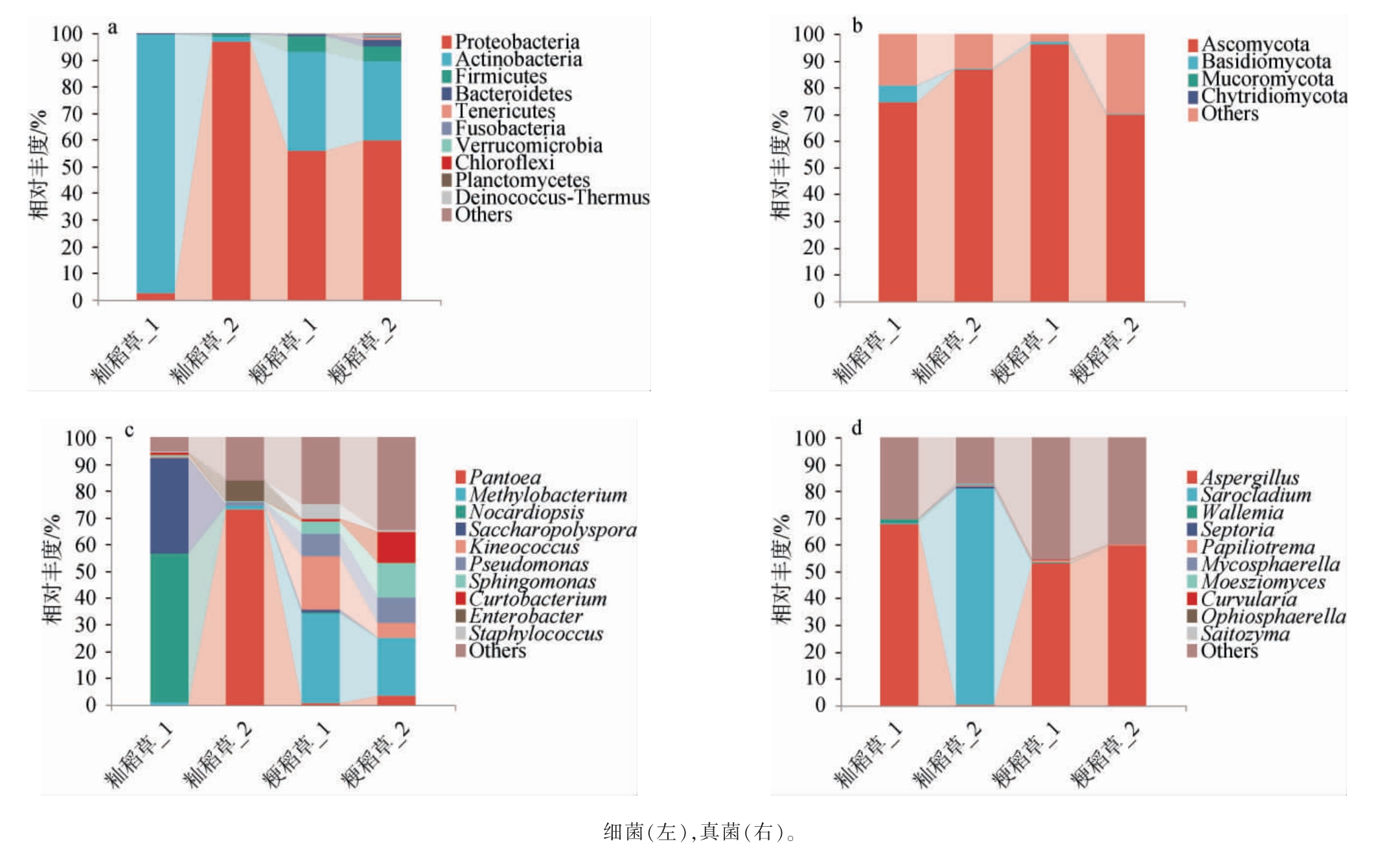
图3 基于门水平(a,b)和属水平(c,d)不同稻草的微生物群落组成
Fig.3 Microbial community composition of different straws based on phylum level (a,b) and genus level (c,d)
2.4 不同稻草样品中微生物群落差异分析
为了进一步解析不同稻草样品中微生物群落差异,利用分类层次树状图分析了物种组成,结果见图4。由图4a可知,经放置3 d后的籼稻草细菌占比较大的主要是α-变形菌纲 (Alpha-proteobacteria)、γ-变形菌纲(Gamma-proteobac teria),粳稻草中细菌占比较大的主要是放线菌纲(Actinobacteria),根据丰度大小可以看出芽孢杆菌纲(Bacilli)和拟杆菌纲(Bacteroidia)在籼稻草中分布占比更高。
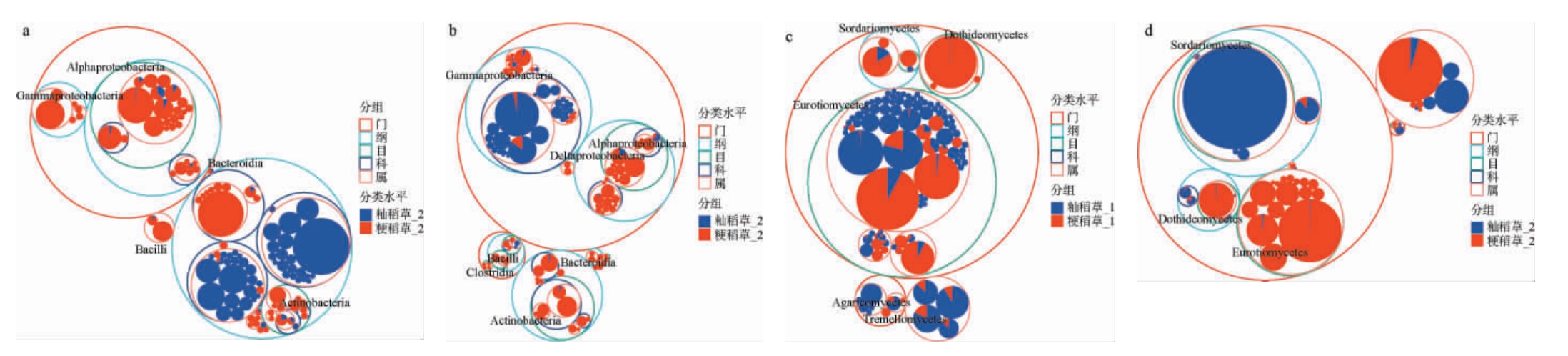
图4 不同稻草微生物分类层次树图
Fig.4 Hierarchy tree of microbial classification in different rice straws
a:放置第3天细菌;b:放置第0天细菌;c:放置第3天真菌;d:放置第0天真菌。
由图4b可知,放置0天的籼稻草细菌分布占比明显高于粳稻草,籼稻草中细菌主要是α-变形菌纲、放线菌纲,而粳稻草中主要是γ-变形菌纲,此外芽孢杆菌纲和拟杆菌纲几乎全部存在于籼稻草中。由图4c可知,经放置3 d后的籼稻草真菌占比较大的主要是座囊菌纲(Dothideomycetes)、粪壳菌纲(Sordariomycetes),而放置3 d后的粳稻草真菌占比较大的主要是散囊菌纲(Eurotiomycetes)、伞菌纲(Agaricomycetes)和银耳纲(Tremellomycetes),其中散囊菌纲基于目水平在不同稻草中分布差异较大。 由图4d可知,放置0 d的籼稻草真菌占比较大的主要是散囊菌纲,粳稻草中真菌占比较大的主要是粪壳菌纲。 综上,籼稻草与粳稻草在细菌和真菌优势菌群上存在显著差异,且经放置3 d对群落结构影响显著,芽孢杆菌纲、拟杆菌纲和散囊菌纲的分布差异可能反映了两种稻草基质理化性质或降解潜力的不同,该结果与张原頔等[8]研究结果相似,正是由于这些差异微生物具备各自独特的功能特性,使得籼、粳稻草的微生物群落对稻草的分解和物质循环等过程产生不同的影响。
2.5 代谢通路的物种组成
通过PICRUSt2标记基因序列预测和挖掘不同稻草样品中相关酶功能和丰度信息,结合发酵相关的代谢通路以此说明不同稻草中微生物潜在功能。细菌和真菌的代谢通路主要集中在生物合成、物质的降解、前体代谢物和能量的产生[23]。 获得不同稻草样品的通路丰度后,进一步使用分层的样本代谢通路丰度进行细菌和真菌物种组成分析[24],结果见图5。

图5 不同稻草样品中代谢通路物种组成
Fig.5 Species composition of metabolic pathways in different straw samples
由图5a可知,放置3 d的籼稻草中拟诺卡氏菌属(Nocardiopsis)、糖多孢菌属(Saccharopolyspora)在糖酵解途径中发挥着主要作用,结合微生物群落组成可以印证拟诺卡氏菌属和糖多孢菌属是籼稻草的优势细菌属;放置0 d的籼稻草中泛菌属(Pantoea)相对丰度较高。 据研究表明[25],泛菌属可能源自种植环境中的水和土壤,它们具有修复和降解农药残留及有毒物质的潜力,同时作为植物内生菌,发挥固氮和促进生长的作用。从功能预测来看,它们还表现出降解葡萄糖和蔗糖的潜力,这些代谢功能与发酵过程密切相关。然而,泛菌属在这些代谢过程中的具体参与程度和机制仍需进一步研究。此外,放置0 d的粳稻草中动球菌属(Kineococcus)和假单胞菌属(Pseudomonas)在糖酵解途径发挥主要作用,通过丰度进一步解析出经发酵的籼稻草中微生物糖酵解能力较强,有助于大曲糖化发酵阶段中微生物代谢,从而影响大曲品质。由图5b可知,籼稻草中富集的微生物在L-酪氨酸降解能力上较少。这可能是稻草自身特性导致富集到的相关微生物较少。粳稻草在L-酪氨酸降解途径中丰度优势明显,其中假单胞菌占据绝对优势,鞘氨醇单胞菌属(Sphingomonas)降解L-酪氨酸能力在放置3d后明显减少。 这两种菌属均是植物根际中常见的细菌,但假单胞菌的适应性更强。 由图5c可知,放置0 d的籼稻草在蔗糖降解途径中丰度优势明显,泛菌属占绝对优势,其代谢特性可能使其在富含可溶性碳水化合物(如蔗糖)的籼稻草中更易繁殖,并高效参与蔗糖降解。具备降解多种复杂有机物能力的假单胞菌在粳稻草的蔗糖降解能力占优势地位。由图5d、5e、5f可知,对于真菌代谢通路物种组成,籼稻草中曲霉属(Aspergillus)在苏氨酸生物合成、转运核糖核酸(transfer ribonucleic acid,tRNA)转载、L-亮氨酸合成途径中所占功能丰度高于粳稻草。放置3 d的籼稻草在这三条通路中,真菌功能丰度均显著高于0 d,说明稻草放置后,真菌从适应稻秆转向氨基酸合成、翻译调控等核心合成代谢。据研究表明曲霉属为大曲中的优势真菌,在发酵过程中会产生许多的风味物质,有助于提升酒体的风味[20,26]。曲霉属在粳稻草放置过程中苏氨酸生物合成、tRNA转载代谢通路贡献变化较小。帚枝霉属(Sarocladium)主要在籼稻草中,并在放置0 d时相对丰度较高,相关研究发现帚枝霉属是水稻的内生真菌,推测原因该菌属可能参与了己酸乙酯合成[6]。整体来讲,放置3 d的稻草真菌在苏氨酸生物合成、tRNA转载、L-亮氨酸合成途径中所占功能丰度高于0 d。
对比发现,籼稻草与粳稻草的微生物代谢功能存在显著差异。籼稻草以糖酵解强化、风味物质合成为核心优势,更适用于大曲发酵品质提升,而粳稻草则侧重复杂物质降解,更利于秸秆预处理或基础代谢。两类稻草的微生物功能差异为其在酿酒、生物降解等领域的差异化应用提供了理论依据。
2.6 籼稻草中微生物群落相关性分析
Mantel test是对两个矩阵相关关系的检验,常在生态学研究中用于分析微生物群落演替与相应阶段之间的相关性。根据上述研究,可以发现籼稻草更适用于大曲发酵品质提升,能够更好的应用于后续大曲培菌糖化发酵,因此利用Mantel test分析籼稻草样品中微生物之间的相关性,优势微生物与微生物群落中的其他微生物相关性结果见图6。
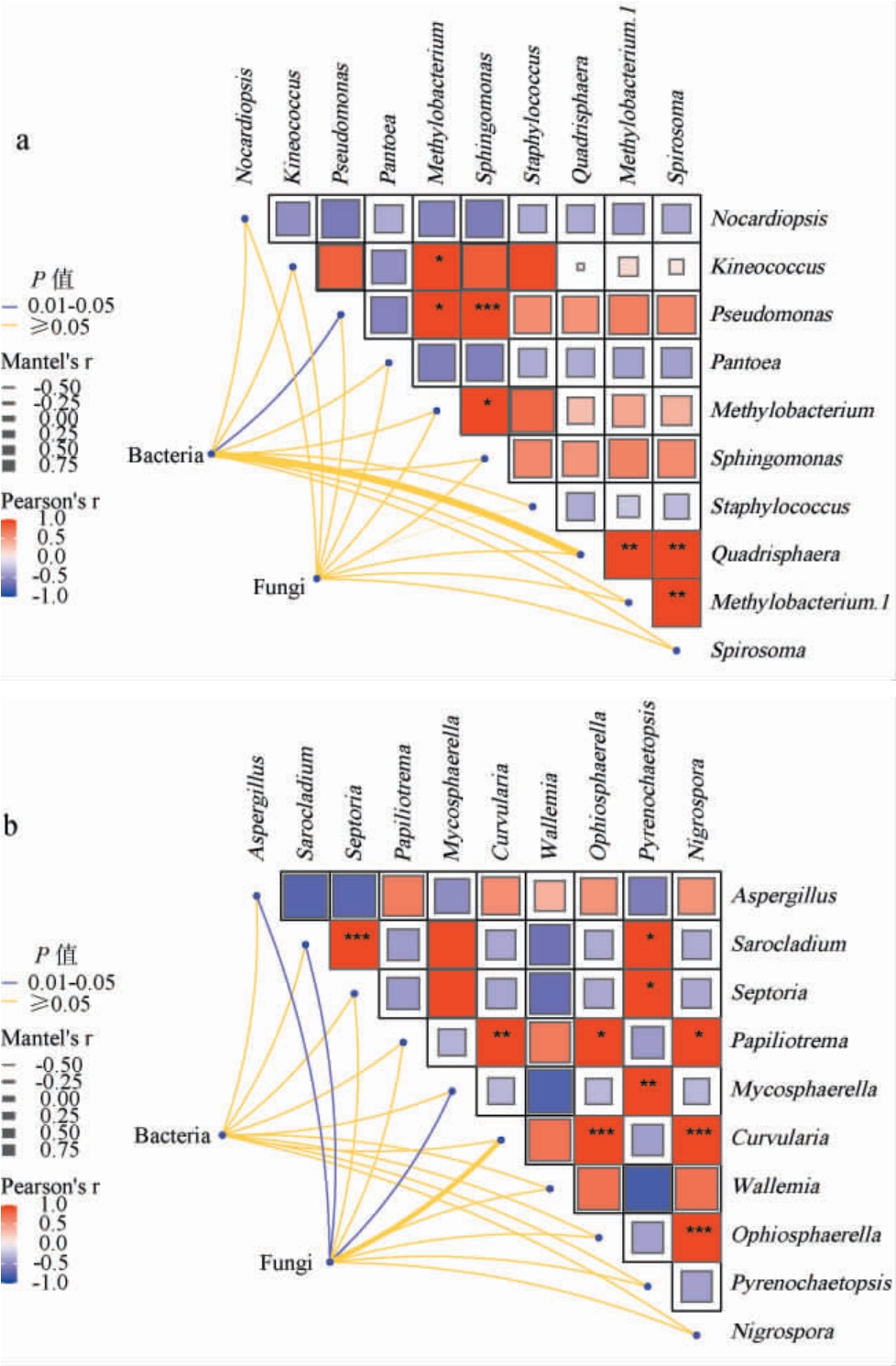
图6 籼稻草细菌(a)和真菌(b)相关性Mantel test热图
Fig.6 Mantel test heat map of correlation between bacteria (a)and fungi (b) of indica rice straw
热图中线条粗细代表群落与环境因子之间相关性大小;不同颜色代表正负相关性,红色代表正相关,紫色代表负相关;颜色深浅代表正负相关性大小;色块中星号代表显著性,“*”表示影响显著,0.01<P≤0.05,“**”表示影响极显著,0.001<P≤0.01,“***”表示影响高度显著,P≤0.001。
由图6a可知,假单胞菌属(Pseudomonas)与其他细菌属有相关性,假单胞菌属与甲基杆菌属(Methylobacterium)呈显著正相关(r>0.75,P<0.05),与鞘氨醇单胞菌属(Sphingomonas)呈高度显著正相关(P<0.001)。 这表明假单胞菌属在发酵过程中可能扮演着重要的角色,通过与其他细菌种群的协同作用,共同推动着发酵过程的进行。 此外,假单胞菌属与细菌群落呈显著相关(P<0.05),这些关系可能涉及到营养竞争、代谢产物交换或共同抵御不利环境等多个方面。由图6b可知,真菌群落中的相关性网络则显得更为复杂和多样。曲霉属(Aspergillus)、帚枝霉属(Sarocladium)和球腔菌属(Mycosphaerella)与真菌群落呈显著相关(r>0.75,P<0.05),作为真菌群落中的优势成员,它们不仅与其他真菌种类存在广泛的相互作用,而且这些相互作用的性质也更为复杂,包括正相关和负相关两种类型。这种复杂的相关性可能反映了真菌群落内部更为精细的生态调控机制,如共生、竞争、拮抗等多种关系的交织。值得注意的是,霉菌在发酵过程中产生的淀粉、纤维素、半纤维等水解酶,不仅是大曲糖化作用的关键,还可能通过影响底物利用和代谢产物积累,间接调控着整个微生物群落的动态平衡[27]。通过揭示籼稻草发酵过程中细菌和真菌群落内部的微生物相关性变化,为深入理解大曲发酵的微生物代谢机制提供重要依据。
3 结论
该研究利用高通量测序技术揭示了籼稻草与粳稻草在微生物群落结构及其代谢功能上的显著差异。 结果表明,籼稻草与粳稻草在微生物群落组成结构上存在显著差异,籼稻草中优势细菌属为泛菌属(Pantoea)和拟诺卡氏菌属(Nocardiopsis),而粳稻草中优势细菌属为甲烷细菌属(Methylobacterium)和动球属(Kineococcus),同时微生物群落差异结果表明,籼稻草经放置3 d后优势细菌门从变形菌门(Proteobacteria)向放线菌门(Actinobacteria)显著转变,同时表现出更高的微生物丰富度与多样性,更有利于大曲糖化发酵,而粳稻草则相对稳定。代谢通路物种组成分析表明,籼稻草中的细菌在糖酵解途径所占功能丰度更高,而粳稻草中的细菌在L-酪氨酸降解和蔗糖降解途径所占功能丰度更高,真菌中的曲霉属在多种代谢途径中功能丰度较高,有助于提升酒体风味。本研究为高温大曲生产中不同稻草品种的选择提供了科学依据,为进一步优化大曲发酵工艺、提升白酒品质奠定基础。
[1]王西,陈波,张亚东,等.高温大曲安曲投入老草的应用研究[J].酿酒科技,2020(12):88-91.
[2]李习,方尚玲,刘超,等.酱香型白酒风味物质主体成分研究进展[J].酿酒,2012,39(3):19-23.
[3]WANG Z M,WANG C T,SHEN C H,et al.Microbiota stratification and succession of amylase-producing Bacillus in traditional Chinese Jiuqu(fermentation starters)[J].J Sci Food Agr,2020,100(8):3544-3553.
[4]ZHANG G Q.Prospects of utilization of inter-subspecific heterosis between indica and japonica rice[J].J Integr Agr,2020,19(1):1-10.
[5]LU B R, CAI X, XIN J.Efficient indica and japonica rice identification based on the InDel molecular method:Its implication in rice breeding and evolutionary research[J].Progr Nat Sci,2009,19(10):1241-1252.
[6]黄瑜.制曲原辅料微生物组成对酱香型白酒风味的影响[D].无锡:江南大学,2021.
[7]陈燕.关于不同辅料在大曲酱香白酒生产中的应用探析[J].中国食品工业,2023(16):88-91.
[8]张原頔,沈毅,程伟,等.三代全长高通量测序分析新稻草和陈稻草的微生物特征[J].中国酿造,2022,41(9):24-30.
[9]吴煜樟,郭敏杰,高宇.酱香型白酒大曲生产使用稻草的研究进展[J].食品科技,2023,48(6):62-66.
[10]阳云.不同产量水稻土壤的微生物群落多样性探究[D].武汉:华中农业大学,2016.
[11]SCHLOSS P D,WESTCOTT S L,RYABIN T,et al.Introducing mothur:Open-source,platform-independent, community-supported software for describing and comparing microbial communities[J].Appl Environ Microbiol,2009,75(23):7537-7541.
[12]BARBERAN A,BATES S T,CASAMAYOR E O,et al.Using network analysis to explore co-occurrence patterns in soil microbial communities[J].ISME J,2012,6(2):343-351.
[13]GAVIN M D,VINCENT J M,JESSE R Z,et al.PICRUSt2 for prediction of metagenome functions[J].Nature Biotechnol,2020,38:685-688.
[14]陈卓,苏伟,母应春,等.洞酿酱香型白酒第三轮次酒醅发酵过程中微生物与挥发性风味物质相关性分析[J].中国酿造,2024,43(5):32-40.
[15]DU H, WANG X S, ZHANG Y H, et al.Exploring the impacts of raw materials and environments on the microbiota in Chinese Daqu starter[J].Int J Food Microbiol,2019,297:32-40.
[16]HOU Q C, WANG Y R, CAI W C, et al.Metagenomic and physicochemical analyses reveal microbial community and functional differences between three types of low-temperature Daqu[J].Food Res Int, 2022,156:111167.
[17]KANG J M,HU Y N,DING Z Y,et al.Deciphering the shifts in microbial community diversity from material pretreatment to saccharification process of Fuyu-flavor Baijiu[J].Front Microbiol,2021,12:705967.
[18]YANG L,CHEN R Y,LIU C,et al.Spatiotemporal accumulation differences of volatile compounds and bacteria metabolizing pickle like odor compounds during stacking fermentation of Maotai-flavor Baijiu[J].Food Chem,2023,426:136668.
[19]沈毅,陈波,王西,等.酱香型郎酒高温大曲、酒醅和窖泥中细菌群落结构分析[J].中国酿造,2020,39(2):89-93.
[20]TONG W H, LI Y Y, YANG Y, et al.Dynamic analysis Caffeic acid production driven by the key physicochemical factor and microbial community succession in Baijiu Daqu:A multi-microorganism fermentation of solid-state fermentation system[J].LWT-Food Sci Technol,2023,190:115542.
[21]陈磊,戴亦军.基于Illumina MiSeq高通量测序技术分析丹阳黄酒酒曲中微生物菌群多样性[J].中国酿造,2024,43(6):141-145.
[22]YAN S B,CHEN X S,GUANG J Q.Bacterial and fungal diversity in the traditional Chinese strong flavour liquor Daqu[J].J I Brewing, 2019,125:443-452.
[23]孔德婷,齐笑含,刘兴蕾,等.不同多年生稻品种内生细菌群落多样性比较分析[J].生物技术通报,2024,40(5):225-236.
[24]ZHAO T,ZHAN L B,ZHOU W,et al.The effects of erchen decoction on gut microbiota and lipid metabolism disorders in zucker diabetic fatty rats[J].Front Pharmacol,2021,12:647529.
[25]ZHANG Y D,XU J G,DING F,et al.Multidimensional profiling indicates the shifts and functionality of wheat-origin microbiota during hightemperature Daqu incubation[J].Food Res Int,2022,156:111191.
[26]杨旭,马歌丽,王光路,等.高通量测序解析白酒中高温大曲细菌和真菌群落结构[J].中国酿造,2020,39(6):119-123.
[27]郭建华,郭宏文,邹东恢,等.白酒酒醅发酵过程中酶活与酯类生成的相关性分析与主成分分析[J].食品与发酵工业,2013,39(11):44-49.