谷氨酸棒杆菌(Corynebacterium glutamicum)是一种重要的工业生产菌株[1-4],常用来合成有机酸等医药、食品等领域的产品[5-7]。目前,谷氨酸棒杆菌常用的pk18mobsacB基因编辑系统因sacB基因本身容易失活,常会导致筛选到假阳性的转化子,产生费时和低效的问题[8]。新型的规律成簇的间隔短回文重复(clustered regularly interspaced short palindromic repeats,CRISPR)技术作为高效的基因编辑工具,已被广泛应用于工业微生物菌株改造[9-10]。其中,CRISPRCpf1基因编辑系统具有脱靶率低等显著优点,在微生物合成生物学领域受到了广泛关注和应用[11-13]。然而,目前研究报道的谷氨酸棒杆菌的CRISPR-Cpf1基因编辑系统主要设计为All-in-one单质粒模式,即将Cpf1蛋白、CRISPR核糖核酸(CRISPR ribonucleic acid,crRNA)表达框及脱氧核糖核酸(DNA)修复片段在同一个质粒中表达,其在进行大片段的基因敲除与整合时,编辑效率仅为5%~15%[14-15]。
谷氨酸棒杆菌CRISPR-Cas基因编辑系统的设计原理是在Cas9或Cpf1蛋白切割染色体基因位点的同时,利用供体DNA分子在断裂位点进行同源重组,导致这种单质粒编辑系统的工作效率较低。PENG F等[16]研究发现,利用多种类型的启动子优化表达框(Cpf1蛋白、DNA修复模板与crRNA),在一定程度上均会影响系统的编辑效率。对于Cpf1的表达方式,本课题组在前期研究中也尝试了利用双质粒分别表达Cpf1和crRNA,即先将携带Cpf1的质粒转化至供试菌株并制备感受态细胞,再将携带crRNA的质粒转化其中,通过统计培养皿中菌落生长情况,结果发现,转化子(漏切的假阳性菌落)数目大幅度增加,说明这种分别表达Cpf1和crRNA的双质粒基因编辑系统严重影响切割致死率,分析原因可能是由于Cpf1基因突变导致的基因编辑效率降低[17]。
为了提高谷氨酸棒杆菌的基因组编辑效率,本研究利用PrpR-PrpD2诱导型启动子表达Cpf1蛋白,将重组和切割事件分离,构建了一种诱导型的CRISPR-Cpf1/dsDNA双质粒编辑方法,并通过比较组成型和诱导型启动子表达Cpf1的效果,统计双质粒系统的基因编辑效率。最后以不产L-丝氨酸的谷氨酸棒杆菌ATCC 13032为出发菌株,利用该基因编辑系统构建筛选一株高产L-丝氨酸的重组菌株,为精准地改变谷氨酸棒杆菌的代谢通路,实现菌株代谢工程的精准改造起到推动作用。
1 材料与方法
1.1 材料与试剂
1.1.1 菌种与质粒
大肠杆菌(Escherichia coli)DH5α、C.glutamicum ATCC 13032(WT)、C.glutamicum WT△cgl1890::kan(M-2)、C.glutamicum WT△xyLA::kan(M-3)、C.glutamicum WT△cgl1890::kans(S-1)、C.glutamicum WT△cgl1890::kan*(X-1):本实验室保藏。
质 粒PEC、pXMJ19、pEC-Ptuf-Cpf1、pXMJ19-Pj23119-crRNA、pXMJ19-Pj23119-crRNA △xyLA、pXTuf-Cpf1-cr-RNAxyLA::kan-xyLA500:本实验室保藏。
1.1.2 引物
本研究所用主要引物见表1。
表1 本研究所用主要引物
Table 1 Main primers used in the study

引物 序列(5'-3')酶切位点PRP-1 PRP-2 PRP-3 PRP-4 PrCpf1-1 PrCpf1-2 PM500-1 PM500-2 PQ500-2 S-1 197A-2 197A-3 S-10 ACB-1 ACB-2 ACB-3 ACB-4 ACB-5 ACB-6 ACB-7 ACB-8 ACB-9 ggcaaatattctgaaatgagctgGAATTCCAATGGAGTGGATGACTGCTTCG AGTCTTTTTTGGGGTGGCCTTTTAGTTGAGGAGTTGGACAGGG CCCTGTCCAACTCCTCAACTAAAAGGCCACCCCAAAAAAGACT tctagaggatccccgGGTACCGGCTGGAATGTGACTCCTTCTC ggtacccggggatccATCGTGTGGTACCATGTGTGGA tgcctgcaggtcgacTCTAGATTAGTTATTGCGGTTCTGGACAAA aaatattctgaaatgagctgTTGACAGCTAGCTCA tctcatccgccaaaacagccAATGGTAGTGAACTACCGTCCCTT tctcatccgccaaaacagccCGCGATTTACTTTTCGACCTC ggaatttctactgttgtagatGTCAACGCTCCTCGCATTGCTGAAGTTTGAGACCACCATTGTTGCT GTTCAAGCAAATGGATCTCTAATTAAGCCAGATCCATCCACACAGC GCTGTGTGGATGGATCTGGCTTAATTAGAGATCCATTTGCTTGAAC tccgccaaaacagcctctagaTGAAACTCACGAGTGAGGTCGA ggaatttctactgttgtagatAAGGTAACGGCATTGGGGTGTTCTTGAATCCTGAAAGCTTCCAGG GTTTTCCTTCGGATCTAAACGATCTCAAACGTCAGGGAATACGACAC GTGTCGTATTCCCTGACGTTTGAGATCGTTTAGATCCGAAGGAAAAC GACGGCCATTCTGGCTCATTGTATGTCCTCCTGGACTTCGTG CACGAAGTCCAGGAGGACATACAATGAGCCAGAATGGCCGTC GAAGTCGGTCATGTCTTCCTCCTTTTTAAGCCAGATCCATCCACACA TGTGTGGATGGATCTGGCTTAAAAAGGAGGAAGACATGACCGACTTC GAATAATCCCCGCAGCACCTTTAACGACGCGCCCTCAGGGGAAACATGGCGAAGAACTGGAACCATG CCCTGAGGGCGCGTCGTTAAAGGTGCTGCGGGGATTATTC EcoR I Kpn I Xba I
续表

注:下划线代表酶切位点,斜体代表引物间重叠区域,小写字母代表同源臂。
引物 序列(5'-3')酶切位点ACB-10 CB1 CB2 CB3 CB4 CB5 CB6 CB7 ZH-1 ZH-2 ZH-3 ZH-4 ZH-5 ZH-6 JS-1 Jsac tccgccaaaacagcctctagaGTGTTAAAGCCGTACCGCTAAC ggaatttctactgttgtagatACGACGCGCCCTCAGGGGAAACATAAAGGAGGAAGACATGACCGACTTC GAGCCTTCAGAGCCTTCGGGGCGAAGAACTGGAACCATG CATGGTTCCAGTTCTTCGCCCCGAAGGCTCTGAAGGCTC GAGTTCAGTCATAAAGTTTCCTCCTTTTTACTTCCTTGCAAAACCGCC GGCGGTTTTGCAAGGAAGTAAAAAGGAGGAAACTTTATGACTGAACTC GAATAATCCCCGCAGCACCTTAGGCATTGGTCAATGGAACG CGTTCCATTGACCAATGCCTAAGGTGCTGCGGGGATTATTC TGCCTGCAGGTCGACTCTAGAGACTAGTGGGGGTTTCTGCTGTT CTTTCTACGTGTTCCGCTTCCTCCTGGCGATAGGTGTCAAGAATTCG GAATTCTTGACACCTATCGCCAGGAGGAAGCGGAACACGTAGAAAG ATAGCGTGGTTGGCGAAGTTTTTACGCTTCGCCAACGCCAGTGCCTCACCATTTTCCACCATGATATTCG Xba I CTGGCGTTGGCGAAGCGTAAAAACTTCGCCAACCACGCTAT TACGAATTCGAGCTCGGTACCAATGGTAGTGAACTACCGTCCCTT CTTCTTTCAACGGTGTGGAAATT CCGTTTAAGGCATTGAAGCAT
1.1.3 培养基
脑心浸液(brian heart infusion,BHI)固体培养基:青岛海博生物技术有限公司。
种子培养基[18-19]:葡萄糖35.0 g/L,玉米浆30.0 mL/L,酵母粉5.0 g/L,豆粕水解液20.0 mL/L,磷酸二氢钾1.5 g/L,硫酸镁0.5 g/L,维生素B1 1.0 mg/L,硫酸铁10 mg/L,硫酸锰10 mg/L,生物素0.2 mg/L,pH 7.1~7.3,121 ℃条件下高压蒸汽灭菌20 min。
发酵培养基[18-19]:葡萄糖100.0 g/L,玉米浆36.0 mL/L,硫酸铵10.0 g/L,豆粕水解液26.0 mL/L,磷酸二氢钾2.6 g/L,硫酸镁0.6 g/L,维生素B1 1.6 mg/L,硫酸铁11 mg/L,硫酸锰10 mg/L,生物素0.2 mg/L,pH 7.1~7.3,121 ℃条件下高压蒸汽灭菌20 min。
玉米浆:挑选新鲜、无菌的玉米粉,将其与水按质量比1∶3混合,搅拌均匀成浆状,煮沸,冷却后,调节pH为6.0~7.0。
豆粕水解液:将豆粕进行清洗、研磨,将豆粕与水按质量比1∶3混合,加入盐酸,保持pH为2.5,加热至90~110 ℃,水解5 h,再用氢氧化钠(NaOH)中和至pH为7.0,去除残渣,得到含有小分子肽、氨基酸和糖类的水解液。
1.1.4 试剂
Primer STAR HS DNA polymerase(2.5 U/μL)、Quick-Cut限制性内切酶(120 U/μL):大连宝生物工程有限公司;Taq聚合酶链式反应(PCR)Master Mix、Clone Express RⅡOne Step Cloning Kit:南京诺维赞生物工程有限公司;质粒提取、DNA回收试剂盒:上海Omega Bio-Tek公司;卡那霉素(kanamycin,Kan)(效价≥750 IU/mg)、链霉素(spectacomycin,Spe)(效价≥720 IU/mg)、氯霉素(chloramphenicol,Cm)(效价≥900 IU/mg):南京杜莱生物技术有限公司;丙酸钠(分析纯):北京索莱宝科技有限公司。其他试剂均为国产分析纯或生化试剂。
1.2 仪器与设备
Mastercycler nexus PCR仪、Eppendorf Eporator电击转化仪:德国Eppendorf公司;GL20A高速冷冻离心机:日本日立有限公司;UV200紫外可见波长检测器:美国Laballiance公司;Agilent 1200高效液相色谱(HPLC)仪:安捷伦科技(中国)有限公司。
1.3 方法
1.3.1 质粒的构建与转化
采用CaCl2介导的化学转化法[20]构建重组质粒,具体方法如下:
重组质粒pEC-PrpR-PrpD2-Cpf1(A质粒)构建:以E.coli DH5α基因组为模板,PRP-1/PRP-2与PRP-3/PRP-4为引物,扩增PrpR-PrpD2的重叠基因。将pEC质粒用限制性内切酶EcoR I与Kpn I进行双酶切线性化,再将重叠基因与其重组连接获得重组质粒pEC-PrpR-PrpD2;以pEC-Ptuf-Cpf1质粒为模板,PrCpf1-1/PrCpf1-2为引物,扩增Cpf1的DNA片段。重组质粒pEC-PrpR-PrpD2经限制性内切酶Xba I单酶切线性化,将Cpf1的DNA片段与pEC-PrpR-PrpD2线性化载体连接获得重组质粒pEC-PrpR-PrpD2-Cpf1,用于诱导型基因编辑系统。
重组质粒pXMJ19-Pj23119-crRNAxyLA::kan-xyLA500(B质粒)构建:以pXTuf-Cpf1-crRNAxyLA::kan-xyLA500质粒为模板,PM500-1/PM500-2为引物扩增DNA片段(crRNAxyLA::kan-xyLA500),将pXMJ19质粒用限制性内切酶EcoR I与Kpn I进行双酶切线性化,再将其与扩增的DNA片段连接,获 得 重 组 质 粒pXMJ19-Pj23119-crRNAxyLA::kan-xyLA500,用于验证基因编辑系统的整合效率。
重组质粒pXMJ19-Pj23119-crRNA△serA197(B质粒)构建:以C.glutamicum ATCC 13032基因组为模板,S-1/197A-2和197A-3/S-10为引物,扩增上下游同源臂,用于serA基因敲除,将pXMJ19-Pj23119-crRNA质粒经Xba I单酶切线性化,再将其与前者基因片段连接,获得重组质粒pXMJ19-Pj23119-crRNA△serA197,用于构建高产L-丝氨酸重组菌株。
重组质粒pXMJ19-Pj23119-crRNA△sdaA::PtufserArC(B质粒)构建:以谷氨酸棒杆菌ATCC 13023基因组为模板,ACB-1/ACB-2、ACB-3/ACB-4、ACB-7/ACB-8、ACB-9/ACB-10为引物,分别扩增上下游同源臂、Ptuf和部分serC的DNA片段。以菌株WT-△serA197基因组为模板,ACB-5/ACB-6为引物,扩增serAr。采用重叠PCR获得含有5部分DNA片段重叠基因。将pXMJ19-Pj23119-crRNA质粒经Xba I单酶切线性化,再将其与重叠基因连接,获得重组质粒pXMJ19-Pj23119-crRNA△sdaA::PtufserArC,用于构建高产L-丝氨酸重组菌株。
重组质粒pXMJ19-Pj23119-crRNAxyLA::serCB(B质粒)构建:以谷氨酸棒杆菌ATCC 13023基因组为模板,CB-1/CB-2、CB-3/CB-4、CB-5/CB-6、CB-7/ACB-10为引物,分别扩增上下游同源臂和serCB的DNA片段。采用重叠PCR获得含有4部分DNA片段的重叠基因。将pXMJ19-Pj23119-crRNA质粒经Xba I单酶切线性化,再将其与重叠基因连接,获得重组质粒pXMJ19-Pj23119-crRNAxyLA::serCB,用于构建高产L-丝氨酸重组菌株。
1.3.2 诱导型CRISPR-Cpf1/dsDNA双质粒系统基因编辑流程
由pEC-PrpR-PrpD2-Cpf1(A质粒)、pXMJ19-Pj23119-crRNA-dsDNA(B质粒)建立诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统,其基因编辑流程见图1,具体方法:①先将A质粒转化至谷氨酸棒杆菌,制备感受态细胞;②再将B质粒转入上述感受态细胞,涂布于含氯霉素抗性(CmR)(50 mg/mL)-链霉素抗性(SpeR)(50 mg/mL)的BHI培养基平板,获得阳性转化子;③再随机挑选平板中阳性转化子的单菌落对点至含有CmR-SpeR-丙酸钠的BHI培养基平板,诱导Cpf1的表达;④挑取对点筛选的阳性转化子,利用菌落PCR进行验证,获得目的菌株。
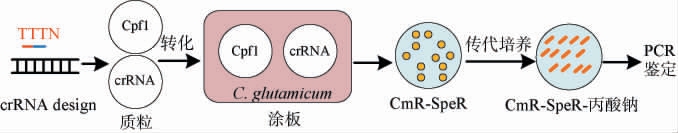
图1 建立诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统的基因编辑流程
Fig.1 Gene editing process for establishment of inducible CRISPRCpf1/dsDNA dual-plasmid gene editing system
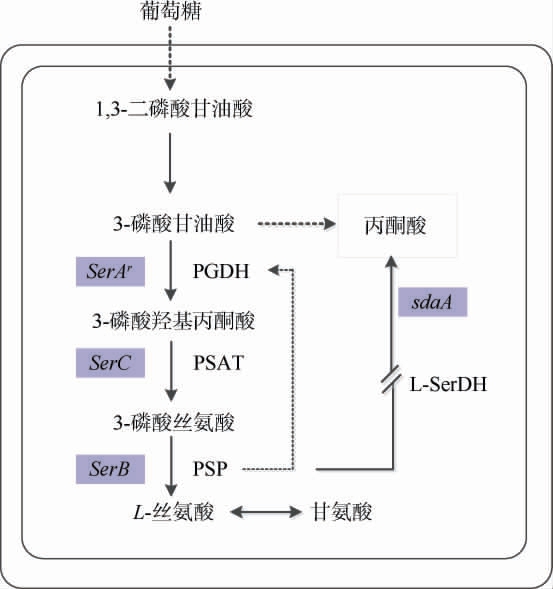
图2 高产L-丝氨酸重组菌株改造策略
Fig.2 Modification strategies for high-producing L-serine recombinant strain
3-磷酸甘油酸脱氢酶(3-phosphoglycerate dehydrogenase,PGDH),磷酸丝氨酸转氨酶(phosphoserine aminotransferase,PSAT);磷酸丝氨酸磷酸酶(Phosphoserine phosphatase,PSP),L-丝氨酸脱氢酶(L-serine dehydrogenase,L-SerDH)。
1.3.3 诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统敲除及整合效率分析
以组合型启动子Ptuf构建的CRISPR-Cpf1/dsDNA双质粒基因编辑系统(pEC-Ptuf-Cpf1(A质粒)、pXMJ19-Pj23119-crRNA-dsDNA(B质粒))为对照,通过表达Cpf1的效果考察诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统的敲除及整合效率。
基因敲除效率分析:采用电转化方法先将A质粒(pECPrpR-PrpD2-Cpf1)转化至敲除模式菌株X-1(C.glutamicum WT△cgl1890::kan*),制备含有A质粒的菌株感受态细胞;再将B质粒(pXMJ19-Pj23119-crRNA△xyLA)转化至上述含有A质粒的感受态细胞,然后涂布于含CmR-SpeR的BHI固体培养基,32 ℃条件下培养24~36 h;在平板中随机挑选10~20个单菌落对点至含有卡那霉素抗性(KanR)(50mg/L)-CmR-SpeR-丙酸钠(100 mg/L)的BHI固体培养基,32 ℃条件下培养24~36 h,统计平板中正常生长的阳性菌株数量,计算基因敲除效率(Kan平板中正常生长的菌株数量/随机挑选的单菌落数量),作为验证基因敲除效率的依据。
基因整合效率分析:除使用整合模式菌株S-1外,方法同上。
1.3.4 诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统的应用
将诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统应用于改造谷氨酸棒杆菌高产L-丝氨酸,具体改造策略:以菌株WT为出发菌,拟采用阻断目的产物分解,增加前体物供应量,加强关键酶表达的策略,构建L-丝氨酸高产菌[21-22]。首先,敲除基因组上serA基因的C端编码197个氨基酸序列区域,解除反馈抑制,构建重组菌株SW-1;再将菌株SW-1的sdaA基因敲除,抑制L-丝氨酸到丙酮酸的支路,整合人工操作子PtufserArserCserB,增加关键酶的表达,最终构建重组菌株SW-3。
重组菌株SW-3的构建:将构建的诱导型双质粒基因编辑系统,即pEC-PrpR-PrpD2-Cpf1(A质粒)和pXMJ19-Pj23119-crRNA△serA197(B质粒),转化至出发菌株WT(C.glutamicum ATCC 13032),获得SW-1(C.glutamicum WT-△serA197)(鉴定引物JS-1/S-10);再将构建的诱导型双质粒基因编辑系统,即pEC-PrpR-PrpD2-Cpf1(A质粒)和pXMJ19-Pj23119-crRNA△sdaA::PtufserArC(B质粒),转化至SW-1,获得重组菌株SW-2(C.glutamicum WT-△serA197△sdaA::PtufserArC)(鉴定引物Jsac/ACB-4);最后将构建的诱导型双质粒基因编辑系统,即pEC-PrpR-PrpD2-Cpf1(A质粒)和pXMJ19-Pj23119-crRNAxyLA::serCB(B质粒)转化至重组菌株SW-2,获得重组菌株SW-3(C.glutamicum WT-△serA197△sdaA::serArCB)(鉴定引物ACB-5/CB4)。
基因编辑效率分析:将重组菌株先涂布于含CmR-SpeR的BHI固体培养基,32 ℃条件下培养24~36 h;在平板中随机挑选10~20个单菌落对点至含CmR-SpeR-丙酸钠的BHI固体培养基,32 ℃条件下培养24~36 h,利用菌落聚合酶链式反应(polymerase chain reaction,PCR)验证阳性目的菌株,分别计算基因敲除和基因整合效率(PCR验证具有正确目的条带的菌株数量/随机挑选的单菌落数量),作为验证基因编辑效率的依据。
重组菌株SW-3的培养:将活化的菌株转接至种子培养基中,32 ℃、230 r/min条件下培养12 h至OD600 nm值约为15,作为种子液;将种子液按10%(V/V)的接种量接种于发酵培养基,在32 ℃、230 r/min条件下培养60 h,每隔12 h取样,采用紫外分光光度计测定菌液在波长600 nm处的吸光度值(OD600nm值)[23-24],并采用高效液相色谱(high performance liquid chromatography,HPLC)测定L-丝氨酸含量[18-19]。
1.3.5 数据处理与统计分析
利用Microsoft Excel 2018、Visio 2019和Origin 2018统计分析数据与绘图。
2 结果与分析
2.1 诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统的基因编辑效率
组成型和诱导型CRISPR-Cpf1/双链脱氧核糖核酸(double stranded deoxyribonucleic acid,dsDNA)双质粒基因编辑系统的基因编辑效率见图3。由图3可知,诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统的基因敲除效率显著高于组成型CRISPR-Cpf1/dsDNA双质粒基因编辑系统(P<0.05),分别为(66.67±1.53)%、(10.00±1.50)%,说明诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统表现较好的编辑效果。推测原因可能是当添加诱导剂丙酸钠后,诱导表达Cpf1蛋白会对非正确编辑菌体的双链DNA位点进行靶向割裂,导致未编辑菌体细胞死亡,而DNA重组片段可修复靶位点,被正确编辑的菌体通过同源重组而存活,目的菌株则表现为培养皿中正常生长[25-26]。结果表明,将基因重组和切割独立开来,提高了基因敲除效率。
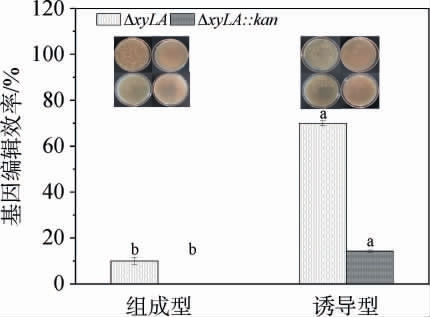
图3 组成型和诱导型启动子对双质粒基因编辑效率的影响
Fig.3 Effect of constitutive and inducible promoters on the gene editing efficiency of dual-plasmids
不同小写字母表示差异显著(P<0.05)。
由图3亦可知,诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统的基因整合效率显著高于组成型CRISPR-Cpf1/dsDNA双质粒基因编辑系统(P<0.05),分别为(14.29±1.50)%、0,说明组成型CRISPR-Cpf1/dsDNA双质粒基因编辑系统不能成功进行基因整合,而诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统可应用于谷氨酸棒杆菌基因组敲除与整合,分析原因可能是复苏时重组DNA模板被高效地插入靶位点,再对没有正确重组的细胞进行了基因组双链DNA切割,致死了绝大部分非编辑细胞,少部分非编辑细胞此时也发生同源重组[27],从而提高了编辑效率,说明本系统根据先重组再切割的原理,利用诱导型启动子PrpR-PrpD2表达Cpf1蛋白的双质粒基因编辑系统具有一定的可行性。
2.2 基于诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统高产L-丝氨酸重组菌株的构建
通过菌落PCR分析诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统在重组菌株SW-1、SW-2、SW-3中的基因编辑效率,鉴定引物PCR扩增结果见图4。serA基因碱基长度为3 368 bp,敲除197个氨基酸序列后,碱基长度为1 197 bp。由图4a可知,随机挑选的12株转化子经菌落PCR鉴定后,仅有2株转化子含有碱基长度为1 103 bp的基因,说明筛选菌株SW-1的基因敲除效率为16.67%。由于操作子serArCB较大,serAr基因的碱基长度为1 002 bp,serC基因的碱基长度为1 144 bp,因此分两次整合在基因组上。第一次先整合serAr和部分serC。由图4b可知,随机挑选的9株转化子经菌落PCR鉴定后,仅有1株转化子含有碱基长度为1 021 bp的serArC基因,说明筛选菌株SW-2基因的整合效率为11.11%。第二次整合另一部分serC和serB,serB基因的碱基长度为1 317 bp。由图4c可知,随机挑选的10株转化子经菌落PCR鉴定后,仅有2株转化子含有碱基长度为2 146 bp的SerCB基因,说明筛选菌株SW-3基因整合效率为20.00%。结果表明,该诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统的基因编辑效率可以满足实际菌株代谢工程改造的需要,在谷氨酸棒状杆菌中的代谢工程应用改造是可行的。
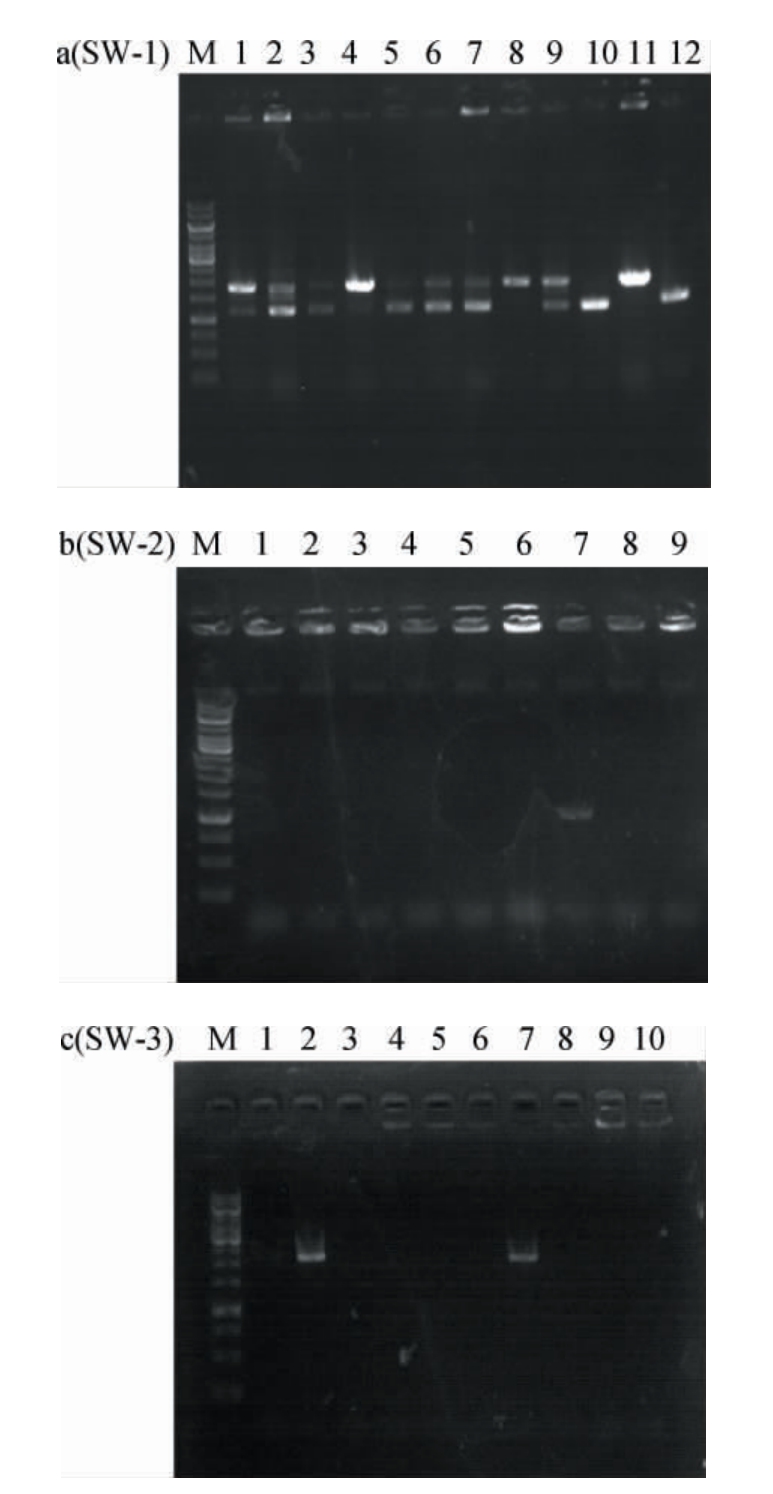
图4 基于菌落PCR诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统在重组菌株SW-1(a)、SW-2(b)及SW-3(c)中的基因编辑效率
Fig.4 Gene editing efficiency of inducible CRISPR-Cpf1/dsDNA dual-plasmid gene editing system in recombinant strains SW-1 (a), SW-2 (b) and SW-3 (c) based on colony PCR
M为DNA Marker,1~12为随机挑选的转化子的菌落PCR鉴定结果。
对菌株WT和SW-3进行摇瓶验证,测定其生长情况和L-丝氨酸产量,结果见图5。由图5可知,经过基因修饰后的菌株SW-3生长明显慢于出发菌株WT,说明L-丝氨酸的积累是以抑制菌体的生长为代价的,此结果与文献[28]报道一致,即谷氨酸棒杆菌经过基因工程改造严重影响菌体的最大比生长速率。出发菌株WT的L-丝氨酸产量未检出,当培养至12 h时,重组菌株SW-3的L-丝氨酸产量逐渐增加,发酵60 h时,达到10.51 mg/L,说明利用强启动子Ptuf表达关键基因serAr、serC和serB增加前体物的供应量,敲除sdaA基因阻断L-丝氨酸分解为丙酮酸的支路的策略是可行的,在一定程度上可增加L-丝氨酸的产量,此结果与文献[29]报道一致。然而,重组菌株L-丝氨酸的产量并没有达到预期,可能是由于大肠杆菌拥有甘氨酸裂解系统,而谷氨酸棒杆菌没有,若切断L-丝氨酸到甘氨酸的代谢路径,将破坏胞内甘氨酸和一碳代谢平衡[30]。因此,推测本研究中合成的L-丝氨酸转化成甘氨酸分泌到胞外维持代谢平衡,导致L-丝氨酸不能大量积累。后续的研究中可尝试弱化L-丝氨酸到甘氨酸的分解代谢的方式解决胞内产物与一碳代谢失衡的问题。
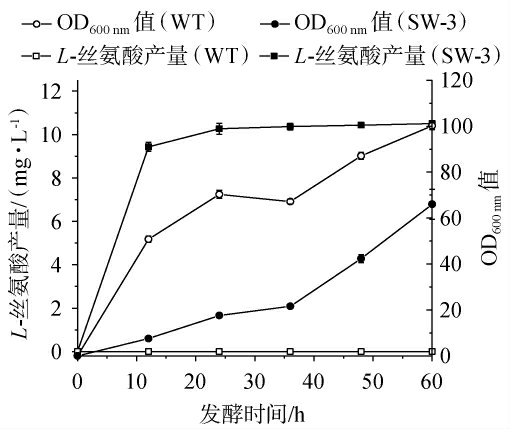
图5 出发菌株WT和重组菌株SW-3的生长曲线及L-丝氨酸产量
Fig.5 Growth curves of starting strain WT and recombinant strains SW-3 and L-serine yield
与谷氨酸棒杆菌现有的基因编辑方法相比,诱导型的双质粒系统分别表达Cpf1和crRNA等元件,利用诱导Cpf1表达介导切割,待菌体细胞充分重组,加入诱导剂丙酸钠促进了非编辑细胞死亡,本研究不仅克服了All-in-one单质粒对于构建重组质粒大小的限制,也有利于表达大片段供体DNA的重组质粒的构建。
3 结论
本研究采用诱导型启动子PrpR-PrpD2表达Cpf1蛋白,将重组和切割事件分离,建立了一种高效的诱导型CRISPRCpf1/dsDNA双质粒基因编辑系统,其基因敲除效率为(66.67±1.53)%,基因整合效率为(14.29±1.50)%,均显著高于组合型启动子组(P<0.05)。此外,以不产L-丝氨酸的谷氨酸棒杆菌(Corynebacterium glutamicum)ATCC 13032为出发菌株,利用诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统成功构建了一株高产L-丝氨酸重组菌株SW-3,其L-丝氨酸产量为10.51 mg/L,说明诱导型CRISPR-Cpf1/dsDNA双质粒基因编辑系统在谷氨酸棒杆菌代谢工程改造中具有可行性,对加速谷氨酸棒杆菌代谢工程改造进程及推动其在氨基酸等高附加值产品研发上的应用将产生深远意义。
[1]谭海,顾阳,卢南巡,等.代谢工程改造谷氨酸棒状杆菌促进L-异亮氨酸发酵合成的研究进展[J].中国酿造,2021,40(9):1-6.
[2]李业,孟丽虹,严豪,等.谷氨酸棒杆菌细胞工厂育种策略以及高通量筛选技术的研究进展[J].食品与发酵工业,2023,49(21):276-283.
[3]上官玲玲,卢慧芳,夏会丽,等.谷氨酸棒杆菌细胞工厂构建与应用的研究进展[J].食品与发酵工业,2022,48(17):313-320.
[4]ZHA J, ZHAO Z, XIAO Z, et al.Biosystem design of Corynebacterium glutamicum for bioproduction[J].Curr Opin Biotechnol,2023,79:102870.
[5]王钰,郑平,孙际宾.谷氨酸棒杆菌的代谢工程使能技术研究进展[J].生物工程学报,2021,37(5):1603-1618.
[6]华珂君.谷氨酸棒杆菌中双产烃路径构建生产纤维素烃类化合物[D].上海:华东理工大学,2021.
[7]王婷,赵兰坤,扈临风,等.基因编辑技术在谷氨酸棒杆菌中的应用研究进展[J].中国酿造,2023,42(4):35-39.
[8]DONG Y,MIAO R,FENG R,et al.Edible and medicinal fungi breeding techniques, a review: Current status and future prospects[J].Curr Res Food Sci,2022,5:2070-2080.
[9]朱华伟,李寅.合成生物制造2025[J].生物工程学报,2025,41(1):10-87.
[10]YANG H,QU J,ZOU W,et al.An overview and future prospects of recombinant protein production in Bacillus subtilis[J].Appl Microbiol Biotechnol,2021,105(18):6607-6626.
[11]XU R, QIN R, LI H, et al.Generation of targeted mutant rice using a CRISPR-Cpf1 system[J].Plant Biotechnol J,2017,15(6):713-717.
[12]DONG D,REN K,QIU X,et al.The crystal structure of Cpf1 in complex with CRISPR RNA[J].Nature,2016,532(7600):522-526.
[13]WU H,LIU Q,SHI H,et al.Engineering CRISPR/Cpf1 with tRNA promotes genome editing capability in mammalian systems[J].Cell Mol Life Sci,2018,75:3593-3607.
[14]JIANG Y,QIAN F,YANG J,et al.CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum[J].Nat Commun,2017,8(1):15179.
[15]WANG Q, ZHANG J, AL MAKISHAH N H, et al.Advances and perspectives for genome editing tools of Corynebacterium glutamicum[J].Front Microbiol,2021,12:654058.
[16]PENG F,WANG X,SUN Y,et al.Efficient gene editing in Corynebacterium glutamicum using the CRISPR/Cas9 system[J].Microb Cell Fact,2017,16:1-13.
[17]LIU J,WANG Y,LU Y,et al.Development of a CRISPR/Cas9 genome editing toolbox for Corynebacterium glutamicum[J].Microb Cell Fact,2017,16(1):205.
[18]郑桂花,邹文安,赵倩.L-丝氨酸生产菌发酵及合成过程优化研究[J].四川农业大学学报,2022,40(6):950-957.
[19]刘岩.L-丝氨酸基因工程菌的构建及发酵条件优化[D].大连:大连工业大学,2015.
[20]赵桂红.谷氨酸棒杆菌发酵法生产L-丙氨酸和D-丙氨酸的研究[D].天津:天津科技大学,2021.
[21]刘安倩,颜文斌,肖文翰,等.氨基脱氧分支酸合成酶对谷氨酸棒杆菌生长和产L-丝氨酸的影响[J].食品与发酵工业,2023,49(19):15-21.
[22]SIEBERT D,WENDISCH V F.Metabolic pathway engineering for production of 1,2-propanediol and 1-propanol by Corynebacterium glutamicum[J].Biotechnol Biofuels,2015,8(1):1-13.
[23]王金多,李澜潇,徐庆阳.L-赖氨酸全营养流加发酵工艺的研究[J].中国调味品,2022,47(10):93-97.
[24]陈晓博,罗雪粤,张淑荣,等.生产L-精氨酸的脯氨酸营养缺陷型菌株的选育[J].北京化工大学学报(自然科学版),2011,38(6):83-86.
[25]SU R, WANG T, BO T, et al.Enhanced production of d-pantothenic acid in Corynebacterium glutamicum using an efficient CRISPR-Cpf1 genome editing method[J].Microb Cell Fact,2023,22(1):1-15.
[26]WANG T,LI Y,LI J,et al.An update of the suicide plasmid-mediated genomeeditingsysteminCorynebacteriumglutamicum[J].Microb Biotechnol,2019,12(5):907-919.
[27]MA W,WANG X,MAO Y,et al.Development of a markerless gene replacement system in Corynebacterium glutamicum using upp as a counterselection marker[J].Biotechnol Lett,2015,37(3):609-617.
[28]PETERS-WENDISCH P, STOLZ M, ETTERICH H, et al.Metabolic engineering of Corynebacterium glutamicum for L-serine production[J].Appl Environ Microbiol,2005,71(11):7139-7144.
[29]LI Y, CHEN G K, TONG X W, et al.Construction of Escherichia coli strains producing L-serine from glucose[J].Biotechnol Lett,2012,34:1525-1530.
[30]来书娟,张芸,刘树文,等.产L-丝氨酸谷氨酸棒杆菌的代谢工程改造和代谢流分析[J].中国科学:生命科学,2012,42(4):295-303.