白酒酿造工艺是一项传统的食品生物加工技术,随着酿造原料在大曲发酵剂的作用下于封闭环境中发酵,酒醅微生物群落呈动态、有规律的演变[1]。发酵过程中微生物在介导特定生态功能时,通过活菌菌群响应[2],其代谢产生影响白酒风味和品质的物质[3]。目前,发酵食品微生物群落的研究主要依赖于脱氧核糖核酸(deoxyribonucleic acid,DNA)的高通量测序技术,但由于其无法区分发酵环境中死细胞和活细胞,往往高估物种多样性与丰度[4]。传统培养法虽能分离活菌,但受限于可培养物种不足(仅占自然界微生物的0.1%~1.0%)及耗时性[5];宏转录组技术虽能通过核糖核酸(ribonucleic acid,RNA)分析活菌功能,但却面临RNA易降解、复杂基质干扰及成本高昂等瓶颈[6]。
叠氮溴化丙锭(propidium monoazide,PMA)作为高亲和力的光反应性DNA交联剂,可与具有细胞膜透过性的死菌细胞DNA进行特异性结合。基于此特性,PMA已被广泛应用于发酵食品中活性微生物的检测。在葡萄酒体系中,有研究者通过优化PMA光活化参数与引物特异性,构建PMACELL-定量聚合酶链式反应(quantitative polymerase chain reaction,qPCR)体系,实现了酿酒酵母(Saccharomyces cerevisiae)活菌的高灵敏度检测(103 CFU/mL),其定量结果与传统平板计数高度一致[6]。在红曲黄酒发酵体系中,研究者通过优化PMA处理条件,成功解析了发酵过程中乳杆菌属(Lactobacillus)的动态演变,其相对丰度从发酵中期的25.7%跃升至发酵后期的68.5%,揭示了活菌群演替与工艺参数的关联机制[7]。LIU D T等[8]在固态醋发酵中整合PMA-16S rDNA测序与PMA-qPCR,不仅实现了活性乳酸菌精准定量(检测限102 CFU/g),还发现环境因子通过调控乳酸脱氢酶基因(ldh)表达,显著影响代谢通路(P<0.05)。目前,鲜见关于白酒酒醅中活微生物群落结构及演替规律的研究报道。
该研究以大曲清香型白酒大米查酒醅为研究对象,以不经过叠氮溴化丙锭(PMA)处理的酒醅为对照组(DT组),以经过PMA处理的酒醅为实验组(DV组),采用扩增子测序技术系统解析酒醅发酵过程中微生物群落组成,并通过线性判别分析效应大小(linear discriminant analysis effect size,LEfSe)对不同酒醅中差异菌群进行分析。以期揭示清香型白酒发酵过程中活微生物群落的演替机制,加深对大曲清香型白酒酒醅发酵机制的认识,为白酒品质提升提供数据支撑。
1 材料与方法
1.1 材料与试剂
清香型白酒大米查酒醅:山西省汾阳市杏花村镇某酒厂;叠氮溴化丙锭(PMA):上海圣尔生物科技有限公司;蛋白酶K(酶活30 U/mg):北京索莱宝科技有限公司;Premix Ex TaqTM荧光定量PCR试剂盒:宝生物工程(大连)有限公司;磷酸缓冲盐溶液(phosphate buffer saline,PBS):武汉普诺赛生命科技有限公司。
1.2 仪器与设备
BL-2200H电子分析天平:岛津国际贸易(上海)有限公司;5804高速低温离心机:德国艾本德公司;New BrunswickTM S41i恒温振荡摇床:美国New Brunswick Scientific公司;PLPMA光解仪:北京普林塞思科技有限公司;NanoDrop 2000分光光度计:赛默飞世尔科技有限公司;CFX ConnectTM 荧光定量PCR仪:美国Bio-Rad公司。
1.3 方法
1.3.1 PMA工作液制备
在避光条件下,将1 mg PMA加入98 μL无菌双蒸水(ddH2O)中,溶解并充分混合,得到20 mmol/L的PMA储存溶液,避光保存在-20 ℃条件下,将PMA储存液稀释10倍后得到PMA工作液。
1.3.2 酒醅样品预处理及DNA提取
对酒醅进行取样,取样时间点分别为0、3 d、7 d、11 d、15 d、21 d、24 d、28 d,每个取样时间采集100 g新鲜酒醅装入无菌塑封袋中。将2.5 g酒醅样品加入10 mL 1 mol/L PBS缓冲液(pH 7.4),并放入5~8粒玻璃珠,涡旋振荡20~30 min后,取上层液体,采用PBS缓冲液制备成25%的酒醅悬浮液。以不经过PMA预处理的酒醅悬浮液为对照组(DT组),将酒醅菌悬液于12 000 r/min条件下离心5 min,收集菌体,采用十六烷基三甲基溴化铵(cetyltrimethylammonium bromide method,CTAB)法提取基因组[9-11]。以经过PMA预处理的酒醅悬浮液为实验组(DV组),向1 mL的25%酒醅悬浮液中加入50μmol/L的PMA工作液,室温下暗孵育,5min后,设置PL-PMA光解仪波长为465 nm,于光解仪中曝光孵育5 min[12],之后于12 000 r/min条件下离心5 min,收集菌体,采用CTAB法提取酒醅基因组,并标记为DV组。
1.3.3 qPCR扩增
将重组质粒标准品进行10倍梯度稀释至10-8后进行qPCR扩增。qPCR扩增体系(20 μL)为:SYBR® Premix Ex Taq TM 10 μL,正反向引物各0.4 μL,DNA模版1 μL,双蒸水(ddH2O)8.2 μL。细菌qPCR扩增程序为:94 ℃预变性10 s;94 ℃变性5 s,58 ℃退火15 s,72 ℃延伸15 s,共40个循环;72 ℃再延伸5 min。真菌qPCR扩增程序为:94 ℃预变性10 s;94 ℃变性5 s,56 ℃退火15 s,72 ℃延伸15 s,共45个循环;72 ℃再延伸5 min,其中总细菌和总真菌的引物序列参考文献[13]进行设计。
1.3.4 扩增子测序
将经过PMA处理后得到的酒醅样本(DV组)和对照样本(DT组)的DNA进行扩增子测序,分析酒醅发酵过程中的活微生物群落多样性与演替规律。通用引物515F(5'-GTGCCAGCMGCCGCGGTAA-3')和806R(5'-GGACTACHVGGGTWTCTA AT-3')扩增细菌16S rRNA基因V4可变区;利用引物ITS1F(5'-CTTGGTCATTTAGAGGAAGTAA-3')和ITS2R(5'-GCTGGTTCTTCATCGATGC-3')扩增真菌内部转录间隔区(internal transcribed spacer,ITS)。PCR扩增条件:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,共27个循环;72 ℃再延伸5 min[14]。DNA纯化后在NovaSeq 6000测序平台进行扩增子测序分析。对Illumina NovaSeq测序得到的下机数据进行拼接和质控,进行嵌合体过滤,以产生适合后续分析的有效数据,使用QIIME2中的分类学习算法对扩增子序列变体(amplicon sequence variation,ASV)进行分类。使用feature-classifier插件中的classify-sklearn中的朴素贝叶斯分类器与16S rRNA基因Green 13.8数据库和真菌ITS UNITE 8.0数据库进行序列比对和鉴定。
1.3.5 统计分析
使用SPSS 21.0进行数据处理分析,通过单因素方差分析(analysis of variance,ANOVA)进行统计比较,P<0.05被认为具有统计学意义,采用Origin 2021、SIMCA 14.0绘图。
2 结果与分析
2.1 PMA处理前后酒醅发酵过程中微生物菌群多样性分析
2.1.1 PMA处理前后酒醅发酵过程中微生物菌群Alpha多样性分析
对经PMA处理的酒醅样品(DV组)及未经PMA处理的对照酒醅样品(DT组)进行扩增子测序,质控、修剪、去噪、拼接、去嵌合体后获得1 739 646条细菌16S rDNA片段有效序列和2 766 046条真菌ITS1 rDNA片段有效序列。PMA处理前后酒醅发酵过程中细菌及真菌Alpha多样性分析结果见表1。Chao1指数可以反映微生物群落的丰富度,其值越大代表物种丰富度越高,主要反映群落中物种的潜在多样性,尤其是对稀有物种的检测较为敏感。Shannon指数反映微生物群落的多样性,其值越大代表群落中物种的多样性越高,即物种不仅数量多,且分布较为均匀。
表1 叠氮溴化丙锭处理前后酒醅发酵过程中细菌及真菌菌群Alpha多样性指数
Table 1 Alpha diversity index of bacterial and fungal communities of fermented grains during fermentation process before and after treatment with propidium monoazide
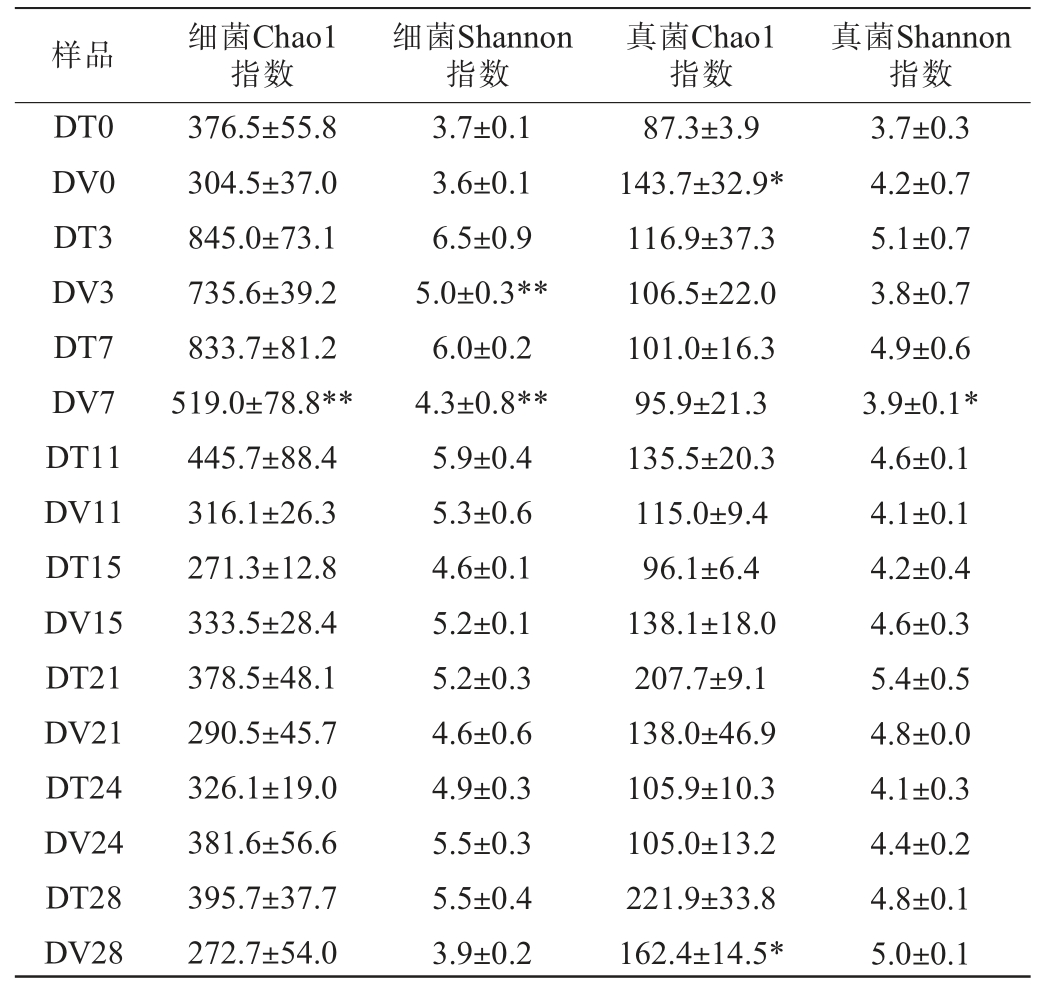
注:“*”表示与DT样本组相比,同一时间点DV样本组差异显著(P<0.05);“**”表示差异极显著(P<0.01)。
样品 细菌Chao1指数细菌Shannon指数真菌Chao1指数真菌Shannon指数DT0 DV0 DT3 DV3 DT7 DV7 DT11 DV11 DT15 DV15 DT21 DV21 DT24 DV24 DT28 DV28 376.5±55.8 304.5±37.0 845.0±73.1 735.6±39.2 833.7±81.2 519.0±78.8**445.7±88.4 316.1±26.3 271.3±12.8 333.5±28.4 378.5±48.1 290.5±45.7 326.1±19.0 381.6±56.6 395.7±37.7 272.7±54.0 3.7±0.1 3.6±0.1 6.5±0.9 5.0±0.3**6.0±0.2 4.3±0.8**5.9±0.4 5.3±0.6 4.6±0.1 5.2±0.1 5.2±0.3 4.6±0.6 4.9±0.3 5.5±0.3 5.5±0.4 3.9±0.2 87.3±3.9 143.7±32.9*116.9±37.3 106.5±22.0 101.0±16.3 95.9±21.3 135.5±20.3 115.0±9.4 96.1±6.4 138.1±18.0 207.7±9.1 138.0±46.9 105.9±10.3 105.0±13.2 221.9±33.8 162.4±14.5*3.7±0.3 4.2±0.7 5.1±0.7 3.8±0.7 4.9±0.6 3.9±0.1*4.6±0.1 4.1±0.1 4.2±0.4 4.6±0.3 5.4±0.5 4.8±0.0 4.1±0.3 4.4±0.2 4.8±0.1 5.0±0.1
由表1可知,在酒醅发酵过程中,DT、DV组酒醅细菌群落Chao1指数和Shannon指数变化趋势相似,Chao1指数均在酒醅发酵第3天时,呈上升趋势;分别在发酵第11天、第7天时呈下降趋势;在之后趋于稳定。在发酵7 d时,DV组酒醅Chao1指数极显著降低(P<0.01),表明在酒醅发酵第7天,活性细菌明显下降。两样本组Shannon指数均在酒醅发酵第3天时呈上升趋势;DT组酒醅Shannon指数在发酵第15天呈下降趋势;之后趋于稳定。DV组酒醅Shannon指数在发酵3天上升后就趋于稳定。两样本组细菌Shannon指数在发酵第3天和第7天差异极显著(P<0.01),这一结果表明在酒醅发酵第3天和第7天,相较于DT组,DV组酒醅中活细菌群落多样性和分布均匀性较低。对于真菌群落而言,DT组酒醅Chao1指数发酵过程中整体呈上升趋势;DV组Chao1指数在整个发酵过程中较为稳定。发酵28 d时,相较于DT组酒醅,DV组酒醅Chao1指数显著降低(P<0.05),表明在酒醅发酵第28天,活性真菌显著下降。两样本组酒醅Shannon指数在整个发酵过程中均没有较大波动;两样本组酒醅真菌Shannon指数在发酵第7天差异显著(P<0.05),这一结果表明在酒醅发酵第7天,相较于DT组酒醅,DV组酒醅中活真菌群落多样性和分布均匀性显著下降。与细菌群落多样性相比,真菌群落Chao1指数和Shannon指数较低,且在酒醅发酵7 d时,微生物死亡更替更加明显。
2.1.2 PMA处理前后酒醅发酵过程中微生物菌群的Beta多样性分析
主坐标分析(principalcoordinateanalysis,PCoA)是一种多维数据分析方法,主要用于探索样本之间的相似性或差异性,Adonis检验可评估不同时间点样本间差异显著性[15]。PMA处理前后酒醅发酵过程中细菌及真菌的主坐标分析结果见图1。
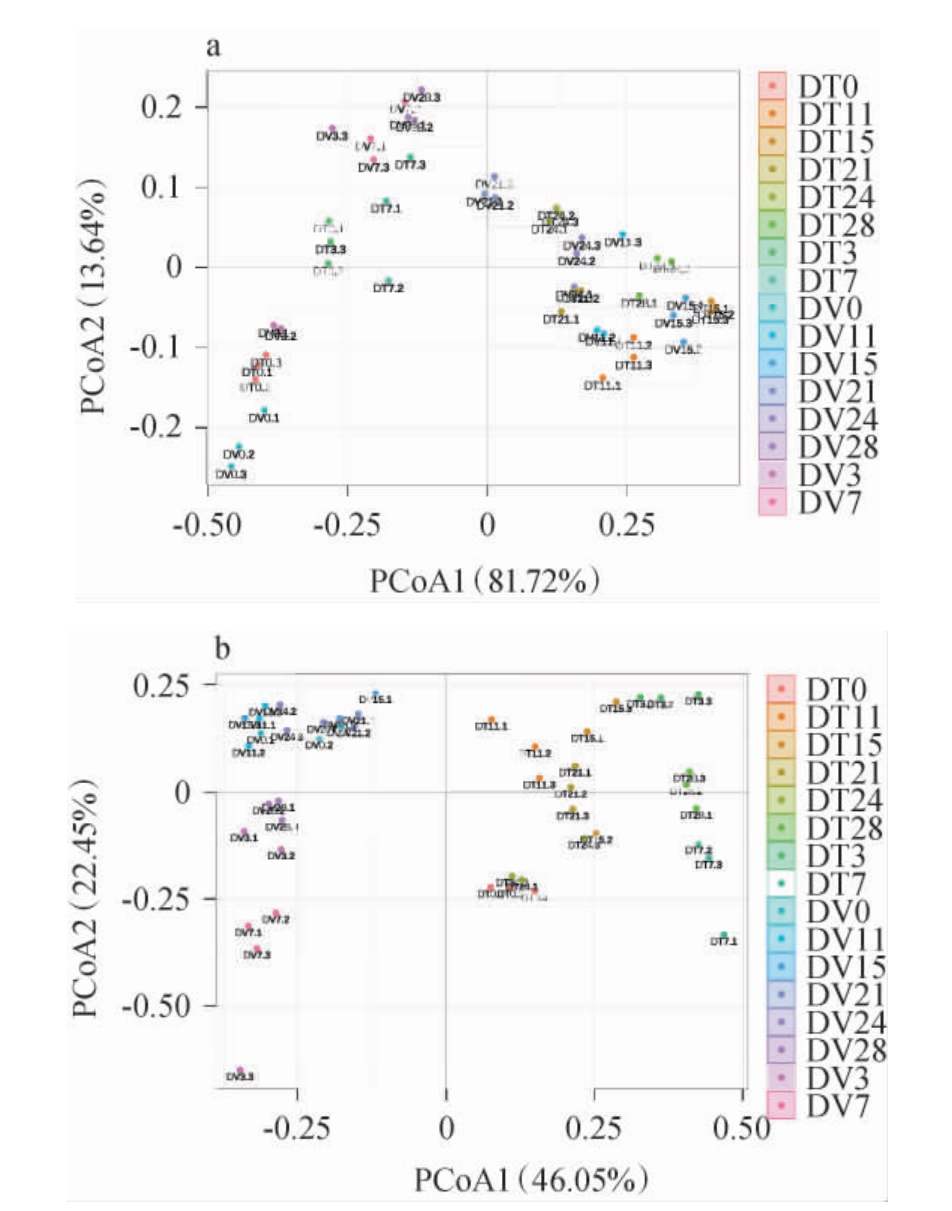
图1 叠氮溴化丙锭处理前后酒醅发酵过程中细菌(a)及真菌(b)的主坐标分析结果
Fig.1 Principal coordinate analysis results of bacteria (a) and fungi(b) of fermented grains during fermentation process before and after treatment with propidium monoazide
由图1a可知,DV、DT组酒醅沿PCoA1轴显著分离(解释率81.72%),表明PMA预处理对酒醅细菌群落的组成和分布具有显著影响(P<0.05),发酵0 d时,DT与DV组酒醅样本点高度重叠,表明初始菌群结构具有一致性;但随着发酵进行,两样本组细菌群落结构逐渐产生差异;发酵3~7 d时,两组样本分化并形成独立的聚类区域;发酵21 d时,两组样本的分离程度达到最大,直至酒醅发酵结束(28 d)。此外,同一处理组内不同发酵天数的酒醅样本沿PCoA2轴逐渐分散,体现了发酵时间对群落结构的塑造作用。由图1b可知,DV组酒醅样品聚集在左侧,DT组酒醅样品聚集在右侧,两样本组的聚类趋势差异相较于细菌群落更明显,且同一处理组内的样本随发酵时间延长沿PCoA2轴扩散。这一结果表明总微生物菌群与活微生物菌群存在显著差异(P<0.05)。
2.2 PMA处理前后酒醅发酵过程中微生物菌群组成
2.2.1 PMA处理前后酒醅发酵过程中细菌菌群组成
对相对丰度排名前10的微生物属进行分析,其他物种为others,将相对丰度>1.0%的菌属定义为优势菌属。基于属水平PMA处理前后酒醅发酵过程中细菌群落结构见图2。由图2可知,PMA处理前后酒醅样品中优势细菌属有乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)、无色杆菌属(Achromobacter)、芽孢杆菌属(Bacillus)、魏斯氏菌属(Weissella)、链球菌属(Streptococcus)、葡萄球菌属(Staphylococcus)、拟杆菌属(Bacteroides)、明串珠菌属(Leuconostoc)、大肠杆菌-志贺菌属(Escherichia-Shigella)。由图2a可知,在DT组酒醅样品中,Lactobacillus的相对丰度在发酵0~7 d稳定,发酵进行至11 d时,相对丰度从20.13%快速增至64.13%,并在发酵15 d达到峰值(75.90%),发酵15~24 d,Lactobacillus的相对丰度略有下降,但基本稳定,在发酵28 d结束时为64.65%;Pediococcus的相对丰度随着发酵的进行不断上升,在发酵7 d时达到最大值(11.81%),随发酵的进行呈波动下降趋势;发酵0~7 d时,Staphylococcus相对丰度在4%~6%范围内波动,发酵7~24 d时,Staphylococcus的相对丰度不断下降,但发酵至28 d时,Staphylococ cus相对丰度上升至9.22%;Bacteroides在发酵0 d相对丰度为7.60%,随着发酵进行,其相对丰度持续降低,至发酵中后期,其相对丰度已降至1%以下;Weissella与Bacteroides变化趋势相似,在发酵初期相对丰度最高(3.79%),随发酵的进行其相对丰度不断降低。由图2b可知,在DV组酒醅样品中,发酵0~15 d时,Lactobacillus的相对丰度增加,发酵15 d时相对丰度最高(71.1%),这一趋势与DT组相似;相较于DT组酒醅样品,Lactobacillus的相对丰度在发酵15~28 d期间内下降幅度较大,在发酵28 d时降至17.02%;Pediococcus的相对丰度同样在发酵7 d 时达到最高值(11.79%),发酵7 d之后相对丰度下降并趋于稳定,至发酵28 d时降低至1.99%;Staphylococcus的相对丰度在发酵3 d时达到最高值(4.77%),发酵3 d之后相对丰度下降并趋于稳定;Bacteroides、Weissella均在发酵初始相对丰度最高(分别为8.02%、3.58%),随着发酵的进行,相对丰度不断降低,Weissella的相对丰度在发酵后期降至1.87%,Bacteroides在发酵中后期未检测到。在两组样本中,Lactobacillus在DT组和DV组酒醅样品中平均相对丰度均为最高的优势菌属(42.4%、32.3%)。但与DT组酒醅样品相比,DV组Lactobacillus、Pediococcus、Staphylococcus平均相对丰度较低。结果表明,在DV组酒醅样本中,死菌细胞DNA被去除,群落中微生物的组成和菌属的相对丰度发生变化,酒醅样本中非活微生物DNA序列的存在遮盖了活菌菌群群落演替的信号[16]。
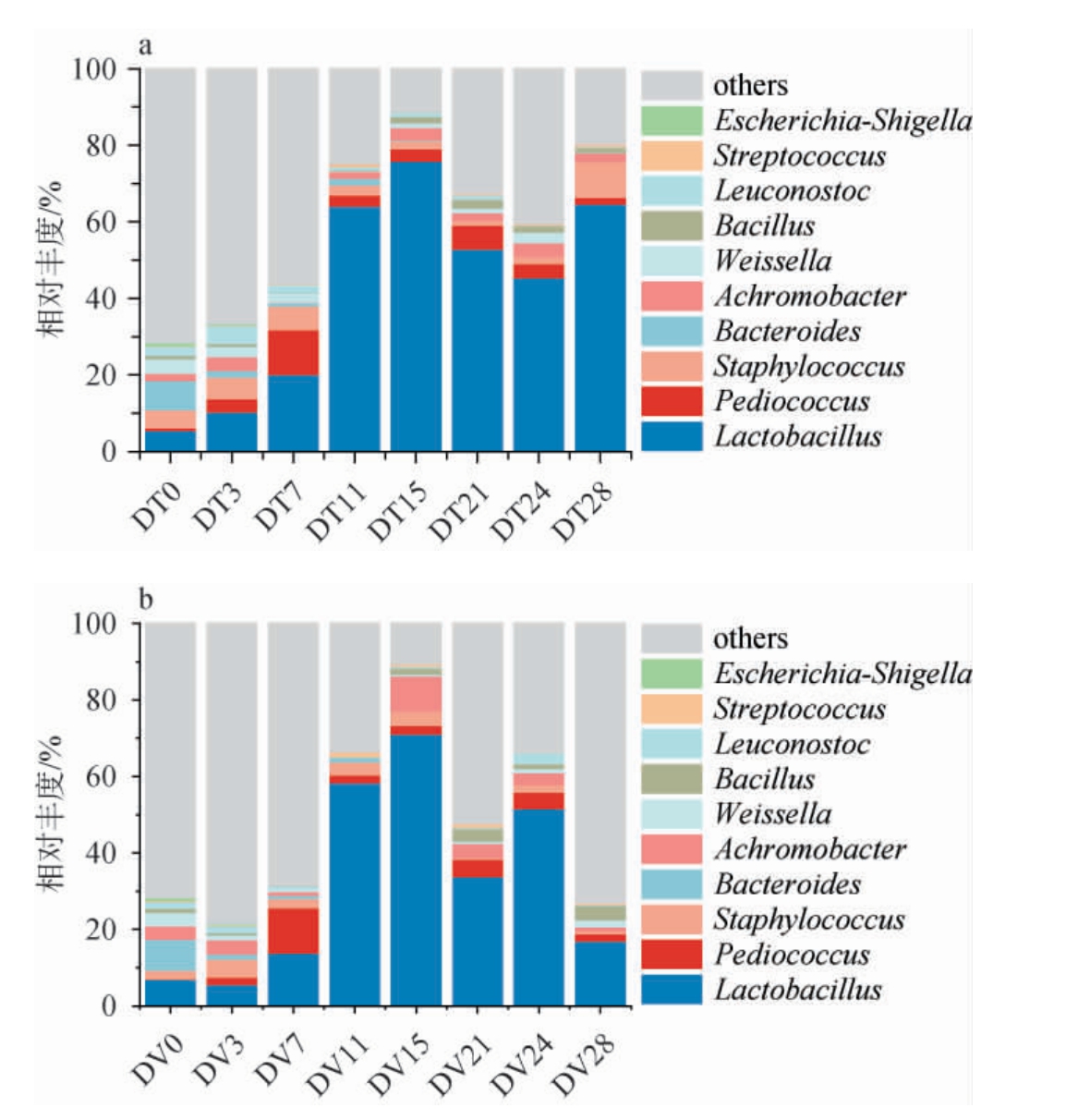
图2 基于属水平叠氮溴化丙锭处理前(a)、后(b)酒醅发酵过程中细菌群落结构
Fig.2 Bacterial community structure of fermented grains during fermentation process before (a) and after (b) treatment with propidium monoazide based on genus level
2.2.2 PMA处理前后酒醅发酵过程中真菌菌群组成
基于属水平PMA处理前后酒醅发酵过程中真菌群落结构见图3。由图3可知,PMA处理前后酒醅发酵过程中优势真菌属有覆膜孢酵母属(Saccharomycopsis)、伊萨酵母属(Issatchenkia)、有孢汉逊酵母属(Hanseniaspora)、曲霉菌属(Aspergillus)。由图3a可知,在DT组酒醅样品中,发酵0~7 d时,Saccharomycopsis的相对丰度从38.93%上升至82.13%;发酵7~24 d时,Saccharomycopsis的相对丰度整体呈下降趋势;发酵结束时,Saccharomycopsis的相对丰度回升至78.4%;Hanseniaspora、Pichia的相对丰度在发酵初始较低(分别为0.16%、0.29%);发酵至11 d时,Hanseniaspora相对丰度增加至28.39%,而Pichia在发酵24 d时相对丰度达到最高值(3.05%),发酵结束时,其相对丰度分别降低至1.37%和1.10%。由图3b可知,发酵初始时,Saccharomycopsis、Aspergillus、Cladosporium、Pichia相对丰度较高,当发酵0~11 d 时,Saccharomycopsis、Aspergillus、Issatchenkia、Hanse niaspora、Cladosporium的相对丰度降低,至发酵15 d时,Saccharomycopsis、Issatchenkia、Hanseniaspora、Cladosporium的相对丰度上升,相对丰度分别为16.39%、7.04%、4.09%、17.77%;发酵15~28 d时,Issatchenkia、Wickerhamomyces等真菌属相对丰度呈下降趋势,这些真菌属的丰度变化呈现了明显的微生物演替特征,表明在发酵过程中存在活菌群的演替活动。 Saccharomycopsis为DT、DV组酒醅共有的相对丰度最高的优势菌属(58.9%、11.3%),与DT组酒醅相比,DV 组 酒 醅 中Saccharomycopsis、Issatchenkia、Hanseniaspora的平均相对丰度较低,而Aspergillus、Cladosporium、Fusarium、Pichia、Thermoascus的平均相对丰度较高。
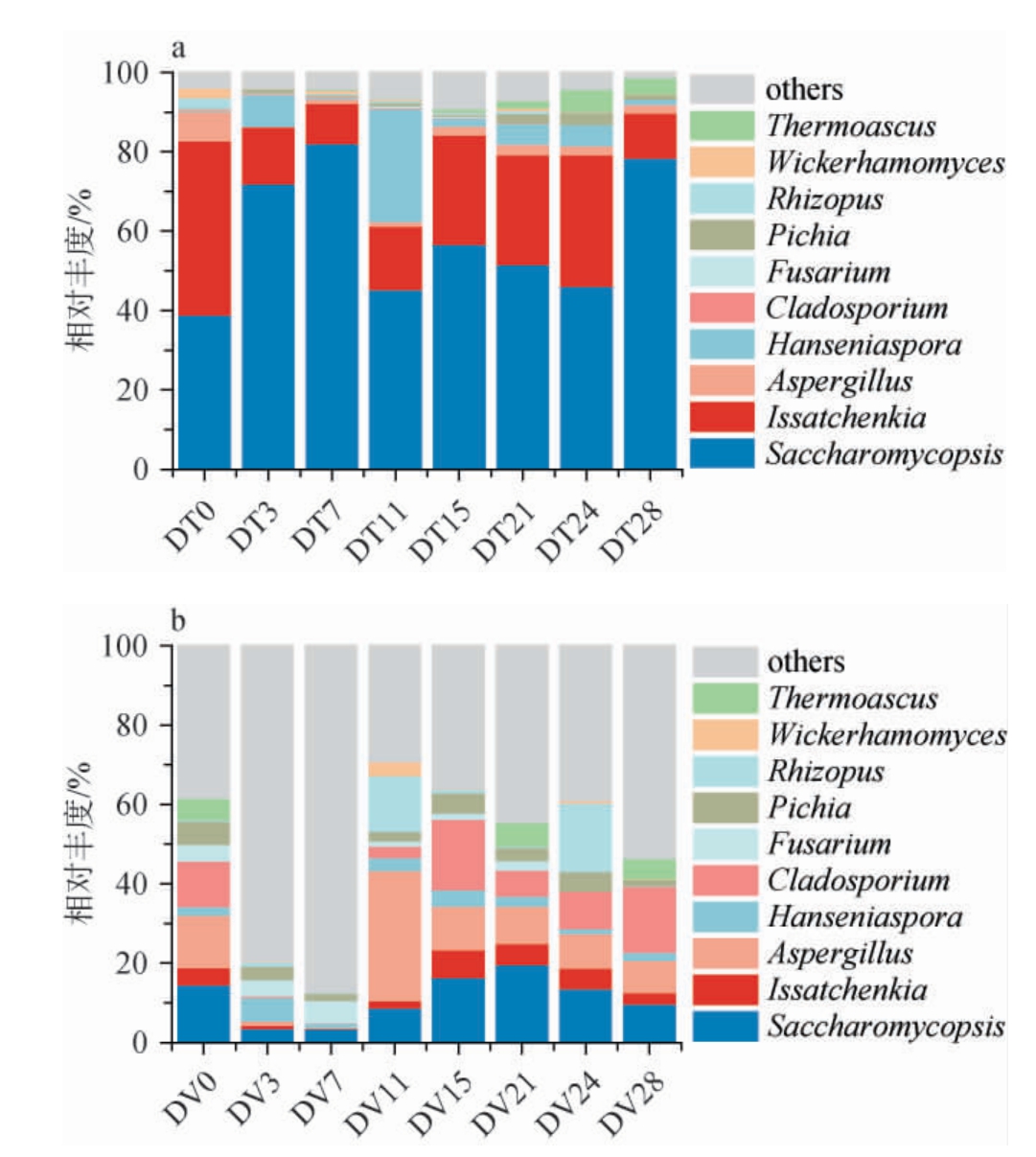
图3 基于属水平叠氮溴化丙锭处理前(a)、后(b)酒醅发酵过程中真菌群落结构
Fig.3 Fungal community structure of fermented grains during fermentation process before (a) and after (b) treatment with propidium monoazide based on genus level
2.3 PMA处理前后酒醅发酵过程中差异微生物菌属分析
2.3.1 差异细菌属分析
线性判别分析效应大小(LEfSe)是微生物组学研究领域用于发现和解释高维度数据中生物标识差异的分析方法,LDA值越大,表明该生物标识在区分不同组别中的贡献越大,LDA值>4.0,P<0.05为显著差异微生物[17]。PMA处理前、后酒醅发酵过程中差异细菌属线性判别分析效应大小分析见图4。由图4a可知,DT组酒醅在发酵初始(0 d)时,差异细菌属为Weissella、Escherichia-shigella、Bacillus;发酵3 d时,差异细菌属为Staphylococcus、Achromobacter、Leuconostoc;发酵7 d时,差异细菌属为Pediococcus;发酵11 d时,差异细菌属为Streptococcus;发酵15 d时,差异细菌属为Lactobacillus。由图4b可知,DV组酒醅在发酵初始(0 d)时,差异细菌属为Bacteroides;发酵11 d时,差异细菌属为Lactobacillus;发酵21 d时,差异细菌属为Streptococcus;发酵28 d时,差异细菌属为Bacillus。采用t-检验对PMA预处理前后发酵过程中酒醅差异细菌属进行统计分析发现,差异细菌属主要有Lactobacillus、Staphylococcus、Bacteroides、Achromobacter、Weissella、Bacillus,这一结 果表明总菌群中检测的菌属丰度信号可能由一部分发酵过程中死菌细胞DNA提供。
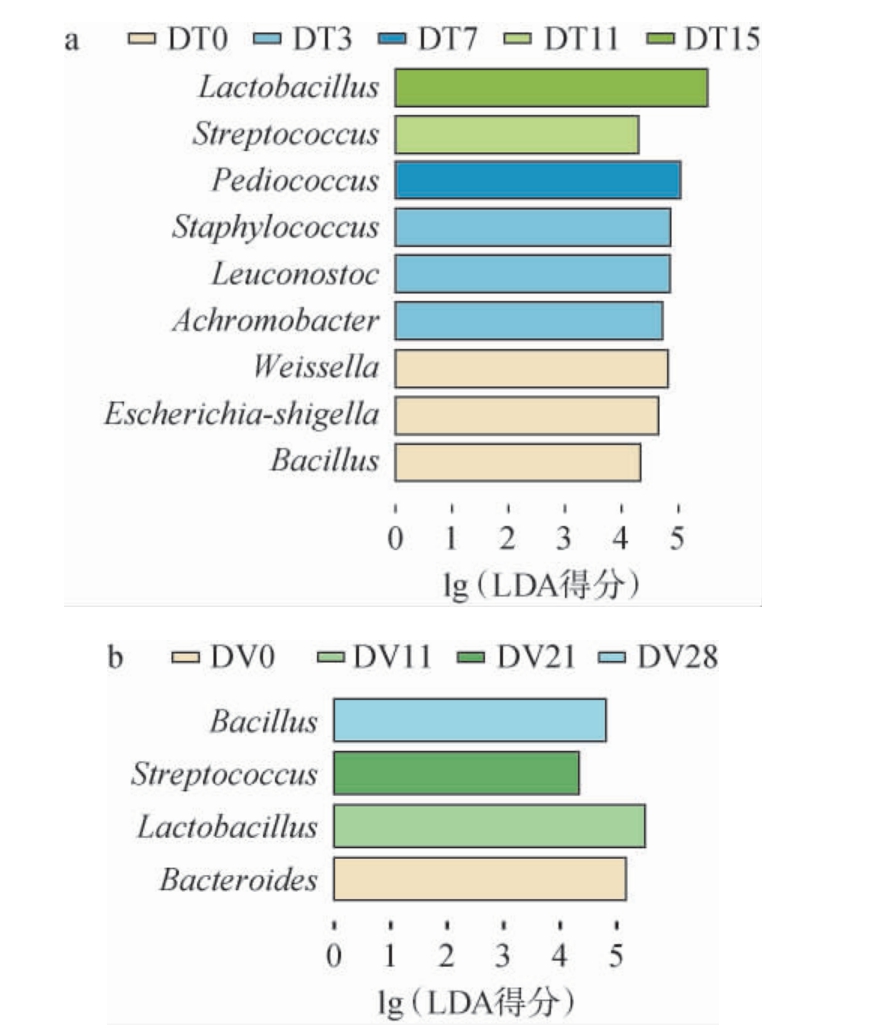
图4 叠氮溴化丙锭处理前(a)、后(b)酒醅发酵过程中差异细菌属线性判别分析效应大小
Fig.4 Linear discriminant analysis effect size of differential bacterial genera of fermented grains during fermentation process before(a) and after (b) treatment with propidium monoazide
2.3.2 差异真菌属分析
PMA处理前、后酒醅发酵过程中差异细菌属线性判别分析效应大小分析见图5。由图5a可知,DT组酒醅发酵0 d时,差异真菌属为Issatchenkia、Aspergillus、Wickerhamomyces;发酵至7 d时,差异真菌属为Saccharomycopsis;发酵至11 d时,差异真菌属为Hanseniaspora、Cladosporium;发酵至24 d时,差异真菌属为Thermoascus、Pichia。由图5b可知,DV组的酒醅样品在发酵11 d时,差异真菌属为Aspergillus;发酵15 d时,差异真菌属为Issatchenkia;发酵24 d时,差异真菌属为Rhizopus;发酵28 d时,差异真菌属为Cladosporium、Thermoascus。主要差异真菌属有Issatchenkia、Aspergillus、Wickerhamomyces等。两组酒醅间仅在Saccharomycopsis、Issatchenkia的相对丰度上存在极显著差异(P<0.01),这可能是因为此时真菌微生物生长代谢旺盛,活真菌属的多样性较高且生物量达到峰值,使群落结构相对稳定,从而减少了组间差异。
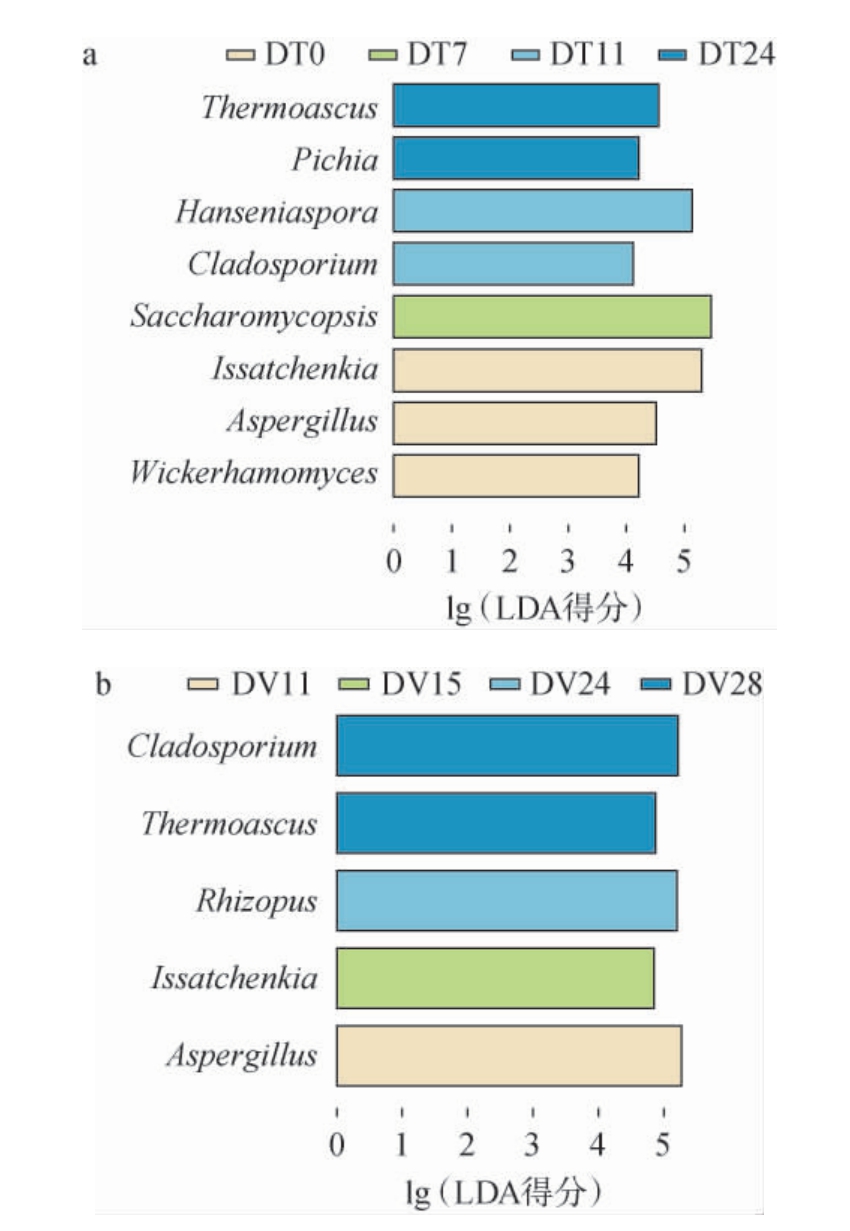
图5 叠氮溴化丙锭处理前(a)、后(b)酒醅发酵过程中差异真菌属线性判别分析效应大小
Fig.5 Linear discriminant analysis effect size of differential fungal genera of fermented grains during fermentation process before (a) and after (b) treatment with propidium monoazide
3 讨论
酒醅为微生物提供了生长所需的营养,同时也在微生物生长环境的变化会影响微生物群落的多样性[18]。Alpha多样性分析结果表明,酒醅发酵过程中,细菌群落的Alpha多样性指数变化幅度较真菌群落更大,且细菌群落的多样性较真菌群落更丰富,这与王喆[19]对小曲清香型白酒的Alpha多样性分析得到的细菌群落的多样性远高于真菌群落的结果一致。总菌群与活菌群中微生物多样性指数主要在发酵3 d和7 d的酒醅中产生了显著差异,可能是由于发酵前期的营养物质丰富,微生物相互竞争,死菌细胞增多,导致微生物多样性降低,而在发酵后期营养物质逐渐被消耗,促进了微生物间的共生互作关系,共生关系中产生的死菌细胞减少[20]。总菌群及活菌群的优势细菌与优势真菌组成相似,并且这些活菌属组成与大曲微生物具有高度相似性[21],这也表明部分活微生物由大曲发酵剂中的微生物迁移而来。在总细菌群落和活细菌群落中,相对丰度较高的细菌属 有Lactobacillus、Pediococcus、Achromobacter、Bacillus、Weissella、Streptococcus、Staphylococcus、Bacteroides、Leuconostoc;总真菌和活真菌群落中优势真菌属有Saccharomycopsis、Issatchenkia、Hanseniaspora、Aspergillus,这些优势活菌属与对浓香型白酒酒醅样本使用宏转录组测序得到的优势活菌属结果相似[22],优势菌属的组成与之前文献报道清香型酒醅优势菌属的组成亦相似[23]。
对细菌群落结构分析发现,Lactobacillus在总、活细菌群中均为优势菌属,是构成细菌群落的主要菌属,总菌群与活菌群中Lactobacillus的相对丰度差异小,仅在发酵后期时,活细菌群中的Lactobacillus相对丰度显著降低,这也表明在酒醅发酵过程中,Lactobacillus生长代谢活跃,保持较高活性,这可能与其为耐酸及兼性厌氧的特性相关[9]。在真菌群落中,发酵3 d和7 d酒醅中存在大量死菌,主要是由于Saccharomycopsis、Issatchenkia、Aspergillus等 菌 属 相 对丰度在发酵至这两个时间点迅速降低,其中酵母类微生物的减少可能由营养物质的竞争和代谢产物的抑制引起[24]。此外,酒醅含水量过高,氧气难以进入物料内部,不利于疏松通气,霉菌和酵母菌的生长也会受到抑制[25]。
PCoA结果显示,在细菌群落分析中,代表总菌群的DT组样本和代表活菌群的DV组样本未有明显的分离,但真菌群落分析中的DT组样本与DV组样本产生了明显的分离。进一步根据t-检验筛选总菌群和活菌群中的显著差异细菌和真菌,结果显示主要在发酵前期(0~7 d)及发酵结束(28 d)时产生的差异性菌属较多,发酵前期产生的差异可能是由于发酵前期部分菌属不能适应极端的发酵环境导致微生物死亡[26],而发酵后期产生较多差异菌属可能是由于酒醅发酵过程中产生的死菌细胞堆积产生[27-28]。
4 结论
本研究以大曲清香型白酒大米查酒醅为研究对象,采用扩增子测序技术系统解析叠氮溴化丙锭(PMA)处理前、后酒醅发酵过程中微生物群落组成,并通过线性判别分析效应大小(LEfSe)分析不同酒醅中差异菌群。Alpha多样性结果表明,酒醅发酵7 d时,DV组酒醅中活细菌、活真菌菌群多样性和分布均匀性均较低。Beta多样性结果表明,总微生物菌群与活微生物菌群存在显著差异(P<0.05)。DV组、DT组酒醅优势细菌属、真菌属(相对丰度>1%)均分别为乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)、无色杆菌属(Achromobacter)等;覆膜孢酵母属(Saccharomycopsis)、伊萨酵母属(Issatchenkia)、有孢汉逊酵母属(Hanseniaspora)等,其中,Lactobacillus、Saccharomycopsis分别为DV组、DT组酒醅中平均相对丰度最高的细菌属及真菌属。两组酒醅差异极显著菌群为Saccharomycopsis和Issatchenkia(LDA值>4.0,P<0.01)。由此,揭示酒醅发酵机制不应只关注总菌群的组成,同时也应该解析活菌菌群结构,这对于准确揭示酒醅微生物发酵机理与风味形成机制十分必要。本研究为其他香型白酒及传统发酵食品活菌群分析提供了参考。
[1]刘凡,周新虎,陈翔,等.洋河浓香型白酒发酵过程酒醅微生物群落结构解析及其与有机酸合成的相关性[J].微生物学报,2018,58(12):2087-2099.
[2]BLAGODATSKAYA E,KUZYAKOV Y.Active microorganisms in soil:critical review of estimation criteria and approaches[J].Soil Biol Biochem,2013,67:192-211.
[3]ZHANG Z Y, WEI Y W, PENG Z H, et al.Exploration of microbiome diversity of stacked fermented grains by flow cytometry and cell sorting[J].Front Microbiol,2023,14:1160552.
[4]ROTH P,HILL-SPANIK K M,MCCURRY C,et al.Propidium monoazidedenaturing gradient gel electrophoresis(PMA-DGGE)assay for the characterization of viable diatoms in marine sediments[J].Diatom Res,2017,32(3):341-350.
[5]SOLDEN L, LLOYD K, WRIGHTON K.The bright side of microbial dark matter:lessons learned from the uncultivated majority[J].Curr Opin Microbiol,2016,31:217-226.
[6]卫博,王杰,陈亦新,等.基于PMA-CELL-qPCR 的葡萄酒发酵中酿酒酵母活菌计数方法的开发与应用[J].食品与发酵工业,2022,48(17):79-86.
[7]吕旭聪,刘志彬,张雯,等.红曲黄酒传统酿造过程中优势细菌活菌检测方法的构建及应用[J].中国食品学报,2018,18(7):210-219.
[8]LIU D T,WANG H,WANG L T,et al.Abiotic factors modulating metabolic pathways of lactic acid in solid-state fermentation of cereal vinegar[J].LWT-Food Sci Technol,2024,214:117092.
[9]LUO A G,YANG N T,YANG J,et al.Effects of microbial interspecies relationships and physicochemical parameters on volatile flavors in sorghumbased fermented grains during the fermentation of Shanxi light-flavored liquor[J].Food Sci Nutr,2023,11(3):1452-1462.
[10]LIU M K,TANG Y M,LIU C Y,et al.Variation in microbiological heterogeneity in Chinese strong-flavor Baijiu fermentation for four representative varieties of sorghum[J].Int J Food Microbiol, 2023, 397:110212.
[11]TANG J,LIN B,SHAN Y M,et al.Effects of sorghum varieties on microbial communities and volatile compounds in the fermentation of light-flavor Baijiu[J].Front Microbiol,2024,15:1421928.
[12]ZHAO X Y,LI J H,DU G C,et al.The influence of seasons on the composition of microbial communities and the content of lactic acid during the fermentation of fen-flavor Baijiu[J].Fermentation,2022,8(12):740.
[13]陶京兰,陆震鸣,王宗敏,等.实时荧光定量PCR监测镇江香醋醋酸发酵过程中微生物变化[J].食品与发酵工业,2013,39(2):156-160.
[14]WANG J,HAO S,REN Q J F.Analysis of bacterial diversity in fermented grains of Baijiu based on culturomics and amplicon sequencing[J].Fermentation,2023,9(3):260.
[15]ANDERSON M J.Permutational multivariate analysis of variance(PERMANOVA)[M].Wiley statsref: statistics reference online, 2014:1-15.
[16]AL M A,CHEN H H,YAN X,et al.Relic DNA confounds the results of DNA-based study on microeukaryotic succession in aquatic biofilms[J].Ecol Ind,2024,158:111350.
[17]WU D L,WANG C J,SIMUJIDE H,et al.Reproductive hormones mediate intestinal microbiota shifts during estrus synchronization in grazing simmental cows[J].Animals,2022,12(14):1751.
[18]ZHU Y L, HUANG Y J, NUERHAMANTI N, et al.The composition and diversity of the rhizosphere bacterial community of Ammodendron bifolium growing in the Takeermohuer Desert are different from those in the nonrhizosphere[J].Microb Ecol,2024,87(1):2.
[19]王喆.特色小曲清香型白酒关键挥发性化合物及其相关微生物分析研究[D].武汉:华中农业大学,2021.
[20]刘小刚.浓香白酒糟长发酵期代谢机理解析及抗氧化醇溶肽的制备与鉴定[D].无锡:江南大学,2023.
[21]ZHENG Y f,LIANG F,WU Y,et al.Unraveling multifunction of lowtemperature Daqu in simultaneous saccharification and fermentation of Chinese light aroma type liquor[J].Int J Food Microbiol,2023, 397:110202.
[22]王康丽.基于宏转录组学的浓香型酒醅微生物群落多样性及代谢特征研究[D].郑州:郑州轻工业大学,2021.
[23]VILLASANTE A,RAMÍREZ C,RODRÍGUEZ H,et al.Dietary carbohydrate-to-protein ratio influences growth performance, hepatic health and dynamic of gut microbiota in atlantic salmon(Salmo salar)[J].Anim Nutr,2022,10:261-279.
[24]WANG D Q,CHEN L Q,YANG F,et al.Yeasts and their importance to the flavour of traditional Chinese liquor:a review[J].J I Brewing,2019,125(2):214-221.
[25]刘霄,黄治国,卫春会,等.扣囊复膜酵母麸曲制备及其在小曲白酒中的应用[J].食品科学,2025,46(1):56-63.
[26]LIU C J,GONG X W,ZHAO G,et al.Liquor flavour is associated with the physicochemical property and microbial diversity of fermented grains in waxy and non-waxy sorghum(Sorghum bicolor)during fermentation[J].Front Microbiol,2021,12:618458.
[27]JIANG X Y,PENG Z,ZHANG J.Starting with screening strains to construct synthetic microbial communities (SynComs) for traditional food fermentation[J].Food Res Int,2024,190:114557.
[28]谭光迅.基于活菌数据的浓香型白酒酿造微生物组成,来源和变化规律研究[D].武汉:华中农业大学,2020.