高温大曲中蕴含芽孢杆菌属(Bacillus)和克罗彭施泰特氏菌属(Kroppenstedtia)等嗜热细菌菌群[1]以及嗜热子囊菌属(Thermoascus)和曲霉菌属(Aspergillus)等嗜热真菌菌群[2]。 微生物群落结构的形成促进了大曲发酵过程中淀粉原料、蛋白质和其他物质的分解利用,从而产生丰富的代谢物,对于白酒风味品质的形成具有重要作用[3]。目前,越来越多的科研人员对大曲中蕴含的功能微生物进行分离、筛选和强化富集,并将其用于大曲的发酵中以提高白酒的品质[4],因此,采用纯培养技术对大曲中微生物进行分离鉴定是获取酿酒微生物菌株资源的主要手段。纯培养技术难以完全模拟微生物生长的原生自然环境,如温度、湿度、氧气、盐浓度和酸碱度等,故而仅能对其部分微生物进行分离,这就导致多数微生物在实验室条件下是不可培养的[5]。因此,实现高温大曲中不可培养微生物的可培养是亟待解决的问题。
第二代高通量测序技术的Illumina MiSeq实现了对于样本中微生物多样性较为全面和客观的揭示[6],在此基础上,根据其微生物组成指导后续选用不同的培养基,并改良培养基的成分和培养温度,尽可能多地从样本中获取常规培养方式无法获取的微生物类群[7]。影响微生物生长的因素包括温度、水分、营养成分和氧气等,其中温度对微生物的酶活和细胞膜的流动性均存在影响[8]。因此,明确高温大曲中各类微生物的适宜生长温度,是实现不可培养微生物可培养策略的必要条件之一。
高温大曲的培菌方式遵循“前缓中挺后缓落”的原则[9],其制曲温度可从室温升至65 ℃[10],温度变化范围较大,然而微生物适宜生长的温度范围相对较小[11],因此依托制曲工艺难以明确高温大曲中各类微生物适宜生长的温度。摇瓶发酵可在实验室条件下,根据菌株的生理特性需要控制不同的培养条件,其在保持恒定制曲温度的同时通过摇床振荡培养赋予氧气,目前在微生物的生长特性评价上有一定应用[12]。基于此,本研究首先将高温大曲与纯水混合、离心制取浸提液,然后接入由4个种高温大曲样品混合而成的曲粉,基于摇瓶实验在40~65 ℃的温度范围内培养,并使用Illumina MiSeq高通量测序技术对不同培养温度下的优势菌群进行解析,同时预测细菌的基因功能。 以期明确高温大曲中特定微生物类群的适宜生长温度,为酿酒微生物菌株的挖掘以及后续微生物强化接种改善大曲和白酒品质提供理论指导。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
高温大曲(成品):采自贵州省仁怀市、河南省永城市、山东省临沂市和潍坊市等地的4个酿酒公司的制曲车间,每个地区各采集10块,共采集40块。
1.1.2 试剂
基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒:德国QIAGEN公司;细菌引物(338F/806R)、真菌引物(ITS3F/ITS4R):武汉天一辉远生物科技有限公司;DNA聚合酶、脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)缓冲液、rTaq酶(5 U/μL)、pMD18-T载体、DL 2 000 Marker、10×聚合酶链式反应(polymerase chain reaction,PCR)缓冲液:宝生物工程大连有限公司;Axygen清洁试剂盒:康宁生命科学吴江有限公司;Illumina MiSeq测序配套试剂:美国Illumina公司。
1.2 仪器与设备
LD-Y300A高速万能粉碎机:上海顶帅电器有限公司;QYC-2102C全温培养摇床:上海新苗医疗器械制造有限公司;CR21N型高速离心机:日本日立金属株式会社;Veriti96-Well梯度基因扩增仪:美国Applied Biosystems公司;164-5050基础电泳仪:美国Bio-Rad公司;UVPCDS8000凝胶成像分析系统:美国UVP公司;MiSeq PE300高通量测序平台:美国Illumina公司;R930机架式服务器:美国DELL公司。
1.3 方法
1.3.1 高温大曲浸提液的制备
将采集的40块高温大曲粉碎,备用。为了使样品中微生物的多样性更高,从所有大曲样品中各取100 g混合成1个样品。将混合高温大曲曲粉与纯水以3∶10(g∶mL)的比例混合,在常温下浸泡30 min后以5 000 r/min的转速离心10 min,过滤取上清液即为浸提液[13]。
1.3.2 基于摇瓶实验不同温度下高温大曲源微生物的培养
在无菌条件下将混合高温大曲曲粉与浸提液以5∶100(g∶mL)的比例混合,装于500 mL三角瓶中,分别于40 ℃、45 ℃、50 ℃、55 ℃、60 ℃和65 ℃条件下在摇床中150 r/min振荡培养7 d,每个温度梯度设置5个平行,样品编号为T40(T40-1~T40-5)、T45(T45-1~T45-5)、T50(T50-1~T50-5)、T55(T55-1~T55-5)、T60(T60-1~T60-5)和T65(T65-1~T65-5)。将不同培养温度下的样品以5 000 r/min的转速离心10 min,过滤,取沉淀置于50 mL离心管中,封口并暂存于-20 ℃冰箱,备用。
1.3.3 宏基因组DNA的提取、PCR扩增和Illumina MiSeq高通量测序
参照基因组DNA提取试剂盒的说明提取不同培养温度样品的DNA,并以其为模板,参照CAI WC等[6]的PCR扩增条件使用引物338F(5'-ACTCCTACGGGAGGCAGCAG-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3')对细菌16S rRNA的V3~V4区基因序列进行PCR扩增,使用引物ITS3F(5'-GCATCGATGAAGAACGCAGC-3')和ITS4R(5'-TCCTCCGCTTATTGATATGC-3')对真菌ITS2区基因序列进行PCR扩增。PCR扩增产物经1.0%琼脂糖凝胶电泳检测后,将合格且纯化后的PCR扩增产物寄往上海美吉生物医药科技有限公司,基于MiSeq PE300高通量测序平台进行Illumina MiSeq高通量测序。
1.3.4 生物信息学分析
参照ZHANG Z D等[14]的方法并基于QIIME平台(v1.90)对Illumina MiSeq测序产生的每个样品的双端序列进行拼接与质控。同时在此平台对不同培养温度样品的微生物多样性进行分析。具体来讲,采用UPARSE软件在≥97%相似度条件下划分操作分类单元(operational taxonomic units,OTU)[15]。 使用ChimeraSlayer检查剔除含嵌合体的OTU[16],使用RDP(v11.5)数据库[17]对细菌物种进行注释,同时使用UNITE(v7.2)数据库[18]对真菌物种进行注释,此外通过计算Chao1指数和香农指数评价各样品中微生物的Alpha多样性(丰富度和多样性),最后参考蛋白质直系同源簇数据库(clusters of orthologous groups,COGs)[19]并基于PICRUSt 2软件对潜在的基因功能进行注释[20]。
1.3.5 数据处理
将5个平行样品测序数据的平均值作为对应培养温度下的测试值,并采用该平均值进行后续的分析。 使用R(v4.1.2)软件进行气泡图和相关性拟合曲线图的可视化,使用Cytoscape(v3.7.2)软件进行共现网络图的可视化。 采用SPSS_29.0.2.0软件进行方差分析,评估不同培养温度下高温大曲微生物及细菌基因功能数据间差异的显著性。
2 结果与分析
2.1 不同培养温度下样品微生物菌群的Illumina MiSeq高通量测序结果及Alpha多样性分析
在60 ℃和65 ℃的培养温度下未见明显的菌群生长,因此,使用Illumina MiSeq高通量测序技术对4个培养温度(40 ℃、45 ℃、50 ℃、55 ℃)条件下样品的微生物类群进行高通量测序及Alpha多样性分析,结果见表1。
表1 不同培养温度下样品微生物菌群Illumina MiSeq高通量测序结果及Alpha多样性分析结果
Table 1 Illumina Miseq high-throughput sequencing and Alpha diversity analysis results of microbial communities in samples at different culturing temperature
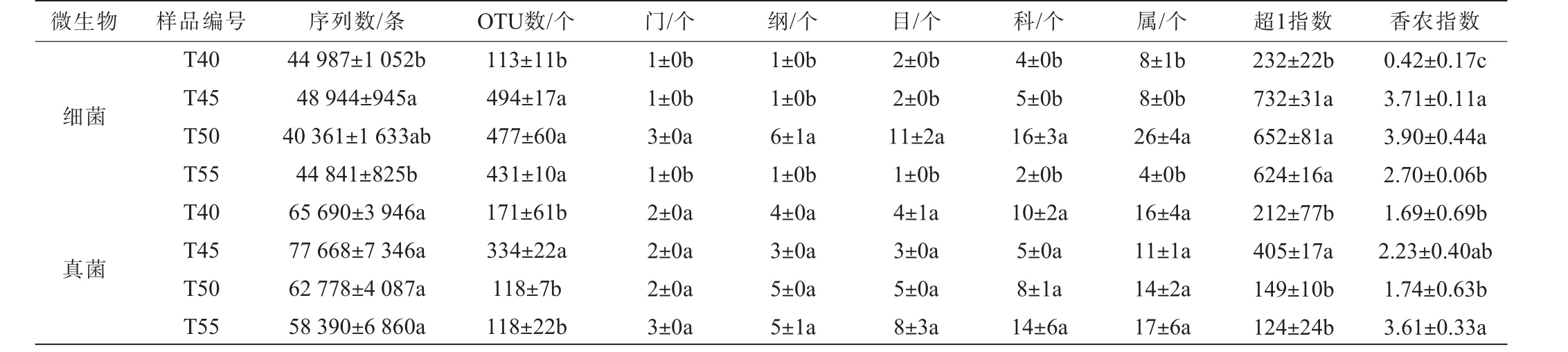
注:细菌超1指数和香农指数均在测序量为41 010条时计算所得;真菌超1指数和香农指数均在测序量为46 010条时计算所得;同列不同字母表示差异显著(P<0.05)。 下同。
微生物 样品编号 序列数/条 OTU数/个 门/个 纲/个 目/个 科/个 属/个 超1指数 香农指数细菌真菌T40 T45 T50 T55 T40 T45 T50 T55 44 987±1 052b 48 944±945a 40 361±1 633ab 44 841±825b 65 690±3 946a 77 668±7 346a 62 778±4 087a 58 390±6 860a 113±11b 494±17a 477±60a 431±10a 171±61b 334±22a 118±7b 118±22b 1±0b 1±0b 3±0a 1±0b 2±0a 2±0a 2±0a 3±0a 1±0b 1±0b 6±1a 1±0b 4±0a 3±0a 5±0a 5±1a 2±0b 2±0b 11±2a 1±0b 4±1a 3±0a 5±0a 8±3a 4±0b 5±0b 16±3a 2±0b 10±2a 5±0a 8±1a 14±6a 8±1b 8±0b 26±4a 4±0b 16±4a 11±1a 14±2a 17±6a 232±22b 732±31a 652±81a 624±16a 212±77b 405±17a 149±10b 124±24b 0.42±0.17c 3.71±0.11a 3.90±0.44a 2.70±0.06b 1.69±0.69b 2.23±0.40ab 1.74±0.63b 3.61±0.33a
由表1可知,平均每个样品的细菌菌群可划分为1 677个OTU,对序列进行比对后显示共隶属于5个门、11个纲、20个目、32个科和50个属,其中不可鉴定到细菌属的序列仅占3.73%。当培养温度为45 ℃、50 ℃和55 ℃时,细菌菌群的超1指数差异不显著(P>0.05),但均显著高于40 ℃(P<0.05)。当培养温度为45 ℃和50 ℃时,细菌菌群的香农指数差异不显著(P>0.05),但均显著高于40 ℃和55 ℃(P<0.05)。由此可见,基于摇瓶实验,在45 ℃和50 ℃时,样品细菌菌群的丰富度和多样性均最高,55 ℃时次之,而40 ℃时最低。
由表1亦可知,平均每个样品的真菌菌群可划分为1 203个OTU,通过比对后发现共隶属于7个门、15个纲、29个目、54个科和76个属,其中不可鉴定到真菌属的序列仅占总序列的6.57%。 当培养温度为45 ℃时,真菌菌群的超1指数显著偏高(P<0.05),而在其余3个培养温度下差异不显著(P>0.05)。 当培养温度为45 ℃和55 ℃时,真菌菌群的香农指数差异不显著(P>0.05),但均明显高于40 ℃和50 ℃。由此可见,基于摇瓶实验,在45 ℃时,样品真菌菌群的丰富度和多样性均最高,而40 ℃和50 ℃时较低。
2.2 不同培养温度下样品微生物类群结构分析
基于属水平对不同培养温度下样品的微生物类群结构进行分析,并对优势细菌属(所有样品中平均相对含量>1.0%)间和优势真菌属间的相关性进行分析,结果见图1。
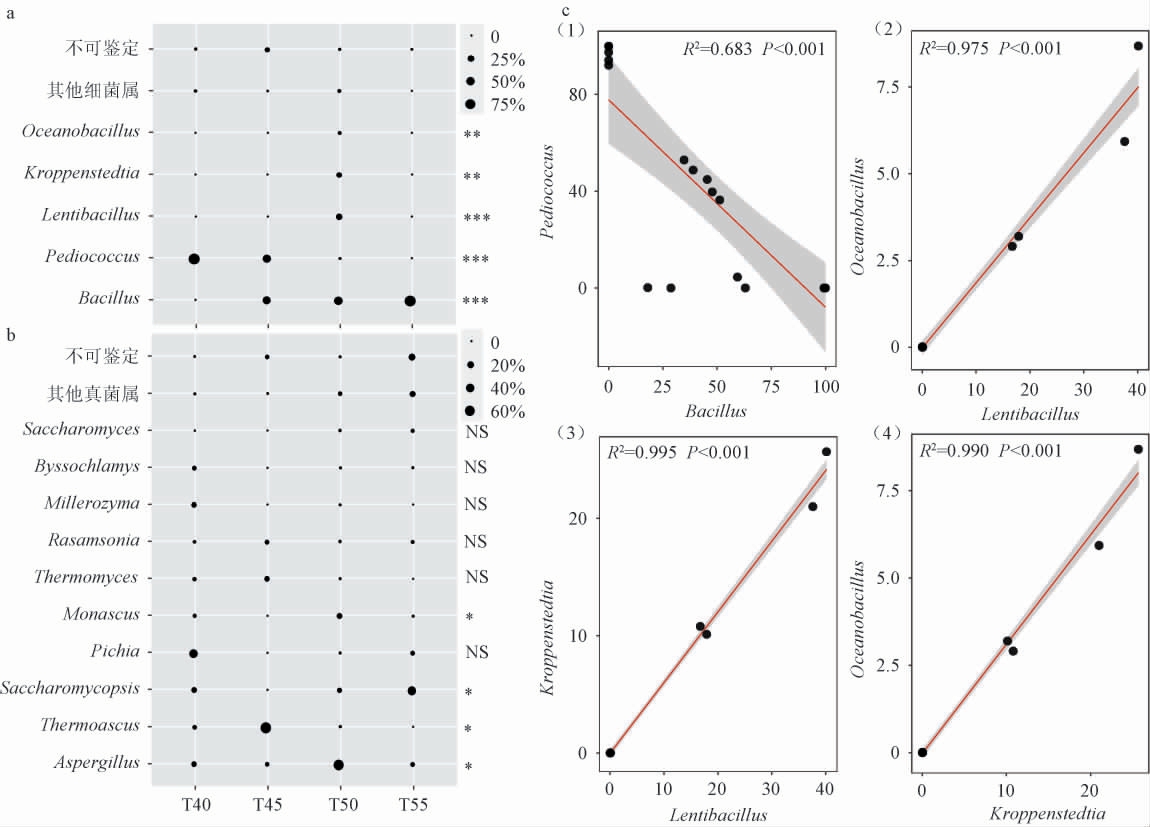
图1 不同培养温度下样品细菌(a)、真菌(b)菌群结构及差异细菌属间相关性分析结果(c)
Fig.1 Bacterial (a) and fungal (b) community structures, and correlation analysis results among different bacterial genera (c) in samples at different culturing temperatures
**代表差异极显著(P<0.01),“*”代表差异显著(P<0.05),NS代表无显著差异(P>0.05)。 下同。
由图1a可知,从不同培养温度下的样品中共注释到5个优势细菌属,分别为Bacillus、片球菌属(Pediococcus)、类芽孢杆菌属(Lentibacillus)、Kroppenstedtia和大洋芽孢杆菌属(Oceanobacillus)。 其中,40 ℃培养温度下样品的细菌菌群以Pediococcus为主,相对含量为96.71%;45 ℃培养温度下样品的细菌菌群以Pediococcus和Bacillus为主,相对含量分别为44.52%和43.61%;50 ℃培养温度下样品的细菌菌群以Bacillus、Lentibacillus和Kroppenstedtia为主,相对含量分别为53.79%、22.49%和13.52%;55 ℃的培养温度下样品的细菌菌群以Bacillus为主,相对含量为99.55%。由图1b可知,Bacillus和Pediococcus的相对含量呈极显著负相关(P <0.01),Lentibacillus、Kroppenstedtia 和Oceanobacillus三者的相对含量之间均呈极显著正相关(P<0.01)。由此可见,随着培养温度的升高,样品中Bacillus的相对含量逐渐增加,而Pediococcus的相对含量却呈现相反的趋势。 究其原因可能一方面是Bacillus中存在耐热孢子,其是微生物对抗高温等不利环境的休眠体[21];另一方面,Bacillus在胁迫压力下可以形成包含聚γ-谷氨酸等多种内源分子的生物被膜,用以抵御高温胁迫[22]。
由图1c可知,从不同培养温度下的样品中共注释到10个优势真菌属,分别为Aspergillus、Thermoascus、复膜孢酵母属(Saccharomycopsis)、毕赤酵母属(Pichia)、红曲属(Monascus)、嗜热丝孢菌属(Thermomyces)、罗山松属(Rasamsonia)、米 勒 酵 母 属(Millerozyma)、丝 衣 霉 属(Byssochlamys)和酵母菌属(Saccharomyces)。 其中,40 ℃培养温度下样品的真菌菌群以Pichia、Saccharomycopsis、Aspergillus和Millerozyma为主,相对含量分别为40.80%、12.44%、11.17%和10.82%;45 ℃培养温度下样品的真菌菌群以Thermoascus为主,相对含量为70.32%;50 ℃培养温度下样品的真菌菌群以Aspergillus和Monascus为主,相对含量分别为66.33%和12.38%;55 ℃培养温度下样品的真菌菌群以Saccharomycopsis为主,相对含量为41.92%。 值得一提的是,10个优势真菌属的相对含量间不存在显著相关性(P>0.05)。
为了判断不同培养温度下高温大曲源微生物生长的共性和差异,将在所有样品中均存在的OTU定义为核心OTU,将仅在某一培养温度样品中存在的OTU定义为特有OTU,并基于OTU水平对不同培养温度下样品微生物类群的结构进行解析,结果见图2。
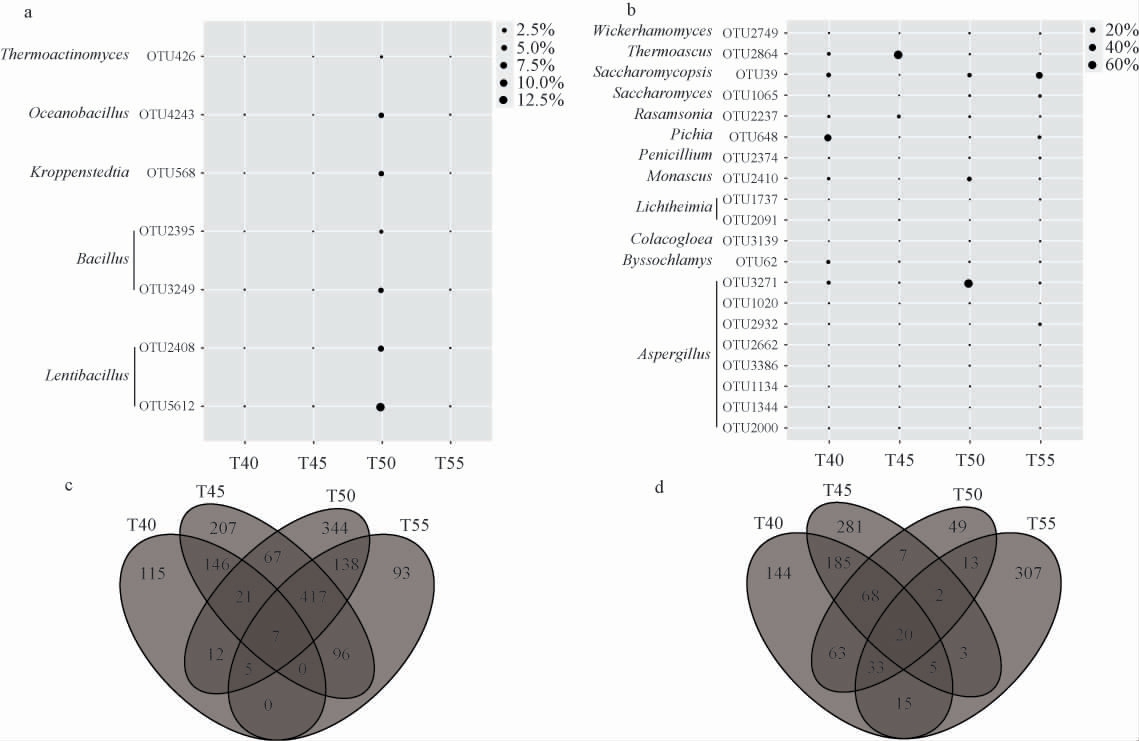
图2 不同培养温度下样品中核心细菌(a)和真菌(b)操作分类单元的气泡图及细菌(c)和真菌操作分类单元(d)的韦恩图
Fig.2 Bubble diagrams of operational taxonomic unit of core bacteria (a) and fungi (b) and Venn diagrams of operational taxonomic unit of bacteria (c) and fungi (d) in samples at different culture temperatures
由图2a可知,不同培养温度下样品的核心细菌OTU有7个,共隶属于5个细菌属,虽其包含的序列数占总序列数的32.01%,但其中18.5%的序列隶属于50 ℃培养的样品,且均被鉴定为Lentibacillus。 由图2b可知,不同培养温度下样品的核心真菌OTU有20个,共隶属于12个真菌属。 40 ℃培养时样品中真菌核心OTU所包含的序列数占总序列数的79.05%,其中40.72%的序列被鉴定为Pichia;45 ℃培养时样品中真菌核心OTU所包含的序列数占总序列数的68.97%,其中62.27%的序列被鉴定为Thermoascus;50 ℃培养时样品中真菌核心OTU所包含的序列数占总序列数的90.00%,其中62.33%的序列被鉴定为Aspergillus;55 ℃培养时样品中真菌核心OTU所包含的序列数占总序列数的53.43%,其中33.83%的序列被鉴定为Saccharomycopsis。 由此可见,在使用某一特定温度对高温大曲源微生物进行培养时,会形成与该温度相对应的优势菌群,这与属水平分析结果一致。
由图2c可知,采用40 ℃、45 ℃、50 ℃和55 ℃对高温大曲进行摇瓶培养时,虽其细菌类群特有OTU分别为115个、207个、344个和93个,包含的序列数依次为845条、885条、7 246条和1 134条,但其在总序列数中的占比仅分别为0.09%、0.96%、0.79%和0.12%。 由图2d可知,采用40 ℃、45 ℃、50 ℃和55 ℃对高温大曲进行摇瓶培养时,虽其真菌类群特有OTU分别为144个、281个、49个和307个,包含的序列数依次为11 537条、10 195条、6 092条和65 898条,但其在总序列数中的占比仅为0.96%、0.85%、0.51%和5.50%。 由此可见,在使用某一特定温度对高温大曲源微生物进行培养时,虽会形成与该温度相对应的优势菌群,但其并未形成与该温度相对应的特有菌群。究其原因可能在于微生物处于不良生长环境时会产生抗逆改良[23],其生长只是受到了抑制但并不是完全停止了生长。
2.3 不同培养温度下样品微生物的共现网络分析
共现网络图的构建是判断微生物群落间共生或竞争等相互作用的重要依据[24]。因此,本研究进一步基于共现网络分析评价了4个培养温度下样品微生物间的相互作用关系,通过Spearman相关性分析挑取了P<0.05且|r|>0.8的细菌属和真菌属绘制了相关性网络图,结果见图3。
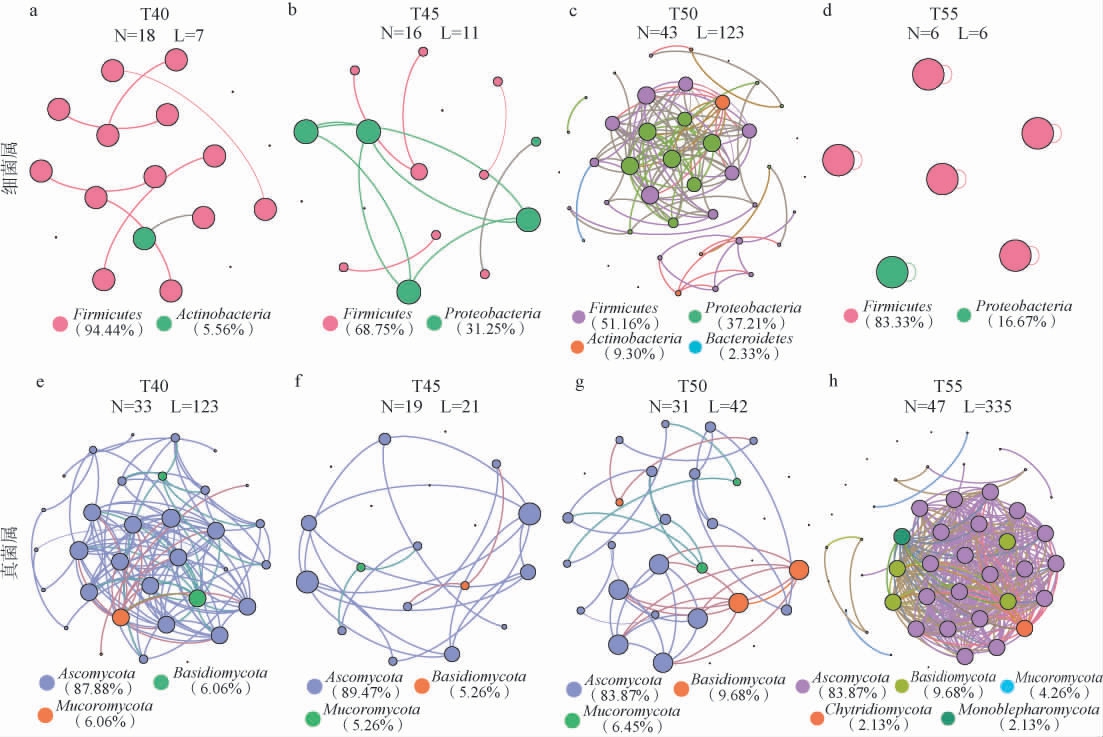
图3 不同培养温度下样品中细菌属(a~d)及真菌属(e~h)间的共现网络分析结果
Fig.3 Co-occurrence network analysis results of bacteria (a-d) and fungi (e-h) genera in samples at different culture temperatures
N代表节点数;L代表边数。
由图3可知,随着培养温度的变化,微生物共生网络的结构发生了一定改变。当培养温度为50 ℃时,样品所蕴含细菌属的种类最为丰富,其在43个细菌属间共存在123个显著相关性(P<0.05)。共生网络的拓扑参数表明,在此培养温度下共生网络图的图密度最高,为0.136。结果表明,在50 ℃的培养温度下构成网络的细菌菌群之间关系更密切,并且网络的稳定性相对更高[25]。
由图3亦可知,在培养温度为55 ℃时,样品所蕴含真菌属的种类最为丰富,其中47个真菌属间共存在335个显著相关性(P<0.05)。共生网络的拓扑参数表明,在此培养温度下共生网络图的图密度最高,为0.310。 结果表明,在55 ℃的培养温度下构成网络的真菌菌群之间联系更密切,网络的稳定性更高。由此可见,分别在50 ℃和55 ℃的培养温度下,样品细菌和真菌菌群组成更丰富,且菌属间相互作用关系更强,此结论与Alpha多样分析结果具有一致性。
2.4 不同培养温度下样品中细菌菌群基因功能的比较分析
进一步基于PICRUSt 2软件预测不同培养温度下样品中细菌菌群的基因功能,结果见表2。
表2 不同培养温度下样品中细菌菌群基因功能预测结果的比较分析
Table 2 Comparative analysis of predicted gene function results for bacterial communities in samples at different culture temperatures

注:“-”表示未检测出,同行不同字母表示差异显著(P<0.05)。
功能分类 功能注释 T40 T45 T50 T55 ABCDEFGHIJKLMNOPQTUVZ核糖核酸(ribonucleic acid,RNA)处理和修饰染色质结构和动力学能量生产和转换细胞周期控制、细胞分裂和染色体分割氨基酸运输和代谢核苷酸运输和代谢碳水化合物运输和代谢辅酶运输和代谢脂质转运与代谢翻译、核糖体结构和生物发生转录复制、重组和修复细胞壁/膜/包膜生物发生细胞运动翻译后修饰、蛋白质转换和伴随蛋白去氧离子运输和代谢次生代谢物的生物合成、运输和分解代谢信号转导机制细胞内运输、分泌和囊泡运输防御机制细胞支架0.00±0.00b 0.00±0.00b 3.72±0.01c 1.06±0.00d 5.67±0.01d 4.35±0.01a 10.38±0.01a 2.47±0.00d 2.18±0.00d 9.82±0.02a 7.53±0.01a 6.56±0.00a 5.76±0.01a 0.00±0.00d 2.74±0.01c 3.97±0.01d 0.56±0.00d 2.46±0.00c 0.98±0.00a 1.92±0.01b 0.00±0.00a 0.00±0.00b-4.20±0.05b 1.18±0.01c 6.95±0.09c 3.55±0.05b 8.29±0.02b 3.02±0.04c 2.49±0.02c 7.86±0.13b 6.85±0.05b 6.18±0.02b 5.16±0.04b 0.40±0.03c 2.97±0.01b 4.51±0.04c 0.73±0.01c 2.95±0.03b 0.89±0.01b 1.81±0.01bc 0.00±0.00b 0.01±0.00a 0.00±0.00a 4.96±0.03a 1.32±0.01a 7.75±0.12b 2.73±0.03c 6.49±0.04c 3.60±0.04a 2.91±0.05a 6.02±0.03c 6.39±0.05c 5.40±0.12d 4.81±0.06c 0.67±0.03b 3.16±0.01a 4.90±0.04b 1.09±0.07a 3.09±0.09b 0.90±0.03b 2.09±0.12a-0.00±0.00b-4.89±0.00a 1.28±0.00b 8.16±0.00a 2.81±0.00c 6.38±0.00c 3.51±0.00b 2.77±0.00b 6.02±0.00c 6.20±0.00d 5.80±0.00c 4.60±0.00d 0.77±0.00a 3.17±0.00a 5.03±0.00a 0.88±0.00b 3.39±0.00a 0.80±0.00c 1.72±0.00c-
由表2可知,在50 ℃的培养温度下样品中细菌菌群在“细胞周期控制、细胞分裂和染色体分割”、“次生代谢物的生物合成、运输和分解代谢”、“辅酶运输和代谢”、“脂质转运与代谢”和“防御机制”等功能的表达上显著高于其他三组(P<0.05),而在“核苷酸运输和代谢”、“碳水化合物运输和代谢”和“复制、重组和修复”等功能的表达上显著偏低(P<0.05)。由此可见,在50 ℃的培养温度下高温大曲源细菌菌群具有较高的生长速率、较强的代谢活性和防御机制,这可能是导致在此温度下样品中细菌菌群的丰富度和多样性均较高的主要原因。 有研究表明,芽孢杆菌中存在的热休克蛋白是其抵抗不利环境的防御机制,该蛋白在细胞内具有分子伴侣的功能,当微生物处于热激状态时胞内蛋白发挥保护作用,可促进产生错误折叠蛋白质的重新折叠,从而使菌株具有一定的耐热性[26]。
3 结论
本研究基于摇瓶实验研究温度对高温大曲微生物类群的影响,结果发现,45 ℃和50 ℃培养时样品细菌菌群的丰富度和多样性均最高,45 ℃培养时样品真菌菌群的丰富度和多样性均最高。当培养温度为40 ℃时,样品中细菌以Pediococcus为主,真菌以Saccharomycopsis、Aspergillus和Millerozyma为主;培养温度为45 ℃时,细菌以Pediococcus和Bacillus为主,真菌以Thermoascus为主;培养温度为50 ℃时,细菌以Bacillus、Lentibacillus和Kroppenstedtia为主,真菌以Aspergillus和Monascus为主;培养温度为55 ℃时,细菌和真菌分别以Bacillus和Saccharomycopsis为主。在使用某一特定温度对高温大曲源微生物进行培养时,虽会形成与该温度相对应的优势菌群,但其并未形成与该温度相对应的特有菌群。 在50 ℃和55 ℃的培养温度下,样品中细菌和真菌的菌群组成更丰富且相互作用关系更强。此外,在50 ℃的培养温度下高温大曲细菌菌群具有较高的防御功能(P<0.05)。 本研究结果可为后续高温大曲中微生物菌株的分离提供理论指导。
[1]WANG Y R,CAI W C,WANG W P,et al.Analysis of microbial diversity and functional differences in different types of high-temperature Daqu[J].Food Sci Nutr,2021,9(2):1003-1016.
[2]HOU Q C,WANG Y R,QU D,et al.Microbial communities,functional,and flavor differences among three different-colored high-temperature Daqu: A comprehensive metagenomic, physicochemical, and electronic sensory analysis[J].Food Res Int,2024,184(5):114257.
[3]LIU Y B,LI H D,DONG S M,et al.Dynamic changes and correlations of microbial communities,physicochemical properties,and volatile metabolites during Daqu fermentation of Taorong-type Baijiu[J].LWT-Food Sci Technol,2023,173(1):114290.
[4]LI H D, LIU S Y, LIU Y B, et al.Functional microorganisms in Baijiu Daqu:Research progress and fortification strategy for application[J].Front Microbiol,2023,14(1):1119675.
[5]李斌斌,吴丹妮,聂国兴,等.未/难培养微生物可培养策略研究:机遇与挑战[J].微生物学通报,2023,50(2):832-844.
[6]CAI W C,TANG F X, WANG Y R, et al.Bacterial diversity and flavor profile of Zha-Chili,a traditional fermented food in China[J].Food Res Int,2021,141(3):110112.
[7]WANG J X,HAO S Y,REN Q.Analysis of bacterial diversity in fermented grains of Baijiu based on culturomics and amplicon sequencing[J].Fermentation,2023,9(3):260.
[8]ZHU M,ZHENG J,XIE J,et al.Effects of environmental factors on the microbial community changes during medium-high temperature Daqu manufacturing[J].Food Res Int,2022,153(3):110955.
[9]崔梦君,王玉荣,侯强川,等.基于三代测序技术高温和中高温大曲真菌多样性解析及酵母菌的分离鉴定[J].食品工业科技,2025,46(2):175-183.
[10]DENG L,MAO X,LIU D,et al.Comparative analysis of physicochemical properties and microbial composition in high-temperature Daqu with different colors[J].Front Microbiol,2020,11(11):588117.
[11]NIE E,HE P,ZHANG H,et al.How does temperature regulate anaerobic digestion?[J].Renew Sust Energ Rev,2021,150(10):111453.
[12]JEZKOVA Z,BINDA E,POTOCAR T,et al.Laboratory scale cultivation of Salinispora tropica in shake flasks and mechanically stirred bioreactors[J].Biotechnol Lett,2021,43(9):1715-1722.
[13]吴建峰,孙莹,季方,等.浓香型721型曲培养周期内风味成分变化的分析研究[J].酿酒科技,2016(12):43-46.
[14]ZHANG Z D,WANG Y R,XIANG F S,et al.Distinct bacterial community of a solid-state fermented Chinese traditional food huase sufu revealed by high-throughput sequencing[J].Food Sci Biotechnol, 2021,30(9):1233-1241.
[15]PRODAN A, TREMAROLI V, BROLIN H, et al.Comparing bioinformatic pipelines for microbial 16S rRNA amplicon sequencing[J].Plos One,2020,15(1):e0227434.
[16]HAAS B J,GEVERS D,EARL A M,et al.Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J].Genome Res,2011,21(3):494-504.
[17]DESANTIS T Z, HUGENHOLTZ P, LARSEN N, et al.Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J].Appl Environ Microbiol,2006,72(7):5069-5072.
[18]ABARENKOV K, NILSSON R H, LARSSON K H, et al.The UNITE database for molecular identification of fungi-recent updates and future perspectives[J].New Phytol,2010,186(2):281-285.
[19]GALPERIN M Y,KRISTENSEN D M,MAKAROVA K S,et al.Microbial genome analysis:The COG approach[J].Brief Bioinform,2019,20(4):1063-1070.
[20]WILKINSON T J,HUWS S A,EDWARDS J E,et al.CowPI:A rumen microbiome focussed version of the PICRUSt functional inference software[J].Front Microbiol,2018,9(5):1095.
[21]吴盛露,杜海,徐岩.生物热对传统固态发酵菌群演替及其代谢影响的研究进展[J].微生物学通报,2022,49(6):2281-2294.
[22]韩钟娆,霍毅欣,郭淑元.芽胞杆菌耐受胁迫条件的机制及工业应用[J].生物技术通报,2024,40(8):24-38.
[23]赵杨,赵心清.生物对逆境环境的适应和抗逆分子育种[J].生物技术通报,2023,39(11):1-5.
[24]MENG L X,XU C X,WU F L.Microbial co-occurrence networks driven by low-abundance microbial taxa during composting dominate lignocellulose degradation[J].Sci Total Environ,2022,845(11):157197.
[25]万凌琳,陈芷凡,郭佳,等.生物共现网络原理及其在淡水生态系统评估中的应用[J].湖泊科学,2022,34(6):1765-1789.
[26]WU Q, XU Y.Transcriptome profiling of heat-resistant strain Bacillus licheniformis CGMCC3962 producing Maotai flavor[J].J Agr Food Chem,2012,60(8):2033-2038.