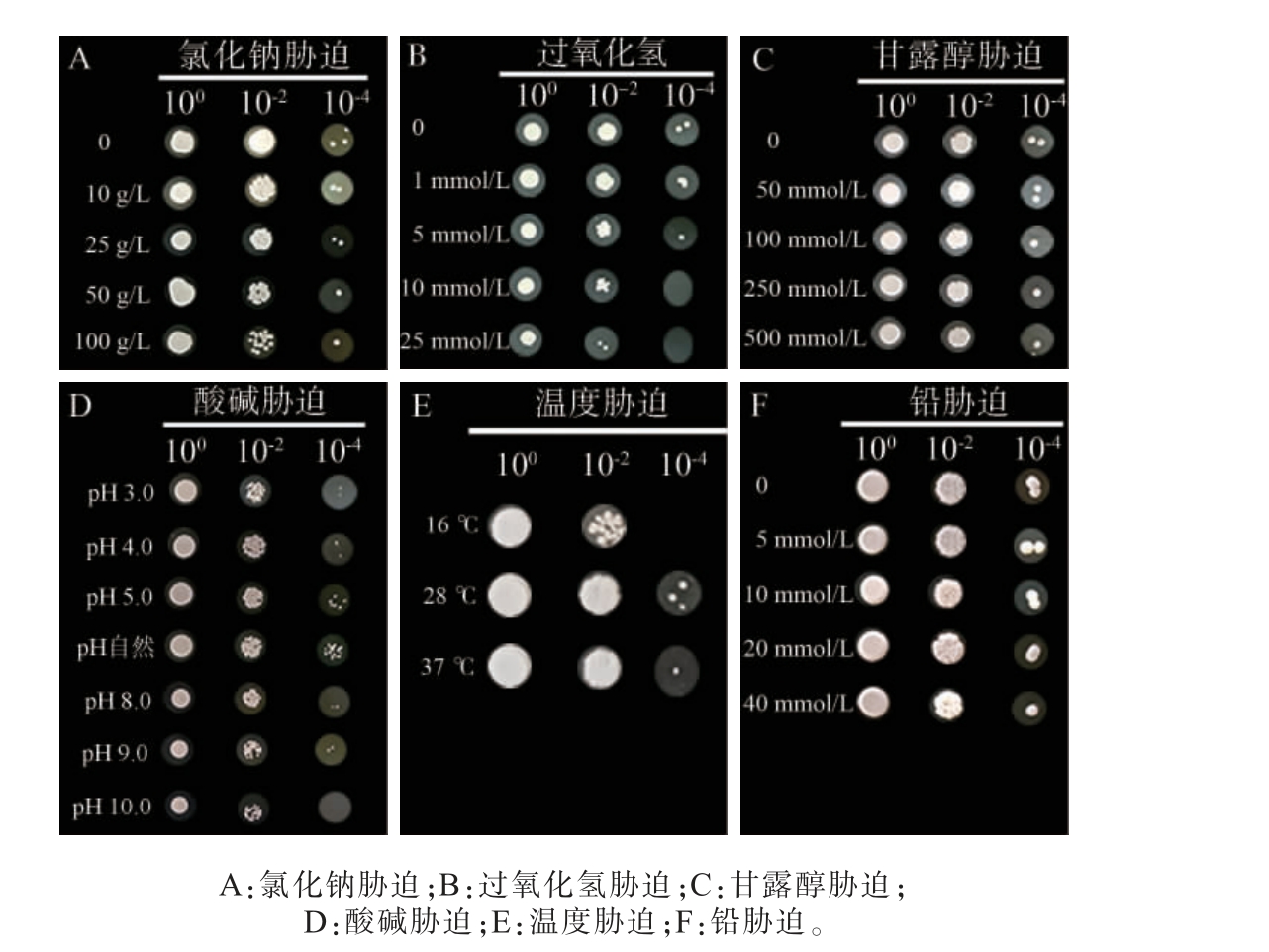
图1 异常威克汉姆酵母C11的逆境胁迫耐受性
Fig.1 Stress tolerance of Wickerhamomyces anomalus C11
Stress tolerance and whole-genome sequencing of an ester-producing Wickerhamomyces anomalus strain
异常威克汉姆酵母(Wickerhamomyces anomalus),也被称为异常毕赤酵母(Pichia anomalus)、异常汉逊酵母(Hansenula anomalus)、膜状假丝酵母(Candida pelliculosa),广泛分布于土壤、植物、水体等各类自然环境中[1]。W.anomalus具有独特的生理特性和代谢特征,如合成多种糖苷酶(α-葡萄糖苷酶、β-葡萄糖苷酶、α-呋喃葡萄糖苷酶、α-木糖酶等),水解底物中结合态风味前体物质,有助于挥发性风味物质的释放[2];合成大量的具有花香和水果香的风味化合物,丰富和改善发酵产品的风味特性[3];代谢产生具有广谱抗菌活性的抗菌物质,有助于防止发酵生产中的有害微生物的污染[4];对多种极端环境(温度胁迫、酸碱胁迫、氧化胁迫等)具有较好的耐受性[5]。此外,W.anomalus安全性高,对人畜无害[6]。因而,W.anomalus是一种具有较高应用价值的工业微生物,被广泛应用于多种发酵体系和发酵生产中[3,7]。 YAN S B等[8]研究发现,在米酒发酵过程中,W.anomalus通过促进酯类、醇类以及氨基酸等风味化合物的生成,增强米酒的香气特征,提高酒体的感官评分;VIANA F等[9]利用W.anomalus发酵葡萄酒时,可增加乙酸乙酯、乙酸异戊酯、2-乙酸苯乙酯等具有花香和水果香酯类物质的种类和含量,提高酒体感官特性值。
随着测序技术的快速发展和测序成本不断地降低,越来越多的微生物基因组被测定,其编码的基因信息也被注释和解析[10]。 李佳等[11]筛选出了一株高效降解棉酚的菌株M-15,采用全基因组测序技术解析了其棉酚降解途径,为微生物发酵棉粕脱毒技术的开发奠定了理论基础;郭宝松等[12]对一株分离自郫县豆瓣的高产氨肽酶枯草芽孢杆菌(Bacillus subtilis)LBJ4-5进行了全基因组测序和信息挖掘,证实该菌株是一株优良的食品发酵生产菌剂;刘芳等[13]利用二代和三代测序技术对一株高产褐藻胶裂解酶的菌株S10的全基因组序列进行了测序分析,从基因水平上挖掘了菌株潜在的产酶机制。目前,已完成基因组序列测定并报道的W.anomalus菌株还相对较少[14-15]。SCHNEIDER J等[14]研究发现,W. anomalus DSM 6766的基因组大小为25.47 Mbp,编码11 512个蛋白;CUNHA A C等[15]研究发现,W.anomalus LBCM1105的基因组大小仅为13.93 Mbp。综上表明,目前对W.anomalus全基因组序列信息的认识还不够充分,仍需更多的研究与分析。
本课题组前期从贵州刺梨果实发酵液中分离出一株产香W.anomalus C11(GenBank序列号:MT705624;中国普通微生物菌种保藏管理中心保藏号:20903)[16-17]。该菌株能够增加刺梨果酒中乙酸乙酯、乙酸苯乙酯、乙酸异戊酯的含量[17],龙眼果酒中乙酸乙酯、棕榈酸乙酯、丁二酸二乙酯含量[18],空心李果酒中壬酸乙酯、棕榈酸乙酯、苯甲酸乙酯含量[19]。同时该菌株还对葡萄糖、乙醇、柠檬酸及二氧化硫具有较好的耐受性[17],但对其他逆境胁迫,如盐胁迫、渗透胁迫、酸碱胁迫等的耐受性还未进行研究。此外,该菌株产酯和逆境胁迫耐受潜在机制还不清楚。因此,本研究采用平板生长法对一株产酯W.anomalus C11的逆境胁迫耐受性进行研究。同时采用全基因组测序技术,从菌株基因水平上挖掘其产香和胁迫耐受机制,为拓宽W.anomalus C11发酵应用领域和菌株的遗传改造奠定理论基础。
1.1.1 菌株
异常威克汉姆酵母(W.anomalus)C11:分离于贵州刺梨果实发酵液,保存于贵州理工学院酵母菌生理生化实验室,具有产酯性能[17]。
1.1.2 试剂
酵母浸粉、蛋白胨、琼脂粉(均为生化试剂)、葡萄糖、三丁酸甘油酯(分析纯)、溴甲酚紫(均为分析纯):贵州博奥瑞杰生物科技有限公司;氯化钠、甘露醇、醋酸铅、盐酸、氢氧化钠(均为分析纯):贵州博奥瑞杰生物科技有限公司。其他试剂均为国产分析纯或生化试剂。
1.1.3 培养基
酵母浸出粉胨葡萄糖(yeast extract peptone dextrose,YPD)培养基[20]:葡萄糖20 g/L,蛋白胨20 g/L,酵母浸粉10 g/L。YPD固体培养基:YPD培养基中添加琼脂粉20 g/L。121 ℃高温蒸汽灭菌15 min。
DHP-420恒温培养箱:天津天泰仪器有限公司;ZD-85A恒温摇床:常州朗越仪器制造有限公司;GR60DA高压蒸汽灭菌锅:北京盛科信德科技有限公司;PHSJ-3F雷磁pH计:上海仪电科学仪器股份有限公司。
1.3.1 异常威克汉姆酵母C11的活化
将超低温冰箱保存的W.anomalus C11划线于YPD固体培养基上,28 ℃条件下活化培养48 h,保存于4 ℃冰箱,备用。
1.3.2 异常威克汉姆酵母C11的耐受性分析
挑取活化的单菌落接种于YPD培养基中,28℃、160r/min条件下培养24 h,按5%(V/V)的接种量分别接种于含不同浓度氯化钠(0、10 g/L、25 g/L、50 g/L、60 g/L和80 g/L)、甘露醇(0、50 mmol/L、100 mmol/L、250 mmol/L、500 mmol/L)、过氧化氢(0、1 mmol/L、5 mmol/L、10 mmol/L、25 mmol/L)铅(0、5 mmol/L、10 mmol/L、20 mmol/L、40 mmol/L)的YPD培养基中,均于28 ℃、160 r/min条件下培养6 h;按5%(V/V)的接种量分别接种于不同pH(3、4、5、8、9、10)的YPD培养基中,于28 ℃、160 r/min条件下培养6 h;按5%(V/V)的接种量接种于YPD培养基中,分别于不同温度(16 ℃、28 ℃、37 ℃)、160 r/min条件下培养6 h。 培养结束后,将各组菌液按10倍梯度分别稀释至10-4,吸取2 μL各组的稀释液涂布于YPD固体培养基上,待菌液干燥后,28 ℃倒置培养48 h,观察菌生长状态。
1.3.3 异常威克汉姆酵母C11的全基因组测序与组装
挑取单菌落接种至YPD培养基中,于28 ℃、160 r/min条件下培养24 h,发酵液经4 ℃、5 000 r/min离心10 min,收集菌体,委托上海美吉生物医药科技有限公司提取菌株基因组脱氧核糖核酸(deoxyribonucleic acid,DNA),并基于illumina novaseq 6000测序平台进行全基因组测序,参考JEON S A等[21]的方法对测序结果进行处理。通过Fastp v0.20.0对测序数据进行质控,去除测序质量较低及质量修剪后长度较小的读数。 采用SOAPdenovo v2.04与GapCloser v1.12对质控后的基因组进行从头组装,采用BUSCO、CEGMA软件对组装后的基因组进行评估。
1.3.4 基因预测及功能注释
采用Maker2对组装的基因组进行编码基因的预测。通过Diamond 3.2将整合基因组所预测的编码基因的蛋白质序列分别与直系同源蛋白簇(clusters of orthologous groups of proteins,COG)数据库、基因本体论(gene ontology,GO)数据库、京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)数据库以及碳水化合物活性酶(carbohydrate-active enzymes,CAZy)数 据 库 中 的 蛋 白质序列采用基本局部比对搜索工具(basic local alignment search tool,BLAST)进行比对分析,从而获取基因组功能注释信息。
1.3.5 致病性分析
采用Diamond 3.2软件以病原体与宿主的相互作用(pathogen host interactions,PHI)数据库为基础,对组装的基因组进行病原菌宿主互作分析,预测菌株的致病性。
1.3.6 数据分析
每个实验均平行重复3次。采用Adobe Photoshop 2020进行数据的辅助作图。全基因组测序分析在上海美吉生物医药科技有限公司云平台上(https://www.majorbio.com/)完成。
已有研究表明,W.anomalus对极端环境胁迫具有较强的耐受性,但菌株间可能具有一定的差异性[5]。 因此,本研究采用平板生长法对W.anomalus C11逆境胁迫耐受性进行分析,结果见图1。 由图1A可知,随着氯化钠质量浓度的逐渐增加,细胞活性也逐渐降低,当氯化钠质量浓度为100 g/L时,细胞存活明显受到抑制,但细胞活性仍较高;W.anomalus C11的氯化钠胁迫耐受性明显优于ZHANG J等[22]从四川泡菜汇中分离出的W.anomalus Y3,该菌株最高仅可耐受14%的盐胁迫。 由图1B可知,1 mmol/L过氧化氢胁迫处理下,细胞活性与未处理组之间没有明显差异;当过氧化氢浓度达到10 mmol/L时,细胞活性明显降低,发酵液稀释度为10-4时已无细胞存活;当过氧化氢浓度为25 mmol/L时,发酵液稀释度为10-2时也仅极个别细胞存活。由图1C可知,W.anomalus C11在50~500 mmol/L甘露醇处理下,细胞仍具有较高的活性,细胞存活与未处理组之间没有明显差别,说明其具有较强的渗透胁迫耐受性。 由图1D可知,W.anomalus C11在酸胁迫下(pH 3.0~5.0)和碱胁迫下(pH 8.0~10.0),菌株仍具有较高的活性,其酸碱胁迫耐受性与菌株P.anomala J121较为接近(pH 2.0~12.0)[23]。由图1E可知,W.anomalus C11在低温(16 ℃)胁迫下,细胞活性较正常培养条件下(28 ℃)有一定程度的降低;但在高温(37 ℃)胁迫下,细胞活性与正常组之间没有明显的区别。 研究发现,菌株P.anomala J121在37 ℃胁迫下仍可以生长,展现出了较强的高温耐受性[23],与本研究菌株W.anomalus C11的高温耐受特性较为一致。由图1F可知,W. anomalus C11细胞对铅胁迫具有较好耐受性,在5~40 mmol/L铅胁迫下,细胞仍具有较高的活性。 综上,W.anomalus C11具有较好的环境耐受性,可耐受100 g/L氯化钠、25 mmol/L过氧化氢、500 mmol/L甘露醇、pH 3.0~10.0、16 ℃低温、37 ℃高温以及40 mmol/L铅的胁迫处理。

图1 异常威克汉姆酵母C11的逆境胁迫耐受性
Fig.1 Stress tolerance of Wickerhamomyces anomalus C11
研究发现,W.anomalus具有较好的产水解酶和产香能力,被较多地应用于了各类酒类的发酵[3,8-9]。课题组前期对W.anomalus C11酿造环境耐受性进行了评价,发现其对葡萄糖、二氧化硫、柠檬酸和乙醇均具有较好的耐受性[17]。本研究发现菌株W.anomalus C11对除了氧化胁迫外的其他胁迫均具有较强的耐受性,这有利于进一步扩大产香W.anomalus的应用领域,如含盐量较高的酱油的发酵生产,酸度较低的醋的发酵生产。李丽杰等[24]研究发现,从包头矿区重金属污染的土壤中分离得到的4株W.anomalus具有较强的Pb2+耐受性和吸附能力,可耐受质量浓度为6 000~7 000 mg/L的Pb2+处理。本研究菌株W.anomalus C11在40 mmol/L(约8 000 mg/L)Pb2+胁迫下,仍具有较高的活性,这为W.anomalus应用于重金属的吸附和重金属污染土壤的治理提供了新思路。
随着测序技术的不断发展,近年来,季也蒙毕赤酵母(Meyerozyma guilliermondii)[25]、布鲁塞尔德克酵母(Dekkera bruxellensis)[26]、土壤哈萨克斯坦酵母(Kazachstania telluris)[27]、粉状米勒酵母(Millerozyma farinosa)[28]等多种酵母菌基因组序列已被测定,为进一步研究这些酵母菌的功能提供了便利。 本研究对贵州本土W.anomalus C11基因组序列进行了测定,结果发现,W.anomalus C11基因组全长为11.48 Mbp,编码基因总长度为8.54 Mbp(占基因组总长度74.39%),鸟嘌呤(guanine,G)胞嘧啶(cytosine,C)含量为34.42%,N含量为0.253%。W.anomalus C11基因组中预测到的编码基因数为4 249个,基因密度为0.37个/kb,编码基因平均碱基长度为2 009.91 bp,基因碱基长度>2 000 bp的有1507个,占比达35.47%;基因碱基长度在1000~2000bp的有1 347个,占比达31.7%;基因碱基长度<1 000 bp的有1 395个,占比达32.83%。W. anomalus C11基因组的大小(11.48 Mbp)与SCHNEIDER J等[14]报道的W.anomalus DSM 6766基因组大小差距较大(25.47 Mbp),但与CUNHA A C等[15]从甘蔗发酵中分离出的W.anomalus LBCM1105基因组大小(13.93 Mbp)较为接近。W.anomalus菌株间基因组大小的差异性可能与基因测序技术和测序深度有关,其具体原因还有待进一步分析。
2.3.1 COG功能注释
采用COG数据库对W.anomalus C11的功能基因进行注释和归类,结果见图2。 由图2可知,共有1 614个基因被注释到COG数据库中的22个功能大类。 其中,被注释到参与翻译、核糖体结构与生物发生的基因数最多,为216个(占比13.38%);其次是参与碳水化合物的运输与代谢,基因数为196个(占比12.14%);被注释到参与氨基酸的转运与代谢的基因数为192个(占比11.90%);注释到参与翻译后修饰、蛋白质迁移的基因数为133个(占比8.24%);注释到参与辅酶的转运与代谢的基因数为125个(占比7.74%);注释到参与染色质结构和动态变化的基因数最少,仅2个。
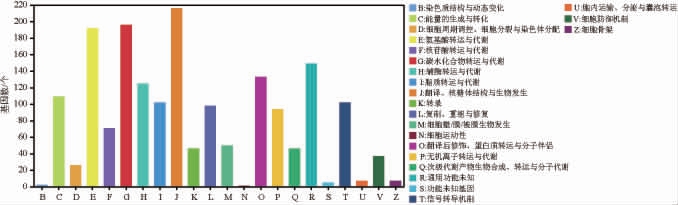
图2 异常威克汉姆酵母C11基因组直系同源蛋白簇功能注释结果
Fig.2 Functional annotation results of homologous protein clusters in the genome of Wickerhamomyces anomalus C11
2.3.2 GO功能注释
GO功能注释主要包括3个方面,分别为生物过程、细胞组成和分子功能[29]。 采用GO数据库对W.anomalus C11的功能基因进行功能注释和归类,结果见图3。
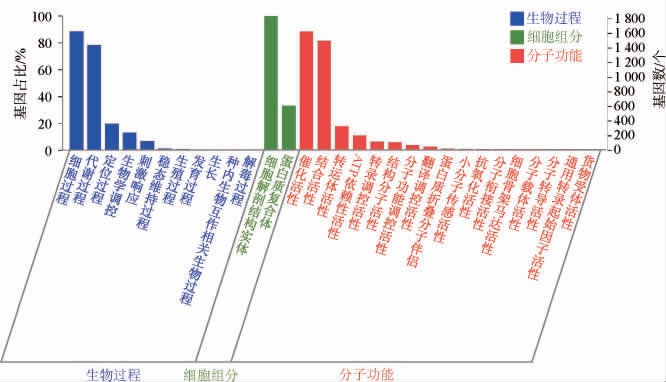
图3 异常威克汉姆酵母C11基因组基因本体论功能注释结果
Fig.3 Functional annotation results of gene ontology in the genome of Wickerhamomyces anomalus C11
由图3可知,W.anomalus C11共有3 346个基因被注释到GO数据库中,其中,与生物过程相关的基因数量为2 095个(占比62.61%);与细胞组成相关的基因数量为2 162个(占比64.61%),与分子功能相关的基因数量为2 594个(占比77.53%)。 生物过程中,被注释最多的类别是细胞过程,有1 626个基因,其次为代谢过程,有1 442个基因;细胞组成中,被注释最多的类别是细胞解剖实体,有1 838个基因;分子功能中,被注释到最多的类别为催化活性和结合过程,基因数分别为1 625个和1 500个。
2.3.3 KEGG功能注释
采用KEGG数据库对W.anomalus C11的功能基因进行功能注释和归类,结果见图4。
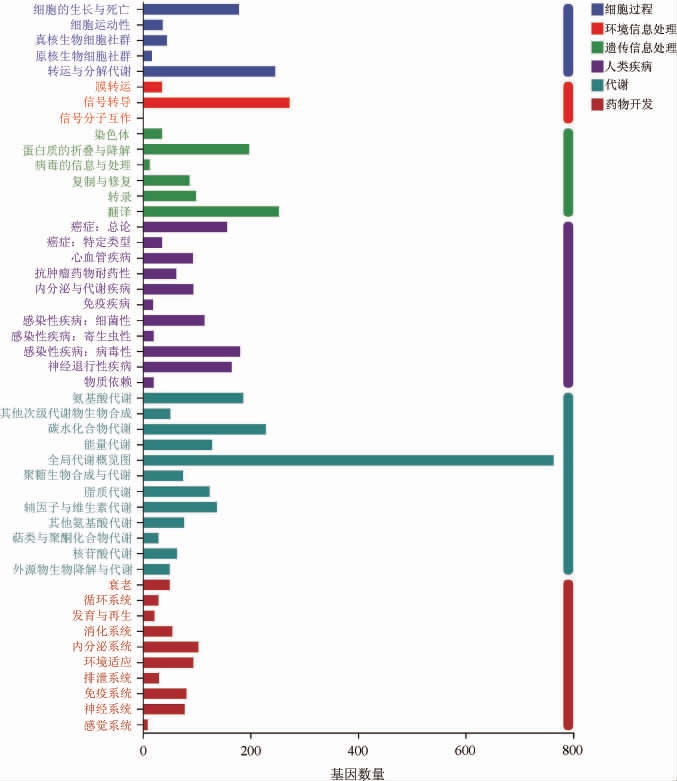
图4 异常威克汉姆酵母C11基因组京都基因与基因组百科全书功能注释结果
Fig.4 Functional annotation results of Kyoto Encycolpedia of Genes and Genomes in the genome of Wickerhamomyces anomalus C11
由图4可知,W.anomalus C11基因组中有3 145个基因被注释到KEGG数据库中,并归属于6大类,分别为细胞过程524个、环境信息处理309个、遗传信息处理686个、人类疾病962个、代谢1 917个、生物体系统551个。 参与代谢的基因中,全局代谢概览图(global and overview maps)的基因数最多,为764个,其次是碳水化合物代谢,有229个。被注释到氨基酸代谢、能量代谢、脂质代谢和核苷酸代谢的基因数分别为187个、129个、124个和64个。
采用CAZy数据库对W.anomalusC11碳水化合物活性酶进行功能注释分析,结果见图5。 由图5可知,从W.anomalus C11基因组中共注释到87个碳水化合物活性酶相关基因,其中糖苷水解酶相关基因32个,糖基转移酶相关基因39个,碳水化合物酯酶相关基因8个。
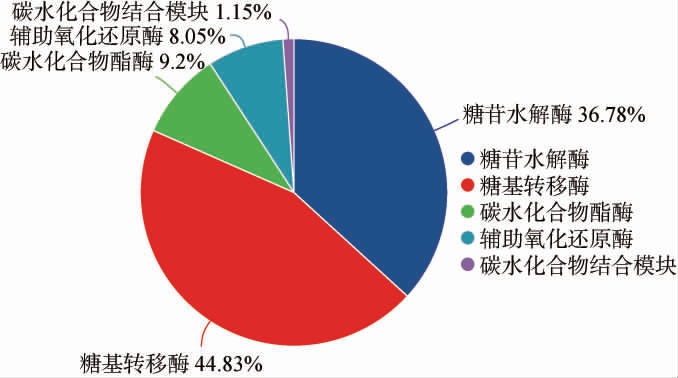
图5 异常威克汉姆酵母C11基因组碳水化合物活性酶注释分析结果
Fig.5 Functional annotation results of carbohydrate active enzyme in the genome of Wickerhamomyces anomalus C11
糖苷水解酶是一大类可水解底物中含糖苷类化合物的酶类,有助于结合态风味化合物的释放,增加食品的风味特性[30]。 通过全基因组测序,W.anomalus C11基因组中共鉴定出32个糖苷水解酶基因,分属于17个糖苷水解酶家族,包括葡聚糖酶、葡聚糖分支酶、葡萄糖苷酶等,具体见表1。前期研究发现,W.anomalus C11具有较强的产β-葡萄糖苷酶性能,其酶活为(41.8±0.22)U/L,显著高于商业化酿酒酵母(Saccharomycescerevisiae)[(25.2±0.31)U/L][17]。由表1可知,在W.anomalus C11基因组序列中注释到了β-葡萄糖苷酶,但葡聚糖酶和甘露糖苷酶在菌株W. anomalus C11中是否真实表达以及具体表达量还有待进一步的测定。 推测这些丰富的糖苷酶基因及其编码的糖苷酶是W.anomalusC11产生浓郁水果香味酯类化合物的主要原因。
表1 异常威克汉姆酵母C11基因组糖苷水解酶基因注释结果
Table 1 Results of gene annotation of glycoside hydrolase of Wickerhamomyces anomalus C11 genome
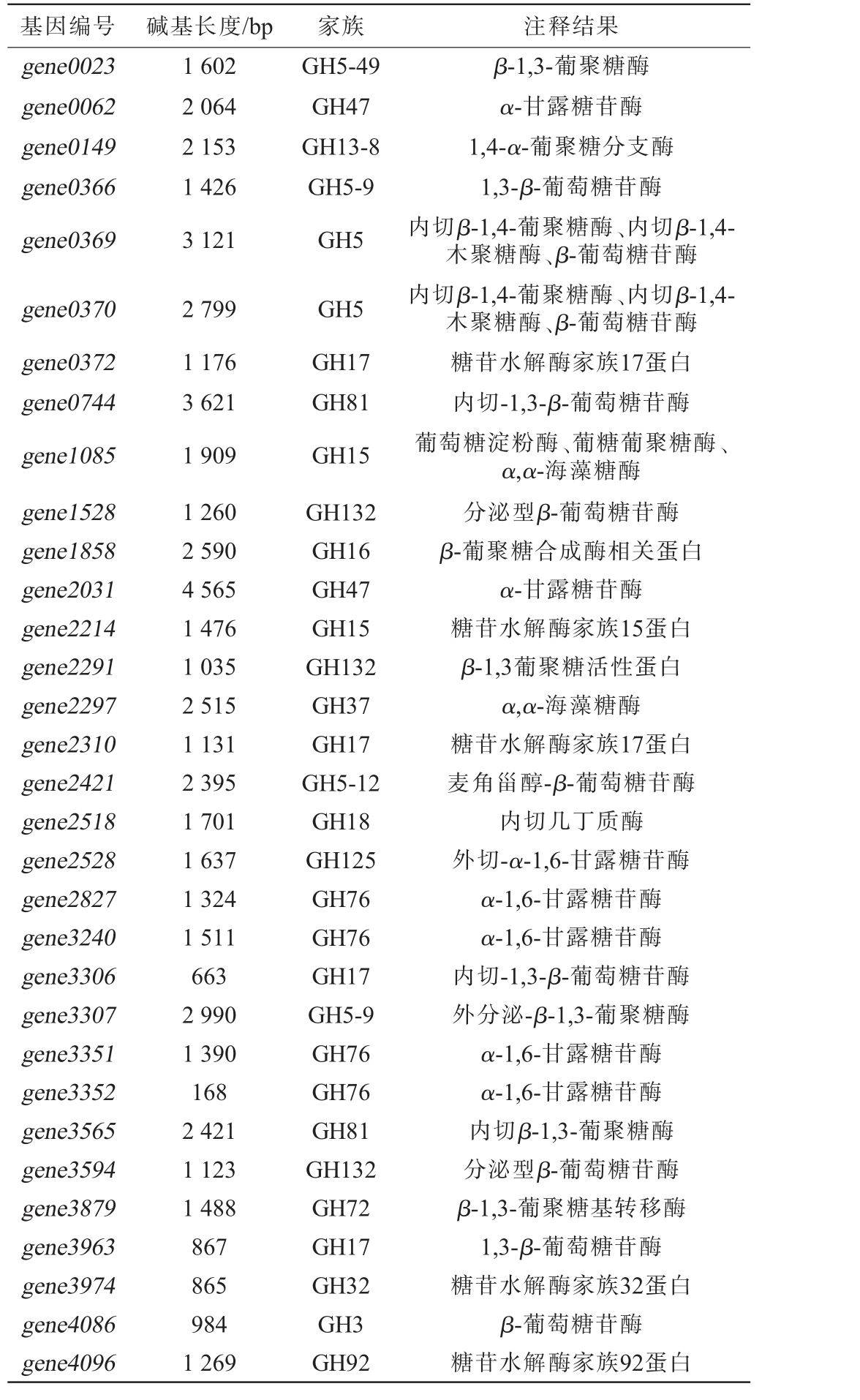
基因编号 碱基长度/bp 家族 注释结果gene0023 gene0062 gene0149 gene0366 gene0369 1 602 2 064 2 153 1 426 3 121 GH5-49 GH47 GH13-8 GH5-9 GH5 gene0370 gene0372 gene0744 gene1085 2 799 1 176 3 621 1 909 GH5 GH17 GH81 GH15 β-1,3-葡聚糖酶α-甘露糖苷酶1,4-α-葡聚糖分支酶1,3-β-葡萄糖苷酶内切β-1,4-葡聚糖酶、内切β-1,4-木聚糖酶、β-葡萄糖苷酶内切β-1,4-葡聚糖酶、内切β-1,4-木聚糖酶、β-葡萄糖苷酶糖苷水解酶家族17蛋白内切-1,3-β-葡萄糖苷酶葡萄糖淀粉酶、葡糖葡聚糖酶、α,α-海藻糖酶gene1528 gene1858 gene2031 gene2214 gene2291 gene2297 gene2310 gene2421 gene2518 gene2528 gene2827 gene3240 gene3306 gene3307 gene3351 gene3352 gene3565 gene3594 gene3879 gene3963 gene3974 gene4086 gene4096 1 260 2 590 4 565 1 476 1 035 2 515 1 131 2 395 1 701 1 637 1 324 1 511 663 2 990 1 390 168 2 421 1 123 1 488 867 865 984 1 269 GH132 GH16 GH47 GH15 GH132 GH37 GH17 GH5-12 GH18 GH125 GH76 GH76 GH17 GH5-9 GH76 GH76 GH81 GH132 GH72 GH17 GH32 GH3 GH92分泌型β-葡萄糖苷酶β-葡聚糖合成酶相关蛋白α-甘露糖苷酶糖苷水解酶家族15蛋白β-1,3葡聚糖活性蛋白α,α-海藻糖酶糖苷水解酶家族17蛋白麦角甾醇-β-葡萄糖苷酶内切几丁质酶外切-α-1,6-甘露糖苷酶α-1,6-甘露糖苷酶α-1,6-甘露糖苷酶内切-1,3-β-葡萄糖苷酶外分泌-β-1,3-葡聚糖酶α-1,6-甘露糖苷酶α-1,6-甘露糖苷酶内切β-1,3-葡聚糖酶分泌型β-葡萄糖苷酶β-1,3-葡聚糖基转移酶1,3-β-葡萄糖苷酶糖苷水解酶家族32蛋白β-葡萄糖苷酶糖苷水解酶家族92蛋白
糖基化为细胞内次级代谢反应,对于维持细胞的稳态平衡和响应逆境胁迫发挥重要作用[31]。糖基转移酶是一类可糖基化修饰小分子化合物的酶类,参与多种细胞生理活动[32]。 对W.anomalus C11基因组进行注释,共注释到39个糖基转移酶基因,大部分基因表达蛋白为甘露聚糖聚合酶、甘露聚糖转移酶和葡聚糖合酶,具体见表2。
表2 异常威克汉姆酵母C11基因组糖基转移酶基因注释结果
Table 2 Results of gene annotation of glycosyl transferases of Wickerhamomyces anomalus C11 genome
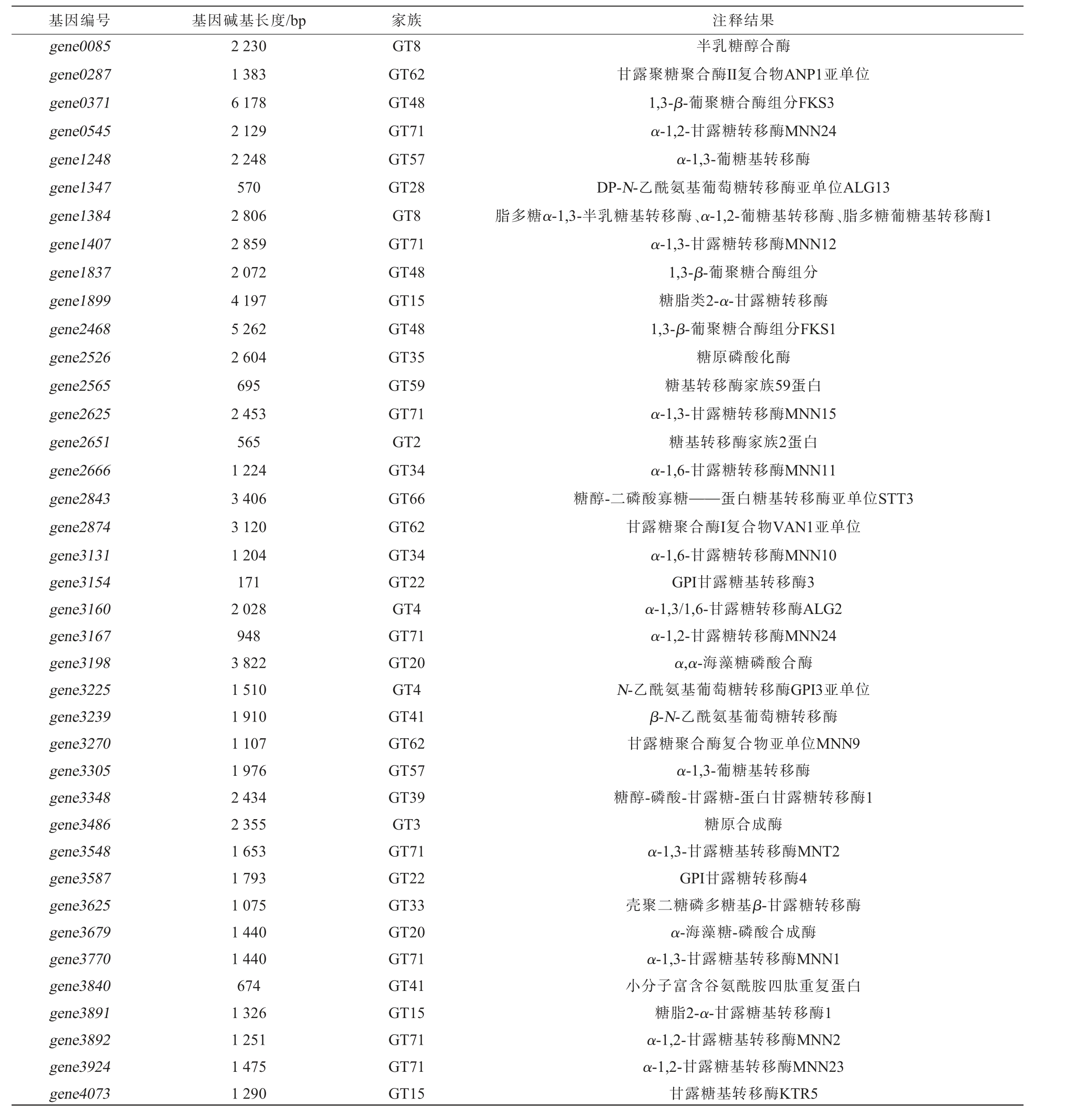
基因编号 基因碱基长度/bp 家族 注释结果gene0085 gene0287 gene0371 gene0545 gene1248 gene1347 gene1384 gene1407 gene1837 gene1899 gene2468 gene2526 gene2565 gene2625 gene2651 gene2666 gene2843 gene2874 gene3131 gene3154 gene3160 gene3167 gene3198 gene3225 gene3239 gene3270 gene3305 gene3348 gene3486 gene3548 gene3587 gene3625 gene3679 gene3770 gene3840 gene3891 gene3892 gene3924 gene4073 2 230 1 383 6 178 2 129 2 248 570 2 806 2 859 2 072 4 197 5 262 2 604 695 2 453 565 1 224 3 406 3 120 1 204 171 2 028 948 3 822 1 510 1 910 1 107 1 976 2 434 2 355 1 653 1 793 1 075 1 440 1 440 674 1 326 1 251 1 475 1 290 GT8 GT62 GT48 GT71 GT57 GT28 GT8 GT71 GT48 GT15 GT48 GT35 GT59 GT71 GT2 GT34 GT66 GT62 GT34 GT22 GT4 GT71 GT20 GT4 GT41 GT62 GT57 GT39 GT3 GT71 GT22 GT33 GT20 GT71 GT41 GT15 GT71 GT71 GT15半乳糖醇合酶甘露聚糖聚合酶II复合物ANP1亚单位1,3-β-葡聚糖合酶组分FKS3 α-1,2-甘露糖转移酶MNN24 α-1,3-葡糖基转移酶DP-N-乙酰氨基葡萄糖转移酶亚单位ALG13脂多糖α-1,3-半乳糖基转移酶、α-1,2-葡糖基转移酶、脂多糖葡糖基转移酶1 α-1,3-甘露糖转移酶MNN12 1,3-β-葡聚糖合酶组分糖脂类2-α-甘露糖转移酶1,3-β-葡聚糖合酶组分FKS1糖原磷酸化酶糖基转移酶家族59蛋白α-1,3-甘露糖转移酶MNN15糖基转移酶家族2蛋白α-1,6-甘露糖转移酶MNN11糖醇-二磷酸寡糖——蛋白糖基转移酶亚单位STT3甘露糖聚合酶I复合物VAN1亚单位α-1,6-甘露糖转移酶MNN10 GPI甘露糖基转移酶3 α-1,3/1,6-甘露糖转移酶ALG2 α-1,2-甘露糖转移酶MNN24 α,α-海藻糖磷酸合酶N-乙酰氨基葡萄糖转移酶GPI3亚单位β-N-乙酰氨基葡萄糖转移酶甘露糖聚合酶复合物亚单位MNN9 α-1,3-葡糖基转移酶糖醇-磷酸-甘露糖-蛋白甘露糖转移酶1糖原合成酶α-1,3-甘露糖基转移酶MNT2 GPI甘露糖转移酶4壳聚二糖磷多糖基β-甘露糖转移酶α-海藻糖-磷酸合成酶α-1,3-甘露糖基转移酶MNN1小分子富含谷氨酰胺四肽重复蛋白糖脂2-α-甘露糖基转移酶1 α-1,2-甘露糖基转移酶MNN2 α-1,2-甘露糖基转移酶MNN23甘露糖基转移酶KTR5
甘露聚糖聚合酶、甘露聚糖转移酶参与甘露糖的合成,葡聚糖合酶是葡聚糖合成的主要酶。甘露糖和葡聚糖是酵母菌细胞壁的主要组成成分。 研究发现,细胞壁的完整性和动态调整对于维持细胞的生理活动和响应逆境胁迫发挥重要作用[33]。 粟酒裂殖酵母(Schizosaccharomyces pombe)重组补体因子H相关蛋白3(recombinant complement factor H related protein 3,Cfh3p)调控葡聚糖合酶Bgs1p的表达,参与响应环境逆境胁迫[34];过表达金针菇(Flammulina filiformis)β-1,6-葡聚糖合酶基因FfGS6可以增强菌丝体细胞的氧化胁迫和机械损伤耐受性[35]。 由表2可知,W.anomalus C11基因组中注释到大量与细胞壁合成相关基因,如葡聚糖合酶、甘露聚糖聚合酶和甘露聚糖转移酶基因。
海藻糖是一种普遍存在于植物、无脊椎动物、真菌以及原核生物中的非还原性双糖,不仅作为一种微生物储备碳源,更是一种天然保护剂,在保护细胞免受胁迫侵害方面发挥着重要功能[36]。前期研究发现,海藻糖参与调控W.anomalus C11响应乙醇胁迫,外源补充海藻糖可增强菌株的乙醇耐受性[37-38]。由表2可知,W.anomalus C11基因组中也注释到少数参与海藻糖合成的酶基因,如gene3198和gene3679。
半乳糖醇合酶参与半乳糖醇生物合成途径,对植物抗逆性发挥调控作用[39]。 由表2可知,在W.anomalus C11基因组中也注释到了半乳糖醇合酶基因gene0085,其在W.anomalus响应逆境胁迫中是否发挥调控作用,在后续研究中有必要进行深入分析。
综上,在W. anomalus C11中被注释的参与细胞壁合成、海藻糖合成以及半乳糖醇合酶相关糖基转移酶基因,可能与W.anomalus C11较强的逆境胁迫耐受性相关。
为评估W.anomalus C11的安全性,进一步对其致病性进行分析,结果见图6。
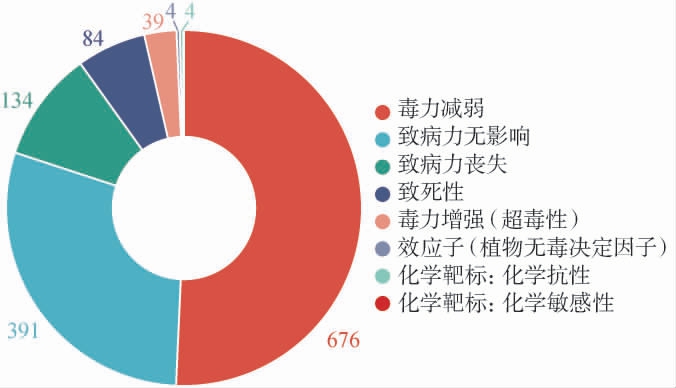
图6 异常威克汉姆酵母C11基因组致病性注释结果
Fig.6 Results of pathogenicity annotation of Wickerhamomyces anomalus C11 genome
由图6可知,从W. anomalus C11基因组中共注释到1 333个致病性相关基因,绝大部分基因被注释为毒性降低相关基因,包括毒性减弱基因676个,致病力无影响基因391个,致病力丧失基因134个;导致菌株致病性的基因为极少数,包括致死性基因84个,毒力增强(超毒力)基因39个。 结果表明W.anomalus C11毒性较低,安全性较高,这与已有的研究报道较为一致[6]。因此,W.anomalus可作为食品发酵菌株,改善食品风味特性,也可作为优良的单细胞饲料蛋白,用于家禽、牲畜的饲养业[40]。
本研究采用平板生长法对一株分离于刺梨果实自然发酵液的产酯W.anomalus C11逆境胁迫耐受性进行分析,结果表明,W.anomalus C11具有较好的环境耐受性,可耐受100 g/L氯化钠、25 mmol/L过氧化氢、500 mmol/L甘露醇、pH 3.0~10.0、16 ℃低温、37 ℃高温以及40 mmol/L铅的胁迫处理。为进一步挖掘W.anomalus C11产酯和逆境胁迫耐受性机制,采用Illumina二代测序技术,对其全基因组进行测序。 结果表明,W.anomalus C11基因组全长11.48 Mbp,GC含量为34.42%,编码基因4 249个基因。 通过COG、GO、KEGG以及CAZy数据库对其功能基因进行注释分析发现,其基因组从COG、GO和KEGG数据库中分别注释到1 641、3 346和3 145个功能基因,从CAZy数据库中注释到32个糖苷水解酶相关基因、39个糖基转移酶相关基因,参与细胞壁的合成,有助于维持细胞壁的完整性。 这从基因层面解释了W.anomalus C11可能的产酯和抗逆胁迫机制。 同时也发现,菌株W.anomalus C11基因组中含有毒性减弱基因676个,致病力无影响基因391个,致病力丧失基因134个,致死性基因84个,毒力增强(超毒力)基因39个,说明其毒性较小,具有一定的安全性。 综上,本研究进一步研究了产酯W.anomalus C11抗逆特性及其潜在分子机制,为进一步扩大该菌株的发酵应用领域提供了理论参考。
[1]LI Y, LONG H, JIANG G, et al.Analysis of the ethanol stress response mechanism in Wickerhamomyces anomalus based on transcriptomics and metabolomics approaches[J].BMC Microbiol,2022,22(1):275.
[2]SABEL A, MARTENS S, PETRI A, et al. Wickerhamomyces anomalus AS1:A new strain with potential to improve wine aroma[J].Ann Microbiol,2014,64:483-491.
[3]谢再斌,王太玉,王茜,等.异常威克汉姆酵母在白酒酿造中的应用研究[J].中国酿造,2022,41(5):18-22.
[4]JUNGES D S B,DELABENETA M F,ROSSETO L R B,et al.Antibiotic activity of Wickerhamomyces anomalus mycocins on multidrug-resistant Acinetobacter baumannii[J].Microb Ecol,2020,80:278-285.
[5]GODANA E A,EDO G S,YANG Q,et al.Wickerhamomyces anomalus:A promising yeast for controlling mold growth and diverse biotechnological applications[J].Trends Food Sci Tech,2024:104649.
[6]PADILLA B, GIL J V, MANZANARES P.Challenges of the non-conventional yeast Wickerhamomyces anomalus in winemaking[J].Fermentation,2018,4(3):68.
[7]李尚逊,姬翔,翟兴媛,等.异常威克汉姆酵母纯种发酵淡豆豉的炮制工艺研究[J].大豆科学,2023,42(4):459-465.
[8]YAN S B,CHEN X S,XIANG X B.Improvement of the aroma of lily rice wine by using aroma-producing yeast strain Wickerhamomyces anomalus HN006[J].AMB Express,2019,9(1):89.
[9]VIANA F, GIL J V, GENOVES S, et al.Rational selection of non-Saccharomyces wine yeasts for mixed starters based on ester formation and enological traits[J].Food Microbiol,2008,25:778-785.
[10]王斌.粉红单端孢全基因组测序及TrPacC和TrNox对其生长发育和致病力的调控[D].兰州:甘肃农业大学,2022.
[11]李佳,徐明洋,高同国,等.棉粕发酵菌株M-15的筛选及全基因组测序分析[J].微生物学通报,2024,51(9):3535-3550.
[12]郭宝松,张玉姣,代艺伟,等.氨肽酶生产菌株Bacillus subtilis LBJ4-5的全基因组测序及基因功能分析[J].食品工业科技,2024,45(24):32-41.
[13]刘芳,舒志强,王共明,等.产褐藻胶裂解酶菌株S10的鉴定、全基因组测序及分析[J].食品科学,2024,45(22):2273-2284.
[14]SCHNEIDER J,RUPP O,TROST E,et al.Genome sequence of Wickerhamomyces anomalus DSM 6766 reveals genetic basis of biotechnologically important antimicrobial activities[J].FEMS Yeast Res,2012,12(3):382-386.
[15]CUNHA A C, SANTOS R A, RIAÑO-PACHON D M, et al.Draft genome sequence of Wickerhamomyces anomalus LBCM1105,isolated from cachaça fermentation[J].Genet Mol Biol,2020,43:e20190122.
[16]刘晓柱,李银凤,于志海,等.刺梨自然发酵过程中非酿酒酵母多样性分析[J].微生物学报,2020,60(8):1696-1708.
[17]LIU X,LI Y,ZHAO H,et al.Identification and fermentative properties of an indigenous strain of Wickerhamomyces anomalus isolated from Rosa roxburghii Tratt[J].Brit Food J,2021,123(12):4069-4081.
[18]LIU X,LI Y,ZHOU J,et al.Effects of co-inoculation and sequential inoculation of Wickerhamomyces anomalus and Saccharomyces cerevisiae on the physicochemical properties and aromatic characteristics of longan (Dimocarpus longan Lour.) wine[J].Qual Assur Saf Crop, 2021,13(2):56-66.
[19]刘晓柱,黎华,李银凤,等.接种异常威克汉姆酵母对空心李果酒理化特性及香气组分的影响[J].食品科技,2020,45(11):21-27.
[20]刘晓柱,黄元敏,杨筱萱,等.一株库尔勒香梨源非酿酒酵母菌的鉴定及其发酵性能分析[J].中国酿造,2023,42(6):128-134.
[21]JEON S A,PARK J L,PARK S J,et al.Comparison between MGI and Illumina sequencing platforms for whole genome sequencing[J].Genes Genom,2021,43:713-724.
[22]ZHANG J, HE Y, YIN L, et al.Isolation of aroma-producing Wickerhamomyces anomalus yeast and analysis of its typical flavoring metabolites[J].Foods,2023,12(15):2934.
[23]FREDLUND E, DRUVEFORS U, BOYSEN ME, et al.Physiological characteristics of the biocontrol yeast Pichia anomala J121[J].FEMS Yeast Res,2002,2(3):395-402.
[24]李丽杰,贺银凤.高吸附Pb2+的异常威客汉姆酵母QF-1-1吸附Pb2+特性及机理[J].食品科学,2020,41(10):152-158.
[25]VALSALAN R, MATHEW D.Draft genome of Meyerozyma guilliermondii strain vkal:A yeast strain with composting potential[J].J Genetic Eng Biotech,2020,18(1):54.
[26]VALDES J, TAPIA P, CEPEDA V, et al.Draft genome sequence and transcriptome analysis of the wine spoilage yeast Dekkera bruxellensis LAMAP2480 provides insights into genetic diversity, metabolism and survival[J].FEMS Microbiol Lett,2014,361:104-106.
[27]MORIO F, O'BRIEN C E, BUTLER G.Draft genome sequence of the yeast Kazachstania telluris CBS 16338 isolated from forest soil in Ireland[J].Mycopathologia,2020,185:587-590.
[28]YOON J A,LEE Y J,PARK E H,et al.Draft genome sequence data of multi-stress tolerant yeast Millerozyma farinosa KCTC27753 isolated from nuruk[J].Data in Brief,2022,42:108030.
[29]BAUER S, GROSSMANN S, VINGRON M, et al.Ontologizer 2.0-a multifunctional tool for GO term enrichment analysis and data exploration[J].Bioinformatics,2008,24(14):1650-1651.
[30]SARRY J E, GÜNATA Z.Plant and microbial glycoside hydrolases:Volatile release from glycosidic aroma precursors[J].Food Chem,2004,87(4):509-521.
[31]BEHR M,NEUTELINGS G,EL JAZIRI M,et al.You want it sweeter:How glycosylation affects plant response to oxidative stress[J].Front Plant Sci,2020,11:571399.
[32]LAIRSON L L,HENRISSAT B,DAVIES G J,et al.Glycosyltransferases:Structures, functions, and mechanisms[J].Annu Rev Biochem, 2008,77(1):521-555.
[33]RUI Y,DINNENY J R.A wall with integrity:Surveillance and maintenance of the plant cell wall under stress[J].New Phytol,2020,225(4):1428-1439.
[34]SHARIFMOGHADAM M R, VALDIVIESO M H.The fission yeast SEL1 domain protein Cfh3p: A novel regulator of the glucan synthase Bgs1p whose function is more relevant under stress conditions[J].J Biol Chem,2009,284(17):11070-11079.
[35]LIU Y Y, MA X B, LONG Y, et al.Effects of β-1,6-glucan synthase gene(FfGS6)overexpression on stress response and fruit body development in Flammulina filiformis[J].Genes,2022,13(10):1753.
[36]IORDACHESCU M,IMAI R.Trehalose biosynthesis in response to abiotic stresses[J].J Integr Plant Biol,2008,50(10):1223-1229.
[37]LI Y F,JIANG G L,LONG H,et al.Contribution of trehalose to ethanol stress tolerance of Wickerhamomyces anomalus[J].BMC Microbiol,2023,23:239.
[38]LI Y F, JIANG G L, LONG H, et al.Exogenous trehalose application promotes survival by alleviating oxidative stress and affecting transcriptome in ethanol-stressed Wickerhamomyces anomalus[J].Brit Food J,2023,125(10):3506-3520.
[39]TAJI T, OHSUMI C, IUCHI S, et al.Important roles of drought-and cold-inducible genes for galactinol synthase in stress tolerance in Arabidopsis thaliana[J].Plant J,2002,29(4):417-426.
[40]HUYBEN D,NYMAN A, VIDAKOVIC A, et al.Effects of dietary inclusion of the yeasts Saccharomyces cerevisiae and Wickerhamomyces anomalus on gut microbiota of rainbow trout[J].Aquaculture, 2017,473:528-537.