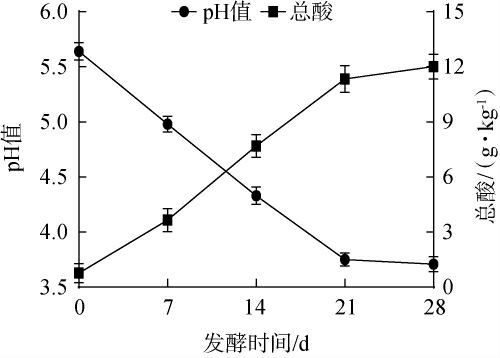
图1 鲜辣椒酱发酵过程中pH值和总酸含量的变化
Fig.1 Changes of pH and total acid contents during the fermentation of fresh chili sauce
Study on the interaction between bacterial community succession and endogenous environment during the fermentation of fresh chili sauce
辣椒(Capsicum annuum L.)是一种重要的蔬菜作物、调味品和辛辣香料,在全球范围内广泛种植和消费,并在农业经济组成中具有重要地位[1-2]。通常,鲜辣椒的贮藏周期较短,而发酵作为一种古老、高效、经济的农产品和食品保鲜技术,可用于增加辣椒的营养和风味成分,延长辣椒的保质期[3-4]。 辣椒酱是全球最受欢迎的辣椒加工产品,尤其是在亚洲[5]。 辣椒酱是通过在新鲜辣椒中添加食盐,由附着在辣椒上的本地微生物自发发酵而成,发酵赋予了辣椒独特的滋味、香气和健康功效益处[6]。微生物通过参与碳水化合物、氨基酸和脂肪酸等营养物质代谢,主导着鲜辣椒酱发酵过程,直接影响产品品质[3]。因此,阐明鲜辣椒酱发酵过程中微生物群落多样性的变化及其作用,有助于实现发酵辣椒生产的人工调控,已成为当下发酵食品工业的研究热点。
传统研究辣椒酱发酵过程中微生物群落多样性的方法大多数都采用独立培养方法,但仅有不足1%的微生物能够被培养,从而极大的限制了对辣椒酱微生物群落多样性的全面认识[7]。 随着高通量测序技术的快速发展,克服了传统微生物独立培养技术的限制,能够更加准确且全面地分析微生物群落多样性和演替变化,揭示发酵食品中更为复杂的微生物组成,包括不可培养的微生物、非优势群落和后期生长的物种[8]。刘颖等[3]利用高通量测序技术揭示了衡阳和韶关地区自然发酵辣椒中细菌群落的多样性和组成的差异。武亚婷等[9]研究发现,乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)、明串珠菌属(Leuconostoc)和魏斯氏菌属(Weissella)是新疆辣椒自然发酵过程中的优势细菌属。 LIAO H等[10]研究发现,辣椒发酵前期的主要优势细菌属为明串珠菌属、魏斯氏菌属和乳杆菌属,而乳杆菌属和片球菌属是发酵后期的主要优势微生物。
微生物群落演替是早期定植的微生物群落被一个或多个后续群落所取代。 微生物群落演替主要发生在微生物对特定环境的适应过程,包括外源环境(温度、湿度和盐度)和内源环境(酸度、pH、还原糖和氨基酸态氮),其中内源环境与原料的营养成分有关[11]。然而,鲜辣椒酱发酵过程中微生物群落和内源环境变化的作用尚不清楚,尤其是细菌群落演替。 阐明内源环境与微生物群落结构之间的相互作用关系有助于深入了解辣椒酱发酵过程中微生物演替的驱动机制。因此,本研究利用高通量测序技术揭示鲜辣椒酱发酵过程中细菌群落的演替及组成,探究内源环境指标的变化规律,揭示各内源环境因子与细菌群落之间的关系,以期为精准调控辣椒酱微生物群落提供理论依据。
新鲜辣椒:本地农副产品市场;氢氧化钠、酚酞、硫酸铜、甲醛、酒石酸钾纳、亚铁氰化钾、乙醇等(均为分析纯):国药集团化学试剂有限公司;Fast脱氧核糖核酸(deoxyri bonucleic acid,DNA)RSPIN Kit for Soil试剂盒、Taq DNA聚合酶:美国MP生物医疗公司;DNA纯化磁珠、MiniBEST Agarose Gel DNA Extraction Kit:宝日医生物技术(北京)有限公司;引物:杭州翱锐基因科技有限公司。其他试剂均为国产分析纯。
FE-28型pH计、XPR204S/AC型电子天平:瑞士梅特勒托利多集团;PBJ-S30E型破壁料理机:江门市贝尔斯顿电器有限公司;Pico17型高速离心机、MINIAMP/Veritipro32型聚合酶链式反应(polymerase chain reaction,PCR)仪:美国赛默飞科技公司;DYCZ-20C型DNA序列分析电泳仪、WD-9413B型凝胶成像分析系统、WD-9403B型紫外透射切胶台:北京六一生物科技有限公司;Illumina Miseq高通量测序仪:美国Illumina公司。
1.3.1 鲜辣椒酱的制备
选取新鲜辣椒,清水洗净表面杂质,沥干,去蒂,置于破壁料理机中高速粉碎30 s,加入12%食盐,混匀,均匀分装于15个1 L玻璃发酵罐中,每罐装800 g,置于30 ℃恒温密闭发酵28 d[12]。每隔7 d随机选取3个发酵罐,收集样品。
1.3.2 内源环境因子检测
参照国标GB 5009.237—2016《食品安全国家标准 食品pH值的测定》测定pH值;参照国标GB 12456—2021《食品安全国家标准食品中总酸的测定》测定总酸含量;参照国标GB 5009.7—2016《食品安全国家标准 食品中还原糖的测定》测定还原糖含量;参照国标GB 5009.235—2016《食品安全国家标准食品中氨基酸态氮的测定》 测定氨基酸态氮含量。
1.3.3 细菌群落多样性分析
采用Fast DNARSPIN Kit for Soil试剂盒提取样品微生物基因组DNA,利用1%琼脂糖凝胶电泳检测DNA的质量,并使用NaneDrop 2000型超微量紫外分光光度计测定DNA浓度和纯度[13]。以提取的基因组DNA为模板,使用引物338F(5'-ACTCCTACGGGAGGCAGCA-3')和806(5'-GGACTACHVGGGTWTCTAAT-3')PCR扩增细菌16S rRNA V3~V4区基因序列。PCR扩增体系:DNA模板(10 ng)1 μL,上下游引物(5 μmol/L)各1 μL,Taq DNA聚合酶1 μL,5×FastPfu缓冲液4 μL,脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTPs)(2.5 mmol/L)2 μL,去离子水10 μL[13]。PCR扩增条件:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,共35个循环;72 ℃再延伸5 min[13]。 每个样品3个重复,合并同一样品的PCR扩增产物,采用2%琼脂糖凝胶电泳检测,使用MiniBEST Agarose Gel DNA Extraction Kit试剂盒切胶回收PCR产物和纯化,纯化后委托杭州翱锐基因科技有限公司进行高通量测序[13]。
参照陆敏等[14]的方法进行序列和生物信息学分析,具体如下:使用FASTQ软件对原始测序序列进行质控,利用USEARCH软件按照默认参数对每个样品的序列进行拼接,对97%相似性的可操作分类单元(operational taxonomic unit,OTU)进行聚类和删除嵌合体,得到优质代表序列,基于SILVA 138数据库对OTU进行物种分类注释,删除不能在门水平上注释的OTU[14]。
1.3.4 统计分析
采用Excel 2019软件进行数据处理,利用SPSS 21.0软件进行单因素方差分析(analysisofvariance,ANOVA),P<0.05表示差异显著,P<0.01表示差异极显著,使用R 4.3.1软件计算微生物Alpha多样性指数,进行细菌群落组成分析、主 坐 标 分析(principal co-ordinates analysis,PCoA)、层 次聚类分析(hierarchical clustering analysis,HCA)、相似性分析(analysis of similarities,ANOSIM)和线性判别分析效应大小(linear discriminant analysis effect size,LEfSe)分析,并对内源环境因子和优势细菌群落属进行冗余分析(redundancy analysis,RDA)、相关性分析和方差分解分析(variance partitioning analysis,VPA)。
pH值和酸度是评价发酵辣椒中微生物代谢活性的重要指标[7],鲜辣椒酱发酵过程中pH值及酸度的变化见图1。由图1可知,鲜辣椒酱的初始pH值和总酸含量分别为5.64和0.77 g/kg。随着发酵的进行,鲜辣椒酱pH值在发酵0~21 d内快速下降,下降至3.75;随后pH值在发酵21~28 d内缓慢下降,于发酵28 d时pH值稳定在3.71。此外,鲜辣椒酱发酵过程中总酸含量的变化与pH值的变化相反,呈现先快速增加(0~21 d)后缓慢增加(21~28 d)的趋势。 在发酵28 d时,鲜辣椒酱的总酸含量可达12.02 g/kg,符合农业标准NY/T 1711—2020《绿色食品辣椒制品》中辣椒总酸含量≤20 g/kg的要求。 鲜辣椒酱发酵过程中pH值和总酸含量的变化与YE Z等[15]的研究结果相一致。 鲜辣椒酱中原生微生物的代谢活性,尤其是乳酸菌,在发酵初期能快速生长繁殖,产生大量酸性物质,抑制致病和腐败等不耐酸微生物的生长[16]。

图1 鲜辣椒酱发酵过程中pH值和总酸含量的变化
Fig.1 Changes of pH and total acid contents during the fermentation of fresh chili sauce
氨基酸态氮含量是评估发酵辣椒滋味品质的重要指标之一[12],还原糖是微生物生长和代谢所需的碳源。 鲜辣椒酱发酵过程中氨基酸态氮和还原糖含量的变化见图2。由图2可知,鲜辣椒酱的初始氨基酸态氮含量仅为0.23 g/kg。随着发酵的进行,鲜辣椒酱的氨基酸态氮含量在发酵0~7 d内快速增加,增加至0.53 g/kg;随后缓慢增加,于发酵21 d达到稳定,其氨基酸态氮含量维持在0.62 g/kg。这一结果与LIAO H等[10]研究结果相一致。氨基酸态氮的主要贡献者是多肽和游离氨基酸,其含量与微生物生长代谢密切相关,微生物分泌的蛋白酶将蛋白质降解为多肽和游离氨基酸,促进发酵前期氨基酸态氮含量迅速增加[12]。 此外,鲜辣椒酱的还原糖含量在发酵前21 d快速下降,从38.37 g/kg下降至15.61 g/kg,随后维持稳定,于发酵28 d时还原糖含量稳定在15.54 g/kg。 较高的还原糖含量可为辣椒酱中乳酸菌生长代谢提供丰富的营养来源,通过糖酵解代谢将还原糖转化为丙酮酸,进一步代谢为酸类、醇类和酯类等风味物质[17]。然而,还原糖的消耗速率减慢,可能是因为较高的酸性环境限制了微生物对糖的利用。 综上,鲜辣椒酱发酵过程中内源环境发生了剧烈变化。
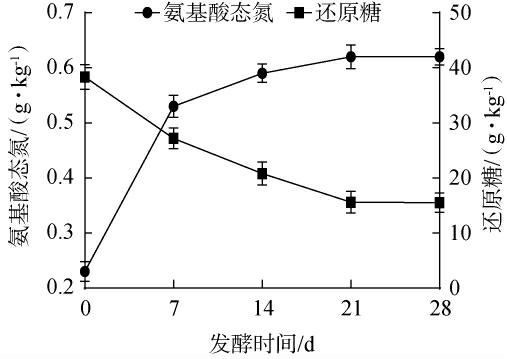
图2 鲜辣椒酱发酵过程中氨基酸态氮和还原糖含量的变化
Fig.2 Changes of amino acid nitrogen and reducing sugar contents during the fermentation of fresh chili sauce
利用高通量测序技术对鲜辣椒酱发酵过程中的细菌菌群多样性进行分析,结果见表1。
表1 鲜辣椒酱发酵过程中细菌群落高通量测序及Alpha多样性分析结果
Table 1 Results of high-throughput sequencing and alpha diversity analysis of bacterial communities during the fermentation of fresh chili sauce
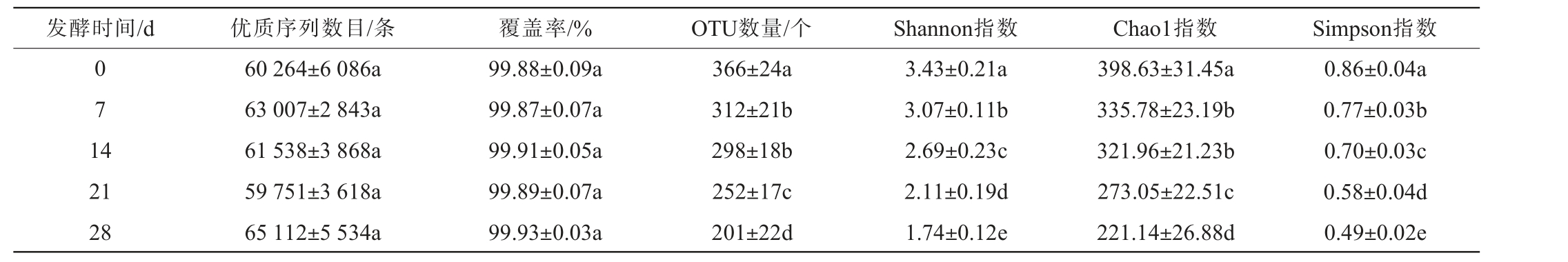
注:同列小写字母不同表示差异显著(P<0.05)。
发酵时间/d 优质序列数目/条 覆盖率/% OTU数量/个 Shannon指数 Chao1指数 Simpson指数0 7 1 4 21 28 60 264±6 086a 63 007±2 843a 61 538±3 868a 59 751±3 618a 65 112±5 534a 99.88±0.09a 99.87±0.07a 99.91±0.05a 99.89±0.07a 99.93±0.03a 366±24a 312±21b 298±18b 252±17c 201±22d 3.43±0.21a 3.07±0.11b 2.69±0.23c 2.11±0.19d 1.74±0.12e 398.63±31.45a 335.78±23.19b 321.96±21.23b 273.05±22.51c 221.14±26.88d 0.86±0.04a 0.77±0.03b 0.70±0.03c 0.58±0.04d 0.49±0.02e
由表1可知,不同时间鲜辣椒酱的优质序列数目和覆盖率范围分别为59 751~65 112条和99.87%~99.93%,各组之间无显著差异(P>0.05),表明测序深度足够解释鲜辣椒酱细菌群落的多样性,绝大多数物种均能检出,能满足进一步分析的要求[18]。所有样品中细菌OTU数量最多的是发酵初始的鲜辣椒酱,其平均OTU数目可达366个,显著高于其他发酵时间的鲜辣椒酱(P<0.05),表明鲜辣椒和发酵器具中含有大量的原生细菌,促进了鲜辣椒酱的发酵进程。 此外,通过Shannon指数、Chao1指数和Simpson指数对鲜辣椒酱细菌群落组成进行Alpha多样性分析,其中Shannon指数和Simpson指数反映细菌群落多样性,其值越大代表细菌群落的多样性越高;而Chao1指数则反映细菌群落的丰富度,其值越小代表细菌物种数越少[19]。鲜辣椒酱初始的Shannon指数、Chao1指数和Simpson指数分别为3.43、398.63和0.86,显著高于其他发酵时间的鲜辣椒酱(P<0.05)。 随着发酵的进行,鲜辣椒酱的Shannon指数、Chao1指数和Simpson指数呈逐渐降低的变化趋势,于发酵28 d 时Shannon 指 数、Chao1 指 数 和Simpson指 数 分 别为1.74、221.14和0.49。通常,盐度和pH是影响细菌群落结构组成和多样性的重要因素,其持续高盐和pH值不断降低的发酵环境导致微生物数量和多样性持续降低[20]。 因此,鲜辣椒酱发酵过程中细菌群落丰富度和多样性呈显著下降趋势(P<0.05)。
基于细菌OTU的相对丰度,利用基于未加权UniFrac距离的PCoA分析,解析鲜辣椒酱发酵过程中细菌群落结构组成的差异,结果见图3A。
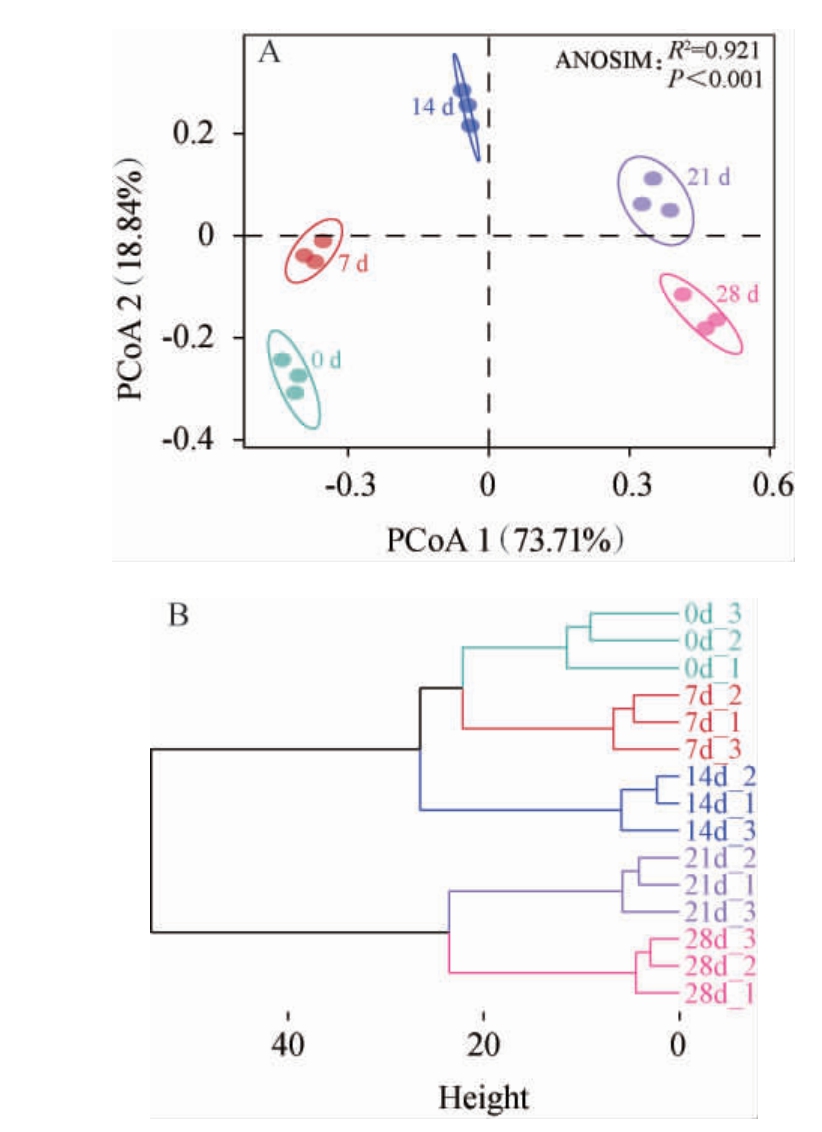
图3 鲜辣椒酱发酵过程中细菌群落组成的主坐标分析(A)和层次聚类分析(B)
Fig.3 Principal coordinate analysis (A) and hierarchical clustering analysis (B) of bacterial community composition during the fermentation of fresh chili sauce
由图3A可知,前两个坐标共解释了鲜辣椒酱发酵过程中细菌群落总变异的92.55%,其中PCoA 1可解释73.71%,PCoA 2可解释18.84%,表明辣椒发酵过程中大部分细菌群落差异可用PCoA前两轴进行解释[5]。此外,除发酵0 d和7 d的鲜辣椒酱位于坐标轴同一象限内,其他不同发酵时间的辣椒分别位于坐标轴的不同象限。 结合ANOSIM分析结果可知,鲜辣椒酱发酵过程中细菌群落结构差异极显著(R2=0.921,P<0.001),表明不同发酵时间的鲜辣椒酱细菌群落结构组成不同,通过改变微生物组成适应持续高盐且逐渐酸化的复杂发酵环境[21]。 随后,利用基于非加权配对算术平均算法构建的HCA分析鲜辣椒酱发酵过程中不同时间细菌群落结构组成的差异性,结果见图3B。由图3B可知,发酵0 d、7 d和14 d的鲜辣椒酱聚类为一簇,而发酵21 d和28 d的鲜辣椒酱则聚类为另一簇,表明发酵时间会显著影响细菌群落结构,导致聚类距离越远。此外,同一发酵时间的鲜辣椒酱细菌群落能较好的聚类在一起,说明组内差异远弱于组间差异。这些结果与PCoA结果相一致,显示出相似的聚集模式。 综上,鲜辣椒酱发酵过程中细菌群落发生了明显变化。
鲜辣椒酱发酵过程中细菌群落的组成见图4。

图4 基于门(A)和属(B)水平鲜辣椒酱发酵过程中细菌群落的演替规律
Fig.4 Succession of bacterial communities during fermentation of fresh chili sauce based on phylum (A) and genus (B) levels
由图4A可知,所有鲜辣椒酱样品共检测到4个细菌门,包括变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Acinobacteriota),其中,变形菌门和厚壁菌门相对丰度之和达到93.77%~99.87%,为绝对优势细菌门。 这一结果与SHI Q等[22]研究结果相一致。随着发酵的进行,不同发酵时间的鲜辣椒酱样品中变形菌门、厚壁菌门、拟杆菌门和放线菌门的相对丰度呈现出差异,其中变形菌门、拟杆菌门和放线菌门的相对丰度逐渐下降,尤其是变形菌门的相对丰度从40.05%下降至4.71%,而厚壁菌门的相对丰度则逐渐上升,相对丰度从54.27%上升至95.15%。因此,厚壁菌门和变形菌门是鲜辣椒酱发酵的主要优势细菌来源。
由图4B可知,所有鲜辣椒酱样品共检测到58个细菌属,其中,相对丰度较高的是乳杆菌属(Lactobacillus)、明串珠菌属(Leuconostoc)、片球菌属(Pediococcus)、葡萄球菌属(Staphylococcus)、魏斯氏菌属(Weissella)、肠杆菌属(Enterobacter)、乳球菌属(Lactococcus)、欧文氏菌属(Erwinia)、普雷沃氏菌属(Prevotella)、不动杆菌属(Acinetobacter)、克雷伯氏菌属(Klebsiella)、鞘氨醇单胞菌属(Sph ingomonas)和沙雷氏菌属(Serratia),其中欧文氏菌属、普雷沃氏菌属、不动杆菌属、克雷伯氏菌属和沙雷氏菌属是常见的腐败致病菌[22]。发酵0 d鲜辣椒酱中优势细菌属(相对丰度>1%)种类最多,分别为明串珠菌属(31.46%)、魏斯氏菌属(9.58%)、乳杆菌属(6.17%)、克雷伯氏菌属(5.32%)、普雷沃氏菌属(4.63%)、乳球菌属(4.48%)、欧文氏菌属(4.31%)、泛菌属(Pantoea)(3.73%)、鞘氨醇单胞菌属(3.49%)、科萨克氏菌属(Kosakonia)(3.29%)和不动杆菌属(2.85%),与表1结果相一致,表明鲜辣椒表面携带的多种细菌共同启动辣椒酱的发酵[6]。 随着发酵的进行,克雷伯氏菌属、普雷沃氏菌属、欧文氏菌属、泛菌属和不动杆菌属的相对丰度逐渐下降,在发酵28 d时,几乎检测不到,表明这些微生物不能在持续高盐和低pH的发酵环境中生长繁殖。 明串珠菌属和魏斯氏菌属的相对丰度在鲜辣椒酱发酵过程中呈先上升后下降的变化趋势,均在发酵7 d时达到最大,其相对丰度分别为38.01%和15.03%,而发酵28 d时,二者的相对丰度均<1%,说明串珠菌属和魏斯氏菌属在鲜辣椒酱发酵前期迅速占据了生态位,与鲊广椒发酵的早期细菌群落演替相一致[23]。这些细菌通过产生淀粉酶、蛋白酶、酯酶和葡萄糖苷酶降解原料中大分子物质,为其他微生物的生长繁殖提供营养源[24]。 值得注意的是,肠杆菌属和葡萄球菌属在鲜辣椒酱发酵过程中生长繁殖滞后于明串珠菌属和魏斯氏菌属,分别在发酵14 d和21 d达到最大,其相对丰度分别为10.43%和17.76%,而发酵28 d时,其相对丰度分别下降至1.84%和4.35%。肠杆菌属和葡萄球菌属利用明串珠菌属和魏斯氏菌属产生的小分子氨基酸、多肽和糖类物质生长代谢,为细菌群落演替创造一个新的生态位[25]。乳杆菌属是鲜辣椒酱发酵过程中的优势细菌属,其相对丰度随着发酵的进行逐渐上升,从4.48%上升至54.67%,而片球菌属则是辣椒发酵后期的优势细菌属,其相对丰度从4.82%(14 d)上升至32.54%(28 d)。 发酵28 d时,乳杆菌属和片球菌属的相对丰度之和达到87.21%,表明这两类细菌能够在低pH和高渗环境中生长。此外,乳杆菌属和片球菌属还可以产生酸类物质和挥发性风味化合物来影响发酵辣椒的口感和风味[15]。 综上,鲜辣椒酱发酵过程中细菌群落结构呈现不同菌属交替演替变化。
从不同水平上应用LEfSe分析鉴别鲜辣椒酱发酵过程中细菌群落标志性差异类群,结果见图5。由图5可知,共27个属水平的细菌类群在辣椒不同发酵时间存在显著差异(P<0.05),分别来自肠杆菌科(Enterococcaceae)、欧文科(Erwiniaceae)、链球菌科(Streptococcaceae)、乳杆菌科(Lactobacillaceae)、鞘脂单胞菌科(Sphingomonadaceae)、芽孢杆菌科(Bacillaceae)、葡萄球菌科(Staphylococcaceae)、醋杆菌科(Acetobacteraceae)、明串珠菌科(Leuconostocaceae)和耶尔辛氏菌科(Yersiniaceae)等。 发酵0 d鲜辣椒酱中细菌群落标志性差异类群最多,主要来自欧文氏菌科、鞘脂单胞菌科和耶尔辛氏菌科。发酵7 d鲜辣椒酱中细菌群落标志性差异类群主要为明串珠菌科和链球菌科,而发酵14 d和21 d鲜辣椒酱分别主要为肠杆菌科和葡萄球菌科。 发酵28 d鲜辣椒酱中细菌群落标志性差异类群主要为醋杆菌科和芽孢杆菌科。 因此,不同发酵时间的不同环境会对细菌类群进行特异性选择,引起生态位差异,导致细菌群落差异巨大。
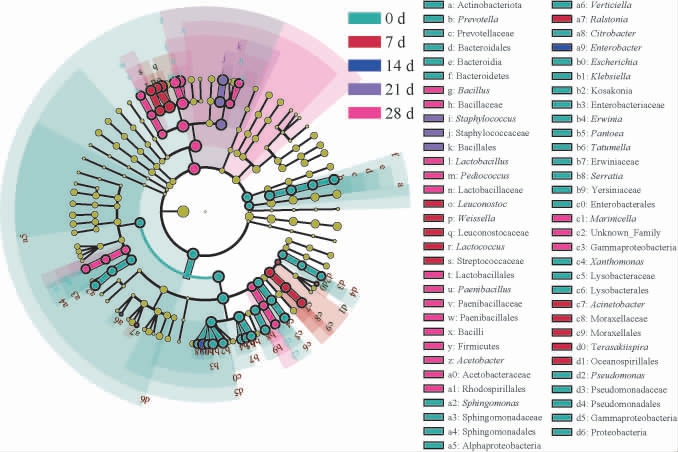
图5 鲜辣椒酱发酵过程中不同发酵时间标志性菌群的分析
Fig.5 Analysis of marker bacterial communities at different fermentation time during fermentation of fresh chili sauce
将发酵过程中总酸含量和pH值作为酸性因子,还原糖和氨基酸态氮含量作为营养因子,利用VPA分析鲜辣椒酱发酵过程中内源环境因子对细菌群落演替的影响,结果见图6。 由图6可知,内源环境因子共同解释鲜辣椒酱发酵过程中细菌群落演替的68.7%变异,而酸性因子和营养因子则分别解释细菌群落演替的5.4%和8.5%,说明大多数鲜辣椒酱的细菌菌种受发酵过程中内源环境变化所调控。此外,仍有17.4%变异不能用所测内源环境指标进行解释,从而表明还有其他因素会影响辣椒发酵过程中细菌群落演替。这一结果与唐艳平等[19]研究结果相似,复杂环境下多种环境因素共同影响微生物群落结构组成。
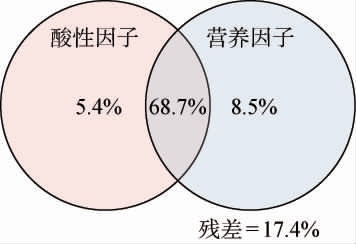
图6 鲜辣椒酱发酵过程中细菌群落演替与内源环境因子的方差分解分析结果
Fig.6 Variance decomposition analysis results of bacterial community succession and endogenous environmental factors during fermentation of fresh chili sauce
进一步采用RDA分析鲜辣椒酱发酵过程中内源环境与细菌群落(平均相对丰度排名前10的细菌属)演替的关系,结果见图7。由图7可知,RDA分析解析了内源环境因子对鲜辣椒酱发酵过程中细菌群落演替贡献度的91.51%,其中RDA 1和RDA 2分别解释了87.55%和3.96%。发酵28 d鲜辣椒酱与其他发酵时间鲜辣椒酱的距离较远,且独自位于一个象限中,与图3A结果相一致。发酵0 d和7 d鲜辣椒酱细菌群落与还原糖和pH值呈正相关,细菌群落主要由明串珠菌属、乳球菌属、魏斯氏菌属、欧文氏菌属和普雷沃氏菌属组成。 发酵21 d鲜辣椒酱细菌群落与氨基酸态氮和总酸呈正相关,细菌群落主要由葡萄球菌属、乳杆菌属和片球菌属组成。总酸含量和pH值呈负相关,这与我们的认知相一致。此外,总酸含量与氨基酸态氮呈正相关,说明鲜辣椒酱中细菌群落通过消耗淀粉和酶解蛋白质,生成氨基酸、多肽和酸性化合物。 因此,鲜辣椒酱发酵过程中多种内源环境因子驱动细菌群落演替。
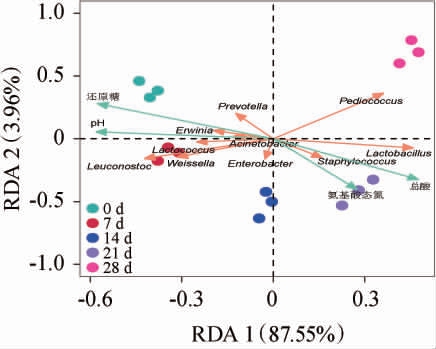
图7 鲜辣椒酱发酵过程中细菌群落演替与内源环境因子的冗余分析结果
Fig.7 Redundancy analysis results of bacterial community succession and endogenous environmental factors during fermentation of fresh chili sauce
为明确内源环境因子对细菌群落的具体影响,采用相关性分析探究二者之间的关系,结果见图8。由图8可知,鲜辣椒酱发酵过程中乳杆菌属与总酸含量和氨基酸态氮含量呈极显著正相关(P<0.01),与还原糖含量和pH值呈极显著负相关(P<0.01)。这一结果与GÄNZLE M G[26]的研究结果相一致,乳酸菌可通过代谢果蔬原料的大分子物质,为后来的微生物提供营养,并使发酵环境逐渐酸化,抵御有害微生物入侵,保障发酵食品安全性。因此,植物乳植杆菌(Lactiplantibacillus plantarum)和发酵乳杆菌(Lactobacillus fermentium)常被用于制作发酵果蔬的发酵剂[27]。此外,葡萄球菌属与氨基酸态氮含量呈显著正相关(P<0.05),与还原糖含量呈显著负相关(P<0.05);而片球菌属则与总酸含量呈显著正相关(P<0.05),与pH值呈显著负相关(P<0.05)。魏斯氏菌属、乳球菌属和明串珠菌属与还原糖含量和pH值呈显著或极显著正相关(P<0.01或P<0.05),与氨基酸态氮含量和总酸含量呈显著负相关(P<0.01或P<0.05)。综上,辣椒发酵过程中不同内源环境因子与优势细菌属的相互作用关系存在差异。
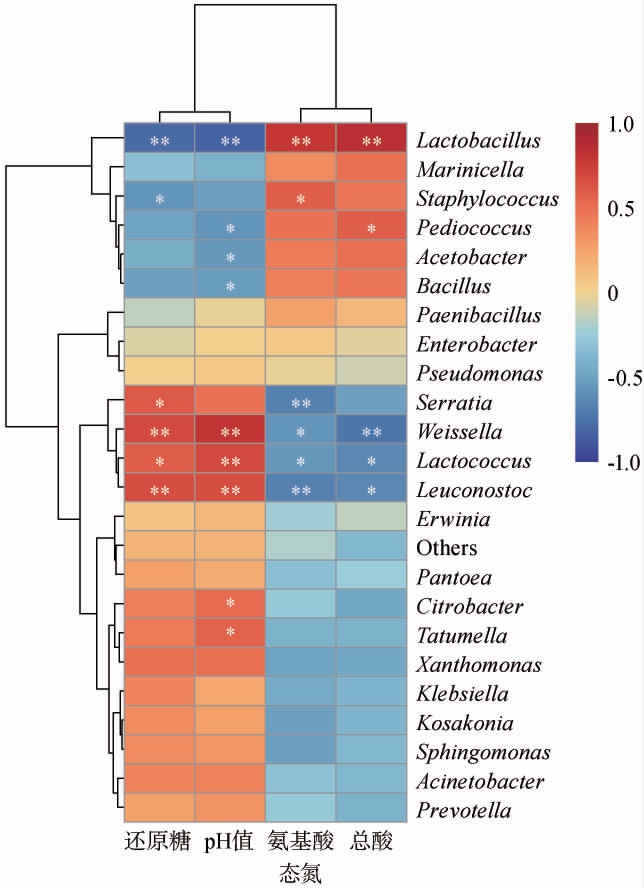
图8 鲜辣椒酱发酵过程中内源环境因子与主要细菌属间的相关性分析结果
Fig.8 Correlation analysis results between endogenous environmental factors and main bacterial genera during fermentation of fresh chili sauce
“*”表示显著相关(P<0.05),“**”表示极显著相关(P<0.01)。
本研究解析了鲜辣椒酱发酵过程中细菌群落及内源环境因子的变化规律,确定了内源环境因子对细菌群落演替的贡献及互作关系。结果表明,随着发酵的进行,鲜辣椒酱中还原糖含量和pH值逐渐下降,总酸及氨基酸态氮含量呈先上升后趋于稳定的趋势,发酵28 d时,鲜辣椒酱的pH、总酸、氨基酸态氮和还原糖含量分别为3.71、12.02 g/kg、0.62 g/kg和15.54 g/kg。鲜辣椒酱发酵过程中细菌群落的丰富度和多样性呈下降趋势,不同发酵时间的鲜辣椒酱细菌群落的结构组成和标志性菌属存在差异,但乳杆菌属贯穿着鲜辣椒酱的整个发酵过程,其平均相对丰度从4.48%上升至54.67%。此外,基于RDA和VPA结果表明,鲜辣椒酱发酵过程中多种内源环境因子共同驱动细菌群落演替,所测内源环境因子共同解释细菌群落演替的68.7%变异,其中,乳杆菌属与总酸和氨基酸态氮含量呈极显著正相关(P<0.01),与还原糖含量和pH值呈极显著负相关(P<0.01)。综上所述,本研究深入探究辣椒发酵过程中细菌群落演替及其内源驱动因素,为发酵辣椒菌群结构的功能调控提供理论基础。
[1]HERNÁNDEZ-PÉREZ T,GÓMEZ-GARCÍA M R,VALVERDE M E,et al.Capsicum annuum(hot pepper):An ancient Latin-American crop with outstanding bioactive compounds and nutraceutical potential.A review[J].Compr Rev Food Sci Food Saf,2020,19(6):2972-2993.
[2]ZOU Z,ZOU X.Geographical and ecological differences in pepper cultivation and consumption in China[J].Front Nutr,2021,8:718517.
[3]刘颖,李晓燕,高静.韶关和衡阳地区自然发酵辣椒微生物群落多样性及功能差异[J].中国食品添加剂,2022,33(3):217-227.
[4]MARCO M L, SANDERS M E, GÄNZLE M, et al.The International Scientific Association for Probiotics and Prebiotics (ISAPP) consensus statement on fermented foods[J].Nat Rev Gastroenterol Hepatol,2021,18(3):196-208.
[5]CAI W,TANG F,WANG Y, et al.Bacterial diversity and flavor profile of zha-chili,a traditional fermented food in China[J].Food Res Int,2021,141:110112.
[6]LI M, XU X, BI S, et al.Identification and validation of core microbes associated with key aroma formation in fermented pepper paste(Capsicum annuum L.)[J].Food Res Int,2023,163:112194.
[7]张睿艺,谢辽辽,田星.辣椒品种对发酵辣椒酱真菌多样性及其理化指标的影响[J].食品科技,2024,49(1):282-289.
[8]贾晶晶,魏爱丽,赵虎威,等.基于高通量测序分析不同食盐浓度的酸菜中微生物多样性[J].中国调味品,2022,47(8):167-170,174.
[9]武亚婷,杜木英,何欢欢,等.基于高通量测序技术分析新疆不同地区自然发酵辣椒酱微生物群落多样性[J].食品与发酵工业,2019,45(21):221-228.
[10]LIAO H,LUO Y,HUANG X,et al.Dynamics of quality attributes,flavor compounds,and microbial communities during multi-driven-levels chili fermentation:Interactions between the metabolome and microbiome[J].Food Chem,2023,405:134936.
[11]LI M, LAO F, PAN X, et al.Insights into the mechanisms driving microbial community succession during pepper fermentation:Roles of microbial interactions and endogenous environmental changes[J].Food Res Int,2024,179:114033.
[12]郑容,何旭峰,熊双,等.不同品种辣椒自然发酵过程中品质变化研究[J].中国酿造,2024,43(3):243-249.
[13]王雪雅,陆宽,殷勇,等.贵州不同地区“生花”糟辣椒中微生物多样性研究[J].中国酿造,2021,40(8):46-53.
[14]陆敏,王雪雅,孙小静,等.贵州南部红酸汤理化指标与菌群结构的相关性分析[J].中国酿造,2023,42(9):109-114.
[15]YE Z,SHANG Z,ZHANG S,et al.Dynamic analysis of flavor properties and microbial communities in Chinese pickled chili pepper (Capsicum frutescens L.):A typical industrial-scale natural fermentation process[J].Food Res Int,2022,153:110952.
[16]THIERRY A, BATY C, MARCHÉ L, et al.Lactofermentation of vegetables: An ancient method of preservation matching new trends[J].Trends Food Sci Technol,2023,139:104112.
[17]杨希,武爱群.酸菜发酵过程中理化因子及原核微生物群落结构差异分析[J].食品与发酵工业,2020,46(22):245-251.
[18]杨晨,柯悦,黄锶钘,等.不同地区鲊广椒细菌多样性比较研究[J].食品工业科技,2023,44(10):145-150.
[19]唐艳平,高秀兵,倪婕,等.白酒发酵副产物黄水原核微生物组成及理化特性研究[J].中国酿造,2024,43(3):72-78.
[20]LIAO H, LUO Y, ASIF H, et al.Novel insights into safety and quality enhancement of low-salt fermented chilies: High-order positively interacting lactic acid bacteria co-fermentation regulates microflora structure,metabolomics,and volatilomics profiles[J].Food Biosci,2024,59:103861.
[21]ZHANG S, BAO X, XIAO Y, et al.Metagenomics reveals microbial communities and functional differences during chili pepper (Capsicum frutescens L.) natural fermentation: Effects of brines and containers[J].Food Front,2024,5:753-770.
[22]SHI Q, TANG H, MEI Y, et al.Effects of endogenous capsaicin stress and fermentation time on the microbial succession and flavor compounds of chili paste(a Chinese fermented chili pepper)[J].Food Res Int,2023,168:112763.
[23]DONG W,SHEN H,LIU H,et al.Unraveling the microbial community and succession during zha-chili fermentation and their relationships with flavor formation[J].Food Res Int,2022,157:111239.
[24]WU Q,ZHU Y,FANG C,et al.Can we control microbiota in spontaneous food fermentation?-Chinese liquor as a case example[J].Trends Food Sci Tech,2021,110(4):321-331.
[25]BLASCHE S, KIM Y, MARS R A, et al.Metabolic cooperation and spatiotemporal niche partitioning in a kefir microbial community[J].Nat Microbiol,2021,6(2):196-208.
[26]GÄNZLE M G.Lactic metabolism revisited: metabolism of lactic acid bacteria in food fermentations and food spoilage[J].Curr Opin Food Sci,2015,4(2):106-117.
[27]ASHAOLU T J,REALE A.A holistic review on Euro-Asian lactic acid bacteria fermented cereals and vegetables[J].Microorganisms,2020,8(8):1176.