白酒是以谷物为原料,以酒曲作为发酵动力源,经蒸煮、发酵、蒸馏、勾调而成[1]。 酒曲是白酒生产的糖化发酵剂,富含微生物,其中微生物不仅是酒发酵过程的动力来源,而且对酒独特香味的形成具有重要影响,曲中的微生物之间相互作用共同维持酒醅发酵的正常进行[2-3]。酒醅是酿酒原料循环、能量流动以及酿酒微生物代谢的重要载体,在其发酵过程中,来自不同酒曲、窖泥、周围环境的微生物起着关键作用,酒曲、环境中的微生物能够分解谷物中的淀粉、纤维素、糖类和蛋白质,产生有机酸和香味[4]。
近年来,高通量测序技术因具有灵敏度高、数据量高、覆盖率广、重复性好、可用于分析不可培养微生物等优点[5],被广泛应用于各种类酒曲及不同香型白酒酿造的微生物群落结构分析中[6-9]。谷晓东等[10]利用高通量测序技术研究6种不同黄酒酒曲中微生物多样性,结果发现,6种酒曲在门水平和属水平上微生物群落结构均存在较大差异。 另外,对于优势菌群的筛选与鉴定也可通过高通量技术来实现,李斌等[11]应用高通量技术分析红曲、大曲、小曲3种曲的微生物菌群多样性,结果发现大曲的菌群多样性最大,红曲的最低,且两者间微生物群落结构差异最大,3种酒曲的优势菌群也不尽相同;贾丽艳等[12]采用高通量技术研究清香型白酒大米查和二米查真菌菌群多样性,也证实了其二者的优势菌群。 目前的研究主要集中于不同类型酒醅发酵过程中或同类型酒醅的不同轮次酒醅微生物菌群演替规律[13-15]、菌群与风味物质的关联性[16-17],而对不同酒曲发酵酒醅微生物菌群的对比研究较少。
本研究基于课题组前期已对4种酒曲(苗家酒曲、安琪酒曲、伍曲香、广西中草药酒曲)微生物菌群多样性研究的基础上,进一步选取这4种酒曲,采用相同发酵工艺发酵酒醅。 利用高通量测序技术分析比较4种不同酒曲发酵酒醅的真菌菌群多样性,考察不同酒曲对酒醅真菌菌群的影响,以期为进一步复合型酒曲的制备、提升酒体风味及质量提供理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
苗家酒曲(MJ):实验室自制;安琪酒曲(AQ):安琪酵母股份有限公司;伍曲香(WQ):凯里市时新酒曲开发有限公司;广西中草药酒曲(ZY):平乐县唐风酒饼加工厂。酒醅:参照文献[18]发酵酒醅,发酵30 d后从发酵缸的上、中、下层3个位置分别采集酒醅样品60 g,混合,装入无菌采样袋密封保存。
1.1.2 试剂
MN NucleoSpin 96 Soi脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒:上海恒斐生物科技有限公司;KOD FX Neo高保真聚合酶链式反应(polymerase chain reaction,PCR)酶(酶活1.0 U/μL):东洋纺(上海)生物科技有限公司;Phusion Plus DNA聚合酶(酶活5 U/μL):美国New England Biolabs(NEB)公 司;Monarch DNA 胶 回 收 试 剂盒:基因生物技术国际贸易(上海)有限公司;脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)、琼脂糖凝胶:生工生物工程(上海)股份有限公司。
1.2 仪器与设备
Nova-Seq6000测序仪:美国Illumina公司;G560E振荡器:美国ScientificIndustries;1795微型离心机:天根生化科技(北京)有限公司;9902 96 well PCR仪:上海艾研生物科技有限公司;5424EQ76675124孔离心机:德国EPPENDORF公司。
1.3 方法
1.3.1 不同酒曲发酵酒醅微生物菌群基因组DNA提取、PCR扩增及高通量测序
参照MN NucleoSpin 96 Soi DNA提取试剂盒说明书提取酒醅中微生物菌群的基因组DNA,利用1%琼脂糖凝胶电泳检测基因组DNA质量。以质量检测合格的基因组DNA为模板,参照文献[19]采用引物ITS1F(5'-CTTGGTCATT TAGAGGAAGTAA-3')和ITS2(5'-GCTGCGTTCTTCATCGATGC-3')对真菌菌群的ITS1区基因序列进行PCR扩增,并委托南昌科畅生物科技有限公司采用IlluminaNovaseq 6000 PE250平台构建文库和高通量测序。
1.3.2 生物信息学分析
利用FLASH(version1.2.11)软件[20]对测序获得的原始数据进行拼接,之后使用Trimmomatic 0.33软件[21]对拼接得到的序列进行质量过滤,并采用UCHIME 8.1软件[22]去除嵌合体,获得高质量的优化序列(Tags)。利用USEARCH 10.0软件[23]在相似度97%的水平上划分操作分类单元(operationaltaxonomicunits,OTUs)[19]。 基于RDP Classifier(http://sourceforge.net/projects/rdpclassifier/)[24]对OTU进行分类学注释分析(阈值为0.8),同时计算该OTU在各样品中的相对丰度,利用Mothur 1.30软件[25]进行Alpha多样性分析。
1.3.3 多元统计分析及数据处理
对最小测序深度下发现物种数和香农指数等α多样性指标进行分析,同时基于UniFrac距离使用主坐标分析(principal coordinates analysis,PCoA)对4种酒醅的真菌类群进行β多样性分析。 采用QIIME 8.1软件生成不同分类水平上的物种丰度表,采用R语言绘制分类学水平下的群落结构图。根据样品物种丰度变化情况,进行斯皮尔曼(Spearman)秩相关性分析,并筛选相关性>0.1且P<0.05的数据构建物种相关性网络图。
2 结果与分析
2.1 不同酒曲发酵酒醅的真菌菌群高通量测序结果及Alpha多样性分析
采用Illumina MiSeq高通量测序技术对4种不同酒曲发酵酒醅共12个样品进行高通量测序,结果共得到1 440 219对Reads,经质控、拼接后共得到667 517条Clean Reads,每个样品最少产生6 734条Clean Reads,平均产生55 626条Clean Reads。 在相似度97%的水平上共聚类得到101个OTUs,采用QIIME2 2020.6计算α多样性指数,结果见表1。 由表1可知,4种酒醅样品的真菌菌群测序结果的覆盖率均>97%,说明测序深度合理,结果能反应酒醅样品中真菌菌群的真实情况。在真菌OTUs数目上,WQ>ZY>MJ>AQ,即真菌菌群物种数目最高的是WQ发酵酒醅,其次是ZY发酵酒醅,最低的为AQ发酵酒醅。在Shannon指数上,AQ>MJ>ZY>WQ,说明AQ发酵酒醅中的物种多样性最高,其次是MJ发酵酒醅,物种多样性最低的为WQ发酵酒醅。 在Chao1指数上,WQ>MJ>ZY>AQ,说明WQ发酵酒醅的丰富度最高,MJ发酵酒醅次之,最低的为AQ发酵酒醅,Chao1指数的结果与OTU的结果相似,进一步证明了WQ、MJ中微生物群落的种群多样性和总体丰度较高,推测可能是不同酒曲的菌种、原料、制作工艺等不同导致其微生物多样性也不尽相同。
表1 4种不同酒曲发酵酒醅样品中真菌菌群操作分类单元数及α多样性指数
Table 1 Operational taxonomic unit number and the diversity index of fungi flora in fermented grains of 4 different Jiuqu
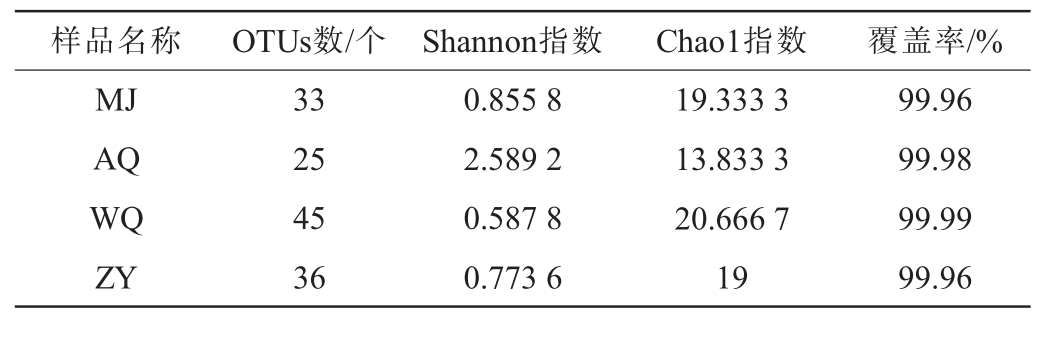
样品名称 OTUs数/个 Shannon指数 Chao1指数 覆盖率/%MJ AQ WQ ZY 33 25 45 36 0.855 8 2.589 2 0.587 8 0.773 6 19.333 3 13.833 3 20.666 7 19 99.96 99.98 99.99 99.96
为了验证测序数据量是否足以反映样品中真菌菌群信息,绘制4种不同酒曲发酵酒醅样品的真菌菌群稀释曲线,结果见图1。由图1可知,随着测序量的升高,所有样品的稀释曲线均呈先急剧升高后趋于平缓的趋势,说明群落中有大量物种被发现,趋于平缓后环境中的物种并不会随测序数量的增加而显著增多,表明测序结果能满足后续分析要求。 AQ发酵酒醅样品的在测序数量达5 000之前就不再增加,这可能是AQ发酵酒醅样品中至少有一个样品测序量不足10 000导致的。
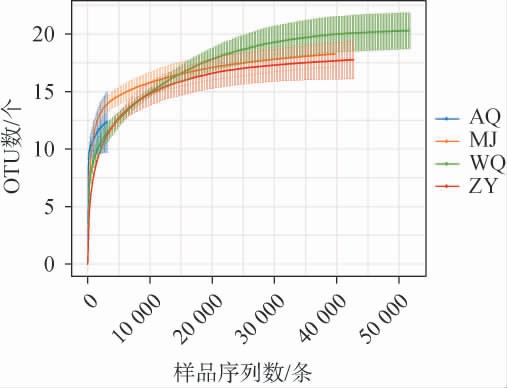
图1 4种不同酒曲发酵酒醅样品真菌菌群的稀释曲线
Fig.1 Dilution curve of fungal flora in fermented grains of 4 different Jiuqu
为对4种酒曲发酵酒醅样品中的真菌群落组成差异进行比较,基于OTUs数绘制韦恩图,结果见图2。
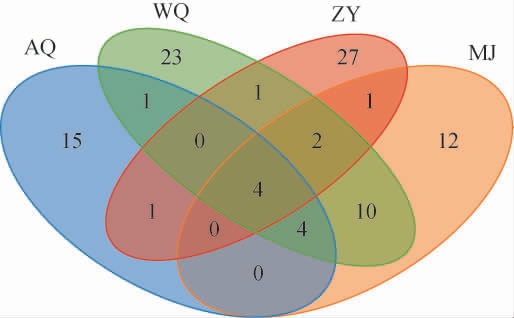
图2 4种不同酒曲发酵酒醅样品中真菌菌群操作分类单元数的韦恩图
Fig.2 4 Venn diagram of operational taxonomic units of fungi flora in fermented grains of 4 different Jiuqu
由图2可知,在4种不同酒曲发酵酒醅样品中共有的真菌OTUs数为4个,其中ZY发酵酒醅中特有的真菌OTUs数为27个,MJ发酵酒醅中特有的真菌OTUs数最低,为12个。结果表明,不同酒曲发酵酒醅间真菌群落组成存在一定特异性,真菌物种的数量受酒曲种类、制备工艺、原辅料等影响较大。
2.2 不同酒曲发酵酒醅真菌菌群结构分析
2.2.1 基于门水平不同酒曲发酵酒醅的真菌群落结构分析
基于Illumina Miseq高通量测序从4个不同酒曲发酵酒醅样品的真菌菌群中共注释到5个门、24个属。在门水平上,不同酒曲发酵酒醅样品的真菌菌群结构见图3。 由图3可知,不同酒曲发酵酒醅的优势真菌门(相对丰度>1%)均为毛霉门(Mucoromycota)和子囊菌门(Ascomycota),且两者在不同酒曲发酵酒醅中占绝对优势地位,相对丰度之和均>99%。子囊菌为多种香型酿造酒醅的主要真菌[26];毛霉门在小曲酒发酵过程中有一定的糖化力作用[27]。
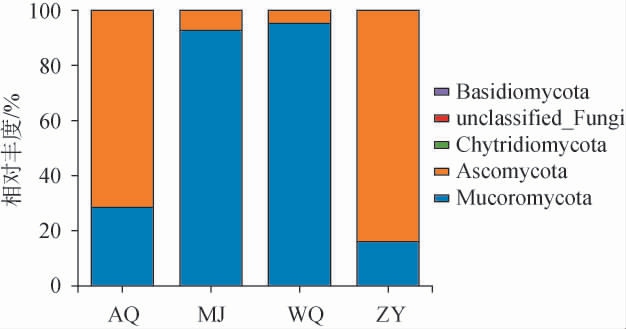
图3 基于门水平4种不同酒曲发酵酒醅样品的真菌群落结构
Fig.3 Fungal community structure of fermented grains of 4 different Jiuqu based on phylum level
2.2.2 基于属水平不同酒曲发酵酒醅的真菌群落结构分析
在属水平上,4种不同酒曲发酵酒醅样品的真菌菌群结构见图4。由图4可知,4种不同酒曲发酵酒醅样品的共同优势真菌属(相对丰度>1%)为根霉属(Rhizopus)和未分类属(unidentified);根霉属(Rhizopus)是常见真菌之一,可分泌多种水解酶,具有糖化和发酵且产生乙酸乙酯等代谢成分的能力[28]。 此外,AQ发酵酒醅样品中的优势真菌属还包括未分类酵母目(unclassified_Saccharomycetales)(44.90%)、假丝酵母属(Candida)(6.01%)、曲霉属(Aspergillus)(2.00%)、毕赤酵母属(Pichia)(1.39%),且毕赤酵母属(Pichia)为AQ发酵酒醅样品中的特有优势真菌属。毕赤酵母属为耐高温酵母菌,主要在酒醅发酵过程中产乙醇和产酯[29]。曲霉属(Aspergillus)为酱香型白酒酒醅和大曲的优势菌属,能产生多种酶类,可调控大曲和酒醅的糖化力、酯化力、液化力,是公认的功能菌[30];同时其可通过代谢有机酸、脂肪酸,促进芳香酯类物质的生成,以提升和改善酒体风味[31]。MJ发酵酒醅样品中的优势真菌属还包括Cyberlindnera(3.88%)、Aspergillus(92.63%)、酵母菌属(Saccharomyces)(2.42%),其中Cyberlindnera和Saccharomyces是其特有的优势真菌属。 WQ发酵酒醅样品中的优势真菌属还包括Aspergillus(95.24%)、红曲霉属(Monascus)(1.86%)、横梗霉属(Lichtheima)(1.86%)。 ZY发酵酒醅样品中的优势真菌属还包括Lichtheimia(6.77%)、Monascus(2.70%)、Candida,其中横梗霉属(Lichtheimia)与芳香类物质呈显著正相关[32];而Candida则是酱香型白酒堆积和窖池发酵酒醅优势真菌属,对提升酒体品质具有重要作用[33]。结果表明,不同酒曲发酵酒醅样品间真菌菌群存在较大差异,可能与酒曲原料自身含有的微生物以及酒曲制作工艺等有关,未分类菌(unclassified)和未注释到属的未知菌在酒醅样品中占比较高,表明发酵是一个微生物种属复杂变化的过程,对于未知菌属的确定还有待深入研究。
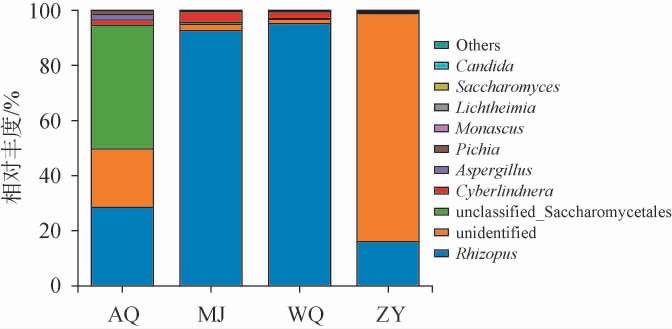
图4 基于属水平4种不同酒曲发酵酒醅样品的真菌群落结构
Fig.4 Fungal community structure of fermented grains of 4 different Jiuqu based on genus level
Others:平均相对丰度排名十之后的真菌属。
2.3 不同酒曲发酵酒醅的真菌群落差异分析
基于优势真菌属的相对丰度对不同酒曲发酵酒醅的真菌菌群进行PCoA,结果见图5。
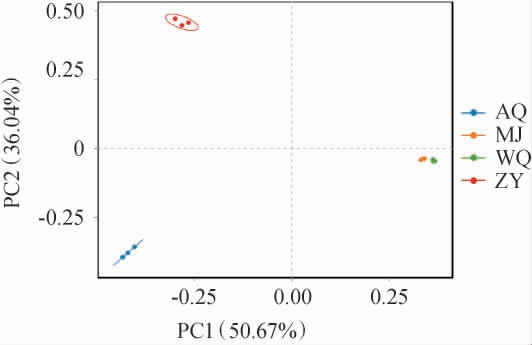
图5 基于优势真菌属相对丰度4种不同酒曲发酵酒醅样品真菌群落的主坐标分析结果
Fig.5 Results of principal coordinates analysis of fungal community of fermented grains of 4 different Jiuqu based on relative abundance of dominant fungi genera
由图5可知,第一主成分的方差贡献率为50.67%,第二主成分的方差贡献率为36.04%,前两个主成分的累计方差贡献率为86.71%,说明两主成分可解释不同酒曲发酵酒醅过程中真菌菌群变化的86.71%。 MJ和WQ发酵酒醅样品距离较为接近,而与AQ、ZY发酵酒醅样品距离较远,说明MJ和WQ发酵酒醅样品中的真菌群落结构相似,这可能与MJ和WQ发酵酒醅样品在属水平上均有曲霉属(Aspergillus)有关,且曲霉属(Aspergillus)在两样品中相对丰度均>90%。AQ、ZY发酵酒醅样品中的真菌群落结构差异较大。
2.4 不同酒曲发酵酒醅样品中真菌菌群相关性分析
基于4种不同酒曲发酵酒醅样品中真菌属的平均相对丰度进行相关性分析,选取相关性较高的前80个真菌属绘制相关性网络图,结果见图6。
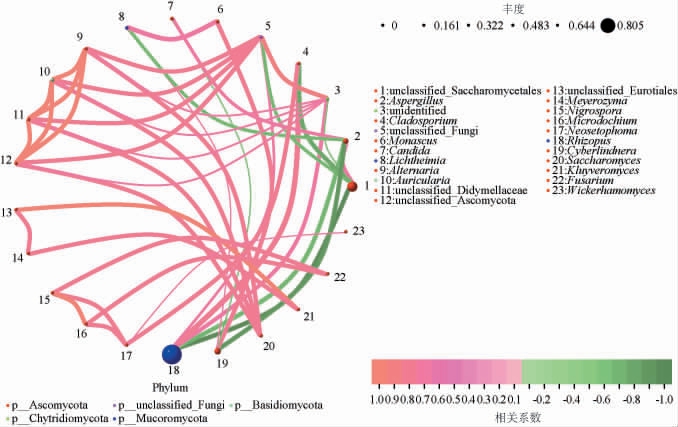
图6 基于属水平4种不同酒曲发酵酒醅样品中真菌菌群相关性网络图
Fig.6 Network plot of fungal flora correlation of fermented grains of 4 different Jiuqu based on genus level
由图6可知,网络图中共有23个节点属,41条边,说明4种不同酒曲发酵酒醅中微生物群落有着复杂的生物学过程。其中,unclassified-Fungi、unidentified、Rhizopus、Cyberlindnera连接节点最多,说明这4个真菌属与其他真菌属联系紧密。4种不同酒曲发酵酒醅样品的共同优势真菌属为Rhizopus,除与Candida、Cyberlindnera以及部分未分类菌属呈负相关外,与其余的真菌属均呈正相关,有文献报道,通过网络中微生物间的相互作用可以推测呈正相关微生物处于互利共生关系,呈负相关的则处于竞争关系,另外,呈正相关的真菌属可能还是酒醅关键物种[34]。 结果说明,Rhizopus与Candida、Cyberlindnera以及部分未分类菌属呈互利共生关系,与其余真菌属处于竞争关系,是酒醅中的关键微生物。 此外,Candida与unidentified、Cladosporium与Rhizopus、Cyberlindnera 与Rhizopus、Monascus 与unclassified_Saccharomycetales的相关性分别为0.770、0.707、0.671、0.662,说明以上两两真菌属间的相关性强。 马琳娜等[35]对不同酒曲酿造黄酒中挥发性风味物质及菌群结构进行分析,发现不同酒曲黄酒特征风味物质组成存在一定差异,不同黄酒酒曲优势微生物与特征风味物质形成有着较好的相关性。
3 结论
基于Illumina MiSeq高通量测序对4种不同酒曲发酵酒醅的真菌群落进行分析,Alpha多样性分析结果表明,AQ发酵酒醅的物种多样性最高,其次为MJ发酵酒醅,WQ发酵的酒醅的物种多样性最低,可能是不同酒曲的菌种、原辅料、制作工艺等不同所致。菌群结构分析表明,从酒醅样品中共注释到5个真菌门和24个真菌属。在门水平上,4种不同酒曲发酵的酒醅真菌优势真菌门(相对丰度>1%)均为Mucoromycota和Ascomycota;在属水平上,4种不同酒曲发酵酒醅的共同优势真菌属为Rhizopus和unidentified,AQ发酵酒醅中的优势真菌属还包括unclassified_Saccharomycetales、Candida、Aspergillus、Pichia;MJ发酵酒醅中优势 真 菌 属 包 括Cyberlindnera、Aspergillus、Saccharomyces;WQ发酵酒醅的中优势真菌属包括Aspergillus、Monascus、Lichtheima;ZY发酵酒醅中优势真菌属包括Lichtheimia、Monascus、Candida。 PCoA结果表明,MJ和WQ发酵酒醅的真菌菌群结构相似,但与AQ、ZY发酵酒醅差异较大。 相关性分析结果表明,unclassified-Fungi、unidentified、Rhizopus、Cyberlindnera与其他物种联系紧密。 本研究为后续探究苗家米酒风味物质形成与微生物群落特征之间的关系提供一定的科学依据,还可为将来筛选苗家米酒酒酿造的优势微生物菌株及酒曲制作奠定基础。
[1]肖冬光.白酒生产技术[M].北京:化学工业出版社,2010:3-4.
[2]WANG B W,WU Q,XU Y,et al.Specific volumetric weight-driven shift in microbiota compositions with saccharifying activity change in starter for Chinese Baijiu fermentation[J].Front Microbiol,2018,9:2349.
[3]ZHANG Y D, XU J G, DING F, et al.Multidimensional profiling indicates the shifts and functionality of wheat-origin microbiota during hightemperature Daqu incubation[J].Food Res Int,2022,156:111191.
[4]赵蒙,孙安,谭功全.白酒酒醅检测技术的研究进展与展望[J].食品科技,2022,47(12):250-257.
[5]邱显平,黄桥,杨静,等.浓香型白酒在新、老窖池发酵过程中酒醅微生物群落结构差异分析[J].中国酿造,2024,43(1):50-56.
[6]王清龙,朱甜甜,刘延波,等.白酒生产不同酒曲微生物群落结构、理化指标及挥发性风味物质研究[J].中国酿造,2023,42(7):93-102.
[7]黎江华,刘超兰,冯兴,等.高通量测序分析五倍子发酵用酒曲与酒糟微生物多样性[J].中成药,2020,42(8):2113-2118.
[8]蔡程山,白飞荣,许玲,等.国井白酒窖池发酵过程中酒醅微生物多样性及理化特性[J].中国酿造,2023,42(11):90-98.
[9]任海伟,孙一帆,王希,等.不同窖龄及位置浓香型白酒窖泥中细菌群落结构的差异性分析[J].食品与发酵工业,2023,49(9):103-111.
[10]谷晓东,刘怡琳,席晓丽,等.基于高通量测序技术对6种黄酒酒曲中微生物多样性的研究[J].食品工业科技,2022,43(16):148-157.
[11]李斌,陈亚楠,孙旭,等.Illumina MiSeq高通量测序分析红谷黄酒酒曲微生物菌群多样性[J].中国酿造,2022,41(9):86-92.
[12]贾丽艳,田宇敏,王晓勇,等.传统清香型白酒发酵过程中真菌群落结构及其动态演替[J].中国食品学报,2020,20(10):309-314.
[13]李斌,闫志鹏,李慧星,等.基于高通量测序技术的浓香型和芝麻香型白酒酒曲细菌群落结构分析[J].中国酿造,2018,37(8):148-152.
[14]胡晓龙,冯大鸿,张勇,等.浓香型酒醅中梭菌群落演替及其与理化性质的相关性分析[J].食品与发酵工业,2023,49(5):94-100.
[15]陈进,陈萍,谭光迅,等.浓香型白酒酿造过程中酒醅微生物区系变化特征分析[J].酿酒科技,2023(8):17-23.
[16]邹恬杏,苏伟,母应春,等.洞酿酱香酒第四轮次酒醅发酵过程中微生物群落与挥发性代谢物分析[J].中国酿造,2023,42(6):64-71.
[17]宋建阳,梁莉莹,岑定运,等.浓香型白酒发酵过程中酒醅微生物群落结构解析及其与风味物质的相关性[J].食品研究与开发,2023,44(14):86-92.
[18]杨玉玲,李术钗,王佳佳,等.传统苗家米酒发酵工艺优化及挥发性风味物质分析[J].中国酿造,2023,42(11):237-242.
[19]BOKULICH NA,SUBRAMANIAN S,FAITH J J,et al.Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J].Nat Methods,2013,10(1):57.
[20]MAGOC T, SALZBERG S L.FLASH: Fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics,2011,27(21):2957-2963.
[21]BOLGERAM,LOHSE M,USADEL B.Trimmomatic:A flexible trimmer for Illumina sequence data[J].Bioinformatics,2014,30(15):2114-2120.
[22]EDGAR R C, HAAS B J, CLEMENTE J C, et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194-2200.
[23]EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nat Methods,2013,10(10):996-998.
[24]WANG Q,GARRITY G M,TIEDJE J M,et al.Naïve Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J].Appl Environ Microbiol,2007,73(16):5261-5267.
[25]SCHLOSS P D,WESTCOTT S L,RYABIN T,et al.Introducing mothur: Open-source, platform-independent, community-supported software for describing and comparing microbial communities[J].Appl Environ Microbiol,2009,75(23):7537-7541.
[26]张春林,杨亮,李哲,等.酱香型白酒二轮次堆积酒醅微生物群落与理化指标相关性研究[J].中国酿造,2021,40(11):31-36.
[27]DZURENDOVA S, LOSADA C B, DUPUY G, et al.Mucoromycota fungi as powerful cell factories for modern biorefinery[J].Appl Microbiol Biotechnol,2021,106:1-15.
[28]刘延波,魏蒙蒙,王琳琳,等.包包曲和平板曲挥发性风味成分及真菌群落结构研究[J].轻工学报,2022,37(5):12-23.
[29]黄永光.酱香型白酒酿造中Aspergillus hennebergii及其分泌酸性蛋白酶的研究[D].无锡:江南大学,2014.
[30]庄名扬,孙达孟.酱香型白酒高温堆积糟醅中酵母菌分离、选育及其分类学鉴定[J].酿酒,2003,30(2):12-13.
[31]申孟林,张超,王玉霞.白酒大曲微生物研究进展[J].中国酿造,2016,35(5):1-5.
[32]余婷婷,赖世强,曹文涛,等.酱香型白酒高温堆积糟醅中耐高温酵母的筛选及鉴定[J].酿酒科技,2014(6):24-26.
[33]王玉荣,葛东颖,蔡文超,等.低温大曲真菌菌群结构解析及其风味品质评价[J].食品与发酵工业,2023,49(14):229-234.
[34]杨玲,刘春,彭佳伟,等.山西老陈醋和怀仁醋酒精发酵阶段细菌菌群多样性分析[J].中国酿造,2023,42(2):63-69.
[35]马琳娜,邱树毅,王啸.不同黄酒酒曲的酿造微生物与风味物质之间的关系[J].食品与发酵科技,2021,57(3):81-89.