黄酮类化合物(flavonoids)是一类存在于自然界的、具有2-苯基色原酮(flavone)结构的化合物,其是植物中一类重要的次生代谢产物,具有显著的抗氧化作用[1]。目前已表征结构的黄酮类化合物超过10 000种[2],包括含有酚羟基的苯环和以硫代元素组成的B环等有机化合物,它们具有多种药物活性[3-7],茯苓薏湿茶、黄芩片、黄芩茎叶解毒胶囊等广泛用于药物开发应用和临床治疗[8-9]。
黄芩素主要存在于黄芩,因此而得名。 黄芩素具有降低脑血管阻力,改善脑血循环、增加脑血流量及抗血小板凝集的作用,临床用于脑血管病后瘫痪的治疗[10]。 研究表明,黄芩素的合成途径主要通过苯丙烷代谢网络将葡萄糖转化为L-苯丙氨酸,在苯丙氨酸解氨酶(phenylalanine ammonia lyase,PAL)的作用下转化为肉桂酸(cinnamic acid,CA)。 随后,相继通过辅酶A连接酶(coenzyme A ligase,CL)(4CL)、查尔酮合成酶(chalcone synthase CHS)、查尔酮异构 酶(chalcone isomerase CHI)、黄 酮 合 成 酶(flavone synthase,FNS)等一系列酶的作用下,转化为白杨素(chrysin)。最后,在黄酮6-羟化酶(flavonoid 6-hydroxylases,F6H)催化下,白杨素发生羟基化生成黄芩素(baicalein)[11-13]。
RODRIGUEZ A等[14]研究团队通过工程改造酿酒酵母(Saccharomyces cerevisiae)的代谢途径和引入相关基因,使其能够直接从葡萄糖中生产六种不同的黄酮类化合物。JI D等[15]在大肠杆菌(Escherichia coli)中设计并组装了一条人工合成途径,成功实现了从前体L-苯丙氨酸及L-酪氨酸到黄芩素的异源合成,黄芩素的产量达到了23.6 mg/L,进一步采用双酶自组装的方式,将黄芩素的产量得到了数倍的提升。LARSSON S等[16]在酿酒酵母中PAD1基因的表达导致菌株将肉桂酸分解成对香豆酸。YAN Y等[17]在酿酒酵母中重构了四基因的基因簇,使大肠杆菌中的乔松素产量达到了37.3 mg/L,相比于HWANG E I等[18]在大肠杆菌中的结果,其乔松素产量提高了22倍。同时据报道,内源性乙酰基辅酶A合成酶(acyl-CoA synthetase,ACS1/ACS2)已被用于丙烷脱氢(propane dehydrogenation,PDH)旁路建立[19-20]。CHEN F等[21]报告了ACS1或ACS2基因在酿酒酵母中的过表达使细胞内的乙酰辅酶A(acetyl coenzyme A,acetyl-CoA)水平增加了2~5倍。JONG-GUBBELS D P等[22]报道称,在酿酒酵母细胞中过表达这两种乙酰辅酶A合成酶(ACS)同工酶,酶活性增加了3~6倍。WILLIAMS I S等[23]在酿酒酵母中重组表达了细胞色素酶P450家族成员1A1重组蛋白(CYP1A1),对白杨素进行选择性的C6羟基化,得到了黄芩素。LIU X等[24]在酿酒酵母中组装了白杨素合成途径,该底盘细胞整合有PAL1、4CL、CHS、CHI、FNS等5种基因,可以苯丙氨酸作为直接原料来合成白杨素,黄芩素产量达到10.06 mg/L。尽管已经可以体外异源合成黄芩素,但合成的效率和产量较低,难以实现规模生产。
本研究为了提高黄芩素产量,在前期构建的酿酒酵母白杨素合成底盘细胞Ch-9基础上,进一步延伸黄芩素合成途径,并对途径中关键基因进行优化改造,将来自拟南芥的辅酶A连接酶(4CL)突变体4AT及同工酶CLL-7基因,整合到肉桂酸脱羧酶PAD1的基因位点,通过同源重组实现基因置换敲除。 将来自苹果的烯脂酰还原酶基因MdECR整合替换到内源性酰烯还原酶的基因DBR位点,同时过表达乙酰辅酶A合成酶的基因ACS来增强乙酰辅酶A的供给。整合基因SbaiCYP82D4,将合成终点延伸到黄芩素,为黄酮类化合物的生产发酵奠定良好基础。
1 材料与方法
1.1 材料与试剂
酿酒酵母白杨素底盘细胞Ch-9、载体Y22(YCplac22)-ATR2-SbaiCYP82D4、质粒Y22-ATR2-SbaiCYP82D4:天津工业生物技术研究所;载体pUC57:武汉金开瑞生物工程有限公司;PHBM368载体:湖北大学;SbaiCYP82D1.1基因序列、SbaiCYP82D4基因序列、ACS基因序列来自文献[21]报道其中SbaiCYP82D1-1基因由金开瑞公司合成并针对酿酒酵母进行密码子优化;TSC13基因序列来自美国国家生物技术信息中心(National Center of Biotechnology Information,NCBI)(NM_001180074.1);MdECR基因序列来自NCBI(XM_008384596.3);本研究所用引物、质粒及菌株分别见表1~表3。
表1 本研究所用引物
Table 1 Primers used in the study

引物 序列(5'-3')CYC1-4AT-F PGK1-4AT-R TDH3-CLL7-F TDH1-CLL7-R Y22反扩-F Y22反扩-R pUC57反扩-F pUC57反扩-R CYC1-MdECR-F PGK1-MdECR-R TDH3-ACS-F TDH1-ACS-R Y22-ATR2-R Y22-ATR2-F 4AT-F 4AT-R MdECR-F MdECR-R CYCI-F PGK1-R AACTAATTACATGAGCGGCCTTATAAACCATTAGCTAACTTAGCCCTC CAAATATAAAAACAGCGGCCATGGCTCCACAAGAACAAG CACATAAACAAACAAACATAATGGAAAAATCTGGTTATGGTAGAG ATCAAGATTGCTTTATCATATTACAATTTAGATCTAACTTTTTCAATTAATTCTCTTC GAAGACAGCCAAAACGCTTAACATGTTATG AACTACGTACGCAATTTGGACTTTCG ATCGGATGCCGGGACCGAC ATCTCGACGCATTCGCGAAGTAC AACTAATTACATGAGCGGCCTTACAAAAATGGTGGCAAAATAACCCATCTTC CAAATATAAAAACAGCGGCCATGAAGGTTACTGTTGTTTCTAGGTCTGG CACATAAACAAACAAACATAATGAGTCAGACCCATAAACATGCC ATCAAGATTGCTTTATCATATTAACTCGGCATTGCAATTGCCTG TGACATAACTAATTACATGATTAATATAAAGTTGGAGATAATCTTGGAGTCAAAATAG TTACAACAAATATAAAAACAATGGAATTGTCTTCTGTTATCTACGGTGC AACTAATTACATGAGCGGCCTTATAAACCATTAGCTAACTTAGCCCTC CAAATATAAAAACAGCGGCCATGGCTCCACAAGAACAAG ATAAAGCAATCTTGATGAGGATAATG TTTGTTTGTTTATGTGTGTTTATTCGAAAC TGTTTTTATATTTGTTGTAAAAAGTAGATAATTACTTCC TCATGTAATTAGTTATGTCACGCTTAC
表2 本研究所用质粒
Table 2 Plasmids used in the study

质粒 特征YCplac22(Y22)Y22-ATR2-SbaiCYP82D4 pUC57 pUC57-4AT pUC57-CLL7 Y22-4AT Y22-CLL7 Y22-4AT-CLL7 pUC57-PAD1-4AT-CLL7 PHBM368(PGK)pUC57-MdECR pUC57-ACS Y22-MdECR Y22-ACS Y22-MdECR-ECR pUC57-TSC13-MdECR-ACS Y22-ATR2-SbaiCYP82D1.1 Centromeric vector,TRP1,AmpR YCplac22 vector,pPGK1-SbarCYP82D-4-tCYC1/pTDH3-ATR2-TDH1 pUC-based Cloning Vector,AmpR pUC57 vector,4AT,AmpR pUC57 vector,CLL-7,AmpR YCplac22 vector,pPGK1-4AT-tCYC1/pTDH3-ATR2-TDH1 YCplac22 vector,pPGK1-SbarCYP82D-4-tCYC1/pTDH3-CLL7-TDH1 YCplac22 vector,pPGK1-4AT-tCYC1/pTDH3-CLL7-TDH1 pUC57 vector,S3,pPGK1-4AT-tCYC1/pTDH3-CLL7-TDH1,TRP1,S4 PHBM368-Based expression vector,PPGK,S,CYC1-TT,AmpR,URA3 pUC57 vector,MdECR,AmpR pUC57 vector,ACS,AmpR YCplac22 vector,pPGK1-MdECR-tCYC1/pTDH3-ATR2-TDH1 YCplac22 vector,pPGK1-SbarCYP82D-4-tCYC1/pTDH3-ACS-TDH1 YCplac22 vector,pPGK1-MdECR-tCYC1/pTDH3-ACS-TDH1 pUC57 vector,S2,pPGK1-MdECR-tCYC1/pTDH3-ACS-TDH1,URA3,S1 YCplac22 vector,pPGK1-SbarCYP82D1.1-tCYC1/pTDH3-ATR2-TDH1
表3 本研究所用菌株
Table 3 Strains used in the study

菌株编号 基因型Ch-9 BA-C1 BA-C2 BA-C3 BA-C4 BA-B1 BA-B2 L11 L12 L13 L14 L15 L16 W303 YORWΔ 17::his3/pADH1-Eb4CL-TPI1t/pHXT7-EbCHS-tTPG1/pPGI1-EbCHI-tADH1/pRPL8A-EbFSII-tCYC1/pADH1-EbPAL-tFBA1/pTDH3-SbCLL-7-tPDC1 Ch-9 Carrying Y22-4AT-CLL7 Ch-9 PAD1Δ::pPGK1-4AT-tCYC1/pTDH3-CLL7-TDH1/TRP1 Ch-9 Carrying Y22-MdECR-ACS Ch-9 TSC13Δ::pPGK1-MdECR-tCYC1/pTDH3-ACS-TDH1/URA3 Ch-9 Carrying Y22-SbarCYP82D-4-ATR2 Ch-9 Carrying Y22-SbarCYP82D1.1-ATR2 pHBM368-pTDH3-ACSseL641P-tTDH1-Trp pHBM368-tCYC1-mPanK1CoA-pPGK1-Trp pHBM368-pTDH3-ACC1-tTDH1-Trp pHBM368-tCYC1-ACSseL641P-pPGK1-pTDH3-ACSseL641P-tTDH1-Trp pHBM368-tCYC1-mPanK1CoA-pPGK1-pTDH3-ACSseL641P-tTDH1-Trp pHBM368-tCYC1-mPanK1CoA-pPGK1-pTDH3-ACC1-tTDH1-Trp
大肠杆菌培养采用LB肉汤培养基、LB琼脂培养基:武汉友名生物技术有限公司。121 ℃灭菌15 min。
酿酒酵母培养采用酵母浸出粉胨葡萄糖(yeast peptone dextrose,YPD)培养基:2%蛋白胨,1%酵母粉,2%葡萄糖。121 ℃灭菌15 min。
酵母浸出粉胨葡萄糖腺嘌呤(yeast peptone dextrose adenine,YPDA)培养基:YPD培养基中添加120 mg/L的腺嘌呤。108 ℃灭菌30 min。
大肠菌株筛选采用含氨苄青霉素LB肉汤培养基:LB肉汤培养基中添加氨苄青霉素(ampicillin,Amp),使其终体积分数为100 μL/100 mL。
酿酒酵母筛选采用完全培养基减去相应的氨基酸制备成营养缺陷型培养基(CM-HIS-TRP/CM-HIS-URA):CM培养基减去组氨酸(histidine,His)、色氨酸(tryptophan,Trp)或尿嘧啶(uracil,Ura)。108 ℃灭菌30 min。
1.2 仪器与设备
LC3100高效液相色谱(high performance liquid chromatography,HPLC)仪:安徽皖仪科技股份有限公司;FE28pH计:梅特勒-托利多仪器有限公司;UV1780紫外分光光度计:日本岛津公司;eBL-100 LED蓝白双光源照胶投射仪:苏州东胜兴业科学仪器有限公司;ZF-288凝胶成像系统:上海嘉鹏科技有限公司;Read Max1900型酶标仪:上海闪谱生物科技有限公司;T100聚合酶链式反应(polymerase chain reaction,PCR)仪:美国BIO-RAD公司。
1.3 方法
1.3.1 样品前处理
对改造菌株使用YPD培养基30 ℃、220 r/min摇瓶发酵84 h,得到发酵液。在超净工作台中使用移液枪取1 mL发酵液样品于EP管中,5 000 r/min离心3 min,取上清300 μL至新的1.5 mL离心管,去除剩余上清液,使用1 mL 1 mol/L磷酸盐缓冲液(phosphate buffer saline,PBS)重悬菌体,然后4 ℃、5 000 r/min条件下离心3 min;去上清,用1 mol/L的PBS重悬菌体,然后4 ℃、5 000 r/min条件下离心3 min;去掉上清,加入1 mL体积分数为70%甲醇重悬菌体;向上清液加入700 μL的甲醇,混匀;将样品放入超声破碎仪中充分振荡30min提取发酵液的黄酮类化合物成分,超声条件为4000Hz,超声时间为30 min,使用0.22 μm尼龙滤膜过滤,所得上清液用于HPLC检测[25]。
1.3.2 质粒的构建
为了构建质粒pUC57-4AT-CLL7,使用引物CYC1-4AT-F/PGK1-4AT-R、TDH3-CLL7-F/TDH1-CLL7-R分别将含有基因片段4AT、CLL-7的质粒pUC57-4AT、pUC57-CLL7扩增,得到4AT、CLL-7基因片段,且每个引物与相邻片段之间具有同源互补序列,用于连接构建质粒。通过Gibson连接将4AT、CLL-7基因片段和线性化载体pUC57组装,构建了质粒pUC57-4AT-CLL7。同理,首先使用引物CYC1-MdECRF/PGK1-MdECR-R、TDH3-ACS-F/TDH1-ACS-R通过扩增获得MdECR、ACS基因片段,以及载体片段pUC57,且基因片段带有相邻片段以及载体片段pUC57的同源臂,通过Gibson连接pUC57载体片段与MdECR、ACS基因片段,构建质粒pUC57-MdECR-ACS,同时将载体片段pUC57与ACS基因片段进行Gibson连接,构建质粒pUC57-ACS,然后以质粒Y22-ATR2-SbaiCYP82D4为模板,使用引物Y22-ATR2-R/Y22-ATR2-F进行PCR扩增,得到Y22-ATR2的基因长链,并带有SbaiCYP82D1.1的同源臂,再通过Gibson连接得到质粒Y22-ATR2-SbaiCYP82D1.1。
1.3.3 同源重组实现基因置换敲除
将10 μL pUC57-4AT-CLL7质粒转入酿酒酵母白杨素合成底盘细胞Ch-9的核糖体脱氧核糖核酸(ribosomal deoxyribonucleic acid,rDNA)位点中,按照酵母化学转化的方法进行转化,筛选培养基使用组氨酸的营养缺陷型培养基(CM-HIS),对长出菌落的重组子提取基因组脱氧核糖核酸(deoxyribonucleic acid,DNA),使用引物对4AT-F/4AT-R进行菌落PCR,并对鉴定正确的重组子接种到YPD培养基上进行二次培养,最终得到强化表达基因4AT-CLL7的酵母菌株BA-C1。同理再取10 μLpUC57-4AT-CLL7质粒转入白杨素合成底盘细胞Ch-9的PAD1基因的位点中,对筛选正确的菌株进行二次培养,得到将PAD1基因置换成4AT-CLL7基因的酵母菌株BA-C2。将pUC57-ACS质粒转入到菌株BA-C2中的核糖体脱氧核糖核酸(rDNA),筛选培养基使用组氨酸、尿嘧啶的营养缺陷型培养基(CM-HIS-URA),使用引物对MdECR-F/MdECR-R进行菌落PCR验证,二次培养后得到将强化表达ACS基因的菌株BA-C3;同理将pUC57-MdECR-ACS质粒转入到BA-C2中的DBR基因的位点中,得到将DBR基因替换成MdECR基因,并强化表达ACS基因的酵母菌株BA-C4。
1.3.4 旁路途经相关菌株构建
针对合成途径中辅酶A、乙酰辅酶A及丙二酰辅酶A的供应及合成调控,分别克隆表达PanK、ACS、ACC1基因,通过构建上述酶基因的单独或不同组合的整合型表达载体:载体pHBM368-ACSseL641P、pHBM368-mPanK1CoA、pHBM368-ACC1、pHBM368-ACSseL641P-ACSseL641P、pHBM368-mPanK1CoA-ACSseL641P、pHBM368-mPanK1CoA-ACC1质粒,通过质粒上的rDNA重组标签,以菌株L0(白杨素底盘细胞CH-9)为基础,将外源基因同源重组至白杨素底盘细胞的rDNA位点,将得到的重组菌株编号为L0~L16,由于菌株L1~L10进行基因整合效果不佳,因此选取mPanK1CoA、ACSseL641P、ACC1、ACSseL641P-ACSseL641P、mPanK1CoA-ACSseL641P、mPanK1CoA-ACC1基因重组菌株L11、L12、L13、L14、L15、L16,来研究强化表达旁路相关基因对白杨素底盘细胞Ch-9的还原糖消耗、乙醇产量、乙酰辅酶A产量的影响。
1.3.5 黄芩素表达菌株构建
将Y22-ATR2-SbaiCYP82D4和Y22-ATR2-SbaiCYP82D1.1基因分别整合到上述的菌株BA-C4中,筛选培养基使用组氨酸、色氨酸的营养缺陷型培养基(CM-HIS-TRP),使用的验证引物为CYCI-F/PGK1-R,得到整合ATR2-SbaiCYP82D4基因、Y22-ATR2-SbaiCYP82D1.1基因的菌株BA-B1、BA-B2。
1.3.6 分析检测
白杨素含量测定采用HPLC法[21]。HPLC色谱条件:使用Waters symmetryC18色谱柱(4.6 mm×250 mm,5 μm),最佳检测波长为210 nm,进样量60 μL,流速0.8 mL/min,柱温30 ℃。 洗脱条件为甲醇∶0.1%磷酸=70∶30(V/V),保留时间10 min。
定性定量方法:按照白杨素标准品保留时间定性。以白杨素标准品含量(x)为横坐标,峰面积(y)为纵坐标绘制白杨素标准曲线,得到标准曲线回归方程y=85.607x+97.814,相关系数R为0.999。按照标准曲线回归方程计算样品中白杨素含量。
黄芩素含量测定采用HPLC法[23]。使用Waters symmetry C18柱(4.6 mm×250 mm,5 μm),最佳检测波长为210 nm,进样量60 μL,流速1 ml/min,柱温30 ℃。 洗脱条件为乙腈∶0.1%磷酸水=30∶70(V/V),保留时间15 min。
定性定量方法:按照黄芩素标准品保留时间定性。 以黄芩素标准品含量(x)为横坐标,峰面积(y)为纵坐标绘制黄芩素标准曲线,得到标准曲线回归方程y=115x-27.469,相关系数R为0.999。按照标准曲线回归方程计算样品中黄芩素含量。

2 结果与分析
2.1 PAD1基因的敲除以及置换4AT-CLL7基因对白杨素
产量的影响
由于PAD1基因的表达会导致酿酒酵母菌株分解肉桂酸为对香豆酸,同时4AT-CLL7又是肉桂酸生成肉桂酰辅酶A的关键基因[26-27]。 因此,对酿酒酵母白杨素合成底盘细胞Ch-9进行了单独的4AT-CLL7基因强化表达,以及在PAD1基因位点进行了4AT-CLL7基因替换,其示意图见图1。
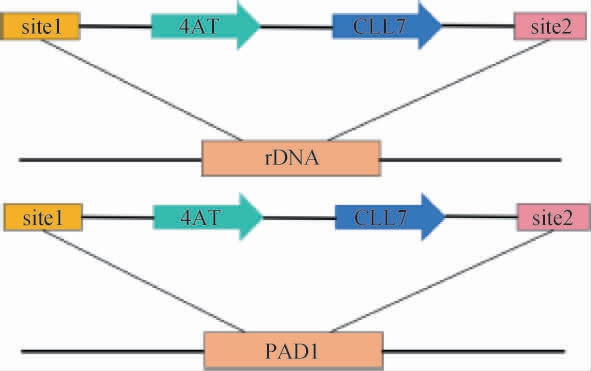
图1 基因4AT-CLL7转入rDNA及PAD1基因位点示意图
Fig.1 Schematic diagram of the transfer of gene 4AT-CLL7 into rDNA and PAD1 sites
通过强化表达4AT-CLL7基因,一共挑选了12株菌,编号为BA-C1-1~BA-C1-12,选取了其中4AT-CLL7基因整合情 况 较 好 的6株 菌(BA-C1-2、BA-C1-3、BA-C1-5、BA-C1-6、BA-C1-7、BA-C1-8),以及PAD1基因敲除置换4AT-CLL7基因一共12个菌株,编号为BA-C2-1~BA-C2-12,同样也选取了其中整合情况较好的6个菌株(BA-C2-1、BA-C2-2、BA-C2-3、BA-C2-4、BA-C2-6、BA-C2-7),同时以未同源重组的底盘细胞Ch-9作为对照组,用YPDA培养基进行摇瓶发酵,发酵84 h后,对上述的12个菌株的发酵液分别取样,采用高效液相色谱分析白杨素含量,结果见图2。
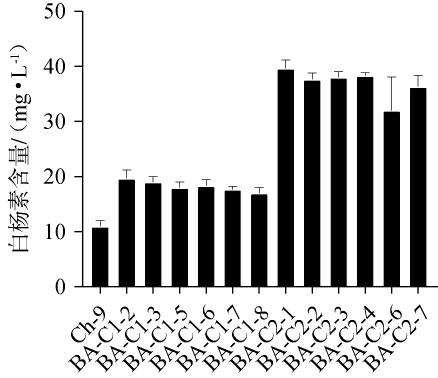
图2 PAD1基因的敲除及置换4AT-CLL7基因重组菌株发酵液中白杨素含量的检测结果
Fig.2 Determination results of chrysin contents in fermentation broth of recombinant strains with knockout PAD1 gene and replacement 4AT-CLL7 gene
由图2可知,在发酵84 h后,与对照组相比,无论是在上清液当中还是菌株中,6株强化表达4AT-CLL7基因菌株(BA-C1-2、BA-C1-3、BA-C1-5、BA-C1-6、BA-C1-7、BA-C1-8)白杨素含量均在不同程度上增加,其中菌株BA-C1-2发酵液白杨素含量达到了19.404 mg/L,相比对照组,白杨素提升了1.9倍。这一结果说明强化4AT-CLL7基因,有促于增加这一反应过程的通量,提高产物产量。
由图2亦可知,4AT-CLL7基因在PAD1基因位点的整合置换则显示了更好的结果,无论是在发酵上清液中还是菌株胞内,PAD1敲除置换4AT-CLL7基因菌株中6株菌株(BA-C2-1、BA-C2-2、BA-C2-3、BA-C2-4、BA-C2-6、BA-C2-7)在白杨素产量上均体现出了不同程度上的增加,其中菌株BA-C2-1的白杨素产量达到了40.210 mg/L,与对照组相比,提升了4倍。 结果表明,敲除PAD1基因后,肉桂酸的降解途径可能收到一定程度的抑制,更有利于产物(白杨素)的合成。
2.2 丙酮酸旁路途径优化
由于目前在酿酒酵母中进行乙酰辅酶A的工程化生产的报道很少,而PDH旁路又是酿酒酵母中提高乙酰辅酶A的关键路径[28-29],因此为了提高胞浆中的乙酰辅酶A水平,整合ACS、ACC1相关基因以及联合使用PanK基因至关重要[30-32],以L0(底盘细胞Ch-9)为原始菌株,利用重组的6株酿酒酵母菌株L11、L12、L13、L14、L15、L16作为实验组,通过检测发酵液的OD600nm值绘制菌的生长曲线,同时测定发酵液的pH、生物量、还原糖消耗、酒精度、乙酰辅酶A及发酵96 h白杨素产量,结果见图3。

图3 丙酮酸旁路途径优化结果
Fig.3 Results of acetoacetate bypass pathway optimization
由图3A可知,菌株L14、L15、L16的发酵OD600 nm值、生物量均高于原始菌株(L0),表明单独导入ACS基因以及联合导入ACS-PanK、PanK-ACC1基因对酿酒酵母菌体生长有显著促进作用。由图3B和图3C可知,L11~L16的葡萄糖消耗速率略微增加,乙醇产量均有一定程度的降低,其中,菌株L11与菌株L15乙醇产量较低,大约为0.13 mmol/L,对比L0菌株大约降低了0.04 mmol/L,表明导入ACS基因以及同时导入ACS-PanK基因显著增加发酵过程中乙醇的消耗。 由图3D可知,菌株L11、L12、L14、L15及L16发酵液中乙酰辅酶A含量均有显著提升,在L0的基础上分别增加了0.62倍、0.64倍、1.9倍、2.34倍及1.11倍。 表明导入ACS、PanK基因对乙酰辅酶A的产生有促进作用。由图3E可知,发酵84 h后菌株L11~L16发酵液白杨素产量增长较为明显,分别增长了0.33%、0.37%、0.43%、0.58%、0.8%、0.73%。 发酵84 h后强化表达ACC1基因的L13菌株产白杨素水平明显高于L11、L12菌株;联合导入ACS-PanK基因的菌株(L15)比联合导入ACS-ACS、Pank-ACC1基因的L14、L16菌株白杨素产量更高。
结果表明,对于单基因强化而言,过表达ACS基因的效果最为显著,多基因强化中联合表达ACS-PanK基因的效果最好,但是鉴于联合表达ACS-PanK基因对于过表达ACS基因的白杨素含量提升不是特别显著,而且在白杨素合成底盘细胞Ch-9中,联合表达ACS-PanK基因的拷贝数比较多,整合到rDNA位点的难度比较大,不易成功。 因此,选择在菌株BA-C2-1中过表达ACS基因来提高白杨素产量。
2.3 DBR基因的敲除置换与ACS基因的强化表达
在上述实验中已经通过对比证明了强化表达ACS基因对于提高PDH旁路途径中乙酰辅酶A的合成有明显的影响,与此同时,由于酵母细胞的产生内源性烯酰还原酶的基因DBR会导致合成途径中间产物肉桂酰CoA的损失。由于DBR基因是参与脂肪酸的合成必需的酶,不能被直接敲除。 因此在菌株BA-C2-1的rDNA位点进行ACS基因强化表达,结果见图4。 验证了12个重组菌株(BA-C3-1到BA-C3-12),选取了其中表达情况较好的6个菌株(BA-C3-4、BA-C3-5、BA-C3-7、BA-C3-8、BA-C3-9、BA-C3-10),同时在DBR基因位点进行了MdECR-ACS基因替换(图4),一共验证了12个菌株(BA-C4-12到BA-C4-24),选取了6株表达情况较好的菌株(BA-C4-17、BA-C4-18、BA-C4-20、BA-C4-21、BA-C4-22、BA-C4-23),同时以PAD1基因置换4AT-CLL7基因的菌株BA-C2-1作为对照组,用YPDA培养基在发酵84 h后,对上述选取的13组菌株的发酵液取样,采用高效液相色谱分析白杨素含量,结果见图5。
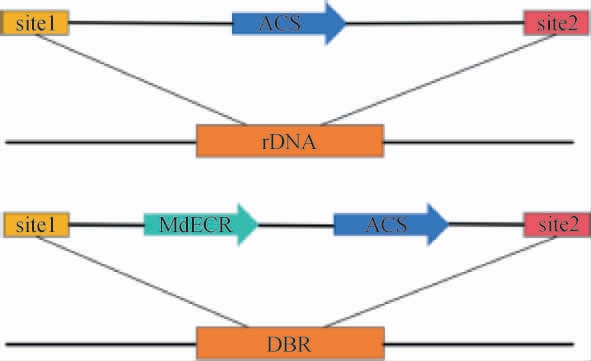
图4 DBR基因的敲除以及置换MdECR-ACS基因位点示意图
Fig.4 Schematic diagram of DBR gene knockout and substitution MdECR-ACS gene site
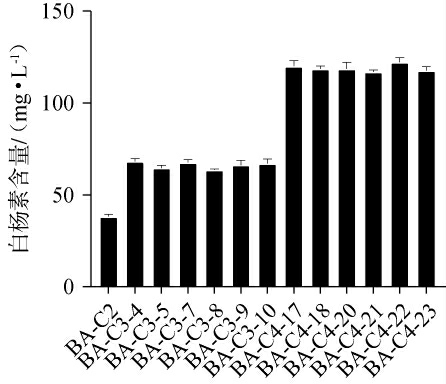
图5 DBR基因的敲除以及过表达ACS基因重组菌株发酵液中白杨素产量的检测结果
Fig.5 Determination results of chrysin contents in fermentation broth of recombinant strains with knockout DBR gene and overexpressing ACS gene
由图5可知,与对照组BA-C2-1相比,ACS基因的引入成功增加了乙酰辅酶A的供应,间接的增加了白杨素的产量。 6组实验组(BA-C3-4、BA-C3-5、BA-C3-7、BA-C3-8、BA-C3-9、BA-C3-10)白杨素的产量均体现出了不同程度上的增加,其中菌株BA-C3-7白杨素含量达到了70.252 mg/L,相比菌株BA-C2,提升了1.75倍。 这一结果说明了ACS基因的强化表达增加了乙酰辅酶A的胞内含量,从而提高了白杨素的产量。
由图5亦可知,6个实验组菌株(BA-C4-17、BA-C4-18、BA-C4-20、BA-C4-21、BA-C4-22、BA-C4-23)中白杨素均体现出了不同程度上的增加,其中菌株BA-C4-22的白杨素含量达到了125.044 mg/L,与对照组BA-C2-1相比,产量提升了3.125倍,这一结果说明了MdECR基因的引入,取代了原本的DBR基因,抑制了肉桂酰辅酶A的降解,使更多的中间产物更有效的转化为白杨素,同时,由于ACS基因的强化表达,使乙酰辅酶A的积累增多,这就导致了更多的肉桂酰辅酶A与乙酰辅酶A被CHS基因转化为下游产物。
2.3 F6H和ATR2基因的筛选和表达
在上述优化的基础上,进一步延长合成途径,研究将催化白杨素合成黄芩素的羟化酶F6H基因及产生其辅助电子传递的酶的基因ATR2导入到BA-C4-22菌株中。 为比较F6H的候选基因SbaiCYP82D4和SbaiCYP82D1.1的催化表达效果,分别构建了含有候选基因表达载体Y22-ATR2-SbaiCYP82D4的菌株BA-B1-1到BA-B1-12,选取了6个表达情况较好的菌株(BA-B1-1、BA-B1-4、BA-B1-5、BA-B1-6、BA-B1-7、BA-B1-8),以及含有候选基因表达载体Y22-ATR2-SbaiCYP82D1.1的菌株BA-B1-13到BA-B1-24,同样也选取了6个表达情况较好的菌株(BA-B2-18、BA-B2-20、BA-B2-21、BA-B2-22、BA-B2-23、BA-B2-24),采用摇瓶发酵方法,用YPDA培养基在发酵84 h后,取上述选取的12个菌株的发酵液,用高效液相色谱测量每个菌株的发酵液中黄芩素和白杨素含量,结果见图6。
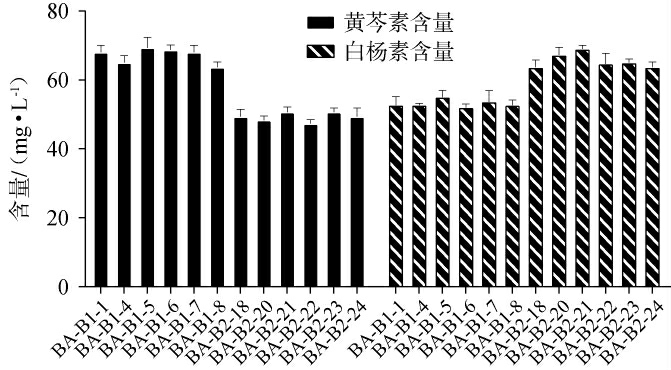
图6 导入SbaiCYP82D4和SbaiCYP82D1.1基因重组菌株中发酵液的黄芩素和白杨素产量检测结果
Fig.6 Determination results of baicalein and chrysin contents in fermentation broth of recombinant strains with delivery SbaiCYP82D4 and SbaiCYP82D1.1 gene
由图6可知,导入SbaiCYP82D4基因的6个菌株发酵液中白杨素的剩余含量在55.373 mg/L左右,其中BA-B1-5菌株的发酵液中白杨素剩余量达到了55.376 mg/L,而在导入SbaiCYP82D1.1基因的6个菌株发酵液中,白杨素剩余量大约在62.354 mg/L左右,其中BA-B2-21菌株发酵液中白杨素剩余量达到了68.729 mg/L。由此可见,导入SbaiCYP82D4基因的菌株比导入SbaiCYP82D1.1基因的菌株对于白杨素的利用量更多。
由图6亦可知,导入SbaiCYP82D4基因的6个菌株发酵液中黄芩素的含量在68.336 mg/L左右,其中BA-B1-5菌株的发酵液中黄芩素的含量最高,达到了70.938 mg/L。而在导入SbaiCYP82D1.1基因的6个菌株的发酵液中,黄芩素的含量在48.746 mg/L左右,其中菌株BA-B2-21发酵液中黄芩素含量最高,达到了51.134 mg/L。由此可见,对比导入SbaiCYP82D1.1基因的菌株,导入SbaiCYP82D4基因的菌株黄芩素的产量明显更多,黄芩素的转化效率也更高,达到了56.15%,因此,SbaiCYP82D4基因编码的羟化酶催化活性相对较好,也将BA-B1-5作为后续实验的菌株。
3 结论
本实验通过使用肉桂酰辅酶A-4CL的突变体4AT,以及4CL的同工酶CLL-7催化肉桂酸缀合,利用同源重组敲除了该酶的PAD1基因,同时强化表达4CL-CLL-7基因来增加肉桂酸的产生和利用。 经过发酵筛选,获得上述改造的酿酒酵母菌株BA-C2-1,白杨素摇瓶发酵产量为40.21 mg/L,比白杨素底盘细胞Ch-9提高4倍。引入MdECR基因来代替DBR基因,并通过强化表达ACS基因来提高乙酰辅酶A的含量,从而提高白杨素产量。经过发酵筛选,获得上述改造的酿酒酵母菌株BA-C4-22,白杨素产量达到了125.04 mg/L,在单纯利用4AT-CLL7基因替换PAD1基因的基础上,又将菌株BA-C2-1发酵液中白杨素含量提高了3.125倍。在BA-C4-22的基础上,再次引入了产生类黄酮6-羟化酶的基因F6H,以及产生细胞色素P450还原酶2的基因ATR2,成功将合成终点有白杨素拓展到了黄芩素,最终菌株BA-B1-5摇瓶发酵的黄芩素的产量到达了70.94 mg/L。
此外,除了对菌株进行上述代谢工程改造,也可以在此菌株其他的rDNA位点进行关键基因的强化表达,如通过荧光定量标记、或者利用Cas9技术来继续强化表达步骤中的关键基因,制备高活性羟基转移酶元件,并解析酶结构与功能之间的关系。
[1]PARMENTER B H, DALGAARD F, MURRAY K, et al.Habitual flavonoid intake and ischemic stroke incidence in the danish diet,cancer,and health cohort[J].Am J Clin Nutr,2021,114(1):348-357.
[2]DIXON R A, PASINETTI G M.Flavonoids and isoflavonoids: from plant biologyto agriculture and neuroscience[J].Plant Physiol,2010,154(2):453-457.
[3]CHEN Y C, SHEN S C, CHOW J M, et al.Flavone inhibition of tumor growth via apoptosis in vitro and in vivo[J].Int J Oncol, 2004, 25(3):661-700.
[4]WAY T D,KAO M C,LIN J K.Apigenin induces apoptosis through proteasomal degradation of HER2/neu in HER2/neu-overexpressing breast cancer cells via the phosphatidylinositol 3-kinase/Akt-dependent pathway[J].J Biol Chem,2004,279(6):4479-4489.
[5]SONG J W,LONG J Y,XIE L,et al.Applications,phytochemistry,pharmacological effects,pharmacokinetics, toxicity of Scutellaria baicalensis Georgi.and its probably potential therapeutic effects on COVID-19:a review[J].Chin Med,2020,15(1):102-112.
[6]ZHAO Q,ZHANG Y,WANG G,et al.A specialized flavone biosynthetic pathway has evolved in the medicinal plant,Scutellaria baicalensis[J].Sci Adv,2016,2(4):150-178.
[7]ALSAADA M,MOHANY M,ALMALKIM S,et al.Baicalein neutralizes hyperchol esterolemia-induced aggravation of oxidative injury in rats[J].Int J Med Sci,2020,17(9):1156-1166.
[8]KIM G,HAN D W,LEE J H.The cytoprotective effects of baicalein on H2O2-induced ROS by maintaining mitochondrial homeostasis and cellular tight junction in HaCaT keratinocytes[J].Antioxidants,2023,12(4):902.
[9]XIAO T, CUI Y, JI H, et al.Baicalein attenuates acute liver injury by blocking NLRP3 inflammasome[J].Biochem Biophys Res Commun,2021,534:212-218.
[10]全海燕,李亚军,孙琼,等.野黄芩素的药理作用研究进展[J].现代药物与临床,2025,40(1):234-241.
[11]ZHAO Q, CHEN X Y, MARTIN C.Scutellaria baicalensis, the golden herb from the garden of Chinese medicinal plants[J].Sci Bull,2016,61(18):1391-1398.
[12]NOEL J P, AUSTIN M B, BOMATI E K.Structure-function relationships in plant phenylpropanoid biosynthesis[J].Curr Opin Plant Biol,2005,8(3):249-253.
[13]YANG L,YANG C,LI C,et al.Recent advances in biosynthesis of bioactive compounds in traditional Chinese medicinal[J].Sci Bull,2016,61(1):3-17.
[14]RODRIGUEZ A,STRUCKO T,STAHLHUT S G,et al.Metabolic engineering of yeast for fermentative production of flavonoids[J].Bioresource Technol,2017,245(1):1645-1654.
[15]JI D,LI J,XU F,et al.Improve the biosynthesis of baicalein and scutellarein via manufacturing self-assembly enzyme reactor in vivo[J].ACS Synth Biol,2021,10(5):1087-1094.
[16]LARSSON S,NILVEBRANT N O,JNSSON L.Effect of overexpression of Saccharomyces cerevisiae Pad1p on the resistance to phenylacrylic acids and lignocellulose hydrolysates under aerobic and oxygen-limited conditions[J].Appl Microbiol Biotechn,2001,57(1-2):167-174.
[17]YAN Y, KOHLI A, KOFFAS M.Biosynthesis of natural flavanones in Saccharomyces cerevisiae[J].Appl Environ Microbiol, 2005, 71(9):5610-5613.
[18]HWANG E I,KANEKO M,OHNISHI Y,et al.Production of plant-specific flavanones by Escherichia coli containing an artificial gene cluster[J].Appl Environ Microbiol,2023,69(5):2699-2076.
[19]CHEN Y, DAVIET L, SCHALK M, et al.Establishing a platform cell factory through engineering of yeast acetyl-CoA metabolism[J].Metab Eng,2013,15(1):48-54.
[20]SHIBA Y, PARADISE E M, KIRBY J, et al.Engineering of the pyruvate dehydrogenase bypass in Saccharomyces cerevisiae for high-level production of isoprenoids[J].Metab Eng,2007,9(2):160-168.
[21]CHEN F, ZHOU J, SHI Z, et al.Effect of acetyl-CoA synthase gene overexpression on physiological function of Saccharomyces cerevisiae[J].Acta Microbiol Sin,2010,50(9):1172-1179.
[22]JONG-GUBBELS D P,BERG V M A,LUTTIK M A H,et al.Overproduction of acetyl-coenzyme A synthetase isoenzymes in respiring Saccharomyces cerevisiae cells does not reduce acetate production after exposure to glucose excess[J].FEMS Microbiol Lett,1998,165(1):15-20.
[23]WILLIAMS I S,CHIB S,NUTHAKKI V K,et al.Biotransformation of chrysin to baicalein:selective c6-hydroxylation of 5,7-dihydroxyflavone using whole yeast cells stably expressing human CYP1A1 enzyme[J].J Agr Food Chem,2017,65(34):7440-7446.
[24]LIU X,CHENG J,ZHU X,et al.De Novo biosynthesis of multiple pinocembrin derivatives in Saccharomyces cerevisiae[J].ACS Synth Biol,2020,9(11):3042-3051.
[25]YU K,PROOST P.Insights into peptidylarginine deiminase expression and citrullination pathways[J].Trends Cell Biol,2022,32(9):746-761.
[26]GEARY B,SUN B,TILVAWALA R R,et al.Peptidylarginine deiminase 2 citrullinates MZB1 and promotes the secretion of IgM and IgA[J].Front Immunol,2023,14:0.
[27]FREITAG M, SCHWERTZ H.A new role of NAP1L1 in megakaryocytes and human platelets[J].Int J Mol Sci,2022,23(23):14694.
[28]EJALONIBU M A, ELRASHEDY A A, LAWAL M M, et al.Probing the dual inhibitory mechanisms of novel thiophenecarboxamide derivatives against Mycobacterium tuberculosis PyrG and PanK:an insight from biomolecular modeling study[J].J Biomol Struct Dyn,2020,40:2978-2990.
[29]KHAN S,ALVI A F,SAIFY S,et al.The ethylene biosynthetic enzymes,1-aminocyclopropane-1-carboxylate (ACC) synthase (ACS) and ACC Oxidase(ACO): the less explored players in abiotic stress tolerance[J].Biomolecules,2024,14(1):90.
[30]LIU S Q,LIU C,LEI Z H,et al.Genome-wide analysis and identification of 1-aminocyclopropane-1-carboxylate synthase (ACS) gene family in wheat(Triticum aestivum L.)[J].Int J Mol Sci,2023,24:80-89.
[31]YOUSAF S,REHMAN T,TABASSUM B,et al.Genome scale analysis of 1-aminocyclopropane-1-carboxylate oxidase gene family in G.barbadense and its functions in cotton fiber development[J].Sci Rep,2023,13:4004.