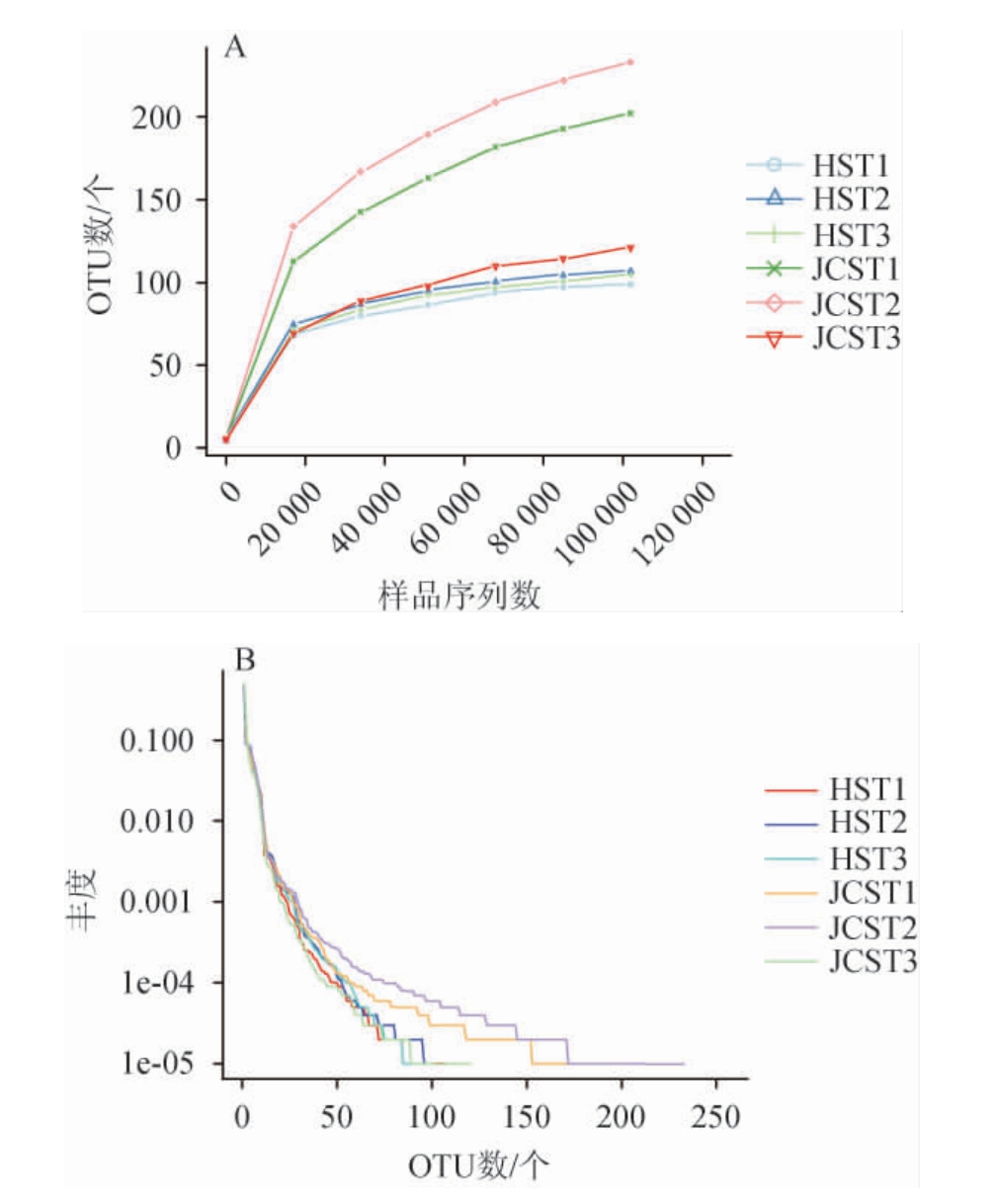
图1 细菌菌群的稀释曲线(A)及丰度等级曲线(B)
Fig.1 Dilution curve(A)and abundance grade curve(B)of bacterial flora
Analysis of microbial flora diversity of leek red sour soup in Guizhou based on Illumina Miseq high-throughput sequencing
酸汤是贵州黔东南地区侗族、苗族人民历经千年的传统发酵食品,因其原料和汤色不同,主要分为两大类,白酸汤和红酸汤[1]。白酸汤的主要原料为大米、糯米等,制作工艺为以米汤为原料,放入密封容器中发酵而成,汤色呈乳白色[2]。红酸汤制作的主要原料为红辣椒、西红柿等,制作工艺为红辣椒、番茄处理干净后剁成酱,加入适量的生姜、大蒜、木姜子以及熬制好的糯米汤后密封发酵而成,汤色呈橙红色[3]。白酸汤和红酸汤均主要作为火锅底料搭配牛肉、鱼肉、排骨、蔬菜等,富含矿质元素、有机酸、氨基酸和多种生物活性成分,是口感鲜美的健康食品[4-6]。
韭菜红酸汤作为红酸汤的一种,盛行于贵州省黔东南州麻江县当地百姓餐桌,其制作工艺是在红酸汤的原料基础上,额外加入韭菜密封发酵而成。蔬菜本身就含有有机酸,酸汤在发酵过程中也会产生有机酸,加入不同的蔬菜,有机酸种类和含量也会不同,从而形成不同的口感[7]。韭菜作为我国特有的药食两用的蔬菜,除含有丰富的胡萝卜素、维生素C(vitamin C,VC)以及矿质元素等营养成分外,韭菜根、叶中还富含挥发性含硫化合物、甾体皂苷、含氮化合物(凝集素、韭子碱甲等)和黄酮类物质,使红酸汤具有抗菌、抗氧化和温肾助阳等保健功能[8]。
为了探究这两种红酸汤微生物菌群结构的差异与联系,本研究采用Illumina Miseq高通量测序技术对韭菜红酸汤和红酸汤的微生物菌群结构进行分析,以期找到韭菜红酸汤中的优势菌群种类,有助于对贵州黔东南州麻江县特色韭菜红酸汤发酵过程进行精准调控和规模指导生产。
1.1.1 样品采集
红酸汤(HST)和韭菜红酸汤(JCST)样品:均采自贵州麻江县裕沃生态农业发展有限公司生产车间,每组样品各采集3个,将采集的样品立即装于密封器皿中,4 ℃短期保存。
1.1.2 试剂
高效植物基因组脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒、TsingkeI5High-FidelityMaster Mix:北京擎科生物科技股份有限公司;Monarch DNA Gel Extraction Kit:美国New England Biolabs公司;琼脂糖:西班牙Biowest公司;MiSeq Reagent Kit v3:美国Illumina公司;Invitrogen Collibri NGS:赛默飞世尔科技(中国)有限公司。其他试剂均为国产分析纯。
NanoDrop2000超微量分光光度计:美国Thermo Fisher Scientific公司;Gel Doc 2000UV凝胶成像仪:美国Bio-Rad公司;ABI Gene 9700聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;Invitrogen Qubit3.0荧光计:美国Life公司;5424R高速台式冷冻离心机:德国Eppendorf公司;NoveSeq6000测序仪:美国Illumina公司。
1.3.1 红酸汤样品宏基因组DNA提取及PCR扩增
使用高效植物基因组DNA提取试剂盒提取红酸汤样品的宏基因组DNA,经1%琼脂糖凝胶电泳检测DNA的提取质量,使用NanoDrop2000超微量分光光度计对DNA浓度、纯度进行检测。以该宏基因组DNA为模板,细菌使用515F(5'-AGAGTTTGATCCTGGCTCAG-3')和806R(5'-GGTTACCTTGTTACGACTT-3')对16S rRNA V4区基因进行PCR扩增;真菌使用ITS5-1737F(5'-GCATATCAATAAGCGGAGGAAAAG-3')和ITS2-2043R(5'-GGTCCGTGTTTCAAGACGG-3')对ITS1区基因进行PCR扩增。PCR扩增体系:TsingkeI5High-FidelityMasterMix25μL,正、反向引物1μL,宏基因组DNA 1 μL,双蒸水(ddH2O)22 μL。细菌PCR扩增条件:98 ℃预变性2 min;98 ℃变性10 s,55 ℃退火15 s,72 ℃延伸15 s,共35个循环;72 ℃再延伸5 min。真菌PCR扩增条件:98 ℃预变性2 min;98 ℃变性10 s,5 ℃退火15 s,72 ℃延伸30 s,共35个循环;72 ℃再延伸5 min。PCR产物经1%琼脂糖凝胶电泳检测。
1.3.2 文库构建和上机测序
将检测合格的PCR扩增产物使用Invitrogen Collibri NGS构建文库,依托贵州微尔来检测技术有限公司经NoveSeq6000测序仪进行高通量测序。
1.3.3 序列分析
根据Barcode序列和PCR扩增引物序列从下机数据中拆分出各样本数据,截去Barcode和引物序列后,使用FLASH(Version 1.2.11)[9]对样品序列进行拼接。使用fastp软件(Version 0.23.1)进行过滤处理得到高质量的数据[10]。使用物种注释数据库(Silva datebase)去除数据嵌合体序列,得到最终的有效序列(Effective Tags)[11]。利用Uparse算法(Uparse v7.0.1001)对所有样本的全部有效序列进行聚类,以97%的一致性将序列聚类成为操作分类单元(operational taxonomic units,OTUs),细菌采用Mothur分类注释算法[12],真菌采用blast分类注释算法[13]。
1.3.4 多样性和统计分析
使用Qiime软件(Version 1.9.1)计算Observed-OTUs、超1(Chao1)、香农(Shannon)、辛普森(Simpson)、Ace指数进行Alpha多样性分析;使用R软件(Version 4.0.3)绘制稀释曲线。为了评估群落组成的复杂性并比较样本(组)之间的差异,使用Qiime软件(Version 1.9.1)进行Beta多样性分析;使用UPGMA进行聚类分析;使用R软件进行主成分分析(principal component analysis,PCA)。
高通量测序结果显示,韭菜红酸汤样品细菌菌群和真菌菌群的有效序列数(194 766±451、114 152±243)均显著高于红酸汤样品(102 466±381、106 084±255)(P<0.05)。数据量的饱和度由稀释曲线表示,测序数量达到要求时,稀释曲线趋于平坦[14]。样本中物种的丰富度和均匀度由丰度等级曲线表示,曲线在X轴跨度越大,物种的丰富度越高,曲线在Y轴曲线越平坦,物种分布越均匀[15]。韭菜红酸汤和红酸汤样品中细菌菌群和真菌菌群的稀释曲线及丰度等级曲线分别见图1和图2。由图1A和图2A可知,随着测序深度的增加,两种红酸汤样品细菌菌群和真菌菌群的稀释曲线趋于平坦,说明本研究测序深度可以较全面的覆盖红酸汤样品的物种。由图1B和图2B可知,韭菜红酸汤样品细菌菌群及真菌菌群丰富度高于红酸汤样品。

图1 细菌菌群的稀释曲线(A)及丰度等级曲线(B)
Fig.1 Dilution curve(A)and abundance grade curve(B)of bacterial flora
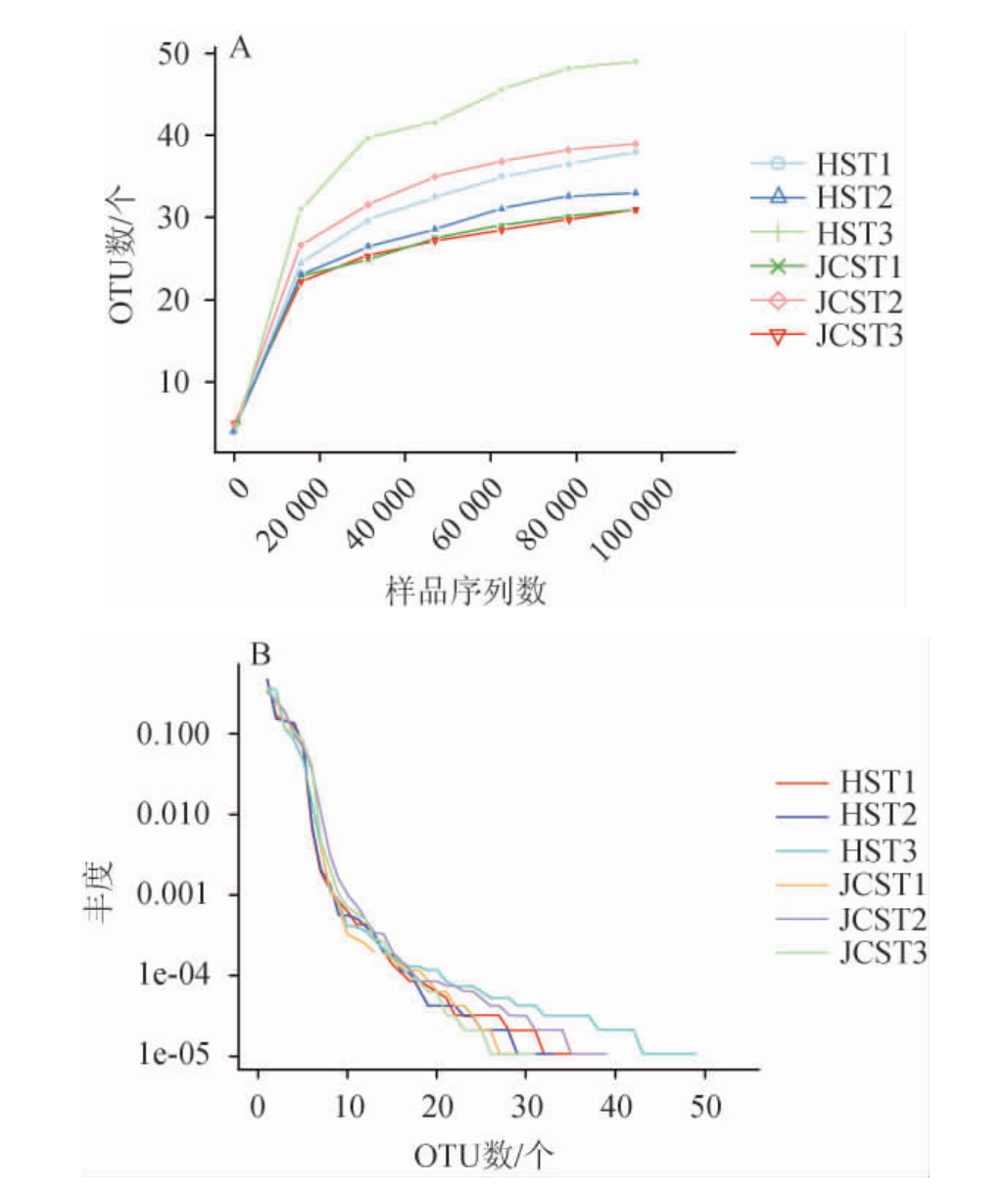
图2 真菌菌群的稀释曲线(A)及丰度等级曲线(B)
Fig.2 Dilution curves(A)and abundance grade curves(B)of fungal flora
红酸汤和韭菜红酸汤样品细菌菌群、真菌菌群OTU韦恩图见图3。由图3可知,从2种红酸汤样品中共获得347个细菌OTUs。其中23个为红酸汤样品特有OTU,202个为韭菜红酸汤样品特有OTU,红酸汤和韭菜红酸汤样品共有OTU有123个。从2种红酸汤样品中共获得72个真菌OTUs。其中23个为红酸汤样品特有OTU,9个为韭菜红酸汤样品特有OTU,红酸汤和韭菜红酸汤样品共有OTU有40个。
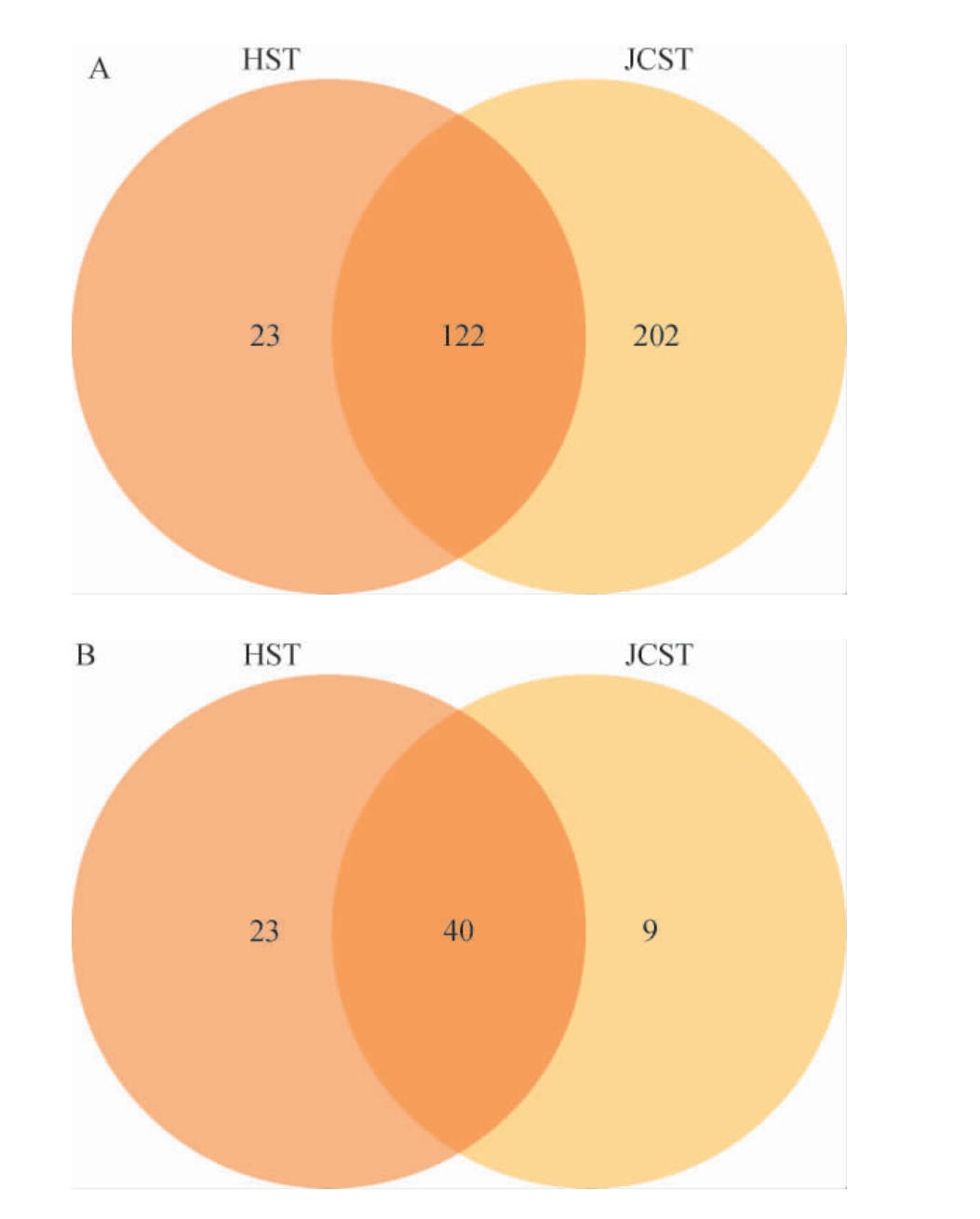
图3 红酸汤样品细菌(A)及真菌(B)菌群操作分类单元韦恩图
Fig.3 Venn diagram of operational taxonomic units of bacteria (A)and fungal (B) flora of red acid soup samples
Alpha多样性用于分析样本内的微生物群落多样性,其中,Ace指数用于评估菌群丰富度,Chao 1指数用于衡量群落物种丰度,Shannon指数和Simpson指数用于衡量群落物种多样性[16]。红酸汤样品微生物菌群的Alpha多样性分析结果见表1。由表1可知,韭菜红酸汤样品细菌菌群的Ace指数和Chao 1指数均显著高于红酸汤样品(P<0.05),Shannon指数和Simpson指数均低于红酸汤样品,但无显著差异(P>0.05);韭菜红酸汤样品真菌菌群的Ace指数、Chao 1指数均显著低于红酸汤样品(P<0.05),Shannon指数和Simpson指数均高于红酸汤样品,且Simpson指数显著更高(P<0.05),说明韭菜红酸汤样品的细菌菌群丰度高于红酸汤样品,多样性低于红酸汤样品,真菌菌群则相反。
表1 红酸汤样品微生物菌群的Alpha多样性分析结果
Table 1 Results of Alpha diversity analysis of red sour soup samples
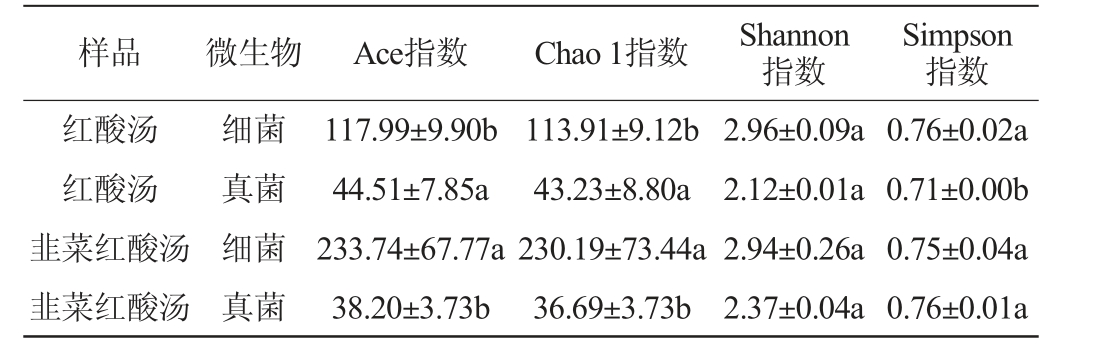
注:不同小写字母表示不同样品同一指标差异显著(P<0.05)。
样品 微生物 Ace指数 Chao 1指数 Shannon指数Simpson指数红酸汤红酸汤韭菜红酸汤韭菜红酸汤细菌真菌细菌真菌117.99±9.90b 44.51±7.85a 233.74±67.77a 38.20±3.73b 113.91±9.12b 43.23±8.80a 230.19±73.44a 36.69±3.73b 2.96±0.09a 2.12±0.01a 2.94±0.26a 2.37±0.04a 0.76±0.02a 0.71±0.00b 0.75±0.04a 0.76±0.01a
从红酸汤样品中共注释到8个细菌门、4个真菌门,从韭菜红酸汤样品中共注释到18个细菌门、3个真菌门。基于细菌、真菌属相对丰度对红酸汤和韭菜红酸汤样品进行PCA,结果见图4。
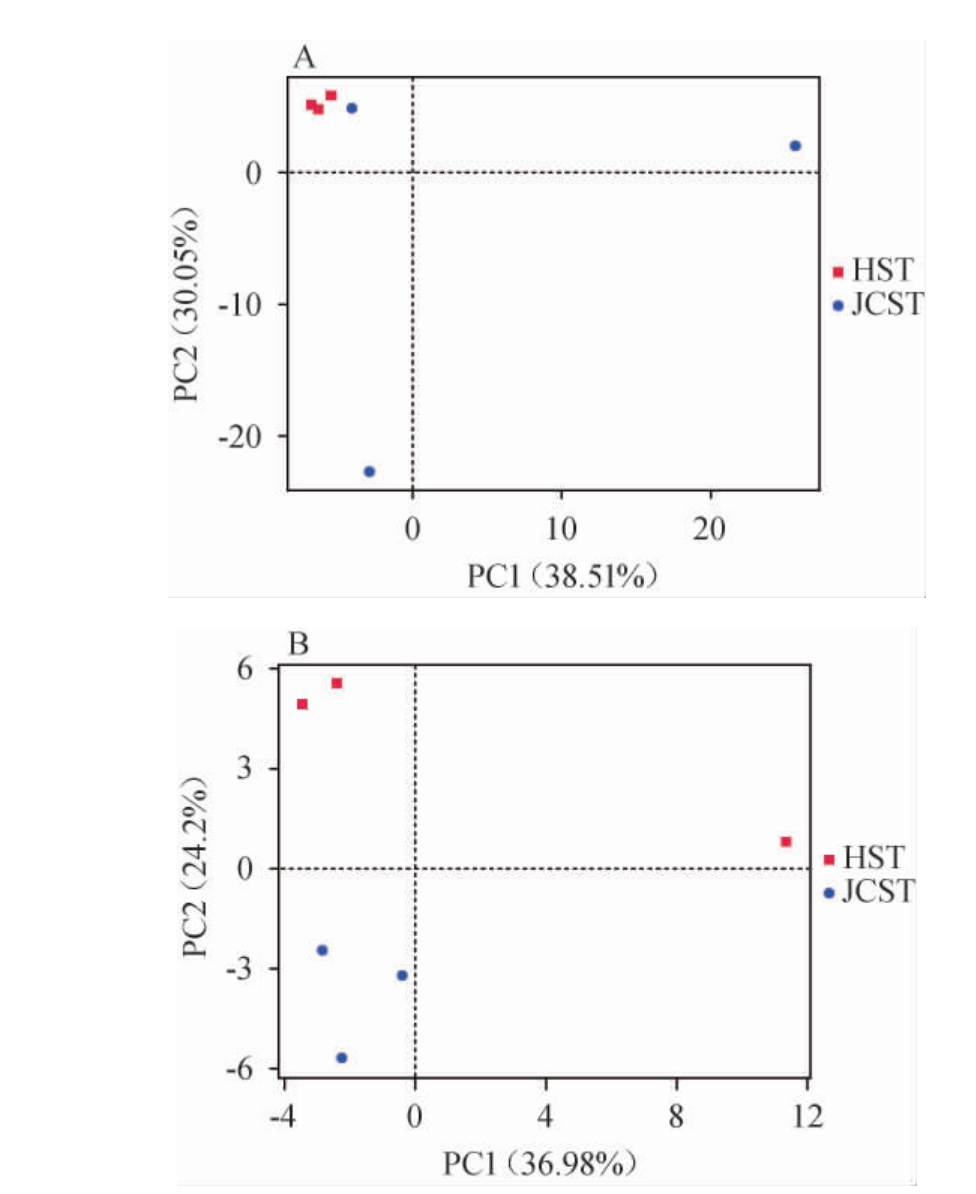
图4 基于细菌(A)及真菌(B)属相对丰度两种红酸汤样品的主成分分析结果
Fig.4 Principal component analysis results of 2 red acid soup samples based on relative abundance of bacteria (A) and fungi (B) genus
由图4A可知,PC1的方差贡献率为38.51%、PC2的方差贡献率为30.05%,累计方差贡献率为68.56%,说明基本可以解释红酸汤样品中细菌菌群绝大数的变异信息。韭菜红酸汤样品中,虽然有一个平行样品与红酸汤样品距离很近,但其他两个平行样品与红酸汤样品距离很远,出现此现象可能是在取平行样时存在偏差,总体说明两种红酸汤样品细菌菌群组成有差异。由图4B可知,PC1方差贡献率为36.98%、PC2方差贡献率为24.20%,累计方差贡献率为61.18%,说明基本可以解释红酸汤样品中真菌菌群绝大数的变异信息。红酸汤样品与韭菜红酸汤样品距离相差较远,说明两种红酸汤样品真菌菌群组成有明显的差异。
2.4.1 基于门水平微生物群落结构分析
基于门水平两种红酸汤样品微生物群落结构见图5。由图5A可知,红酸汤与韭菜红酸汤样品中的优势细菌门(相对丰度>1%)均为厚壁菌门(Firmicutes)(85.30%、86.03%)、变形菌门(Proteobacteria)(8.22%、8.59%)、蓝菌门(Cyanobacteria)(6.46%、4.90%)。由图5B可知,红酸汤与韭菜红酸汤样品中的优势真菌门均为子囊菌门(Ascomycota)(77.58%、82.16%)、担子菌门(Basidiomycota)(22.39%、17.82%)。结果表明,在门水平,两种红酸汤样品的菌群结构相似,具体差异主要体现在菌群的相对丰度上。
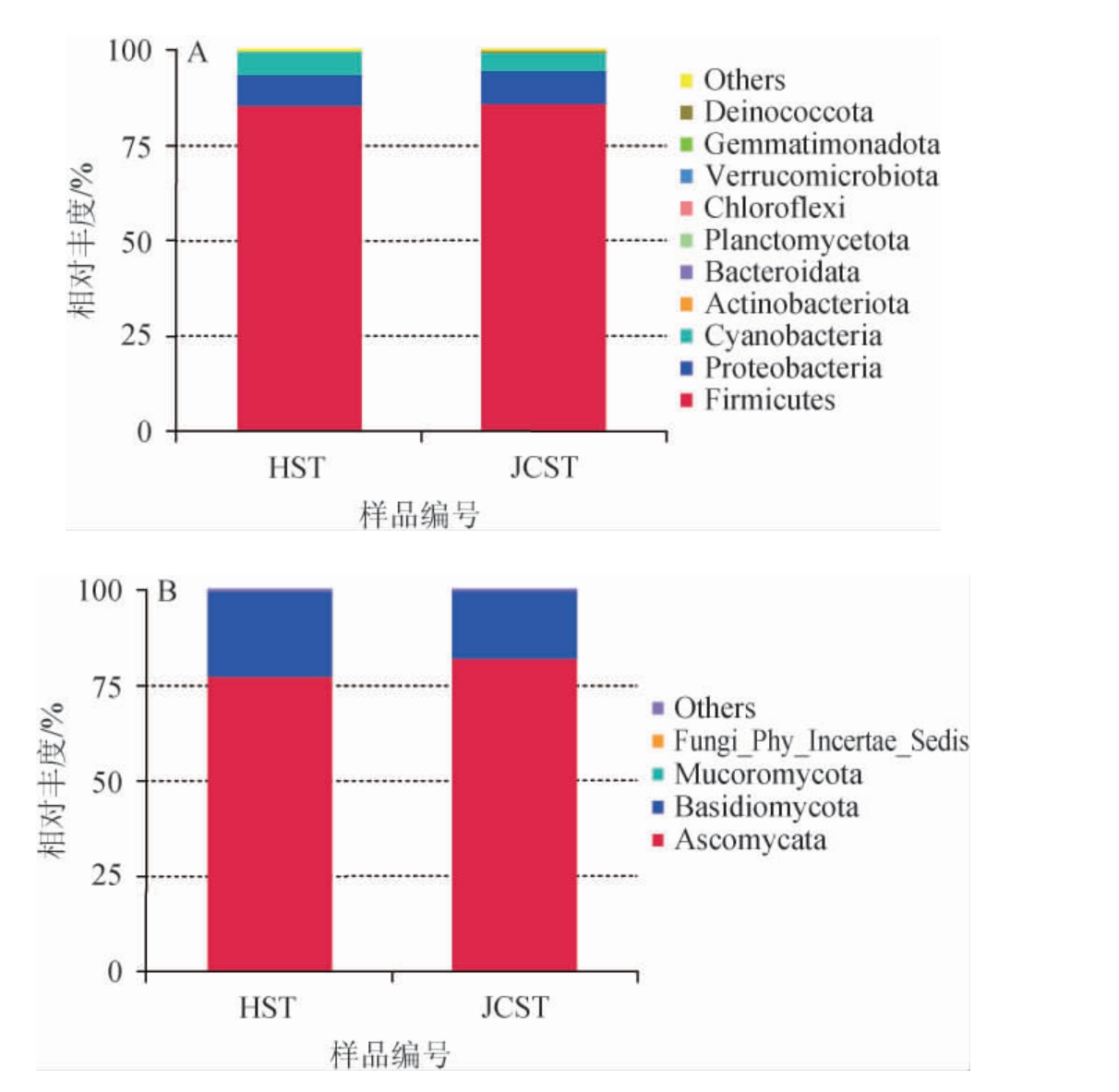
图5 基于门水平两种红酸汤样品细菌(A)及真菌(B)群落结构
Fig.5 Community structures of bacterial (A) and fungal (B) of 2 red acid soup samples based on phylum level
Others:未被注释到的和相对丰度较低的其他门。
2.4.2 基于属水平微生物群落结构分析
基于属水平两种红酸汤样品微生物群落结构见图6。由图6A可知,红酸汤与韭菜红酸汤样品中的优势细菌属(相对丰度>1%)均为乳酸杆菌属(Lactobacillus)(50.00%、49.53%)、迟缓乳杆菌属(Lentilactobacillus)(9.56%、8.99%)、片球菌属(Pediococcus)(9.11%、7.23%)、小坂菌属(Kosakonia)(4.67%、5.04%)、乳酪杆菌属(Lacticaseibacillus)(3.63%、3.09%)、乳植杆菌属(Lactiplantibacillus)(2.62%、2.08%)。韭菜红酸汤样品中乳酸杆菌属、小坂菌属的相对丰度均高于红酸汤样品,乳酸杆菌属是一类能促进醇类、酯类、酸类等物质生成,具有良好环境耐受性,对发酵食品的风味有显著影响的菌属[17-18];小坂菌属能够产阿魏酸酯酶,降解植物中的细胞壁,产生具有保健功能的阿魏酸[19]。红酸汤样品中迟缓乳杆菌属、双球菌属、乳酪杆菌属、乳植杆菌属(Lactiplantibacillus)的相对丰度均高于韭菜红酸汤样品,迟缓乳杆菌属大多模式菌株分离自青贮和发酵蔬菜,已被证明具有抗氧化、降低胆固醇含量和调节肠道菌群结构与数量等益生功效;双球菌属是一种能够耐酸耐盐环境,具有亚硝酸盐降解能力,产酸能力很强的菌属[20];乳酪杆菌属具有良好的耐酸及胆汁抗性,其不仅可以降低血浆胆固醇,增强宿主对微生物病原体的非特异性抵抗力,还可以加快清除肠道内病原体,治疗肠道菌群紊乱和增强肠道透性,从而防止食物过敏和急性腹泻,此外,还可增加抗低密度氧化脂抗体和淋巴细胞,使粒细胞的噬菌作用明显增强,对宿主进行免疫调节,防止肿瘤的产生[21];乳植杆菌属(Lactiplantibacillus)广泛存在于发酵食品中,具有调节肠内菌群平衡、促进消化吸收、降胆固醇、预防心血管疾病、控制体重、增强免疫力等多种保健功能[22-24]。
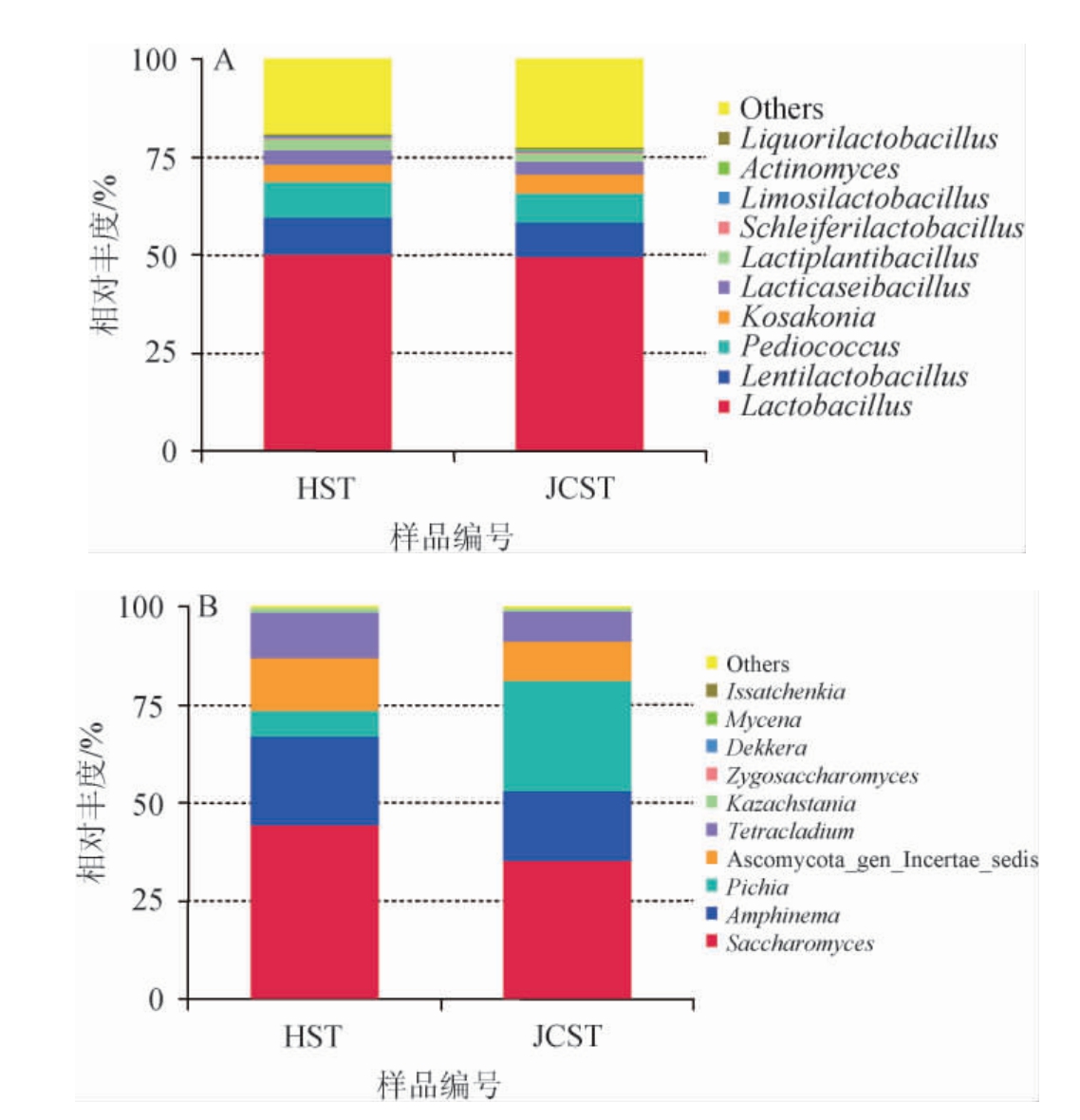
图6 基于属水平两种红酸汤样品细菌(A)及真菌(B)群落结构
Fig.6 Community structures of bacterial (A) and fungal (B) of 2 red acid soup samples based on genus level
Others:未被注释到的和相对丰度较低的属。
由图6B可知,红酸汤与韭菜红酸汤样品中的优势真菌属均为酵母属(Saccharomyces)(44.47%、35.20%)、糙缘腺革菌属(Amphinema)(22.36%、17.82%)、毕赤酵母属(Pichia)(6.58%、27.98%)、Ascomycota_gen_Incertae_sedis(13.51%、10.31%)、Tetracladium(11.63%、7.56%)、哈萨克斯坦酵母属(Kazachstania)(1.26%、0.95%)。红酸汤样品中酵母菌属、糙缘腺革菌属、子囊菌门、Tetracladium、哈萨克斯坦酵母属的相对丰度均高于韭菜红酸汤样品,酵母菌菌属可以在发酵过程中增加酯类物质的种类和相对含量,降低乙醇相对含量、pH值以及亚硝酸盐峰值,使得发酵产品风味更加醇厚协调,品质更好[25];糙缘腺革菌属能够发酵产生多糖,具有免疫调节、降低胆固醇、降血压等生理活性[26];子囊菌门中的属作为优势菌株普遍存在于发酵食品中[27-29];Tetracladium广泛存在于土壤中[30-31],在发酵食品中的作用尚不知晓,可能是在酸汤制作过程中引入的外源微生物;哈萨克斯坦酵母属在一些发酵食品中可以起到改善风味的作用[32]。韭菜红酸汤样品中毕赤酵母属的相对丰度高于红酸汤样品,毕赤酵母属在发酵过程中能够产生酸、醇、酯以及一些独特的呈香物质,使得发酵产品风味成分更加丰富,增加发酵产品的品质[33]。韭菜红酸汤样品中毕赤酵母属的相对丰度高于红酸汤样品,毕赤酵母属在发酵过程中能够产生酸、醇、酯以及一些独特的呈香物质,使得发酵产品风味成分更加丰富,增加发酵产品的品质[34]。
结果表明,在属水平,两种红酸汤样品的菌群结构相似,具体差异主要体现在菌群的相对丰度上。
采用高通量测序技术对红酸汤和韭菜红酸汤微生物菌群多样性及群落结构进行分析。Alpha多样性结果表明,韭菜红酸汤的细菌菌群丰度高于红酸汤,多样性低于红酸汤,真菌菌群相反。主成分分析结果表明,两种样品细菌菌群以及真菌菌群组成均有差异,这种差异主要体现在相对丰度上。从红酸汤样品中共注释到8个细菌门、4个真菌门,从韭菜红酸汤样品中共注释到18个细菌门、3个真菌门,优势细菌门(相对丰度>1%)均为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、蓝菌门(Cyanobacteria),优势真菌门均为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)。从红酸汤样品中共注释到43个细菌属、25个真菌属,从韭菜红酸汤样品中共注释到103个细菌属、18个真菌属,优势细菌属(相对丰度>1%)均为乳酸杆菌属(Lactobacillus)、迟缓乳杆菌属(Lentilactobacillus)、片球菌属(Pediococcus)、小坂菌属(Kosakonia)、乳酪杆菌属(Lacticaseibacillus)、乳植杆菌属(Lactiplantibacillus);优势真菌属均为酵母属(Saccharomyces)、糙缘腺革菌属(Amphinema)、毕赤酵母属(Pichia)、Ascomycota_gen_Incertae_sedis、Tetracladium、哈萨克斯坦酵母属(Kazachstania)。本研究结果有助于对贵州黔东南州麻江县特色韭菜红酸汤发酵过程进行精准调控和规模指导生产。
[1]蒋文周.新时代标准化助推特色产业高质量发展——以凯里酸汤产业为例[N].黔东南日报,2023-08-09.
[2]陈中爱,熊江,石庆楠,等.黔东南地区传统发酵白酸汤中乳酸菌的益生特性及应用[J].食品安全质量检测学报,2021,12(14):5815-5821.
[3]袁野,李云成,孟凡冰,等.贵州红酸汤研究进展[J].粮食与油脂,2022,35(6):19-23.
[4]鲁杨,王楠兰,李贤,等.凯里红酸汤主要营养和功能成分的分析研究[J].食品研究与开发,2019,40(7):163-166.
[5]周绍琴,李凤兰,吴映梅,等.红酸汤的抗氧化活性研究[J].中国调味品,2021,46(7):17-21.
[6]鲁杨.凯里红酸汤成分及其对大鼠血脂调节作用研究[D].贵阳:贵州医科大学,2019.
[7]郝明玉.直投式发酵泡菜与自然发酵泡菜的比较研究[D].南昌:南昌大学,2013.
[8]赵丽芬,王青青,奥宁,等.不同韭菜品种营养品质评价分析[J].中国蔬菜,2024(6):81-88.
[9]MAGOCˇT, SALZBERG S L.FLASH: fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics, 2011, 27(21):2957-2963.
[10]BOKULICH N A,SUBRAMANIAN S,FAITH J J,et al.Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J].Nat Methods,2012,10(1):57-59.
[11]EDGAR R C, HAAS B J, CLEMENTE J C, et al.Improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194-2200.
[12]QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J].Nucleic Acids Res,2013,41:D590-D596.
[13]HERR J R, QBIK M, HIBBETT D S.Towards the unification of sequence-based classification and sequence-based identification of hostassociated microorganisms[J].New Phytol,2015,205(1):27-31.
[14]杨玲,刘春,彭佳伟,等.山西老陈醋和怀仁醋工艺中酒精发酵阶段细菌种群分析[J].中国酿造,2023,42(2):63-69
[15]LUNDBERG D S,YOURSTONE S,MIECZKOWSKI P,et al.Practical innovations for high-throughput amplicon sequencing[J].Nat Methods,2013,10(10):999-1002.
[16]韩国强,孙协平,吴鹏飞,等.基于高通量测序分析复配小曲白酒发酵过程中微生物群落结构及多样性[J].食品科学,2021,42(18):80-85.
[17]林麟,杜如冰,吴群,等.基于比较基因组学解析耐酸乳杆菌G10的多碳源利用特征[J].微生物学通报,2022,49(8):3279-3292.
[18]PANG X N,CHEN C,HUANG X N,et al.Influence of indigenous lactic acid bacteria on the volatile flavor profile of light-flavor Baijiu[J].LWTFood Sci Technol,2021,147:111540.
[19]章宇丹,温雪瓶,李丽,等.考氏科萨克氏菌产阿魏酸酯酶条件优化、酶学性质及其初步应用[J].中国酿造,2021,40(2):47-52.
[20]高逸,张健,陈国辉,等.乳酸菌CICC 6282的鉴定、生物学特性及其在发酵辣椒中的应用[J].中国酿造,2024,43(2):53-59.
[21]张文娟,陈安特,韩宇琴,等.酿酒酵母对萝卜泡菜发酵的影响[J].食品与发酵工业,2017,43(8):134-137.
[22]徐永霞,白旭婷,赵洪雷,等.植物乳杆菌在水产品中的应用研究进展[J].中国调味品,2022,47(2):195-199.
[23]刘超楠.同来源植物乳杆菌基因组以及对碳水化合物利用的比较分析[D].北京:中国农业科学院,2021.
[24]SHUKLA P K,MEENA A S,MANDA B,et al.Lactobacillus plantarum prevents and mitigates alcohol-induced disruption of colonic epithelial tight junctions,endotoxemia,and liver damage by an EGF receptor-dependent mechanism[J].FASEB J,2018,32(11):6274-6292.
[25]王艳,胡跃,方红美,等.白萝卜泡菜加工工艺的研究[J].食品工业科技,2021,42(11):185-191.
[26]赵盼.罗耳阿太菌胞外多糖经Nrf2通路防护小鼠肝肾铅损伤[D].长春:吉林农业大学,2022.
[27]ZANG J H,XU Y S,XIA W S,et al.Dynamics and diversity of microbial community succession during fermentation of Suan yu, a Chinese traditional fermented fish.determined by high throughput sequencing[J].Food Res Int,2018,111:565-573.
[28]乌仁图雅.应用454焦磷酸测序技术对传统发酵乳制品微生物多样性的研究[D].呼和浩特:内蒙古农业大学,2014.
[29]姚灿,张倩,张霞,等.古顺大曲微生物群落结构的分析[J].酿酒科技,2020(1):20-23.
[30]王伟,布丽根·加冷别克,胡晓东,等.基于高通量测序技术的酿酒葡萄产区土壤微生物多样性[J].新疆农业科学,2020,57(5):859-868.
[31]王立鹏.藏东南冰川区锤舌菌纲真菌系统学及抑菌活性研究[D].保定:河北大学,2023.
[32]SPANOGHE M,JARA M G,RIVIÈRE J,et al.Development and application of a quantitative real-time PCR assay for rapid detection of the multifaceted yeast Kazachstania servazzii in food[J].Food Microbiol,2017,62:133-140.
[33]邢晓莹,刘毅,霍乃蕊,等.山西老陈醋中优良酵母菌株筛选及其在木枣果酒发酵中的应用[J].中国酿造,2021,40(3):64-72.
[34]曾骏.传统四川泡菜发酵过程中酵母菌的动态变化规律及发酵性能研究[D].雅安:四川农业大学,2013.