酵母蛋白质组学分析与制备技术研究进展
赵 焱1,2,李 宁3,朱绪春1,2,周麟依1,2,韩朝玮1,2,刘晓永4,刘红芝1,2*
(1.北京工商大学 食品与健康学院,北京100080;2.北京工商大学 老年营养与健康教育部重点实验室,北京100080;3.内蒙古蒙牛乳业(集团)股份有限公司,内蒙古 呼和浩特011599;4.河南农业大学 食品科学技术学院,河南 郑州450002)
摘 要:我国传统蛋白的获取大量依赖于种植业和养殖业,存在严重的土地和水资源依赖性,难以应对巨大的蛋白缺口。酵母中蛋白含量超过50%,且必需氨基酸占氨基酸总量的47.58%,是一种完全优质蛋白。目前,工业废酵母多用于提取酵母蛋白,如果可以通过酵母蛋白质组学分析,筛选高营养的酵母菌株,就能为酵母蛋白资源的利用奠定原料基础。绿色高效的酵母蛋白制备技术直接影响酵母蛋白利用,目前多采用酸碱制备酵母蛋白,亟待开发高效清洁的酵母蛋白制备技术。该文介绍了蛋白质组学相关技术,并探讨了不同种类、不同培养条件以及不同生长阶段的酵母菌株的蛋白质组差异和多种不同酵母蛋白制备工艺的比较,旨在为高产蛋白酵母菌株的筛选和酵母蛋白资源的高值化利用奠定理论基础,并勾勒出未来的发展前景。
关键词:酵母蛋白;蛋白质组学;制备技术;研究进展
中图分类号:TS261.1
文章编号:0254-5071(2025)03-0008-07
doi: 10.11882/j.issn.0254-5071.2025.03.002
引文格式:赵焱,李宁,朱绪春,等.酵母蛋白质组学分析与制备技术研究进展[J].中国酿造,2025,44(3):8-14.
收稿日期:2024-02-27 修回日期:2024-06-13
基金项目:北京市高层次人才队伍建设项目(19008023229)
作者简介:赵 焱(1997-),男,助理工程师,硕士研究生,研究方向为功能配料开发与健康食品创制。
*通讯作者:刘红芝(1980-),女,研究员,博士,研究方向为功能配料开发与健康食品创制。
Research progress of yeast proteomic analysis and preparation techniques
ZHAO Yan1,2,LI Ning3,ZHU Xuchun1,2,ZHOU Linyi1,2,HAN Zhaowei1,2,LIU Xiaoyong4,LIU Hongzhi1,2*
(1.School of Food and Science,Beijing Technology and Business University,Beijing 100080,China;2.Key Laboratory of Geriatric Nutrition and Health,Ministry of Education,Beijing Technology and Business University,Beijing 100080,China;3.Inner Mongolia Mengniu Dairy(Group)Co.,Ltd.,Hohhot 011599,China;4.College of Food Science and Technology,Henan Agricultural University,Zhengzhou 450002,China)
Abstract:The acquisition of traditional protein in China depends heavily on planting and aquaculture,and there is a serious dependence on land and water resources, which is difficult to cope with the huge protein gap.The protein content in yeast is more than 50%, and the essential amino acids account for 47.58% of the total amino acids, which is a completely high-quality protein.At present, industrial waste yeast is mostly used to extract yeast protein.If yeast strains with high nutrition can be screened through yeast proteomic analysis,it can lay a foundation for the utilization of yeast protein resources.The green and efficient yeast protein preparation technology directly affects the utilization of yeast protein.At present,most of the yeast proteins are prepared by acid and alkali, and it is urgent to develop efficient and clean yeast protein preparation technology.In this paper, the proteomic techniques were introduced,and the proteomic differences of yeast strains with different species,different culture conditions and different growth stages,as well as the comparison of various yeast protein preparation processes were discussed,in order to lay a theoretical foundation for the screening of high-protein yeast strains and the high-value utilization of yeast protein resources,and outline the future development prospects.
Key words:yeast protein;proteomics;preparation technique;research progress
我国有超50%的居民蛋白质摄入不足,约2.7亿在校生蛋白摄入量仅为国家标准的65%。传统动植物蛋白的获取大量依赖于种植业和养殖业,存在严重的土地和水资源依赖性,难以应对巨大的蛋白缺口。酵母中蛋白质含量超过50%,其氨基酸种类齐全,包括人体必需的8种氨基酸和2种半必需氨基酸,属于完全蛋白质[1-2]。此外,酵母蛋白中必需氨基酸占氨基酸总量的47.58%,必需氨基酸与非必需氨基酸含量的比值达到了0.91,符合联合国粮农组织和世界卫生组织标准规定的40%和0.6[3]。因此,酵母蛋白可以作为优质的新型替代蛋白来填补动植物蛋白的缺口。另外,酵母蛋白也是一种低成本的食品原料,因为食品企业通常是从一些廉价的食品工业废酵母中提取的酵母蛋白[4]。因此,深入研究酵母蛋白对缓解我国蛋白质缺口,推动我国食品工业的高质量发展,保障全民营养健康具有深远的意义。
1 酵母蛋白质组学分析
蛋白质在生物体中既是重要的组成部分,同时也是构建细胞结构的主要成分,在代谢和细胞信号通路中起到至关重要的作用[5]。随着科学技术的不断进步,蛋白质组学作为一种全面了解生物体内蛋白质种类、结构、功能以及相互作用的工具,为酵母蛋白的研究提供了全新的视角。蛋白质组学分析涉及蛋白质表达、蛋白质结构和蛋白质功能分析,特别是致力于研究每个蛋白质在生理代谢过程中承担的多种功能,同时能够对蛋白质进行识别、鉴定和量化[6]。
1.1 蛋白质组学分析技术
1.1.1 二维凝胶电泳
1975年,O'Farrel提出二维凝胶电泳可以用来检测蛋白质[7]。第一向电泳根据蛋白质的不同等电点分离各种蛋白质,即等电聚焦法(isoelectric focusing,IEF)。第二向电泳根据蛋白质的不同分子质量进一步分离等电点相同的蛋白质,即十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)。二维凝胶电泳可以将某一蛋白质与其翻译加工后的蛋白质形式同蛋白酶水解后的蛋白质形式分离开,为研究不同活化功能状态的蛋白质提供便利[8]。但通过二维凝胶电泳将蛋白质分离后只能检测到几百个蛋白质,因此二维凝胶电泳方法基本被用来检测一些结构简单的蛋白质。20世纪70年代后期,研究者开始利用二维凝胶电泳技术建立蛋白质数据库,对凝胶中的蛋白质斑点进行了鉴定和分类,建立了所有表达蛋白质数据库[7]。
1.1.2 质谱技术
质谱技术因其高灵敏度、高分辨率、高通量常被用于蛋白质组学研究,并且还可以通过电荷比来对蛋白质定性和定量[9]。1987、1988年发明的基质辅助激光解吸电离(matrixassisted laser desorption ionization,MALD)[10]和电喷雾电离(electron spray ionization,ESI)[11]使质谱从有机化学的应用过渡到生物大分子的检测[12]。质谱技术在蛋白质组学的研究中四个关键参数分别是分辨率、灵敏度、质量准确度和从肽段生成信息丰度的离子质谱的能力[13]。质谱技术的核心是质量分析器,目前蛋白质组学的研究中最常用的四种质量分析器分别是离子阱、飞行时间、四级质量分析器和傅里叶变换离子回旋加速分析器,这四种质量分析器可以单独使用,也可以串联在一起使用[14]。
蛋白质组学最初以定性测定为主,但随着对蛋白质组学研究的深入,为了获取更准确的蛋白质组数据,当前的研究越来越注重定量测定[15]。因此,为了对蛋白质组进行更深入的准确分析,研究者采用了多种技术手段,包括非标记定量蛋白质组学(label-free)[16]、串联质谱标签法(tandem mass tag,TMT)[17]以及同位素标记的相对和绝对定量技术(isobaric tags for relative and absolute quantification,iTRAQ)[18]等。各种蛋白质组学分析方法的优缺点总结结果见表1。这些技术的应用使得蛋白质组学研究能够更全面、更精确地了解蛋白质的表达及变化,推动了该领域的发展,研究中应根据样品类型和实验条件来选择分析方法以获得更准确的数据。
表1 各种蛋白质组学分析方式的优缺点
Table 1 Advantages and disadvantages of various proteomic analysis methods

蛋白质组学分析方法 优点 缺点二维凝胶电泳 高分辨率;能检测翻译和修饰;适用各种样品非标记定量法串联质谱标签法操作复杂;灵敏性不足;重叠和漂移;蛋白质大小有限制无法提供绝对丰度;易受其他因素干扰;样品类型会影响分析结果样品制备繁琐;成本较高;可能有假阳性现象同位素标记的相对和绝对定量技术实验流程简化;适用于复杂样品;成本低高灵敏度高通量定量;适用于复杂样品;定性和定量一体化可以同时对四种不同样本中的蛋白质进行定量比较易被样本中杂质蛋白和缓冲液污染
1.1.3 生物信息学技术
生物信息学是利用应用数学、信息学、统计学、计算机科学以及生命科学等方法处理生物大分子数据的新兴学科。利用生物信息学对大量的组学数据进行计算分析,揭示基因的功能,完善代谢调控网络,为研究生物体的各种生命过程提供了重要启示和新的机遇[19]。
生物信息学分析主要包括蛋白序列比对、蛋白功能分析及结构预测以及蛋白分子相互作用等方面。随着研究的不断深入,蛋白质数据库的建立和分析软件的不断改进升级为生物信息学的发展提供了广泛的技术支持。蛋白质组数据库是有机体、组织或细胞中所有表达蛋白质的详尽信息的存储库[20]。目前,广泛应用的蛋白质组学研究数据库包括Swiss-Prot、ProFound、MSQuant、PROSITE等,其中Swiss-Prot数据库是世界两大蛋白序列数据库之一,收录了真实存在的蛋白质信息资源;PROSITE数据库收录了蛋白质保守结构域,包含重要生物学意义的位点、模式、轮廓和翻译后修饰位点等[21]。
1.2 蛋白质组学技术在酵母蛋白研究中的应用
1.2.1 不同品种酵母菌株蛋白质组学分析
研究不同品种酵母菌株的蛋白质组学是理解它们在代谢、应激响应和生长等方面的差异的关键。这种研究有助于揭示菌株之间的功能差异,为工业、生物技术和医学等领域提供有用的信息。常用的酵母菌株种类有:酿酒酵母(Saccharomyces cerevisiae)、毕赤酵母(Pichia pastoris)以及马克斯克鲁维酵母(Kluyveromyces marxianus)等。
酿酒酵母是特征最广泛的模式生物之一,对酿酒酵母蛋白质组的研究已经十分全面[22]。GAO Y等[22]基于高分辨率质谱鉴定了酿酒酵母中5 610个蛋白质并建立了蛋白质组数据库,所有鉴定的蛋白质中有94.1%是核心蛋白质,即使是此类实验中难以鉴定的膜蛋白也能达到93.4%的覆盖率,此外,该蛋白质组数据集涵盖了91%以上的基因本体(gene ontology,GO)分析和98%的京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)分析通路中的蛋白质。UEBAYASHI K等[23]通过靶向蛋白质组分析比较了七种酿酒酵母菌株发酵特性,七种酵母菌株之间蛋白质表达水平的变化导致了它们不同的发酵特性,对不同的蛋白质进行基因富集分析,分析显示生物学过程最丰富的类别与代谢过程相关。ROSSIO V等[24]对实验室中常用的两种酵母菌株W303和BY474在对数生长期和平稳生长期进行蛋白质组学分析,使用等压标签质谱法突显了这两种酵母菌株蛋白质组的复杂性和差异性,对4 400种蛋白质进行了量化处理,发现其中有数百种蛋白质在不同菌株和不同生长阶段之间存在丰度的差异。这些差异可能表明在指数和平稳生长阶段,这些蛋白质受到不同的基因调节,这种蛋白质丰度的改变可能有助于研究不同类型菌株中特异性基因的表达。SANTOS R M等[25]应用定量蛋白质组学方法比较了影响PE-2和CAT-1两种酵母菌株发酵性能的特征,结果表明,菌株CAT-1中对氧化应激有关的蛋白质(Sod1、Trx1)和海藻糖合成有关的蛋白质(Tps3)比菌株PE-2更丰富,因此菌株CAT-1具有更好的发酵性能。
LI P S等[26]为了解马克斯克鲁维酵母在高温乙醇发酵后期对应激条件的细胞反应,对马氏酵母进行了基于同位素标记相对和绝对定量(isobaric tags for relative and absolute quantification,iTRAQ)的定量蛋白质组学分析,鉴定了25 022个肽段和3 331个蛋白质并从中找出差异表达蛋白,还通过GO分析和KEGG通路分析对所有已鉴定的蛋白质进行功能注释,这些蛋白质被归类到GO术语中的催化活性、代谢过程等功能类别以及KEGG途径中的代谢过程、次生代谢产物的生物合成、抗生素生物合成等。
毕赤酵母是目前一种成熟的蛋白质表达宿主,主要应用与生物制药和工业生产,具有严格调控的启动子并且可以实现高密度培养[27]。SHEVCHENKO A等[28]在2001年使用从头测序方法对毕赤酵母进行了蛋白质组学分析。RENUSE S等[29]表征了毕赤酵母GS115的蛋白质组,分析了46 889个肽段,映射到3 914个独特的蛋白质组,对研究中鉴定的蛋白质进行GO分析,基于分子功能的注释中28%的蛋白质参与了催化,30%的已鉴定蛋白质参与了结合,还有约26%的蛋白质没有可用注释;根据GO中注释的生物过程分类,有39%的蛋白质参与代谢过程,而其他蛋白分别参与了运输过程(8%)、生物过程调控(6%)以及生物合成过程(3%),但还有约42%的蛋白质没有注释。总的来说,还有大约40%的蛋白质没有被GO注释分类,接下来应着重对这些未分类的蛋白质进行研究。
1.2.2 酵母发酵条件对蛋白质组的影响
酵母细胞的生长和代谢受到培养基组分、温度等多方面因素的调控,这些因素将可能导致酵母蛋白质组产生差异。
GARCIA-ALBORNOZ M等[30]使用无标记蛋白质组学方法,对在葡萄糖、半乳糖、麦芽糖和海藻糖等作为唯一碳源上生长的酵母菌的蛋白质丰度进行定量,与在葡萄糖作为碳源相比,共有892个蛋白质丰度产生显著性差异,对蛋白进行GO富集分析,结果见表2。由表2可知,这项研究揭示了在不同碳源条件下酿酒酵母菌株代谢相关途径的发现和酵母细胞蛋白质水平的差异,可能存在一组对生长条件敏感的蛋白质,在不同生长条件下会出现显著不同的表达。
表2 不同碳源培养条件下GO富集的差异
Table 2 Difference of GO enrichment under different carbon source cultivation conditions

培养基中碳源种类 差异蛋白GO富集分析结果麦芽糖半乳糖海藻糖电子传递链;ATP合成等代谢相关碳水化合物降解;细胞蛋白修饰翻译过程;脂肪酸代谢;细胞应激
PAULO J A等[31]研究了在使用不同碳源的最小生长培养基生长的培养物引起视觉蜡样酵母蛋白质组的变化,大多数发生改变的蛋白质被归类为代谢过程中的功能蛋白质,研究表明,碳源不同会导致蛋白质组发生改变,可能影响功能蛋白的表达。ZHAO S H等[32]研究了不同氮源条件下培养的酵母菌株蛋白质组学差异,对差异蛋白进行GO分类分析结果表明,差异蛋白与前体代谢产物和能量产生、细胞氨基酸代谢过程、翻译和应激反应相关。该研究为了解酵母如何适应不同营养条件提供了线索。
酵母是研究不同应激因素对真核细胞稳态影响的重要模型[33]。刘秋影等[34]研究了毕赤酵母单独热胁迫和热-盐共胁迫组之间的蛋白质组差异,利用串联质谱标签定量蛋白质组学技术筛选与耐热性相关的关键蛋白。研究表明,盐胁迫显著提高麦角固醇合成相关酶的表达,有利于维持胞内生物大分子结构和功能的稳定,热胁迫下酵母细胞内能量代谢的多种酶在盐胁迫下表达显著提高,以维持细胞正常新陈代谢。FAKANKUN I等[35]对氮和氧限制条件下的油性胡萝卜素酵母(R.diobovata)的蛋白质组进行了分析,在被鉴定和定量的4 408种蛋白质中,有1 126种蛋白质被考虑用作初始功能生物学分析。在表达上调的蛋白质中,GO分析表明,这些蛋白与组氨酸合成、信使核糖核酸(messenger ribonucleic acid,mRNA)加工和糖酵解过程相关。在低氮环境下,组氨酸合成提高有助于释放氨以维持生物合成过程。JUSTINIC′ I等[36]以吡虫啉农药为胁迫因子研究了其对酿酒酵母蛋白质组的影响。研究发现,吡虫啉农药的毒性引起27种蛋白质表达发生改变,其中包括跨膜转运蛋白、催化蛋白以及细胞应激相关蛋白。蛋白质表达改变、代谢活性改变和脂质含量变化共同作用确保细胞稳态并保护酵母细胞。
综上,培养条件可以从很大程度上影响酵母的蛋白质组,特定的培养条件可以使特定的基因表达产生显著的上调或下调。如果可以通过控制培养条件使酵母细胞过量表达需要的基因,提高发酵的效率。
1.2.3 酵母菌株生长过程中蛋白质组学变化
酵母菌株的生长过程是一个极为复杂而精密的生物学过程,涉及到细胞生命周期、代谢调控以及应对外部环境变化的机制。蛋白质组学为深入研究基因功能提供了一套有效的手段,使人们能够直接在蛋白质水平上进行探索。
XU W N等[37]探究了酿酒酵母自溶过程中蛋白质水平的变化。研究发现,共有49种蛋白质参与了自溶过程,其中8种上调,41种下调,并且出现了7种新的蛋白质。蛋白质功能分析表明,8种上调的蛋白质可以被分为能量代谢、蛋白质降解、应激反应以及氨基酸代谢几大类,这8种蛋白质上调表明酵母在抵抗自溶时受到很大影响。下调的41种蛋白质中除了一个未知功能的蛋白质,其余的可被归为能量代谢、应激响应、氨基酸代谢及核苷酸代谢几大类,这些蛋白质的抑制可能影响能量的利用或产生。SUN Y等[38]使用胞外稳定同位素标记法(stable isotope labeling with amino acids in cell culture,SILAC)定量蛋白质组学分析比较了正常条件和热量限制条件下年轻和衰老酵母细胞的蛋白质组。分析发现,衰老蛋白质组中产生了不同的特征蛋白。此外,衰老细胞和热量限制细胞蛋白质组之间存在显著的相似性,这可能表明热量限制通路参与了细胞的衰老。这些发现可以为研究细胞衰老的影响因素开拓新的思路。
酵母自溶可以使酵母细胞内部多种活性物质更好的释放,以此来提高酵母提取物的品质和产量[39]。PORRASAGÜERA J A等[40]对两种发酵常用的工业酵母菌株进行蛋白质组学分析。研究发现,两株酵母的自溶均发生在发酵阶段,而且在二次发酵条件下表达上调的特异性蛋白质如:Bcy1p、Sec2p、Sec13p、Sec18p、Shp1p和Vps15p可作为加速酵母自溶的生物标志物。根据这项研究,后续可以通过基因工程或其他手段促进酵母自溶从而降低生产成本和时间。
2 酵母蛋白制备技术
2.1 酵母破壁技术研究
酵母蛋白的制备涉及细胞破壁和蛋白提取两个过程[41]。酵母细胞结构复杂,且具有坚硬的细胞壁[42]。细胞壁将酵母细胞内的营养物质以及蛋白质包裹在细胞内,提取酵母蛋白之前要先破坏酵母细胞的细胞壁,使细胞内的蛋白暴露出来。目前主要依靠物理、化学和生物的方法对酵母细胞进行破壁。物理破壁的方法有机械法破壁、超声法破壁、高压均质法破壁、脉冲电场法破壁等,通过物理作用将酵母细胞壁破坏[43];化学方法有高温碱热法、离子液体法等,利用化学物质的作用破坏酵母细胞壁或改变酵母细胞的通透性;生物法常利用酶法和自溶法等方式破坏酵母细胞壁[44]。虽然破壁方法很多,但应用到大规模生产中会出现破壁率低、处理量低、操作复杂等问题。
2.1.1 自溶法破壁
酵母在发酵过程中,会不可避免地发生自溶现象。因为酵母细胞内存在能降解自身细胞膜和细胞壁的酶,当这类酶作用于酵母细胞自身时会破坏细胞膜和细胞壁,使酵母细胞中的大分子物质释放出来[39]。BERLOWSKA J等[45]研究了五种常用于发酵过程的酵母菌株:酿酒酵母、马克斯克鲁维酵母LOCK 0026、马克斯克鲁维酵母NCYC 179、谢佛酵母NCYC 1541和毕赤酵母NCYC 495,在皂苷的存在下以50 ℃进行48 h的自溶过程,再使用光谱法和色谱法检测了酵母自溶过程中蛋白质和游离氨基酸的含量,发现在酿酒酵母和毕赤酵母NCYC 495的自溶物中蛋白质和氨基酸含量最高,分别为44.9 g/L及40.53 g/L。该研究使用了功能成分皂苷作为促进酵母自溶的物质为之后的酵母自溶研究开辟了新的方向。酵母自溶法破壁原理简单,易于操作,破壁效果好,但需要的处理时间过长且处理所需的温度较高。因此,自溶期间可能会被致病菌和蛋白水解微生物所污染,引起腐烂从而降低酵母蛋白的质量[46]。如何加速酵母自溶,减少致病菌污染成了重点问题。FUNDO J F等[47]研究发现,将自溶时的pH值从5提高到8,在50 ℃时,可将自溶时间从4 h缩短至2 h。DIMOPOULOS G等[48]采用脉冲电场处理酵母细胞悬浮液使酵母细胞自溶加速来制备酵母提取物,最终氨基酸和总固体释放分别与未处理相比增加了37%和20%。
2.1.2 超声法破壁
超声法破壁是一种利用超声波的机械效应来破坏细胞结构的方法。这个过程通过超声波的振荡和产生的微小气泡爆破的效应,可以有效地破坏细胞壁,从而释放细胞内的物质。这一技术通常应用于细胞解裂、组织破碎、乳化、均质化等领域。EOM S J等[49]利用超声技术破裂细胞壁从而迅速提取酵母提取物,与使用常规化学提取方法相比总提取率提高了73.6%。ZHU L S等[50]用超声法提取废弃啤酒酵母中的蛋白质和多肽,其最大产率可达73.94%,且从废弃啤酒酵母中提取的蛋白质可作为高质量氨基酸的来源用于食品和饲料工业。综上,超声法破壁具有破壁率高、操作简单和产品无污染等特点,但超声法破壁也存在设备成本高、单次处理量低等问题需要改进。
2.1.3 微珠涡流法破壁
机械法破壁通过施加机械力来破坏细胞结构,从而释放细胞内的物质。这种方法的原理基于机械力对细胞壁或膜的物理作用,使其发生破裂和断裂,从而使细胞内的成分得以释放。HOMSI C等[51]开发了一种从酵母细胞中快速提取组蛋白的方法,使用酸性乙醇并且结合珠磨法从酵母细胞中提取组蛋白,这种方法提高了组蛋白的提取效率、减少了高分子质量蛋白质污染且简化了蛋白制备步骤。AVRAMIA I等[52]为了从废弃的酿酒酵母中提取β-葡聚糖和蛋白质,使用玻璃珠与酵母细胞悬浮液一起进行涡旋从而达到细胞破碎的目的,通过响应面法确定了最佳参数为:细胞悬浮液浓度5%,酵母细胞与玻璃珠比例为1∶2,涡旋循环次数为3。在此条件下破裂效率可达99.8%。机械破碎法虽然破碎效率高,但存在能量利用率低、机械装置提供的剪切力可能会导致目标蛋白变性失活、产生大量细胞碎片增加分离纯化的难度等问题。
2.1.4 高温碱热法破壁
高温碱热法是通过添加氢氧化钠或其他碱性物质破坏酵母细胞壁的结构,从而达到破壁的效果。此外,高温也能够加速生物化学反应,有助于更快的破坏酵母细胞壁。李强等[53]采用圆红冬孢酵母进行油脂发酵,并研究以其湿菌泥为原料,采用氢氧化钠热辅助破壁进行油脂提取的方法,研究发现,当氢氧化钠的添加量为0.2 g/g时,油脂提取率可达到99.3%。石豪磊等[54]通过正交试验优化碱法破壁工艺条件,即处理温度60 ℃,碱液添加量70 g/L,破壁时间1.5 h。此条件下得到的蛋白质含量为163.21 mg/L。FAUSTINO M等[55]研究从废弃酿酒酵母中提取甘露聚糖,使用碱热水解法获得了最高的生产产率(58.82%)。碱热法具有良好的破壁效果,破壁率高于其他方法,而且碱热法设备简单,反应时间短,所需化学试剂也相对便宜,但碱热法处理后的菌液无法长时间保存,同时加热的过程会导致一些风味氨基酸的流失,酵母菌液发生变质后也会影响后续蛋白的提取利用。尽管高温碱热法存在这些问题,但因其简单高效,仍被广泛运用于大规模工业化生产。
2.1.5 生物酶法破壁
对于添加外源酶裂解酵母细胞壁的研究最早要追溯到1988年Hunter和Asenjo对裂解酵母细胞的研究[56-58]。酵母细胞的细胞壁主要成分是β-葡聚糖(占酵母细胞壁干质量的60%)和少量的壳聚糖,内层结合着甘露蛋白(约占酵母细胞壁干质量的40%)[59]。可以利用特定的生物酶分解掉酵母细胞壁中的成分从而破坏酵母细胞壁,使细胞内物质能够释放出来。TAKALLOO Z等[60]比较了自溶、等离子体裂解和酶解三种方法对酵母裂解产生的可溶性蛋白含量的影响。研究表明,48 h后,酶解法的可溶性蛋白质含量为27.9%,分别为自溶法和等离子体裂解法的1.3倍和1.9倍。MARSON G V等[61]研究了废啤酒酵母的重新利用,研究发现酵母化合物在酶水解后释放效率更高,粗蛋白增加50%,蛋白质回收率提高83%,抗氧化能力提高63%。综上,酶法提取对蛋白质的提取更有效,但酶法具有处理时间长和成本相对较高的问题使之不能在大规模的工业生产中应用。
2.1.6 破壁工艺总结
在酵母破壁方法的研究中,探讨了从超声法到酶解法等不同的提取策略的优劣,汇总结果见表3。由表3可知,物理破壁方法一般具有更高的破碎效果和更多的处理量,但外力导致的蛋白变性和破碎产生的大量细胞碎片仍然会影响后续对蛋白质的利用。碱热法、自溶法和酶解法由于条件温和或处理量小更适合实验室规模的蛋白提取。
表3 各类破壁方法的优缺点比较
Table 3 Comparison of advantages and disadvantages of various cell wall breaking methods
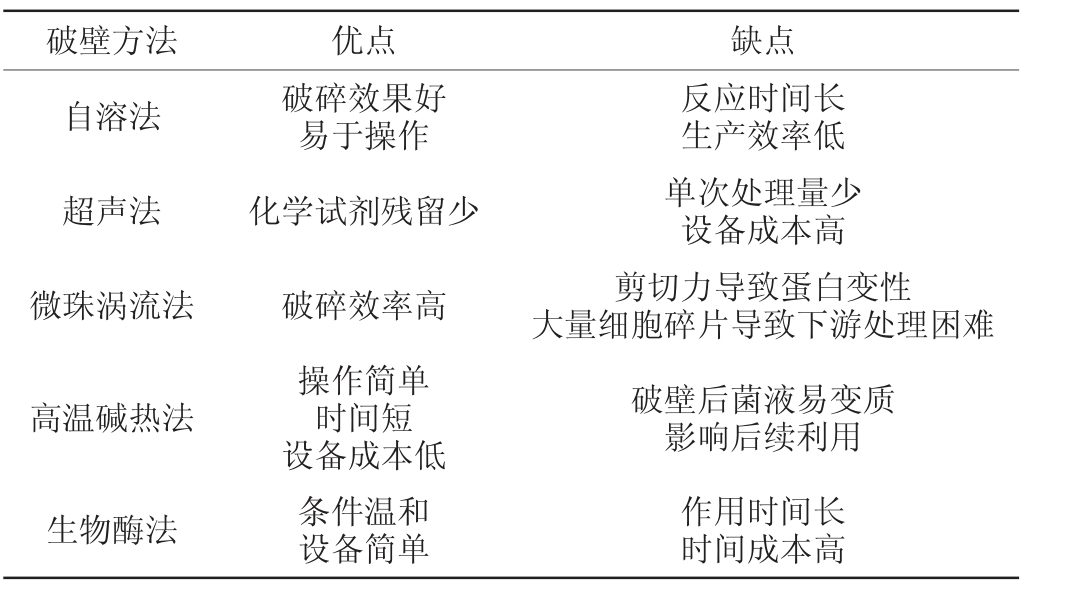
破壁方法 优点 缺点自溶法 破碎效果好易于操作超声法化学试剂残留少微珠涡流法高温碱热法生物酶法破碎效率高操作简单时间短设备成本低条件温和设备简单反应时间长生产效率低单次处理量少设备成本高剪切力导致蛋白变性大量细胞碎片导致下游处理困难破壁后菌液易变质影响后续利用作用时间长时间成本高
2.2 酵母蛋白提取技术研究
酵母细胞被破坏后,通过一定的方式除去细胞壁碎片,再用酸、碱、盐或加热进行进一步处理可以实现酵母蛋白的分离纯化[62]。
酵母蛋白可以通过碱溶酸沉的方法提取。酵母蛋白可以在碱性条件下溶解,因为碱性环境中,蛋白可以与脂质及其他不溶性组分分离[2]。随着提取液中氢氧化钠浓度的增加,酵母蛋白的溶解度和提取率均有提高[41]。再通过调节溶液pH值使达到蛋白质的等电点,使蛋白质溶解度降低从而使蛋白质溶液中沉淀出来完成分离。龚迪等[63]研究发现,在使用氢氧化钠法提取酵母蛋白质时加入适量的十二烷基磺酸钠可以抑制酵母中内源性酶对蛋白质的酶解,从而有效的增加酵母蛋白质的提取量。
除了碱溶酸沉的方法,琥珀酰化法也可以用来提取蛋白质。琥珀酰化是一种翻译后修饰,它可以在蛋白质赖氨酸残基上以共价键的方式添加一个琥珀基(-CO(CH2)2COOH)。酵母蛋白的琥珀酰化是由琥珀酰辅酶A的介导下将一个带负电荷的四碳琥珀酰基转移到蛋白质中赖氨酸残基的伯胺上的过程。这种修饰会改变蛋白质的功能、结构和稳定性。VANANUVAT P等[64]研究发现,琥珀酰化可以使蛋白质的溶解度提高,并使蛋白质的等电点从pH 4.5降低到4.0。与传统的碱溶酸沉法相比,琥珀酰化结合碱溶酸沉具有更高的蛋白提取率、更高的纯度以及更好的蛋白质稳定性和溶解度[65-66]。
3 总结与展望
酵母蛋白因其接近理想的氨基酸组成在未来一定会占据越来越多的市场份额。但现在对酵母蛋白的研究和利用仍然存在如下的问题:
①待开发一种无需水解处理的蛋白质组学分析方法。现在对蛋白质进行组学分析需要先对蛋白进行水解,水解的过程会影响酵母蛋白质组,导致蛋白质组学分析的结果出现不同。
②蛋白质组学现在更多被用在分析不同条件下酵母菌株的差异表达蛋白,而对于筛选酵母菌株方面则使用很少。
③目前对于不同种类的酵母菌株和不同条件下生长的酵母菌株的蛋白质组学研究很多,但对于不同方法提取后的酵母蛋白质组的研究较少,有些破壁提取方法势必影响酵母的蛋白质组,如生物酶法破壁、碱热法等。此外,酵母蛋白的提取仍然存在着如单次处理量少、设备成本高、生产效率低等问题,急需开发一种产业化可行的绿色高效的酵母蛋白制备方法。
参考文献:
[1]JACH M E,SEREFKO A,ZIAJA M,et al.Yeast protein as an easily accessible food source[J].Metabolites,2022,12(1):63.
[2]MA J R,SUN Y F,MENG D M,et al.Yeast proteins:The novel and sustainable alternative protein in food applications[J].Trend Food Sci Technol,2023,135:190-201.
[3]唐晓荞,武宇,樊军,等.酵母蛋白的营养质量评价[J].公共卫生与预防医学,2020,31(6):100-104.
[4]RODRIGUEZ L M,CAMINA J L,BORRONI V,et al.Protein recovery from brewery solid wastes[J].Food Chem,2023,407:134810.
[5]GRAHAM R L J,GRAHAM C,MCMULLAN G.Microbial proteomics:a mass spectrometry primer for biologists[J].Microb Cell Fact,2007,6:1-14.
[6]GANDHI A,SHAH N P.Integrating omics to unravel the stress-response mechanisms in probiotic bacteria:Approaches,challenges,and prospects[J].Crit Rev Food Sci Nutr,2017,57(16):3464-3471.
[7]O'FARRELL P H.High resolution two-dimensional electrophoresis of proteins[J].J Biol Chem,1975,250(10):4007-4021.
[8]龙娟,杨晓红,于桂宝.微生物蛋白组学的发展及前景[J].生物技术通报,2006(S1):91-94,99.
[9]MATTHIESEN R,BUNKENBORG J.Introduction to mass spectrometrybased proteomics[J].Mass spectrometry data analysis in proteomics,2013:1-45.
[10]KARAS M,BAHR U,GIEßMANN U.Matrix-assisted laser desorption ionization mass spectrometry[J].Mass Spectrom Rev, 1991, 10(5):335-357.
[11]FENN J B, MANN M, MENG C K, et al.Electrospray ionization for mass spectrometry of large biomolecules[J].Science,1989,246(4926):64-71.
[12]季美超,付斌,张养军.基于质谱的蛋白质组学方法新进展[J].质谱学报,2021,42(5):862-877.
[13]MANN M,HENDRICKSON R C,PANDEY A.Analysis of proteins and proteomes by mass spectrometry[J].Annu Rev Biochem, 2001, 70:437-473.
[14]AEBERSOLD R, MANN M.Mass spectrometry-based proteomics[J].Nature,2003,422(6928):198-207.
[15]DOMON B,AEBERSOLD R.Options and considerations when selecting a quantitative proteomics strategy[J].Nat Biotechnol,2010,28(7):710-721.
[16]HAQQANI A S,KELLY J F,STANIMIROVIC D B.Quantitative protein profiling by mass spectrometry using label-free proteomics[J].Methods Mol Biol,2008,439:241-256.
[17]LIU D T,YANG S,KAVDIA K,et al.Deep profiling of microgram-scale proteome by tandem mass tag mass spectrometry[J].J Proteome Res,2021,20(1):337-345.
[18]ZIESKE L R.A perspective on the use of iTRAQTM reagent technology for protein complex and profiling studies[J].J Exp Bot, 2006, 57(7):1501-1508.
[19]张茂娜,姜亮,张焱.硒代谢网络与硒蛋白质组的生物信息学研究进展[J].生物技术进展,2017,7(5):537-543.
[20]黄姗,时文六,白天,等.蛋白质组学技术在速冻食品中的应用研究进展[J].食品研究与开发,2023,44(22):180-185.
[21]马骏骏,王旭初,聂小军.生物信息学在蛋白质组学研究中的应用进展[J].生物信息学,2021,19(2):85-91.
[22]GAO Y, PING L Y, DUONG D, et al.Mass-spectrometry-based nearcomplete draft of the Saccharomyces cerevisiae proteome[J].J Proteome Res,2021,20(2):1328-1340.
[23]UEBAYASHI K,SHIMIZU H,MATSUDA F.Comparative analysis of fermentation and enzyme expression profiles among industrial Saccharomyces cerevisiae strains[J].Appl Microbiol Biotechnol, 2018, 102:7071-7081.
[24]ROSSIO V,LIU X Y,PAULO J A.Comparative proteomic analysis of two commonly used laboratory yeast strains:W303 and BY4742[J].Proteomes,2023,11(4):30.
[25]SANTOS R M, NOGUEIRA F C S, BRASIL A A, et al.Quantitative proteomic analysis of the Saccharomyces cerevisiae industrial strains CAT-1 and PE-2[J].J Proteom,2017,151:114-121.
[26]LI P S,FU X F,CHEN M,et al.Proteomic profiling and integrated analysis with transcriptomic data bring new insights in the stress responses of Kluyveromyces marxianus after an arrest during high-temperature ethanol fermentation[J].Biotechnol Biofuels,2019,12:1-13.
[27]AHMAD M,HIRZ M, PICHLER H, et al.Protein expression in Pichia pastoris: recent achievements and perspectives for heterologous protein production[J].Appl Microbiol Biotechnol,2014,98:5301-5317.
[28]SHEVCHENKO A,SUNYAEV S,LOBODA A,et al.Charting the proteomes of organisms with unsequenced genomes by MALDI-quadrupole time-of-flight mass spectrometry and BLAST homology searching[J].Anal Chem,2001,73(9):1917-1926.
[29]RENUSE S,MADUGUNDU A K,KUMAR P,et al.Proteomic analysis and genome annotation of Pichia pastoris,a recombinant protein expression host[J].Proteomics,2014,14(23-24):2769-2779.
[30]GARCIA-ALBORNOZ M, HOLMAN S W, ANTONISSE T, et al.A proteome-integrated,carbon source dependent genetic regulatory network in Saccharomyces cerevisiae[J].Mol Omics,2020,16:59-72.
[31]PAULO J A,O'CONNELL J D,GAUN A,et al.Proteome-wide quantitative multiplexed profiling of protein expression:carbon-source dependency in Saccharomyces cerevisiae[J].Mol Biol Cell,2015,26(22):4063-4074.
[32]ZHAO S H,ZHAO X R,ZOU H J,et al.Comparative proteomic analysis of Saccharomyces cerevisiae under different nitrogen sources[J].J Proteom,2014,101:102-112.
[33]NIGHTINGALE D J H, GELADAKI A, BRECKELS L M, et al.The subcellular organisation of Saccharomyces cerevisiae[J].Curr Opin Chem Biol,2019,48:86-95.
[34]刘秋影,李春生,杨贤庆,等.基于TMT的定量蛋白质组学技术解析盐胁迫提高库德毕赤酵母耐热性机制[J].食品科学,2022,43(14):102-110.
[35]FAKANKUN I, SPICER V, LEVIN D B.Proteomic analyses of the oleaginous and carotenogenic yeast Rhodotorula diobovata across growth phases under nitrogen-and oxygen-limited conditions[J].J Biotechnol,2021,332:11-19.
[36]JUSTINIC′ I,KATIC′ A,URŠICˇIC′ D,et al.Combining proteomics and lipidanalysistounravelConfidorstressresponse in Saccharomycescerevisiae[J].Environ Toxicol,2020,35(3):346-358.
[37]XU W N, WANG J J, LI Q.Comparative proteome and transcriptome analysis of lager brewer's yeast in the autolysis process[J].FEMS Yeast Res,2014,14(8):1273-1285.
[38]SUN Y,YU R F,GUO H B,et al.A quantitative yeast aging proteomics analysis reveals novel aging regulators[J].Geroscience, 2021, 43(5):2573-2593.
[39]苏星,张政,张棋炜,等.固态发酵下酵母自溶的工艺优化[J].生物工程学报,2019,35(4):726-736.
[40]PORRAS-AGÜERAJ A,MORENO-GARCÍA J,GONZÁLEZ-JIMÉNEZ M D C, et al.Autophagic proteome in two Saccharomyces cerevisiae strains during second fermentation for sparkling wine elaboration[J].Microorganisms,2020,8(4):523.
[41]岳红卫,韩富亮.酿酒酵母蛋白提取工艺优化[J].食品工业科技,2015,36(16):304-307,318.
[42]SZYMANSKI E P,KERSCHER O.Budding yeast protein extraction and purification for the study of function,interactions,and post-translational modifications[J].J Vis Exp,2013(80):50921.
[43]肖海芳,马海乐,孙进良,等.基于藻红蛋白提取的条斑紫菜脉冲超声破壁方法研究[J].食品科学,2007,28(11):199-202.
[44]易弋,容元平,程谦伟,等.不同破壁方法提取酵母菌总RNA的比较[J].食品科学,2011,32(11):161-164.
[45]BERLOWSKA J,DUDKIEWICZ-KOŁODZIEJSKA M,PAWLIKOWSKA E,et al.Utilization of post-fermentation yeasts for yeast extract production by autolysis:the effect of yeast strain and saponin from Quillaja saponaria[J].J I Brewing,2017,123(3):396-401.
[46]BATAEVA D S,MAKHOVA A A,KRYLOVA V B,et al.Clostridium spp.detection in food samples using 16S rDNA-based PCR method[J].IOP Conf Ser: Earth Environ Sci,2020,421(5):052025.
[47]FUNDO J F,DEUCHANDE T,RODRIGUES D A,et al.Induced autolysis of engineered yeast residue as a means to simplify downstream processing for valorization-a case study[J].Fermentation,2023,9(7):673.
[48]DIMOPOULOS G,STEFANOU N,ANDREOU V,et al.Effect of pulsed electric fields on the production of yeast extract by autolysis[J].Inn Food Sci Emerg Technol,2018,48:287-295.
[49]EOM S J,LIM T G,JHUN H,et al.Inhibitory effect of Saccharomyces cerevisiae extract obtained through ultrasound-assisted extraction on melanoma cells[J].Ultrason Sonochem,2021,76:105620.
[50]ZHU L S, WANG J F, FENG Y C, et al.Process optimization, amino acid composition, and antioxidant activities of protein and polypeptide extracted from waste beer yeast[J].Molecules,2022,27(20):6825.
[51]HOMSI C,RAJAN R E,MINATI R,et al.A rapid and efficient method for the extraction of histone proteins[J].J Proteome Res,2023,22(8):2765-2773.
[52]AVRAMIA I,AMARIEI S.A simple and efficient mechanical cell disruption method using glass beads to extract β-glucans from spent brewer's yeast[J].Appl Sci,2022,12(2):648.
[53]李强,黄其田,赵宗保.碱热法辅助提取圆红冬孢酵母油脂[J].可再生能源,2020,38(11):1421-1426.
[54]石豪磊,赵国正,孔祥君,等.碱法破壁-酶法提取葡萄酒废酵母细胞壁多糖的工艺研究[J].中国酿造,2018,37(12):82-86.
[55]FAUSTINO M, DURÃO J, PEREIRA C F, et al.Comparative analysis of mannans extraction processes from spent yeast Saccharomyces cerevisiae[J].Foods,2022,11(23):3753.
[56]HUNTER J B,ASENJO J A.Kinetics of enzymatic lysis and disruption of yeast cells:I.Evaluation of two lytic systems with different properties[J].Biotechnol Bioeng,1987,30(4):471-480.
[57]HUNTER J B,ASENJO J A.Kinetics of enzymatic lysis and disruption of yeast cells:II.A simple model of lysis kinetics[J].Biotechnol Bioeng,1987,30(4):481-490.
[58]HUNTER J B,ASENJO J A.A structured mechanistic model of the kinetics of enzymatic lysis and disruption of yeast cells[J].Biotechnol Bioeng,1988,31(9):929-943.
[59]YAMMINE M,BRAY F,FLAMENT S,et al.Reliable approach for pure yeast cell wall protein isolation from Saccharomyces cerevisiae yeast cells[J].ACS Omega,2022,7(34):29702-29713.
[60]TAKALLOO Z,NIKKHAH M,NEMATI R,et al.Autolysis,plasmolysis and enzymatic hydrolysis of baker's yeast(Saccharomyces cerevisiae):a comparative study[J].World J Microbiol Biotechnol,2020,36:1-14.
[61]MARSON G V,MACHADO M T D C,DE CASTRO R J S,et al.Sequential hydrolysis of spent brewer's yeast improved its physico-chemical characteristics and antioxidant properties:A strategy to transform waste into added-value biomolecules[J].Process Biochem,2019,84:91-102.
[62]JACOB F F,HUTZLER M,METHNER F J.Comparison of various industrially applicable disruption methods to produce yeast extract using spent yeast from top-fermenting beer production:influence on amino acid and protein content[J].Eur Food Res Technol,2019,245:95-109.
[63]龚迪,毕阳,王军节,等.基于蛋白组学的水果蛋白质提取方法[J].食品工业科技,2014,35(3):376-379.
[64]VANANUVAT P, KINSELLA J E.Preparation of succinylated yeast protein:Composition and solubility[J].Biotechnol Bioeng,1978,20(9):1329-1344.
[65]EINHORN-STOLL U, KRETZSCHMAR U, LIPPERT E.Enzymatic modification of yeast protein isolates[J].Acta Biotechnol,1994,14(4):379-385.
[66]SCENI P,PALAZOLO G G,VASALLO M D C,et al.Thermal and surface behavior of yeast protein fractions from Saccharomyces cerevisiae[J].LWT-Food Sci Technol,2009,42(6):1098-1106.


