中国白酒是世界六大蒸馏酒之一,在不同地理环境与酿造工艺条件下,形成了以浓香型、清香型、酱香型、米香型为主的4大基本香型白酒[1]。 浓香型白酒以泥窖发酵、续糟配料、混蒸混烧为特征工艺酿造而成,具有窖香浓郁、口感绵柔、余味悠长等特点,深受广大消费者喜爱[2]。白酒质量通常由自然环境、酿造工艺、窖泥质量等因素共同决定,其中窖泥质量是影响白酒风味的重要因素[3-4]。
窖泥通常是指附着在窖壁与底部的一层富含酿酒微生物的特殊土壤,是浓香型白酒发酵的重要载体[5]。人工老熟窖泥是以优质泥土为基础,添加模拟自然老窖微生态环境的复合微生态菌群,并辅以微生物生长所需的营养物质,营造有益微生物的生长环境,经过发酵培养得到的一种接近自然老熟窖泥质量的窖泥,具有快速富集微生物,缩短窖泥老熟时间等优点,可作为自然老熟窖泥替代品[6-7]。近年来,窖泥研究主要集中在自然老熟窖泥微生物菌群组成,CHEN C等[8]研究发现,浓香型白酒自然窖泥中常见的细菌属为己酸菌属(Caproiciproducens)、乳酸杆菌属(Lactobacillus)等;HE G Q等[9]研究发现,5年、20年、50年窖龄窖泥的主要优势菌门为拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和子囊菌门(Ascomycota);任海伟等[10]利用高通量测序技术检测浓香型白酒窖泥中细菌菌群,结果表明,己酸菌属是50年自然成熟老窖泥的绝对优势菌属,能量代谢、氨基酸代谢、碳水化合物代谢是窖泥微生物代谢的主要方式;但有关于人工老熟窖泥与自然老熟窖泥间微生物群落差异及代谢功能对比研究较少,明确人工老熟窖泥与自然老熟窖泥中的微生物群落组成差异与功能代谢对进一步提升人工老熟窖泥质量有重要意义。
高通量检测技术是目前微生物多样性检测常用的方法之一,其准确性高,重复性好,具有高灵敏度、高精度等特点,能够更加全面反映样品群落信息组成情况,对于全面揭示窖泥微生物组成信息具有重要作用[11-13]。 本研究基于高通量测序技术检测人工老熟窖泥与自然老熟10年、50年窖泥的微生物多样性,并使用PICRUSt2软件对窖泥细菌菌群的代谢功能进行预测,分析人工老熟窖泥与自然老熟10年、50年窖泥之间微生物群落组成及代谢功能的差异,以期为浓香型白酒窖池养护与人工窖泥质量提升提供理论依据与数据支撑。
1 材料与方法
1.1 材料与试剂
1.1.1 窖泥样品
窖泥样品取自西北某浓香型酒厂酿酒窖池,随机取5个窖池作为平行样品,采用5点取样法从每个窖池底部采集窖泥,混匀。自然老熟10年的窖泥标记为Ypm、自然老熟50年的窖泥标记为Opm,人工老熟1年的窖泥标记为Apm,放入-80 ℃冰箱保存,待测。
1.1.2 试剂
TransStart聚合酶试剂盒:北京全式金生物技术有限公司;Novaseq试剂盒:美国Illumina公司;VAHTS脱氧核糖核酸(deoxyribonucleic acid,DNA)Clean Beads试剂盒:南京诺唯赞生物科技有限公司;GeneScan-LIZ120:美国Thermo Fisher Scientific公司。
1.2 仪器与设备
Qubit 3.0荧光定量仪、ABI 2720 Thermal Cycler自动化聚合酶链式反应(polymerase chain reaction,PCR)仪:美国Thermo Fisher Scientific公司;Vortex Genius 3漩涡混合器:德国IKA公司;Agilent 2100 bioanalyzer生物分析仪:美国Agilent Technologies公司。
1.3 方法
1.3.1 窖泥细菌菌群宏基因组DNA的提取及Illumina MiSeq高通量测序
采用TransStart聚合酶试剂盒提取窖泥细菌菌群宏基因组DNA,通过1.0%琼脂糖凝胶电泳检测宏基因组DNA的完整性,采用Qubit 3.0荧光定量仪定量检测宏基因组DNA质量。以提取的宏基因组DNA为模板,采用引物515F(5'-GTGCCAGCMGCCGCGGTAA-3')与907R(5'-CCGTCAATTCMTTTRAGTTT-3')PCR扩增细菌菌群16S rRNA基因的V4-V5区基因序列。PCR扩增体系(25 μL):2×Trans Start FastPfu Mix 12.5 μL,515F(10 μmol/L)0.5 μL,907R(10 μmol/L)0.5 μL,基因组DNA模板1.0 μL(10~50 ng),双蒸水(ddH2O)10.5 μL。PCR扩增条件:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,共30个循环;72 ℃再稳定延伸10 min。PCR扩增产物经2%琼脂糖凝胶电泳检测合格后,基于Illumina MiSeq平台进行高通量测序[5]。
1.3.2 数据处理
使用FastQC进行原始数据质量评估,通过DADA2进行质控、去噪和嵌合体过滤后生成扩增子序列变体(amplicon sequence variants,ASVs)。 随后基于SILVA数据库(v138)进行物种注释,并采用R语言进行Alpha多样性和Beta多样性分析。基于京都基因与基因组百科全书(KyotoEncyclopedia of Genes and Genomes,KEGG)数据库采用PICRUSt2软件进行代谢功能预测。使用Origin 2022绘图,所有试验均重复3次,结果以“平均值±标准差”表示。
2 结果与分析
2.1 窖泥样品中细菌菌群高通量测序结果分析
基于IlluminaMiSeq平台对不同窖泥样品中的细菌群落进行高通量测序,优化后共得到1735628条有效序列,覆盖率为99.99%。为了评估测序深度是否足以反映样品中细菌群落的真实多样性,基于Shannon指数绘制稀释曲线,结果见图1。
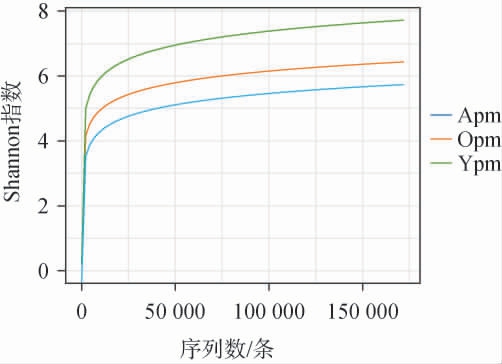
图1 人工老熟窖泥与自然老熟窖泥中细菌菌群的稀释曲线
Fig.1 Rarefaction curves of bacteria community in artificially and naturally aged pit mud
由图1可知,随着测序深度的增加,所有窖泥样品细菌菌群的稀释曲线均呈先急剧升高后逐渐趋于平缓的趋势,表明本次测样量充足,测序数据合理,可进行后续分析[14]。
2.2 窖泥样品中细菌群落Alpha多样性分析
人工老熟窖泥与自然老熟窖泥中细菌群落的Alpha多样性分析结果见表1。Chao1指数与ACE指数可反映微生物群落的丰富度,其数值越大,表示微生物群落丰富度越高;Shannon与Simpson指数代表微生物群落多样性,Shannon指数越大,Simpson指数越小,表示微生物群落多样性越高[15]。 由表1可知,自然老熟50年窖泥中细菌群落的Chao1指数、ACE指数及Shannon指数最高,Simpson指数最低,其次为自然老熟10年窖泥,说明自然老熟50年窖泥细菌菌群丰富度与多样性最高,自然老熟10年窖泥次之,人工老熟1年窖泥最低,这与任海伟等[10]研究的浓香型白酒不同窖龄窖泥微生物群落多样性结果一致。人工老熟培养配料工艺与发酵方式形成的生长环境可能存在营养物质匮乏以及对微生物适宜的环境条件难以精确控制,是造成人工老熟窖泥中微生物多样性较低的主要原因[16]。
表1 人工老熟窖泥与自然老熟窖泥中细菌群落的Alpha多样性分析结果
Table 1 Alpha diversity analysis results of bacterial communities in artificially and naturally aged pit mud
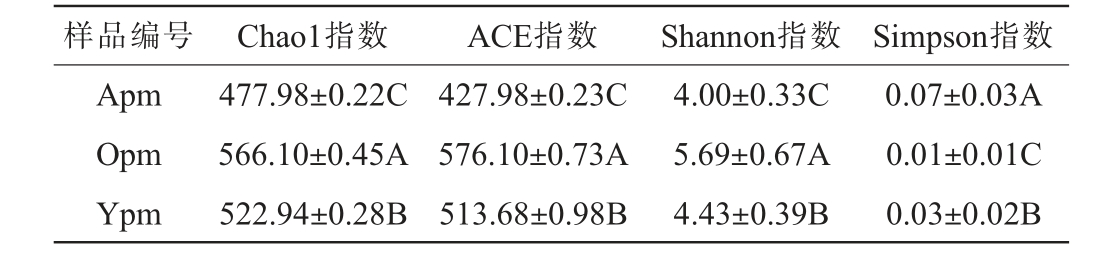
注:同列不同大写字母表示差异显著(P<0.05)。
样品编号 Chao1指数 ACE指数 Shannon指数 Simpson指数Apm Opm Ypm 477.98±0.22C 566.10±0.45A 522.94±0.28B 427.98±0.23C 576.10±0.73A 513.68±0.98B 4.00±0.33C 5.69±0.67A 4.43±0.39B 0.07±0.03A 0.01±0.01C 0.03±0.02B
2.3 窖泥样品中细菌群落Beta多样性分析
2.3.1 基于门水平人工老熟窖泥与自然老熟窖泥细菌菌群结构分析
基于门水平,人工老熟窖泥与自然老熟窖泥中细菌群落结构见图2。由图2可知,从人工老熟窖泥与自然老熟窖泥样品中共注释到38个细菌门,包括4个优势细菌门(相对丰度>1%[17]),人工老熟1年窖泥中优势细菌门为变形菌门(Proteobacteria)(77.26%)、拟杆菌门(Bacteroidetes)(9.25%)、厚壁菌门(Firmicutes)(7.53%)、放线菌门(Actinobacteriota)(1.49%);自然老熟10年窖泥中优势细菌门为厚壁菌门(70.99%)、放线菌门(1.91%)、拟杆菌门(1.09%);自然老熟50年窖泥中优势菌门为厚壁菌门(80.60%)、拟杆菌门(15.86%)、放线菌门(1.49%)。这与肖琴等[14]研究的浓香型白酒不同窖龄窖泥中优势菌门基本相似。厚壁菌门是衡量窖泥老熟程度的标志菌门,在人工老熟窖泥与自然老熟窖泥中均有分布,但在自然老熟50年窖泥中相对丰度最高,自然老熟10年窖泥次之,人工老熟1年窖泥中相对丰度最低。人工老熟1年窖泥中细菌菌群以变形菌门为主,厚壁菌门、拟杆菌门相对丰度偏低,这与姜雨函[18]研究的人工老熟窖泥主要菌群结果一致。
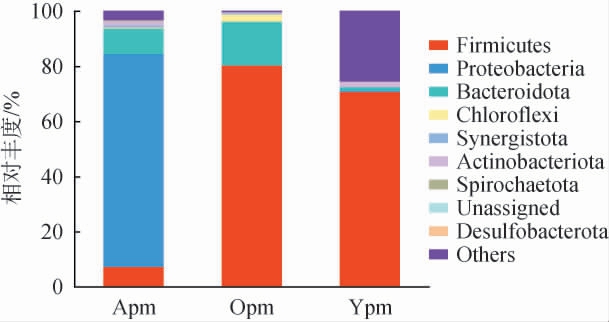
图2 基于门水平人工老熟窖泥与自然老熟窖泥中细菌群落结构
Fig.2 Bacterial community structure in artificially and naturally aged pit mud at the phylum level
窖泥成熟过程受到多种因素共同影响,如环境温度、湿度、水分含量、营养成分等[5]。任海伟等[10]研究发现,厚壁菌门是老窖泥独有的菌门之一,能够参与有机物的水解和酸化以及淀粉、纤维素等复杂有机物的降解,除此之外还能产生芽孢,平衡窖泥水分与酸碱度,保持窖池环境稳定,因此窖龄越长厚壁菌门丰度越高,这与本试验结果一致[4]。CHEN C等[8]研究发现,拟杆菌门相对丰度随着年份增加呈上升趋势,这与本试验结果一致,拟杆菌门微生物能够发酵糖、有机酸和氨基酸,生成与白酒风味密切相关的乙酸或丙酸等,为白酒提供独特的口感和风味,同时拟杆菌门微生物能够在代谢过程中将窖池中的Fe3+转化为Fe2+,使窖泥从黄色转化为老熟窖泥的青灰色,是从感官判断窖泥老熟程度的方法之一[19-20]。人工老熟1年窖泥培养过程中菌群富集方式及营养物质添加的不同导致其菌群结构复杂,变形菌门在人工老熟窖泥中处于绝对优势地位,变形菌门中的许多菌属偏好较丰富的有机质和氧气,而新培养的人工窖泥的理化环境更适合其生长[17]。
2.3.2 基于属水平人工老熟窖泥与自然老熟窖泥细菌菌群结构分析
基于属水平人工老熟窖泥与自然老熟窖泥中细菌群落结构见图3。
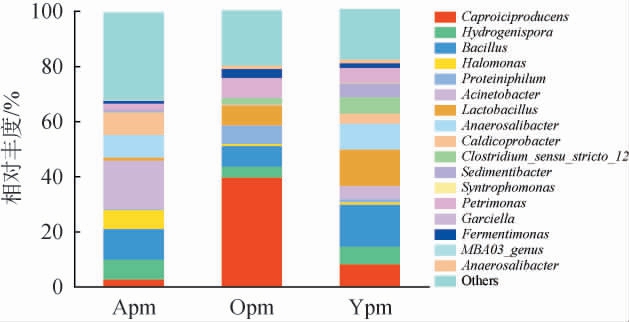
图3 基于属水平人工老熟窖泥与自然老熟窖泥中细菌菌群结构
Fig.3 Bacterial community structure in artificially and naturally aged pit mud at the genus level
由图3可知,从人工老熟窖泥与自然老熟窖泥样品中共注释到488个细菌菌属,包括18个优势细菌属(相对丰度>1%)。 人工老熟1年窖泥中的优势细菌属为不动杆菌属(Acinetobacter)(17.56%)、芽孢杆菌属(Bacillus)(11.04%)、厌氧杆菌属(Anaerosalibacter)(8.19%)、氢孢菌属(Hydrogenispora)(7.22%)、乳酸杆菌属(Lactobacillus)(7.09%)、盐单胞菌属(Halomonas)(7.00%)、己酸菌属(Caproiciproducens)(2.98%)等;自然老熟10年窖泥中优势细菌属为乳酸杆菌属(13.23%)、己酸菌属(10.44%)、厌氧杆菌属(9.45%)、氢孢菌属(6.45%)、芽孢杆菌属(5.25%)等;自然老熟50年窖泥中的优势细菌属为己酸菌属 (39.91%)、芽孢杆菌属(7.53%)、乳酸杆菌属(7.12%)、嗜蛋白菌属(Proteiniphilum)(6.52%)、氢孢菌属(4.04%)等。 人工老熟1年窖泥中不动杆菌属相对丰度最高,自然老熟窖泥中己酸菌属相对丰度最高,且自然老熟50年窖泥中己酸菌属远高于自然老熟10年窖泥,人工老熟1年窖泥己酸菌属相对丰度更低。 从人工老熟1年的窖泥到自然老熟50年的窖泥菌属丰度来看,菌群结构类似但菌群丰度发生着巨大变化,在窖泥老熟过程中,窖泥高酸厌氧环境不断筛选着优势菌群,菌群逐渐适应窖池环境,窖泥功能模块逐渐完善[21]。
己酸菌属是浓香型白酒窖泥的优势功能菌群,吴玉轩[22]研究发现,窖龄时间越长,己酸菌属相对丰度越高,窖泥功能菌群越稳定,更有利于浓香型白酒风味物质形成,与本研究检测结果一致,己酸菌属的相对丰度在一定程度上可表明窖泥质量[23];曾波等[24]研究发现,乳酸杆菌属是老熟时间短的窖泥中的优势菌属之一,窖泥质量越高,乳酸杆菌属的相对丰度逐渐下降,这与本研究结果相似,乳酸杆菌属是窖泥中一类非常重要的兼性厌氧微生物,可代谢产生有机酸,降低发酵体系的pH值,为酵母菌等有益微生物提供适宜的生长环境,有利于白酒的发酵和保持酿酒微生态环境[25];芽孢杆菌属在自然老熟50年窖泥中相对丰度最高,自然老熟10年窖泥次之,这可能与芽孢杆菌属作用机理有关,芽孢杆菌属在浓香型白酒发酵过程中承担着水解淀粉、蛋白质与产生有机酸等重要功能,承担着产酸与酯化前体物质的生成[26];不动杆菌属是一类兼性厌氧菌,环境适应性更强,此外人工老熟窖泥培养过程中的特定理化条件更适合不动杆菌生长最适条件,因此成为人工老熟窖泥的优势菌群[27]。
2.4 窖泥样品中细菌菌群KEGG代谢功能预测结果
基于KEGG数据库对不同窖泥中细菌菌群的代谢功能进行预测,结果见图4。由图4可知,窖泥中的细菌菌群主要涉及12种代谢通路,丰度较高的为碳源代谢、氨基酸代谢、能量代谢、辅助因子和维生素代谢、脂类代谢。 碳源代谢主要包括淀粉、有机酸代谢、糖类物质代谢等,承担着能量转化、风味物质形成等重要作用,碳源代谢强弱一定程度上也能够表示窖泥质量[28],氨基酸代谢主要包括氨基酸的生物合成及转化利用,氨基酸代谢能够为微生物提供氮源与反应所需酶类,是浓香型白酒风味物质形成的重要条件之一[29];能量代谢是窖泥微生物生命活动的核心,为菌群生长、物质合成及风味物质形成提供能量,如糖酵解途径等[29];辅助因子和维生素代谢包括微生物分泌酶类参与脂肪酸代谢及部分微生物合成维生素,辅助因子和维生素代谢直接影响窖泥功能微生物的活性,进而调控白酒风味物质[30];脂类代谢主要包括脂肪酸合成与降解,直接参与窖泥中风味脂类的形成[26]。
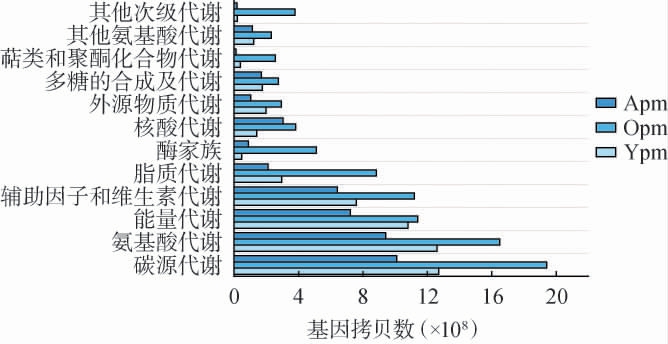
图4 人工老熟窖泥与自然老熟窖泥细菌群落KEGG代谢功能预测结果
Fig.4 Prediction results of KEGG metabolic functions of bacterial communities in artificially and naturally aged pit mud
人工老熟窖泥与自然老熟窖泥细菌菌群整体代谢能力强弱排序为自然老熟50年窖泥>自然老熟10年窖泥>人工老熟1年窖泥。自然老熟窖泥碳代谢功能更强是因为其中微生物群落长期适应高酸的窖池环境,形成了稳定的碳利用网络(如梭菌属降解纤维素、芽孢杆菌代谢淀粉),代谢效率更高,而人工老熟的窖泥老熟时间短,菌群结构简单,碳利用网络转化能力不足[28];自然老熟窖泥氨基酸代谢功能更强是因为其长期富集的微生物具备更完整的氨基酸代谢途径,而人工老熟的窖泥中功能菌群简单、老熟程度不足限制了复杂氨基酸代谢途径的建立[31]。自然老熟窖泥长期驯化的微生物能量代谢效率高,能协同利用复杂碳源(如淀粉、纤维素),维持窖池微生态稳定,而人工老熟窖泥能量代谢途径单一(如仅依赖糖酵解),三磷酸腺苷(adenosine triphosphate,ATP)供应不足,限制风味物质合成[15];自然老熟窖泥功能微生物协同作用强,能够高效合成并循环利用辅酶和维生素支撑复杂的代谢网络,而人工老熟1年窖泥微生物多样性低、互作弱,且缺乏长期驯化,导致辅助因子合成不足、代谢途径不完整,整体代谢效率较低[32];自然老熟窖泥富含己酸菌等功能菌群,脂类代谢能力强,风味物质积累多,而人工老熟窖泥功能菌群不完善,脂肪酸合成效率低,需外源添加专一性酶[26]。由此可见,自然老熟50年窖泥代谢能力最强,功能结构更加完善,自然老熟10年窖泥正处于优化时期,功能结构逐渐完善,人工老熟1年窖泥由于培养方式对窖泥功能菌定向培养不足,与酿酒相关的功能结构并不完整。
3 结论
基于高通量测序结果来看,自然老熟50年窖泥细菌菌群丰富度与多样性最高,其次是自然老熟10年窖泥,人工老熟1年窖泥最低。 在门、属水平,人工老熟窖泥与自然老熟窖泥的细菌菌群结构组成并无明显差异,但相对丰度差异明显。在门水平上,人工老熟1年窖泥样品中变形菌门(Proteobacteria)相对丰度最高,自然老熟窖泥样品中厚壁菌门(Firmicutes)相对丰度最高。 人工老熟1年窖泥样品中关键功能菌门的相对丰度还未达到自然老熟10年窖泥样品水平;在属水平上,人工老熟1年窖泥样品中不动杆菌属(Anaerosalibacter)相对丰度最高,己酸菌属(Caproiciproducens)作为评价窖泥老熟程度的关键菌属,在自然老熟窖泥样品中相对丰度最高,人工老熟1年窖泥样品中最低。窖泥细菌菌群主要涉及12个主要代谢通路,其中碳源代谢、氨基酸代谢、能量代谢、辅助因子和维生素代谢、脂质代谢能力最强,且窖泥细菌菌群代谢强弱排序为自然老熟50年窖泥>自然老熟10年窖泥>人工老熟1年窖泥,自然老熟50年的窖泥代谢能力更强,窖泥功能结构更完善。本研究结果对于揭示人工老熟窖泥与自然老熟窖泥的细菌群落结构差异、解析窖龄对微生物群落和代谢功能的影响、优化人工窖泥制备工艺、改进人工窖泥的性能具有重要的数据支撑与理论依据。
[1]LI J Y, DING Z Q, DONG W, et al.Analysis of differences in microorganisms and aroma profiles between normal and off-flavor pit mud in Chinese strong-flavor Baijiu[J].J Biosci Bioeng,2024,137(5):360-371.
[2]胡景辉,陈禹锜,薛新新,等.浓香型白酒发展概述[J].中国酿造,2022,41(6):24-30.
[3]刘治国,方超,张晓元,等.浓香型白酒窖泥理化因子、微生物群落、感官质量以及相关性研究进展[J].食品科学,2023,44(21):351-358.
[4]HUANG Z G,ZENG B,DENG J,et al.Succession of microbial community structure in fermented grains during the fermentation of strong-flavor Baijiu and its impact on the metabolism of acids, alcohols, and esters[J].Food Sci Biotechnol,2024,33(15):3501-3513.
[5]靳喜庆,冯亮,张无疾,等.白酒人工窖泥培养方法研究进展[J].中国酿造,2024,43(7):7-13.
[6]MAO F J,HUANG J,et al.Succession of microbial community of the pit mud under the impact of Daqu of Nongxiang Baijiu[J].J Biosci Bioeng,2023,136(4):304-311.
[7]SUN Z K,CHEN C,HOU X G,et al.Prokaryotic diversity and biochemical properties in aging artificial pit mud used for the production of Chinese strong flavor liquor[J].3 Biotech,2017,7(5):335-337.
[8]CHEN C,YANG H Q,ZHANG K Z,et al.Revealing microbiota characteristics and predicting flavor-producing sub-communities in Nongxiangxing Baijiu pit mud through metagenomic analysis and metabolic modeling[J].Food Res Int,2024,118(5):190-193.
[9]HE G Q,GAO L,DENG Y,et al.Microbial succession in different years of pit mud from a distillery in Sichuan for Nong-xiang Baijiu fermentation[J].Food Sci Biotechnol,2024,33(13):3083-3092.
[10]任海伟,孙一帆,王希,等.不同窖龄及位置浓香型白酒窖泥中细菌群落结构的差异性分析[J].食品与发酵工业,2023,49(9):103-111.
[11]LIU Y B, SUN M X, HOU P, et al.Analysis of microbial community structure and volatile compounds in pit mud used for manufacturing Taorong-type Baijiu based on high-throughput sequencing[J].Sci Rep,2022,12(1):7347.
[12]GUTIÉRREZ-GONZÁLEZA A,HIERRO I D,CARIAGA-MARTÍNEZ A E,et al.Advancements in multiple myeloma research:High-throughput sequencing technologies,omics,and the role of artificial intelligence[J].Biology,2024,13(11):923-925.
[13]QUIJADA N M, HERNÁNDEZ M, RODRÍGUEZ-LÁZARO D.Highthroughput sequencing and food microbiology[J].Adv Food Nutr Res,2020,54(8):295-300.
[14]肖琴,何平,周瑞平,等.不同窖龄及位置浓香型白酒窖泥微生物群落多样性与理化因子的比较分析[J].食品科学,2023,44(20):165-174.
[15]REN D L,LIU S P,QIN H,et al.Heterogenetic mechanism in multidimensional pit mud with different fermentation years: From microbial structure dynamic succession to metabolism phenotypes[J].Food Res Int,2024,192:114770.
[16]马龙,林一心,尉军强,等.西北地区人工窖泥与老窖泥细菌群落结构差异[J].中国酿造,2023,42(11):104-108.
[17]MU Y,HUANG J,ZHOU R Q,et al.Effects of Daqu attributes on distribution and assembly patterns of microbial communities and their metabolic function of artificial pit mud[J].Foods,2022,11(18):2922.
[18]姜雨函.不同配方人工窖泥的特性比较及应用研究[D].成都:四川大学,2021.
[19]CHEN S Q,HUANG J,QIN H,et al.Characterizing the interaction relationship of the microbial communities between Zaopei and pit mud disturbing by Daqu[J].Food Sci Biotechnol,2021,30(10):1357-1367.
[20]ZHOU W,LIAO Z M,WU Z Y,et al.Analysis of the difference between aged and degenerated pit mud microbiome in fermentation cellars for Chinese Luzhou-flavor Baijiu by metatranscriptomics[J].J Sci Food Agr,2021,101(11):4621-4631.
[21]王乐,王耀,韩怀斌,等.不同窖龄窖泥产酒发酵前后的理化指标、风味物质含量和细菌菌群结构差异分析[J].酿酒科技,2023(7):22-33,43.
[22]吴玉轩.浓香型白酒窖泥己酸菌丰度分析与应用研究[D].济南:齐鲁工业大学,2023.
[23]FLAIZ M,BAUR T,BRAHNER S,et al.Caproicibacter fermentans gen.nov.,sp.nov.,a new caproate-producing bacterium and emended description of the genus Caproiciproducens[J].Int J Syst Evol Micr,2020,70(7):4269-4279.
[24]曾波,蒲吉洲,陈秋旭,等.不同位置窖泥酶活力及其细菌群落结构相关性分析[J].食品与发酵工业,2024,50(4):102-109.
[25]ZHOU H,XU S S,XU B Y,et al.Effect of Caproicibacterium lactatifermentans inoculation on the microbial succession and flavor formation of pit mud used in Chinese Baijiu fermentation[J].Food Res Int,2024,28(8):1219-1290.
[26]CHAI L J,QIAN W,ZHONG X,et al.Mining the evolutionary patterns of pit mud microbiome and driving factors under the impact of longterm strong-flavor Baijiu production[J].Appl Environ Microbiol,2021,87(17):521-533.
[27]LIU M K,TANG Y M,GUO X J,et al.Structural and functional changes in prokaryotic communities in artificial pit mud during Chinese Baijiu production[J].mSystems,2020,5(2):e00829-19.
[28]周文,舒学香,张崇军,等.宜宾地区不同性状窖泥的细菌群落结构解析及代谢功能预测[J].中国酿造,2022,41(8):110-114.
[29]毕天然,黄钧,张宿义,等.不同窖龄及位置窖泥微生物群落和代谢组分的差异[J].食品与发酵工业,2022,48(2):231-237.
[30]MU Y,HUANG J,ZHOU R Q,et al.Response and assembly of abundant and rare taxa in Zaopei under different combination patterns of Daqu and pit mud: from microbial ecology to Baijiu brewing microecosystem[J].Food Science and Human Wellness,2024,13(3):1439-1452.
[31]CAO L B,SUN H M,XU Z Y,et al.Metagenomic and physicochemical profiling reveal microbial functions in pit mud for Jiang-Nong Jianxiang Baijiu fermentation[J].BMC Microbiol,2025,25(1):190-209.
[32]WAN Y,HUANG J,TANG Q,et al.Characterizing the contribution of functional microbiota cultures in pit mud to the metabolite profiles of fermented grains[J].Foods,2024,13(11):1150-1163.