越南米酒在越南地区是最具乡土特色的酒以糯米等谷物为原料,以米酒曲为糖化发酵剂,经蒸煮、发酵而成。米酒曲微生物菌群丰富,在米酒的发酵过程中扮演着重要角色,对其产品品质的形成具有重要的作用[1]。越南米酒曲多以大米或草本植物为原料,经自然发酵、挤压成型等传统手工工序制成,蕴含根霉、毛霉及少量酵母等微生物,其中根霉产生的糖化酶能将糯米淀粉高效水解为葡萄糖[2]。
近年来,对米酒曲的研究集中在理化指标、微生物菌群等方面,其受制作原料、工艺和地域环境等因素的影响,不同地区的米酒曲其蕴含的微生物类群各不相同[3]。通过采用纯培养技术,发现陕北和苏州等地区米酒曲中相关优势真菌为霉菌和酵母菌[4-5]。在采用纯培养技术的基础上,通过采用变性凝胶梯度电泳技术对孝感地区酒曲微生物进行解析发现,其优势细菌属为乳酸杆菌属和魏斯氏菌属[6-7]。以MiSeq为代表的第二代高通量测序技术以其检测数据量大、准确快速的特点,成为研究微生物群落结构和功能的重要工具[8]。通过采用该技术对各地区的米酒曲进行解析,其中朝鲜族传统米酒曲优势真菌属为假单胞菌属(Pseudomonas)、肠杆菌属(Enterobacter),优势菌种为普鲁蓝久浩酵母(Guehomyces pullulans)[9];海南山兰米酒曲真菌属主要有根霉属(Rhizopus)、假丝酵母属(Candida)等,细菌属主要有片球菌(Pediococcus)等[10];孝感凤窝酒曲优势真菌属主要有淀粉霉(Amylomyces)和扣囊复膜孢酵母属(Saccharomycopsis)等[11],优势细菌属为魏斯氏菌属(Weissella)和乳酸杆菌属(Lactobacillus)[12],甘晋铭等[13]研究表明,越南地区米酒曲霉菌及细菌总数高于中国苏州米酒曲。目前对越南米酒曲的相关研究较少,通过对越南米酒曲的微生物及理化指标进行分析,旨在科学阐释其独特风味的微生物成因,并挖掘具有优良糖化发酵特性的功能菌种。
为分析越南地区米酒曲中微生物群落结构组成差异,本研究以越南5个地区米酒曲样品(YB、Y2G、YNNB、YNG1、YNG2)为研究对象,采用常规方法测量其理化指标,通过MiSeq高通量测序技术解析了的微生物群落结构,并基于检测结果进行主坐标分析(principal coordinate analysis,PCoA)及相关性分析,旨在解析越南地区不同米酒曲样品中微生物菌群,为米酒品质提升及工业生产提供理论依据及技术支撑。
1 材料与方法
1.1 材料与试剂
米酒曲样品(YB、Y2G、YNNB、YNG1、YNG2):样品YB来自越南北部河内市,样品Y2G来自越南中部岘港市,样品YNNB来自越南南部胡志明市,样品YNG1来自越南海防市,样品YNG2来自越南芽庄市。
FastDNA Spin Kit for Soil脱氧核糖核酸(deoxyribonucleic acid,DNA)抽提试剂盒:美国Mp Biomedicals公司;NEXTFLEX Rapid DNA-SeqKit建库试剂盒:北京柏奥泰科技有限公司;MiSeq Reagent Kit测序试剂盒:美国Illumina公司。
Spin Kit for Soil脱氧核糖核酸(deoxyribonucleic acid,DNA)抽提试剂盒:美国Mp Biomedicals公司;NEXTFLEX Rapid DNA-SeqKit建库试剂盒:北京柏奥泰科技有限公司;MiSeq Reagent Kit测序试剂盒:美国Illumina公司。
1.2 仪器与设备
5430 R型高速台式冷冻离心机:德国Eppendorf公司;Wonbio-L型粉碎研磨仪:上海万柏生物科技有限公司;QuantusTM型微型荧光计:美国Promega公司;DYY-16D型电泳仪:北京市六一仪器厂;GeneAmp 9700型聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司;MS-102-3003型MiSeq测序仪:美国Illumina公司。
1.3 方法
1.3.1 样品预处理
将从越南5个地区的米酒厂收集到的5种用于实际酿酒的成品曲块用粉碎机粉碎至颗粒直径约为0.15~0.25 mm,过40目筛网进行筛分处理。每种样品取3个重复,分别装入无菌容器中,放入-20 ℃超低温冰箱中保藏以待用。
1.3.2 DNA的提取、PCR扩增及Illumina高通量测序
使用FastDNA Spin Kit for Soil试剂盒从0.5g米酒曲样本中提取总DNA,并通过1%琼脂凝胶电泳检验其完整性。为鉴定细菌,采用通用引物341F/806R扩增16S rRNA的V3-V4高变区进行PCR反应。为鉴定样本中的真菌,使用ITS1F/ITS2R引物对18S rRNA V5~V7区进行PCR扩增。PCR扩增体系(50 μL):DNA 10 ng,通用引物各1 μL,PCR混合液25 μL,采用双蒸水(ddH2O)定容至50 μL;PCR扩增条件:95 ℃预变性5 min;95 ℃变性30 s,58 ℃退火30 s,72 ℃延伸30 s,35次循环;最终72 ℃延伸5 min[14]。确认扩增产物质量合格后基于MiSeq PE300平台进行测序。
Spin Kit for Soil试剂盒从0.5g米酒曲样本中提取总DNA,并通过1%琼脂凝胶电泳检验其完整性。为鉴定细菌,采用通用引物341F/806R扩增16S rRNA的V3-V4高变区进行PCR反应。为鉴定样本中的真菌,使用ITS1F/ITS2R引物对18S rRNA V5~V7区进行PCR扩增。PCR扩增体系(50 μL):DNA 10 ng,通用引物各1 μL,PCR混合液25 μL,采用双蒸水(ddH2O)定容至50 μL;PCR扩增条件:95 ℃预变性5 min;95 ℃变性30 s,58 ℃退火30 s,72 ℃延伸30 s,35次循环;最终72 ℃延伸5 min[14]。确认扩增产物质量合格后基于MiSeq PE300平台进行测序。
1.3.3 序列质控和生物信息学分析
使用Fastp 20.0软件对双端原始测序序列进行质控过滤,包括截除低质量碱基、剔除过短读段及含N过高的序列;使用FLASH 1.2.7软件根据双端读段之间的重叠区将成对序列拼接为一条完整序列;最后,基于样本特异的Barcode和引物序列区分样本并校正序列方向,最终获得优化序列。基于97%序列相似度通过Uparese(v7.0.1090)软件划分可操作 分类单元(operational taxonomic unit,OTU)[15],对不同来源的微生物群落样品进行比较,去除测序过程中的错误序列提高分析的准确性[16]。
1.3.4 理化指标的检测
糖化力、液化力的检测:参照QB/T 4257—2011《酿酒大曲通用分析方法》测定[17],均以酒曲的干质量计。
1.3.5 数据处理与统计分析
所有的数据分析均在美吉云平台(https://analysis.majorbio.com/)上进行,采用Mothur 1.30.2计算Alpha多样性指数;采用Networkx 1.11计算物种之间的相关性,构建出物种相关性网络;采用“Spearman相关性分析”衡量两个变量之间非线性关系的相关程度;使用R语言(v3.3.1)绘图;使用Venn图来描述群落之间的相似性和差异性;采用热图以颜色梯度来呈现群落物种组成及物种的丰度信息。
2 结果与分析
2.1 米酒曲样品理化指标检测结果
米酒曲样品理化指标检测结果见表1。由表1可知,5种米酒曲的糖化力与液化力有较大的差异,样品YNG2、YB和Y2G的糖化力与液化力差异不大;样品YNNB的糖化力、YNG1的液化力最高,分别为306.63mg/(g·h)、1.02 g/(g·h)。因此,不同米酒曲样品发酵能力有一定差异。
表1 米酒曲样品理化指标检测结果
Table 1 Determination results of physicochemical indexes of rice wine koji samples

样品 糖化力/[mg·(g·h)-1] 液化力/[g·(g·h)-1]YNG1 YNG2 YNNB YB Y2G 274.57±1.8 136.52±1.2 306.63±1.2 130.82±2.1 135.66±2.1 1.02±0.01 0.29±0.03 0.46±0.08 0.23±0.05 0.25±0.08
2.2 米酒曲微生物菌群多样性分析
2.2.1 米酒曲微生物菌群高通量测序结果及Alpha多样性分析
不同米酒曲样品共获得286428条细菌序列,其中YNG1、YNG2、YNNB、YB和Y2G的序列数分别为46 840条、69 312条、66 702条、46 766条、56 808条。YNG1、YNG2、YNNB、YB和Y2G3产生的18S rRNA V5~V7区真菌序列分别为56 110条、110 678条、139 538条、69 765条和56 566条。米酒曲细菌和真菌菌群的Shanon指数曲线见图1。由图1可知,随着测序数量增加,不同米酒曲样品Shannon指数呈现出先增加后趋于平缓的趋势,这说明测序数据量相对合理,测序深度能够有效呈现大曲样品中绝大多数微生物物种的信息[18]。

图1 不同米酒曲样品细菌(a)和真菌(b)菌群的Shannon指数稀释曲线
Fig.1 Rarefaction curves of Shannon indexes of bacterial (a) and fungal (b) community in rice wine koji samples
ACE指数和Chao1指数均反映群落中物种丰富度,其值越高表示丰富度越高;Shannon指数和Simpson指数反映物种多样性,其值越高表明物种多样性越大[19]。 米酒曲细菌、真菌菌群Alpha多样性分析结果见表2。
表2 米酒曲样品细菌、真菌菌群Alpha多样性分析结果
Table 2 Alpha diversity analysis results of bacterial and fungal flora of rice wine koji samples
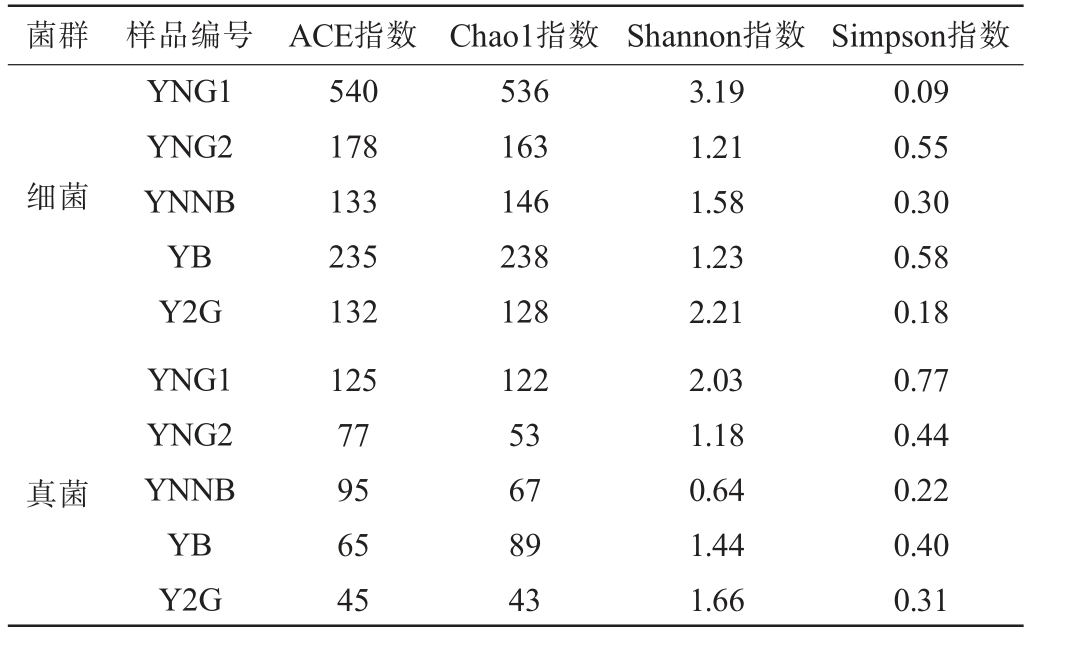
菌群 样品编号 ACE指数 Chao1指数 Shannon指数 Simpson指数细菌真菌YNG1 YNG2 YNNB YB Y2G YNG1 YNG2 YNNB YB Y2G 540 178 133 235 132 125 77 95 65 45 536 163 146 238 128 122 53 67 89 43 3.19 1.21 1.58 1.23 2.21 2.03 1.18 0.64 1.44 1.66 0.09 0.55 0.30 0.58 0.18 0.77 0.44 0.22 0.40 0.31
由表2可知,样品YNG1的细菌菌群ACE指数、Chao1指数、Shannon指数均最高,表明其细菌物种丰富度、多样性最优;样品YNG2、YB的细菌菌群ACE指数、Chao1指数较高;样品YB的细菌菌群Simpson指数最高,Shannon指数较高;样品YNNB、Y2G细菌菌群的多样性指数差异不大。
样品YNG1的真菌菌群多样性指数均最高,表明其真菌物种丰富度和多样性最优;样品YB真菌菌群Chao1指数、Shannon指数较高,样品YNNB真菌菌群的ACE指数较高,样品Y2G真菌菌群ACE指数、Chao1指数最低,表明其真菌物种丰富度较低。
结果表明,不同米酒曲样品中微生物菌群Alpha多样性有明显差异,且样品YNG1微生物丰富度和多样性最优。
2.2.2 不同米酒曲样品基于OTU的Venn图分析
不同米酒曲中细菌和真菌OTU的Venn图见图2。由图2a可知,样品YNG1、YNG2、YNNB、YB和Y2G的细菌OTU数分别为486个、131个、62个、205个、124个,5种样品共有的细菌OTU为18个,占细菌OTU总数的2.73%。 样品YNG1、YNG2、YNNB、YB和Y2G分别有294个、39个、9个、44个和52个特有的细菌OTU。 其中YNG1和YB共有的细菌OTU数为78个,两者的细菌物种相似度最高;样品YB和YNNB的共有细菌OTU仅为1个,细菌物种相似度最低;由图2b可知,样品YNG1、YNG2、YNNB、YB和Y2G的真菌OTU数分别为119个、46个、55个、59个、41个。5种样品共有的真菌OTU为6个,占真菌OTU总数的3.51%,样品YNG1、YNG2、YNNB、YB和Y2G分别有46个、6个、16个、11个和15个特有的真菌OTU。其中YB和YNG1的共有真菌OTU为11个,两者真菌物种相似度最高;样品YB和YNNB、Y2G和YNG2、Y2G和YB的共有OTU均为0,真菌物种相似度最低。
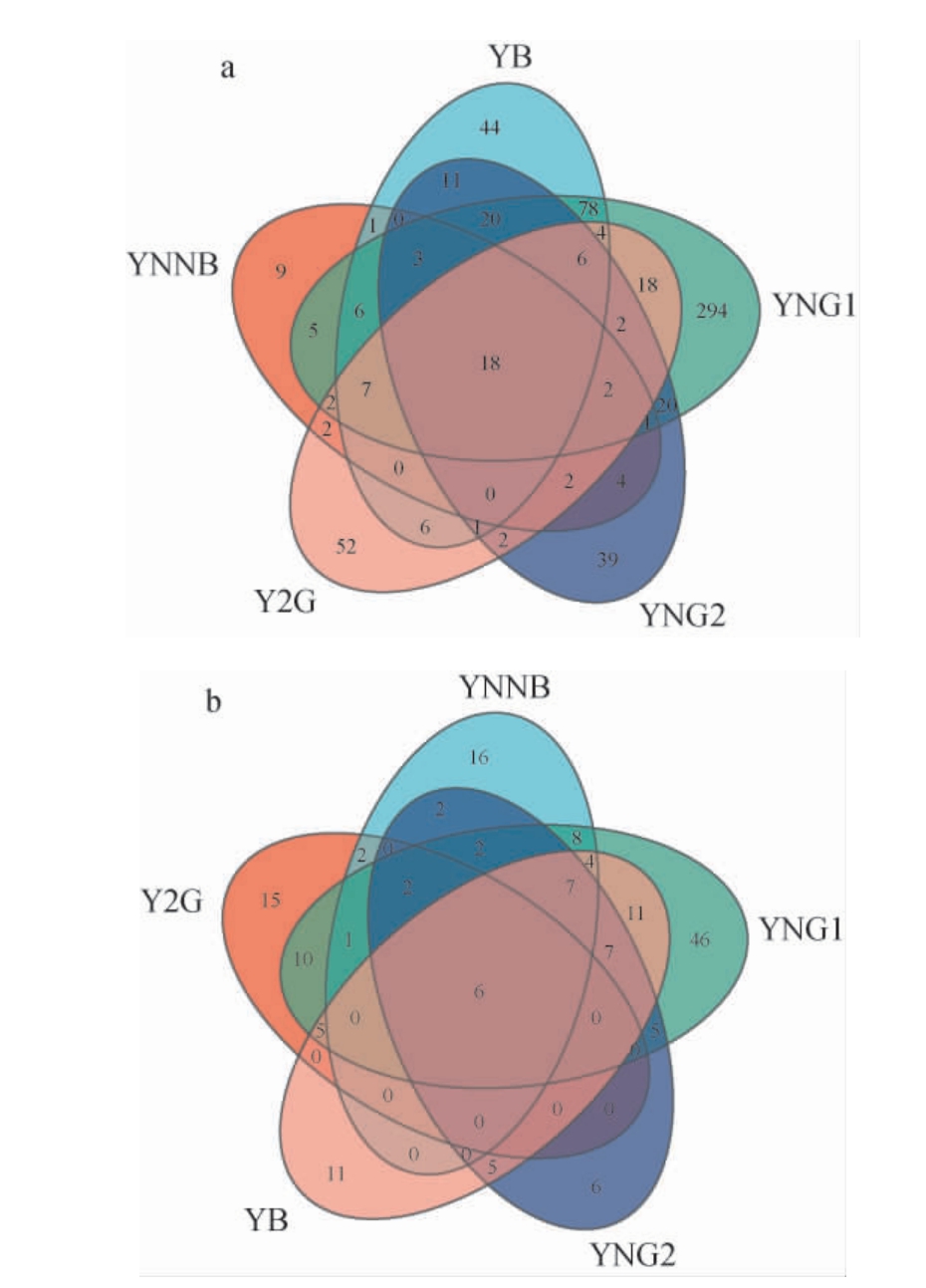
图2 米酒曲样品中细菌(a)和真菌(b)操作分类单元的Venn图
Fig.2 Venn diagram of operational taxonomic unit of bacteria (a) and fungi (b) in rice wine koji samples
2.3 不同米酒曲微生物群落结构组成
2.3.1 基于门水平微生物群落组成
不同地区的米酒曲蕴含的微生物类群受制作原料、工艺和地域环境等因素的影响,优势菌属在不同环境下表现也存在差异[20]。按样品中细菌和真菌的相对丰度划分为优势菌,将相对丰度>1%的微生物定义为优势菌[21]。 基于门水平不同米酒曲样品细菌、真菌菌群结构见图3。由图3a可知,5个越南米酒曲样品在门水平上的细菌群落结构较为相似,共检出5个优势细菌门(相对丰度>1%),分别为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteriota)、蓝 藻 门(Cyanobacteria)和 拟 杆 菌 门(Bacteroidota)。其中,厚壁菌门和变形菌门为所有样品中共有,但相对丰度差异显著,厚壁菌门在YNNB、YNG2、YB和Y2G样品中占绝对优势,相对丰度分别为98.34%、94.38%、81.60%和82.00%;而YNG1中变形菌门为最优势菌门,达59.05%。罗莉等[22]研究结果表明,厚壁菌门和变形菌门为宜昌和广西酒曲中的优势微生物。由图3b可知,5个越南米酒曲样品中共检出3个优势真菌门(相对丰度>1%),分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和毛霉门(Mucoromycota)。 其中,子囊菌门在全部样品中占据绝对优势,相对丰度均达到95%以上。杨甜甜等[23]研究表明,南宁和孝感地区酒曲中优势菌门均为子囊菌门和毛霉菌门。
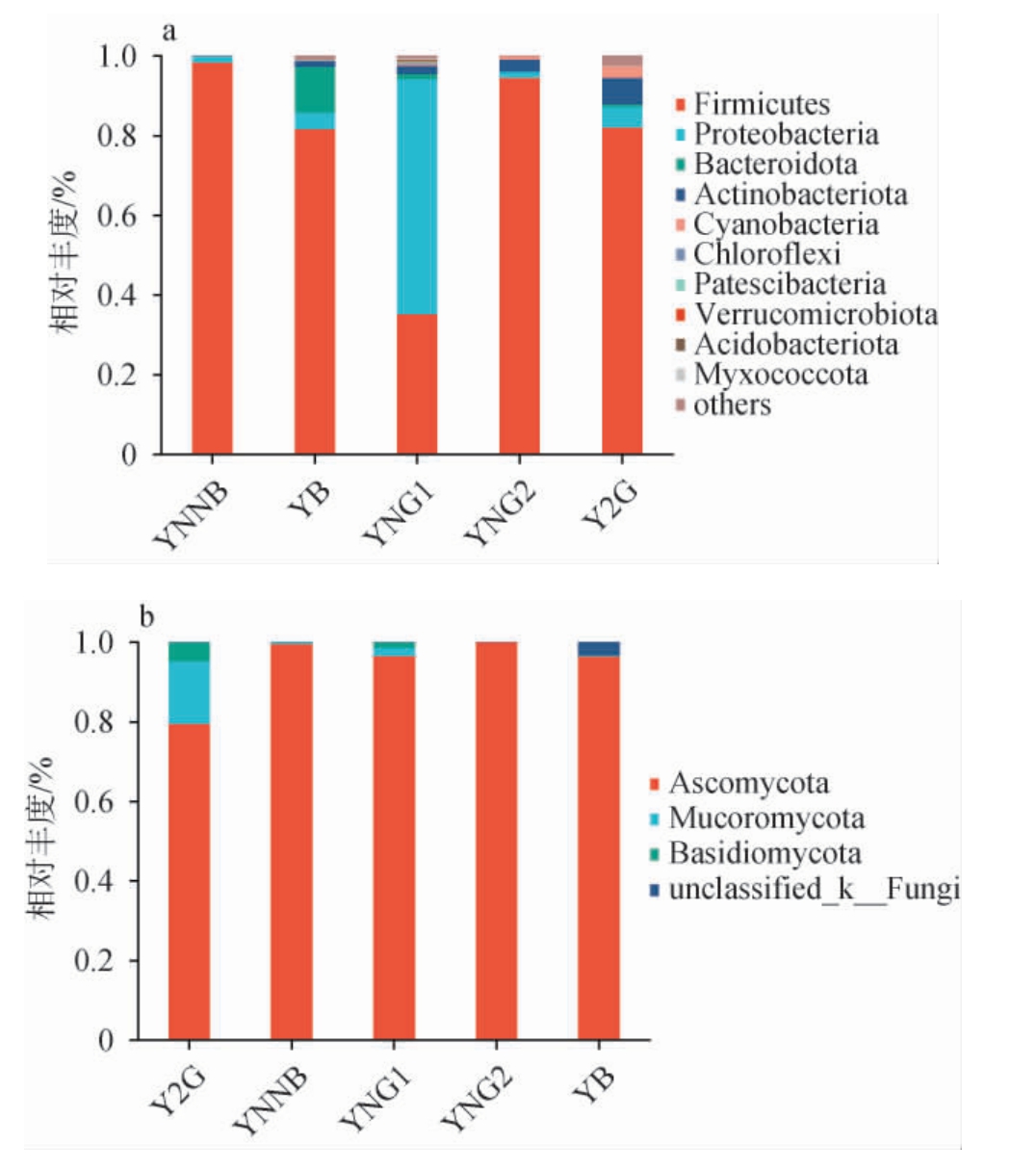
图3 基于门水平不同米酒曲样品细菌(a)和真菌(b)菌群结构
Fig.3 Bacterial (a) and fungal (b) community structure of different rice wine koji samples based on phylum level
2.3.2 基于属水平米酒曲微生物群落组成分析
基于属水平,对米酒曲中相对丰度前50的细菌、真菌菌群进行聚类分析,结果见图4。由图4a可知,共检测出10个优势细菌属(相对丰度>1%),包括乳酸杆菌属(Lactobacillus)、片球菌属(Pediococcus)、乳球菌属(Lactococcus)、泛菌属(Pantoea)、芽孢杆菌属(Bacillus)和魏斯氏菌属(Weissella)等。所有样品共有的细菌属为乳酸杆菌属(Lactobacillus),其在样品YNG1、YNG2、YNNB、Y2G、YB中的相对丰度分别为10.81%、13.73%、52.73%、49.87%、2.49%。样品YNG1中主要优势菌属为泛菌属(Pantoea)(11.1%)、乳酸杆菌属(Lactobacillus),与其他样品相比还存在克罗诺杆菌属(Cronobacter)(19.07%)等优势细菌属;样品YNG2中主要优势菌属为葡萄球菌属(Staphylococcus)(73.43%)及乳酸杆菌属(Lactobacillus);样品YB中主要优势菌属为魏斯氏菌属(Weissella)(75.66%)和Apibacter(11.10%),其中,拟杆菌属在该样品中独有;而样品YNNB主要优势菌属为乳酸杆菌属(Lactobacillus)和片球菌属(Pediococcus)(35.41%);样品Y2G中主要优势菌属为乳酸杆菌属(Lactobacillus)和乳球菌属(Lactococcus)(15.19%),其中乳球菌属相对丰度高于其他样品。与谢玲等[24-25]的研究结论一致,表明乳酸杆菌属、芽孢杆菌属和魏斯氏菌属等米酒曲中常见优势菌属在米酒发酵过程中具有重要功能。 研究表明,芽孢杆菌能够降解蛋白质与淀粉,促进风味物质如丁酸乙酯和己酸乙酯的形成[26];乳酸菌则直接影响酸味组成并可通过细菌素抑制杂菌[27],但其丰度需控制在合理范围内,以免对酒质产生负面影响[28]。不同样品中优势菌属的显著差异源于各地制作工艺、原料及气候环境的不同,体现出越南米酒曲微生物群落的地域特性。

图4 米酒曲样品中相对丰度排名前50的细菌(a)和真菌(b)菌群的聚类分析热图
Fig.4 Heat map of cluster analysis of bacterial (a) and fungal (b)community with relative abundance ranking in top 50 in rice wine koji samples
由图4b可知,共检出根霉属(Rhizopus)、假丝酵母属(Candida)、曲霉属(Aspergillus)、毛霉属(Mucor)、扣囊复膜酵母属(Saccharomycopsis)、异常威克汉姆酵母属(Wickerhamomyces)、耐干霉菌属(Xeromyces)、阿萨丝孢酵母属(Trichosporon)和奥默柯达酵母属(Kodamaea)等13个优势真菌属(相对丰度>1%)。 样品YNNB和Y2G中共有的优势真菌属为假丝酵母属(Candida),其相对丰度分别为88.73%和51.2%。样品YNG1、YNG2和YB中共有的优势真菌属为曲霉属(Aspergillus),其相对丰度分别为40.79%、68.71%和74.55%。不同样品的主要优势真菌属组成呈现差异,其相对丰度在不同样品中相对丰度明显高于其他样品。样品YNG1中主要优势属为曲霉属(Aspergillus)、耐干霉菌属(Xeromyces)(24.98%)和扣囊复膜酵母属(Saccharomycopsis)(12.94%),其中耐干霉菌属相对丰度明显高于其他样品;样品YNG2中主要优势菌属为曲霉属(Aspergillus)和耐干霉菌属(Xeromyces)(20.53%);样品YB中主要优势菌属为曲霉属(Aspergillus)和扣囊复膜酵母属(Saccharomycopsis)(16.24%);而样品YNNB中主要优势菌属为假丝酵母属(Candida),其相对丰度明显高于其他样品;样品Y2G中主要优势菌属为假丝酵母属(Candida)、根霉属(Rhizopus)(15.68%)和奥默柯达酵母属(Kodamaea)(12.72%),且根霉属、奥默柯达酵母属相对丰度明显高于其他样品。 这些真菌在米酒发酵过程中承担重要功能,根霉可分泌丰富的淀粉酶,有效催化淀粉糖化[29];米曲霉与黄曲霉具有较强的α-淀粉酶及糖化酶活性[30];毛霉则能产生淀粉酶与脂肪酶,协同促进糖化过程[31]。 假丝酵母可通过与酿酒酵母共发酵影响乙醇含量[32],这可能是造成越南米酒风味特征的因素之一。
2.3.3 不同米酒曲微生物群落物种主坐标分析
基于OTU水平对5种样品中细菌、真菌群落进行主坐标分析(PCoA),降低样本距离矩阵的维数简化数据结构,结果见图5。 图中不同形状的点代表不同的样本,两点之间的距离越近,说明两个样品微生物群落结构差异性小相似性高[33]。 由图5a可知,PC1和PC2的方差贡献率分别为30.24%和39.9%,累计方差贡献率为70.14%,说明两个主成分可解释5种样品中细菌群落变化的70.41%。样品YNNB与Y2G细菌群落之间的距离较近,与其他3个样品细菌群落差异明显,与样品YNG2细菌群落有较大差异,说明样品YNNB与Y2G细菌群落结构相似,这可能与样品YNNB和Y2G在属水平上均有乳酸杆菌属(Lactobacillus)有关,且乳酸杆菌属(Lactobacillus)在两样品中相对丰度均接近50%。由图5b可知,PC1和PC2方差贡献率分别为24.81%和66.03%,累计方差贡献率为90.84%,样品YNG2和YB、Y2G和YNNB的真菌群落组成相似,而样品YNG1的真菌群落组成与其他样品差异明显。这可能与样品YNG2和YB在属水平上主要优势菌属类似有关。综上,PCoA可以有效区分不同地区米酒曲样品。
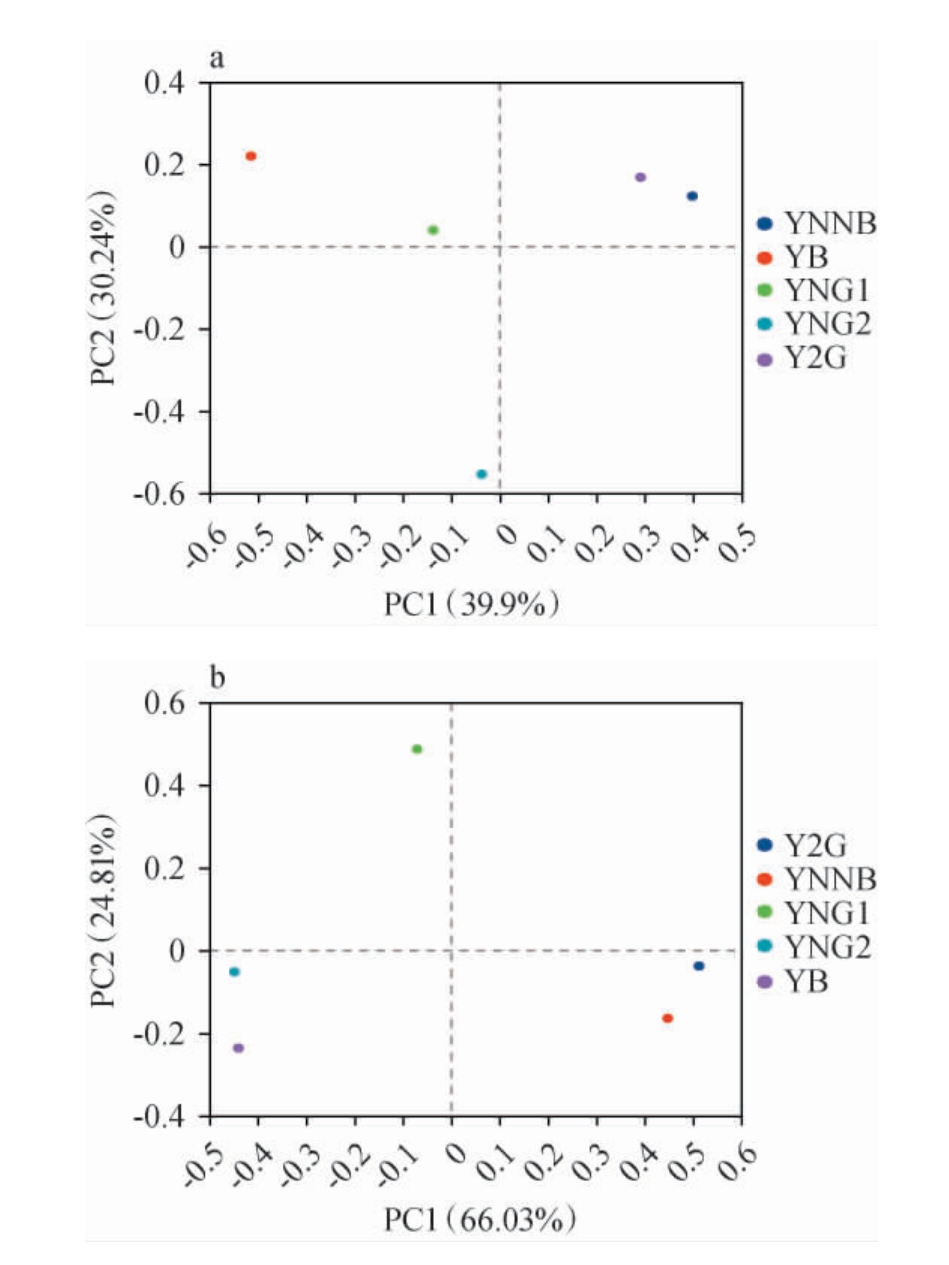
图5 米酒曲样品的细菌属(a)和真菌属(b)主坐标分析
Fig.5 Principal coordinate analysis of the bacterial genera (a) and fungal genera (b) of rice wine koji samples
2.3.4 微生物属相关性网络分析
对米酒曲中相对丰度前50的细菌属、真菌属进行相关性网络分析,结果见图6。

图6 米酒曲样品中细菌属(a)和真菌属(b)相关性网络分析
Fig.6 Correlation network analysis of bacterial genera (a) and fungal genera (b) in rice wine koji samples
图中节点代表不同微生物属,红色连线表示正相关,绿色连线表示负相关,连线粗细代表相关性系数的大小[34-35]。
在米酒曲细菌属水平相关性网络中,节点的度表示与某节点相连的数目,节点越多,节点的度越大,节点的聚类系数表示该节点的相邻节点之间的连接情况,若相关性网络中节点与相邻节点之间完全相连,聚类系数为1,若相邻节点之间几乎没有连接关系,聚类系数接近0,聚类系数越大节点越重要[36-37]。 由图6a可知,正、负相关关系分别为133个、12个,节点46个,其中微杆菌属(Microbacterium)、动胶菌属(Zoogloea)等在网络图中节点的度均>10,且聚类系数分别为0.64和0.82;醋酸杆菌属(Acetobacter)、芽孢杆菌属(Bacillus)等节点在网络中节点的度<3,且聚类系数为0,说明其功能较独立,可能在特定代谢途径发挥专一性功能。
由图6b可知,正、负相关关系分别为94个、4个,节点共47个,其中聚类系数为1且度<3的节点33个,说明多数节点在网络中连接度较低且分布分散,未出现具有高度集中连接特征的核心节点,但在网络中形成多个若干结构稳定的紧密簇,如曲霉属(Aspergillus)、拟青霉属(Paecilomyces)、假丝酵母属(Candida)等,这些节点的相邻节点之间的连接非常紧密。芽生葡萄孢酵母属(Blastobotrys)、丝孢毕赤酵母属(Hyphopichia)等节点在网络中的度>5,聚类系数均为1,在维持真菌群落局部结构与功能联系中具有重要地位。
从整体上看,细菌属间节点联系紧密,真菌属间连接度较低,在细菌网络中呈现“核心-边缘”结构,而真菌网络的呈现为“聚类子群+桥梁节点”。反映出两类微生物在米酒曲中功能不同,细菌核心节点在全局代谢起作用,真菌通过小团体协作与桥梁节点连接实现精细调控。
2.4 理化指标与优势菌属聚类分析
通过使用Spearman系数法对糖化力、液化力2个理化指标与相对丰度>1%的优势细菌属、真菌属进行相关性分析,结果见图7。
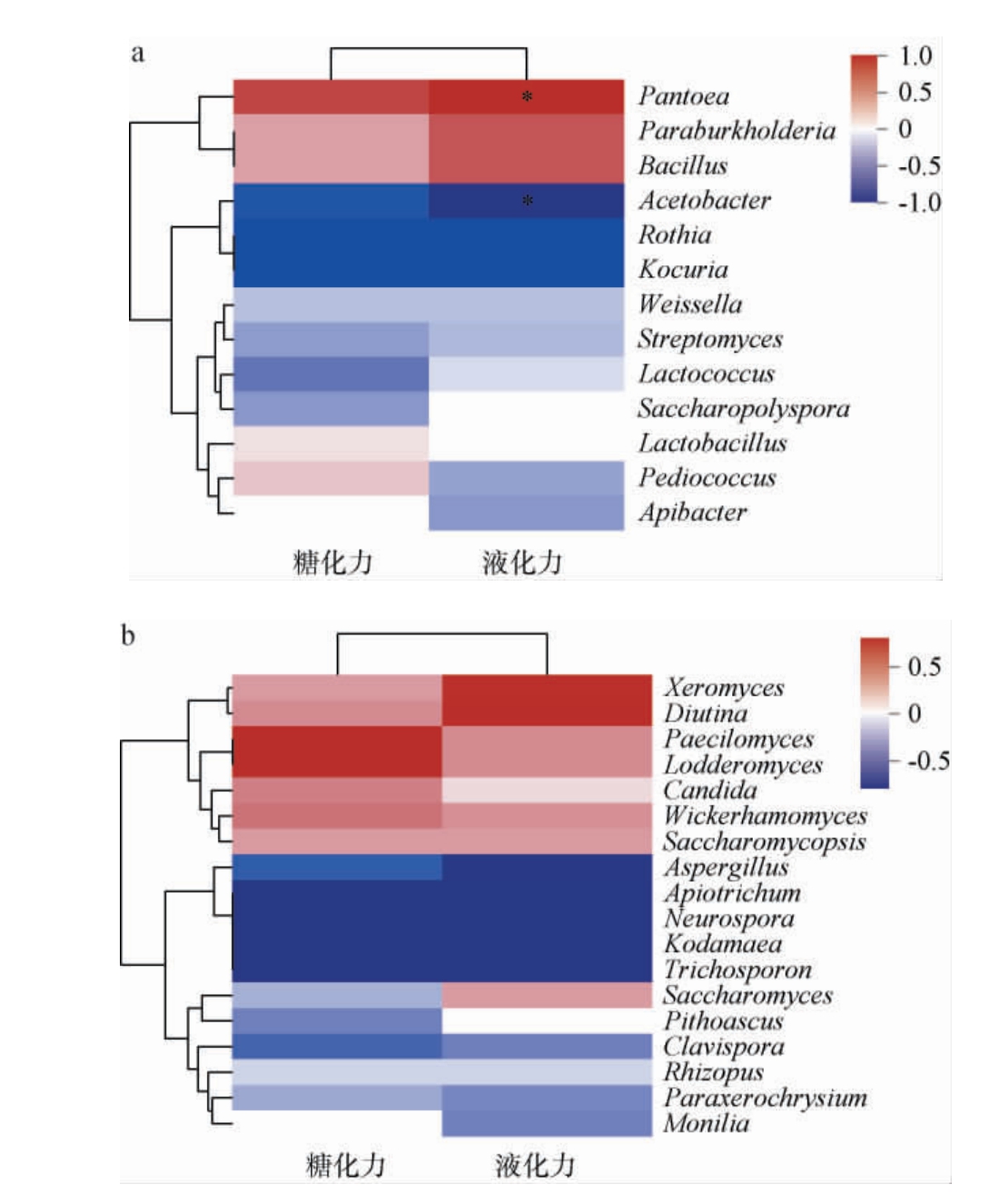
图7 理化指标及优势细菌属(a)、真菌属(b)的相关性分析热图
Fig.7 Heat map of correlation analysis between physicochemical indicators and dominant bacterial genera (a) and dominant fungal genera (b)
“*”代表相关性显著(P<0.05)。
由图7a可知,泛菌属(Pantoea)与液化力呈显著正相关(P<0.05),且与糖化力亦呈正相关,米酒曲制作过程中使用的草本植物带有木质素、纤维素等成分,而泛菌属(Pantoea)拥有大量能分泌降解复杂多糖的酶[38],从而增强糖化力和液化力;芽孢杆菌属(Bacillus)与糖化力及液化力呈正相关,其拥有能破坏植物或纤维中结构的酶[39];乳酸杆菌属(Lactobacillus)和片球菌属(Pediococcus)通过直接或者间接的方式参与糖化过程[40],对糖化力呈现正相关;醋酸杆菌属(Acetobacter)与糖化力呈现负相关,与液化力呈显著负相关(P<0.05),当与酵母共培养时,能消耗乙醇产酸导致体系酸化,从而抑制液化酶的活性或相关功能菌的生长[41]。魏斯氏菌属(Weissella)、乳球菌属(Lactococcus)等与糖化、液化呈负相关或者无显著作用,这些菌主要通过产酸及风味前体物质在米酒曲中发挥作用,而非直接参与糖化、液化过程[42-43]。
由图7b可知,假丝酵母属(Candida)、异常威克汉姆酵母属(Wickerhamomyces)等非酿酒酵母与糖化力、液化力均呈正相关,耐干霉菌属(Xeromyces)等的液化能力较强,而拟青霉属(Paecilomyces)、洛德酵母属(Lodderomyces)等糖化能力较强,扣囊复膜酵母属(Saccharomycopsis)促进淀粉水解与糖生成[44];阿萨丝孢酵母属(Trichosporon)、曲霉属(Aspergillus)、奥默柯达酵母属(Kodamaea)、根霉属(Rhizopus)等与糖化、液化均呈负相关,这可能因为其代谢活动可能产生过多酸性代谢物或竞争性底物消耗,从而对群落中其他功能菌形成抑制作用,导致整体糖化与液化能力下降[45-46]。
3 结论
不同越南地区米酒曲样品理化指标有明显差异,样品YNNB糖化力最高(306.63 mg/(g·h)),样品YNG1液化力最高(1.02 g/(g·h))。 高通量测序结果表明,不同米酒曲微生物菌群有明显差异,样品YNG1细菌和真菌丰富度及多样性均最高;不同米酒曲样品中共检出10个优势细菌属和13个优势真菌属(相对丰度>1%),共有优势细菌属为乳酸杆菌属(Lactobacillus),假丝酵母属(Candida)为样品YNNB和Y2G中共有优势真菌属,曲霉属(Aspergillus)为样品YNG1、YNG2和YB中共有优势真菌属,且主要优势菌属的相对丰度在不同样品中差异较大。主坐标分析(PCoA)可以有效区分不同地区米酒曲样品。菌群相关性网络分析表明,细菌属间节点联系紧密,真菌属间连接度较低;泛菌属(Pantoea)与液化力呈显著正相关(P<0.05),醋酸杆菌属(Acetobacter)与液化力呈显著负相关(P<0.05)。 本研究可为越南米酒曲的微生物解析提供科学依据,为米酒工艺优化与功能菌株的定向选育提供理论基础。未来可通过深入挖掘越南米酒曲中优势菌属的代谢功能及协同机制,进一步聚焦越南特色菌属与国内优良菌群的复配潜力,推动米酒风味与品质的协同提升。
[1]李丝桐,裴阳阳,向思妮,等.南方地区米酒曲中微生物群落结构及功能菌的筛选与应用[J/OL].食品与发酵工业,1-12[2025-07-03].https://doi.org/10.13995/j.cnki.11-1802/ts.042427.
[2]DUNG N T P,ROMBOUTS F M,NOUT M J R.Functionality of selected strains of moulds and yeasts from Vietnamese rice wine starters[J].Food Microbiol,2006,23(4):331-340.
[3]向凡舒,朱媛媛,邓风,等.建始地区米酒曲细菌和真菌多样性研究[J].食品工业科技,2021,42(1):126-131.
[4]郝莹,王卫卫,王莉娟,等.陕北传统米酒曲中优势菌种的分离、纯化及鉴定[J].检验检疫学刊,2010,20(2):44-47,20.
[5]蔡海莺,盛宇华,沈灵智,等.不同原料的米酒多酚及其抗氧化性能比较[J].中国食品学报,2021,21(2):327-333.
[6]王玉荣,张俊英,胡欣洁,等.湖北孝感和四川成都地区来源的酒曲对米酒滋味品质影响的评价[J].食品科学,2015,36(16):207-210.
[7]王丹丹,倪慧,赵慧君,等.凤窝酒曲中乳酸菌的分离及其作用下的米酒品质评价[J].中国酿造,2018,37(6):80-84.
[8] HE M Y, XING S G, YAO G H, et al.Application of next generation semiconductor based sequencing for species identification and meat derived products authentication[J].Food Control,2024,165:110639.
[9]宁亚丽,吴跃,何嫱,等.基于高通量测序技术分析朝鲜族传统米酒及其酒曲中微生物群落多样性[J].食品科学,2019,40(16):107-114.
[10]杨春敏,黄建初,查丽艳.海南山兰米酒酒曲微生物多样性分析[J].食品工程,2019(4):44-48.
[11]王丹丹,沈馨,董蕴,等.孝感凤窝酒曲真菌多样性评价[J].中国酿造,2017,36(11):38-42.
[12]沈馨,尚雪娇,董蕴,等.基于MiSeq高通量测序技术对3个孝感凤窝酒曲细菌多样性的评价[J].中国微生态学杂志,2018,30(5):525-530,544.
[13]甘晋铭,朱俊颖,罗煜皓,等.越南米酒曲与国内米酒曲发酵特性对比分析[J].酿酒,2025,52(5):91-98.
[14]毛文定,冯文聪,孙威,等.不同香型白酒酿造大曲微生物群落与理化指标关联分析[J].中国酿造,2024,43(7):147-153.
[15]EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nat Methods,2013,10(10):996-998.
[16]YADAV D, DUTTA A, MANDE S S.OTUX: V-region specific OTU database for improved 16S rRNA OTU picking and efficient cross-study taxonomic comparison of microbiomes[J].DNA Res,2019,26(2):147-156.
[15]中华人民共和国工业和信息化部.QB/T 4257—2011 酿酒大曲通用分析方法[S].北京:中国标准出版社,2011.
[18]ZHAO P, LI C, RAHAMAN M M, et al.EMDS-6: Environmental microorganism image dataset sixth version for image denoising,segmentation, feature extraction, classification, and detection method evaluation[J].Front Microbiol,2022,13:2022.
[19]张二豪,奉综涛,何萍,等.西藏地区青稞酒曲微生物群落结构及多样性分析[J].中国食品学报,2024,24(5):381-392.
[20]王倩,李振双,杨富成,等.广西凭祥红锥-马尾松混交林菌根际微生物群落结构[J].菌物学报,2021,40(6):1343-1356.
[21]王清龙,朱甜甜,刘延波,等.白酒生产不同酒曲微生物群落结构、理化指标及挥发性风味物质研究[J].中国酿造,2023,42(7):93-102.
[22]罗莉,甘晋铭,向亮亮,等.基于高通量测序技术对不同地区米酒曲微生物群落结构的分析[J].酿酒科技,2024(9):82-86.
[23]杨甜甜,杨少勇,侯强川,等.基于高通量测序技术分析比较南宁和孝感地区米酒曲中真菌多样性[J].中国酿造,2021,40(9):110-114.
[24]谢玲,龚小会,张东亚,等.贵州特色米酒产区酒曲微生物群落多样性分析[J].中国酿造,2024,43(8):170-176.
[25]CAI H Y,ZHANG T,ZHANG Q,et al.Microbial diversity and chemical analysis of the starters used in traditional Chinese sweet rice wine[J].Food Microbiol,2018,73:319-326.
[26]WANG X, WANG Z X, ZHANG X T, et al.Bioinformatics-assisted mining and design of novel pullulanase suitable for starch cold hydrolysis[J].J Biotechnol,2025,398:106-116.
[27]葛东颖,何梦雪,张振东,等.南宁地区米酒曲源乳酸菌在糯米汁中氨基酸和有机酸代谢特征研究[J].中国酿造,2020,39(9):53-57.
[28]XU M L, YU Y, RAMASWAMY H S, et al.Characterization of Chinese liquor aroma components during aging process and liquor age discrimination using gas chromatography combined with multivariable statistics[J].Sci Rep,2017,7(1):39671.
[29]ANCHUNDIA M,LEÓN-REVELO G,SANTACRUZ S,et al.Polyphasic identification of Rhizopus oryzae and evaluation of physical fermentation parameters in potato starch processing liquid waste for β-glucan production[J].Sci Rep,2024,14(1):14913.
[30]LV X C,HUANG X L,ZHANG W,et al.Yeast diversity of traditional alcohol fermentation starters for Hong Qu glutinous rice wine brewing,revealed by culture-dependent and culture-independent methods[J].Food Control,2013,34(1):183-190.
[31]CHU L F, ZAN X Y, TANG X, et al.The role of a xylose isomerase pathway in the conversion of xylose to lipid in Mucor circinelloides[J].RSC Adv,2016,6(81):77944-77952.
[32]ENGLEZOS V, RANTSIOU K, CRAVERO F, et al. Starmerella bacillaris and Saccharomyces cerevisiae mixed fermentations to reduce ethanol content in wine[J].Appl Microbiol Biotechnol,2016,100(12): 5515-5526.
[33]WANG Y,SUN F Z,LIN W,et al.AC-PCoA:Adjustment for confounding factors using principal coordinate analysis[J].PLoS Comput Biol,2022,18(7):e1010184.
[34]郑巳芳,程驰新笑,龙树瑺,等.基于理化指标和MiSeq高通量测序技术红曲品质及微生物类群的解析[J].食品工业科技,2023,44(18):131-138.
[35]SI J, YOU H J, YU J, et al.Prevotella as a hub for vaginal microbiota under the influence of host genetics and their association with obesity[J].Cell Host Microbe,2017,21(1):97-105.
[36]MIYASHITA R, HIRONAKA S, SHUDO K.Clustering coefficient reflecting pairwise relationships within hyperedges[J].Sci Rep,2025,15(1):20729.
[37]CHEN D, SU H S.Identification of influential nodes in complex networks with degree and average neighbor degree[J].IEEE J Emerg Sel Top Circuits Syst,2023,13(3):734-742.
[38]MA J S,ZHANG K K,LIAO H D,et al.Genomic and secretomic insight into lignocellulolytic system of an endophytic bacterium Pantoea ananatis Sd-1[J].Biotechnol Biofuels,2016,9(1):25.
[39]OUATTARA H G, REVERCHON S, NIAMKE S L, et al.Molecular identification and pectate lyase production by Bacillus strains involved in cocoa fermentation[J].Food Microbiol,2011,28(1):1-8.
[40]REDDY G,ALTAF M,NAVEENA B J,et al.Amylolytic bacterial lactic acid fermentation-A review[J].Biotechnol Adv,2008,26(1):22-34.
[41]ALBERS E,JOHANSSON E,FRANZÉN C J, et al.Selective suppression of bacterial contaminants by process conditions during lignocellulose based yeast fermentations[J].Biotechnol Biofuels,2011,4(1):59.
[42]XU J L,WU H J,WANG Z W,et al.Microbial dynamics and metabolite changes in Chinese Rice Wine fermentation from sorghum with different tannin content[J].Sci Rep,2018,8(1):4639.
[43]FUSCO V,QUERO G M,CHO G,et al.The genus Weissella:taxonomy,ecology and biotechnological potential[J].Front Microbiol,2015,6:155.
[44]ZOU J,CHEN X H,WANG C Y,et al.Microbial communities and correlation between microbiota and volatile compounds in fermentation starters of Chinese sweet rice wine from different region[J].Foods,2023:12(15):2932.
[45]ZHANG Y,ZHANG Z,HUANG J,et al.Characterizing the contribution of strain specificity to the microbiota structure and metabolites of Muqu and fresh high-temperature Daqu[J].Foods,2024,13(19):3098.
[46]ZHAO C,PENTTINEN P,ZHANG L Z,et al.A novel high-level phenyllactic acid fungal producer,Kodamaea ohmeri w5 screened from fermented broad bean-chili-paste[J].Int J Food Microbiol,2025,426:110923.