酯酶(esterases,EC 3.1.1)作为水解酶超家族中的重要成员,是一类具有重要工业应用价值的生物催化剂[1]。其中,羧酸酯酶(carboxylesterase,EC 3.1.1.1)作为酯酶的典型代表[2],能够在不依赖辅因子的条件下特异性催化酯键的水解反应,将底物分解为水溶性酰基化合物和短链脂肪酸甘油酯,同时还可介导酯交换、酯合成、多肽合成与拆分以及手性化合物的立体选择性转化等重要生化过程[3]。从极端环境中分离的羧酸酯酶,在具备酯酶的通用特性之外,通常还具有耐盐、耐酸碱和耐有机溶剂等特性[4],在食品加工[5]、造纸工业、洗涤剂生产、纺织加工[6]、药物合成[3]、化妆品制造[7]以及环境治理[8]等领域均展现出不可替代的应用价值,已成为绿色生物制造的核心工具酶之一。 耐盐羧酸酯酶因其独特的理化特性,能够在高盐浓度条件下保持稳定的催化活性,特别适用于传统高盐发酵食品的加工工艺优化[9]。以酱油发酵为例,添加耐盐羧酸酯酶可以促进大豆油脂的水解过程,不仅能够缩短整体发酵周期,还能有效提升终产品中酯类风味物质的含量[10]。
尽管羧酸酯酶因其优异的催化性能,在工业生产和食品加工等领域具有重要的应用价值,但其在原始宿主菌株中的表达水平普遍偏低,严重制约了该酶在工业化生产中的规模化应用[11]。 近年来,异源表达系统的开发已成为提升酶的生产效率、优化生物工艺经济性的重要策略[12]。 相较于天然宿主,异源表达可突破原始菌株的生长限制,实现目的酶的高效合成。其中,大肠杆菌(Escherichia coli)表达系统因其基因组信息明确、遗传稳定且遗传操作成熟、培养成本低、抗污染能力强、蛋白表达量高等优势[13],特别适用于酯酶等工业用酶的生产。李全发等[14]将灰色链霉菌(Streptomyces griseus)来源的新型酯酶EstE在E.coli系统中高效异源表达,获得的重组酯酶具有优异的催化特性、热稳定性、碱稳定性和有机溶剂耐受性;邓名等[15]从金色马赛菌(Massilia aurea)中成功鉴定并克隆了酯酶Est2的编码基因,借助大肠杆菌(E.coli)表达系统,实现了该酯酶的高效异源表达。 此外,优化发酵培养基和发酵条件可进一步提高重组工程菌所产酯酶的产量和活性。常用的优化方法包括正交试验法和响应面分析法等[16-17]。 正交试验法能够系统地考察多个因素及其不同水平对发酵效果的影响,从而筛选出关键因素并确定其最佳水平组合[18]。 响应面分析法则通过建立数学模型, 进一步精确地优化关键因素的水平,最大程度地提升目标物的产量和活性[19]。这些方法的综合运用,为提高重组工程菌酯酶的生产效率和质量提供了有力的技术支持。
本研究通过人工合成技术获得嗜盐菌Vreelandella sp.CH40中耐盐羧酸酯酶Est40的编码基因,经聚合酶链式反应(polymerase chain reaction,PCR)引入酶切位点和标签序列后[20],将其整合至pET-22b(+)表达载体,并导入大肠杆菌(E.coli)Rosetta(DE3)表达宿主中,构建能够高效表达该酯酶的重组工程菌株。进一步以重组羧酸酯酶Est40活力为响应值,采用单因素试验、Plackett-Burman(PB)及Box-Behnken(BB)响应面试验优化重组工程菌株的发酵条件,为后续重组酯酶的工业化生产提供技术支撑。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
Vreelandella sp.CH40:分离自盐湖极端环境,中度嗜盐,本实验室保存。E.coli Rosetta(DE3)、质粒pET-22b(+):生工生物工程(上海)股份有限公司。
1.1.2 试剂
异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactoside,IPTG)、氨苄青霉素、蔗糖、葡萄糖、酵母粉、蛋白胨、MgSO4、(NH4)2SO4、K2HPO4(均为分析纯):北京普西唐生物科技有限公司;对硝基苯酚乙酸酯(p-nitrophenyl acetate,pNPA):美国Sigma公司;考马斯亮蓝R-250:上海雅酶生物医药科技有限公司;柱式脱氧核糖核酸(deoxyribonucleic acid,DNA)胶回收试剂盒、质粒DNA小量抽提试剂盒、Ni-NTA Sepharose 6HP(His-Tag)蛋白纯化琼脂糖树脂、50 mmol/L咪唑缓冲液、500 mmol/L咪唑缓冲液:上海生物工程有限公司;10×Ex Taq Buffer、10×脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)Mixture、Taq DNA聚合酶(5 U/μL):北京全式金生物技术股份有限公司。
1.1.3 培养基
LB液体培养基[17]:酵母浸粉5.0 g/L,胰蛋白胨10.0 g/L,NaCl 10.0 g/L,pH 7.0。121 ℃高压蒸汽灭菌20 min。LB固体培养基:在LB液体培养基中添加琼脂粉20 g/L。
发酵培养基[21]:酵母膏2.0 g/L,蛋白胨10.0 g/L,柠檬酸钠3.0 g/L,MgSO4·7H2O 20.0 g/L,KCl 2.0 g/L,无水CaCl2 0.2 g/L,葡萄糖5.0 g/L,NaCl 10.0 g/L,pH 7.0。121 ℃高压蒸汽灭菌20 min。
1.2 仪器与设备
IMARK多功能酶标仪、T100 PCR扩增仪:伯乐生命医学产品(上海)有限公司;V2200可见分光光度计:上海美谱达仪器有限公司;ZQZY-78C型振荡培养箱:上海知楚仪器有限公司;HPH型数字化酸度测定仪:上海佑科仪器仪表有限公司;3K15离心机:德国Sigma公司。
1.3 方法
1.3.1 耐盐羧酸酯酶Est40基因的克隆与重组质粒pET-22b-Est40的构建
委托生工生物工程(上海)股份有限公司采用基因合成技术获取Vreelandella sp.CH40的耐盐羧酸酯酶Est40基因序列,以其为模板,使用包含NcoI酶切位点(CCATGG)的正向引物(5'-CCATGGATGAGCGAACCGGGCGAAC-3')和包含XhoI酶切位点(CTCGAG)的反向引物(5'-CTCGAGTTAGTGGTGGTGGTGGTGGTGCTCGAGTCT-3')PCR扩增耐盐羧酸酯酶Est40的基因序列。在反向引物中,终止密码子TGA前设计了6×His标签编码序列(CATCATCATCATCAT),以便后续重组蛋白的纯化。PCR扩增体系(25 μL):模板基因组DNA 0.2 μL,上下游引物各0.5 μL,10×Ex Taq Buffer 2.5 μL,Taq DNA聚合酶0.25 μL,10×dNTP Mixture 2.5 μL,无菌双蒸水(ddH2O)18.55 μL。PCR扩增条件:95 ℃预变性3 min;30个循环(95 ℃变性30 s,45 ℃退火30 s,72 ℃延伸1 min);72 ℃再延伸6 min。利用1%琼脂糖凝胶电泳技术对PCR扩增产物进行分析检测。PCR扩增产物经NcoI和XhoI双酶切后,与经相同酶处理的pET-22b(+)载体进行连接,构建重组表达质粒pET-22b-Est40。
1.3.2 重组工程菌的构建与重组羧酸酯酶Est40的诱导表达
采用热激转化法(42 ℃短时刺激)将重组质粒导入E.coli Rosetta(DE3)感受态细胞,均匀涂布于含50 μg/mL氨苄青霉素的LB固体培养基,37 ℃、180 r/min条件下恒温培养12~16 h。随机挑选阳性单菌落,采用质粒小提试剂盒法提取重组质粒进行双酶切验证,并将验证正确的单菌落接种至含相同抗生素的LB液体培养基,37 ℃、180 r/min条件下振荡培养至OD600nm值=0.6,加入终浓度为0.5 mmol/L的IPTG在20 ℃条件下过夜诱导表达。
取发酵液于4 000 r/min条件下离心10 min,收集菌体。菌体沉淀用预冷磷酸缓冲盐溶液(phosphate buffer saline,PBS)(pH 7.4)重悬后超声破碎(功率300 W,工作3 s,间隔5 s,总时间10 min),4 ℃、6 000 r/min条件下离心10 min,取上清;Ni-NTA Sepharose 6HP(His-Tag)蛋白纯化琼脂糖树脂经PBS平衡后与上清孵育,收集穿出液并再次平衡;采用50 mmol/L咪唑洗涤,500 mmol/L咪唑洗脱,收集洗脱液。将洗脱液通过十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecylsulfate-polyacrylamidegelelectrophoresis,SDS-PAGE)[22]分析重组羧酸酯酶Est40是否表达。 获得的重组工程菌阳性转化子E.coli Rosetta(DE3)/pET-22b-Est40至于-80 ℃甘油保藏。
1.3.3 重组工程菌种子液的制备及初始发酵条件
从-80 ℃甘油保藏管中取出重组工程菌,依次置于-20 ℃和4 ℃梯度解冻。取100 μL菌液接种至含50 μg/mL氨苄青霉素的50 mL LB液体培养基,37 ℃、180 r/min条件下振荡培养12 h,制备种子液。
将重组工程菌种子液按2%(V/V)接种量接入装液量为50 mL/100 mL含50 μg/mL氨苄青霉素的发酵培养基,37 ℃、180 r/min条件下振荡培养至OD600 nm值为0.6~0.8,加入0.5 mmol/L IPTG于20 ℃条件下诱导发酵24 h。 发酵液经6 000 r/min条件下离心10 min,收集菌体,采用PBS(pH 7.4)重悬后测定酯酶活力。
1.3.4 重组羧酸酯酶活力的测定
以p-NPA为底物,采用酶标仪法[23]测定酯酶的酶活性。酯酶活性的单位(U)定义为在pH 8.0、37 ℃条件下每分钟催化生成1 μmol p-NP所需的酶量。
1.3.5 重组工程菌发酵产羧酸酯酶条件优化
(1)单因素试验
在发酵培养基基础上,采用单因素轮换法分别考察不同碳源种类(葡萄糖、乙酸钠、蔗糖、可溶性淀粉、甘露醇,质量浓度为10g/L)及最佳碳源添加量(10 g/L、20 g/L、30 g/L、40 g/L、50 g/L和60 g/L)、不同氮源种类(酵母粉、蛋白胨、硫酸铵、尿素、硝酸钾,质量浓度为10 g/L)及最佳氮源添加量(5 g/L、10 g/L、15 g/L、20 g/L、25 g/L和30 g/L)、不同无机盐种类(CaCl2、CuSO4、MgSO4、FeSO4、ZnSO4、K2HPO4,质量浓度为5 g/L)及最佳无机盐添加量(5 g/L、10 g/L、15 g/L、20 g/L、25 g/L和30 g/L)对重组酯酶Est40活力的影响。
同时,在液体发酵培养基基础上,分别考察NaCl添加量(10 g/L、20 g/L、30 g/L、40 g/L、50 g/L和60 g/L)、诱导温度(20 ℃、24 ℃、28 ℃、32 ℃和36 ℃)、培养基初始pH值(6.5、7.0、7.5、8.0、8.5和9.0)、接种量(2%、4%、6%、8%、10%和12%)对重组酯酶Est40活力的影响。
(2)PB试验
基于前期单因素试验结果,选取蔗糖添加量(A)、酵母粉添加量(B)、MgSO4添加量(C)、诱导温度(D)、初始pH(E)、接种量(F)和NaCl添加量(G)为考察因素,以重组羧酸酯酶Est40活力(Y1)为评价指标,通过Design-Expert 13.0软件设计PB试验,试验因素与水平见表1。
表1 发酵条件优化Plackett-Burman试验因素与水平
Table 1 Factors and levels of the Plackett-Burman experiments for fermentation condition optimization
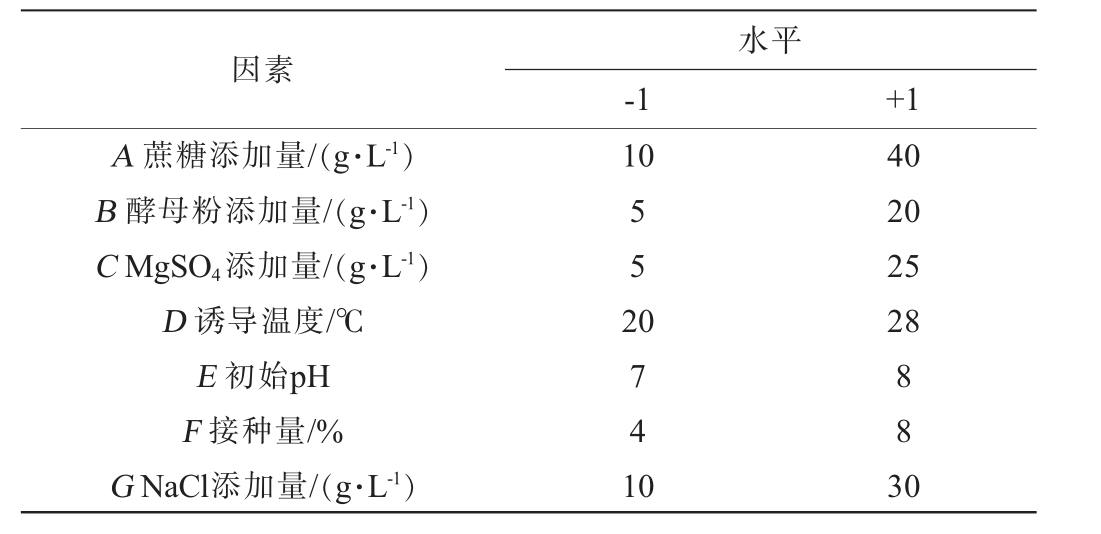
因素A 蔗糖添加量/(g·L-1)B 酵母粉添加量/(g·L-1)C MgSO4添加量/(g·L-1)D 诱 导 温 度/℃ E 初始pH F 接种量/%G NaCl添加量/(g·L-1)水平-1 +1 10 552 0741 0 40 20 25 28 883 0
(3)BB试验
基于PB试验结果,选取对重组酯酶Est40活性影响较大的因素蔗糖添加量(X1)、MgSO4添加量(X2)和接种量(X3)为考察因素,以重组羧酸酯酶Est40活性(Y2)为响应值,采用Design-Expert 13.0软件设计3因素3水平的BB响应面试验,试验因素与水平见表2。
表2 发酵条件优化BB试验因素与水平
Table 2 Factors and levels of the BB experiments for fermentation condition optimization

因素 水平-1 30 10 6 0+1 X1蔗糖添加量/(g·L-1)X2MgSO4添加量/(g·L-1)X3接种量/%20 5 4 40 15 8
1.3.6 数据处理
每组试验设置3次重复。单因素试验采用Origin 2020软件进行数据分析处理和绘图;PB和BB多因素试验采用Design-Expert 13.0软件对试验数据进行回归分析和方差分析,并绘制响应面3D曲面图。
2 结果与分析
2.1 重组工程菌的构建及重组羧酸酯酶的诱导表达分析
重组工程菌经过IPTG诱导表达和酯酶纯化后进行SDS-PAGE电泳分析,结果见图1。由图1可知,在泳道3中可观察到一条明显的蛋白条带,分子质量约为30 kDa,与Vreelandella sp.CH40耐盐羧酸酯酶Est40的预测分子质量高度吻合,表明耐盐羧酸酯酶Est40的基因已在大肠杆菌表达系统中成功实现表达,进一步说明重组菌株E.coli Rosetta(DE3)/pET-22b-Est40构建成功。
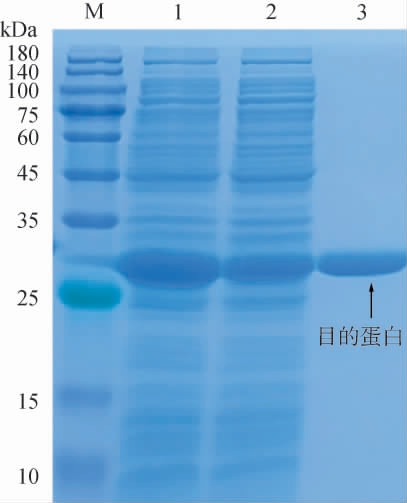
图1 重组羧酸酯酶Est40的十二烷基硫酸钠-聚丙烯酰胺凝胶电泳结果
Fig.1 Sodium dodecyl sulfate-polyacrylamide gel electrophoresis results of the recombinant carboxylesterase Est40
M表示蛋白Marker;泳道1和泳道2为纯化前总蛋白;泳道3为纯化后的蛋白。
2.2 重组工程菌发酵条件优化
2.2.1 单因素试验
碳源作为微生物生长和代谢的基础营养物质,为细胞构建和代谢产物的合成提供必需的碳骨架[24]。考察不同碳源及其最佳碳源添加量对重组工程菌产重组羧酸酯酶Est40活力的影响,结果见图2。由图2a可知,以蔗糖作为碳源时羧酸酯酶活力最高,为(312.19±4.44) U/mL;其次为葡萄糖、甘露醇和乙酸钠;可溶性淀粉作为碳源时羧酸酯酶的活力最低。 分析原因可能是蔗糖的分解速度适中,既能持续供能,又不会分解太快导致代谢抑制,且相比葡萄糖(容易抑制代谢)和乙酸钠(会产生酸性物质)对pH和渗透压的影响更小[25]。因此,确定最佳碳源为蔗糖。由图2b可知,随着蔗糖添加量的增加,羧酸酯酶活力呈先升高后下降的趋势,分析原因可能与蔗糖浓度过高时会引起渗透压升高、从而影响菌体生长有关[26]。当蔗糖添加量为30 g/L时,羧酸酯酶活力最高,为(320.38±2.09)U/mL。因此,确定蔗糖的最适添加量为30 g/L。
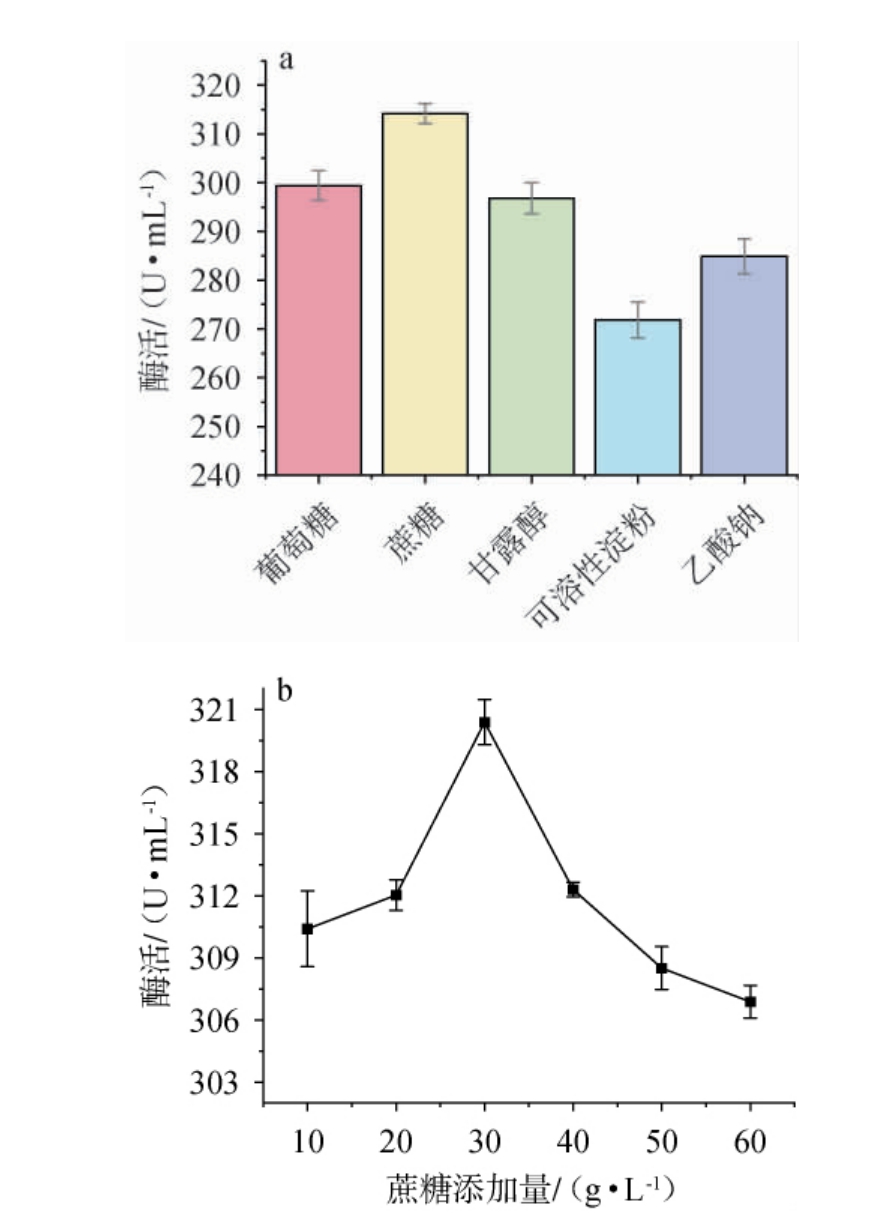
图2 不同碳源(a)及蔗糖添加量(b)对重组羧酸酯酶Est40活力的影响
Fig.2 Effect of different carbon sources (a) and sucrose addition (b)on the activities of recombinant carboxylesterase Est40
氮源是微生物生长必需的营养组分,主要参与细胞物质合成和含氮代谢产物的生成[27]。因此,考察不同氮源及最佳氮源添加量对重组工程菌产重组羧酸酯酶Est40活力的影响,结果见图3。
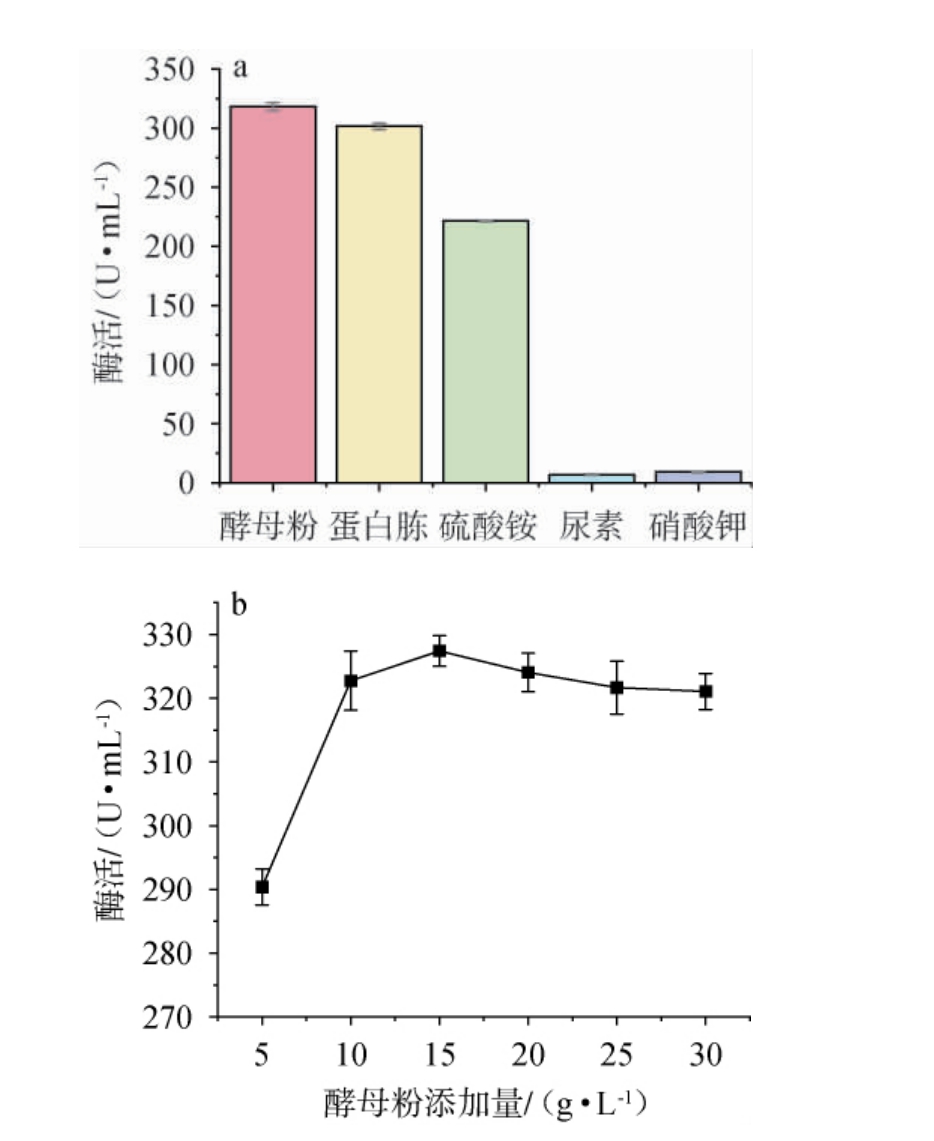
图3 不同氮源(a)及酵母粉添加量(b)对重组羧酸酯酶Est40活力的影响
Fig.3 Effect of different nitrogen sources (a) and yeast extract addition (b) on the activities of recombinant carboxylesterase Est40
由图3a可知,当酵母粉作为氮源时,羧酸酯酶活力最高,为(318.32±4.87)U/mL;其次是蛋白胨和硫酸铵;以尿素和硝酸钾作为氮源时,羧酸酯酶活力较低。分析原因可能是酵母粉初期提供优质营养与生长因子助菌体生长,进而提升后期酶活[28]。因此,确定最佳氮源为酵母粉。由图3b可知,随着酵母粉添加量的增加,羧酸酯酶活力呈先升高后下降的趋势,分析原因可能是酵母粉可以提供适中的氮释放速率,使菌株能够快速生长繁殖;但添加量过高会与胺类等含氮副产物积累而产生抑制作用[29];当酵母粉添加量为15 g/L时,羧酸酯酶活力最高,为(327.42±3.57)U/mL。因此,确定酵母粉的最适添加量为15 g/L。
无机盐是微生物生长必需的重要营养物质,其通过提供必需元素参与细胞结构和代谢功能的构建[30]。不同无机盐及最佳无机盐添加量对重组工程菌产重组羧酸酯酶Est40活力的影响见图4。
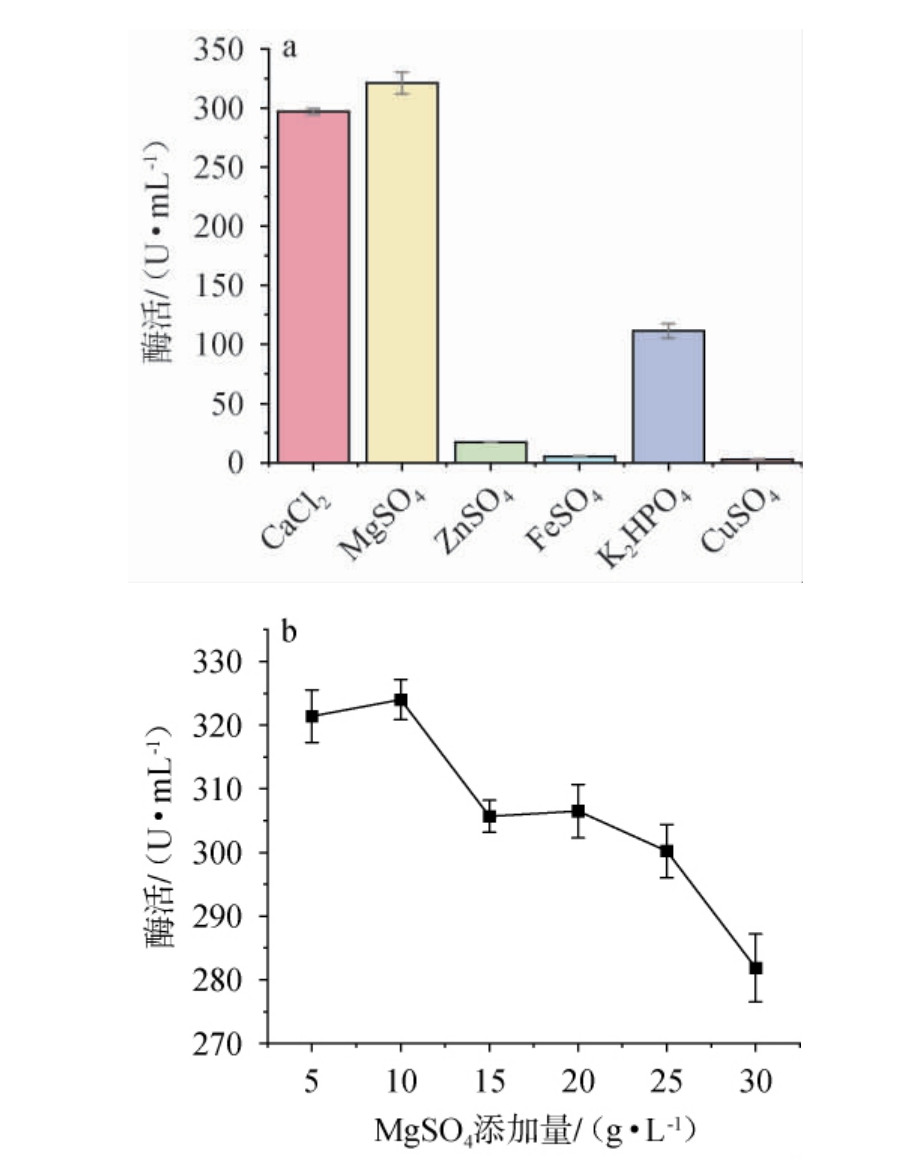
图4 不同无机盐(a)及MgSO4添加量(b)对重组羧酸酯酶Est40活力的影响
Fig.4 Effect of different inorganic salts (a) and MgSO4 addition (b)on the activities of recombinant carboxylesterase Est40
由图4a可知,当无机盐为MgSO4时,羧酸酯酶活力最高,为(321.87±5.74)U/mL,其次为CaCl2和K2HPO4。分析原因可能是Mg2+是许多酶的必需辅因子,对酯酶活性有促进作用[31]。 因此,确定最佳无机盐种类为MgSO4。 由图4b可知,随着MgSO4添加量的增加,羧酸酯酶活力呈先升高后下降的趋势;当MgSO4添加量为10 g/L时,羧酸酯酶活力最高,为(326.51±4.98)U/mL。分析原因可能是MgSO4质量浓度过高时会增加溶液的渗透压,破坏酯酶蛋白表面的水化层,降低其活性[32]。因此,确定MgSO4的最适添加量为10 g/L。
考察NaCl添加量、诱导温度、初始pH值及接种量对重组工程菌产重组羧酸酯酶Est40活力的影响,结果见图5。
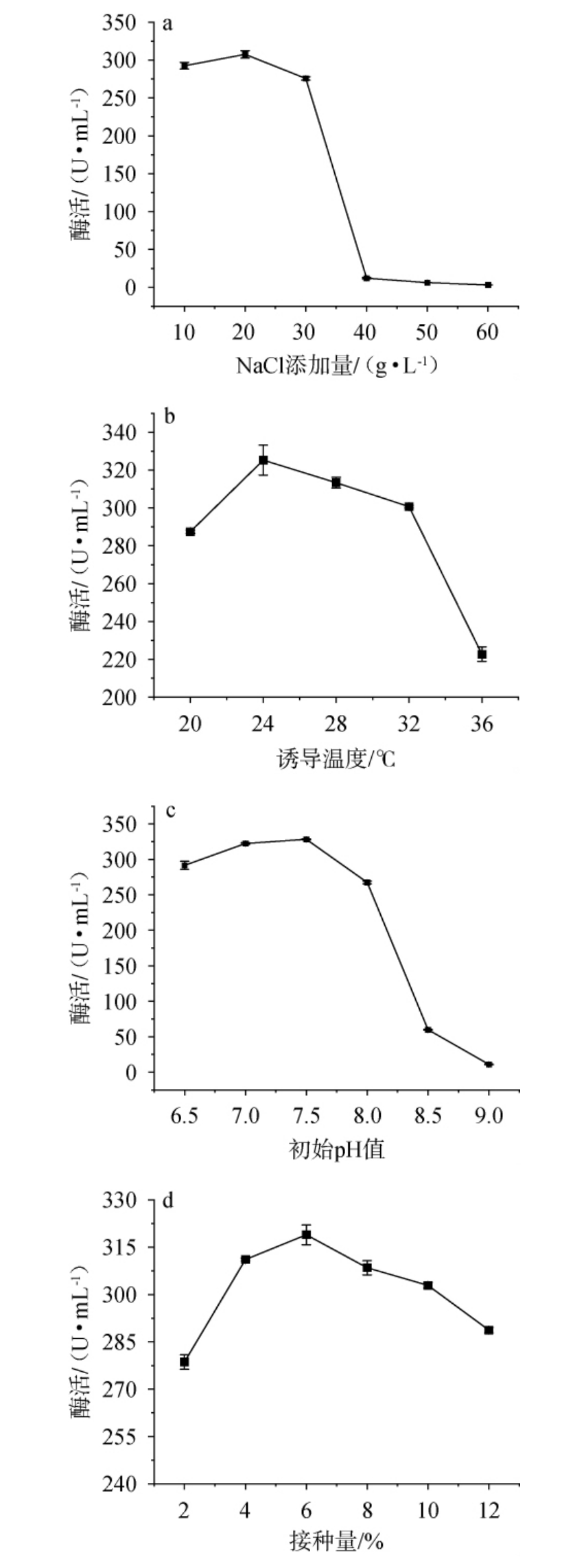
图5 NaCl添加量(a),诱导温度(b),初始pH值(c)及接种量(d)对重组羧酸酯酶Est40活力的影响
Fig.5 Effect of NaCl addition (a), induction temperature (b), initial pH value (c) and inoculum on the activities of recombination carboxylesterase Est40
由图5a可知,随着NaCl添加量的升高,羧酸酯酶活力呈先升高后下降的趋势;当NaCl添加量为20 g/L时,羧酸酯酶活力最高,为(306.12±8.56)U/mL。过高的NaCl含量则会对菌体生长产生抑制作用,导致细胞脱水变形,进而破坏酶蛋白的高级结构,降低酶活[33]。因此,确定NaCl的最佳添加量为20 g/L。
由图5b可知,随着诱导温度的升高,羧酸酯酶活力呈先上升后下降的趋势。 当诱导温度为24 ℃时,羧酸酯酶活力最高,为(322.92±13.76)U/mL。分析原因可能是,过高的诱导温度可导致重组酯酶错误折叠或聚集,形成无活性的包涵体,影响酶的活性和稳定性,不利于重组工程菌酯酶的积累[34]。因此,确定最佳诱导温度为24 ℃。
微生物的最适生长pH值因菌种而异,通常细菌的最适pH值范围为7.0~8.0[35]。 环境pH值通过影响微生物的代谢活动,进而调控其生长速率和胞内酶活性[36]。 由图5c可知,随着初始pH的升高,羧酸酯酶活力呈先升高后下降的趋势,当初始pH值为7.5时,羧酸酯酶活力达到最大,为(328.23±1.41)U/mL。分析原因可能是由于极端pH条件会引起酶蛋白分子构象的改变,导致活性中心结构破坏[37]。因此,确定最佳初始pH值为7.5。
接种量显著影响菌体生长和产酶效率,适宜接种量可缩短延滞期,促进菌体生长并抑制杂菌;过高接种量则会导致溶氧不足,影响酶合成[38]。由图5d可知,随着接种量的升高,酯酶活力呈先升高后下降的趋势,当接种量为6%时,酯酶活力最高,为(317.83±4.98)U/mL。因此,确定最佳接种量为6%。
2.2.2 PB试验设计及结果
以蔗糖添加量(A)、酵母粉添加量(B)、MgSO4添加量(C)、诱导温度(D)、初始pH(E)、接种量(F)和NaCl添加量(G)为考察因素,以重组羧酸酯酶Est40活力(Y1)为评价指标,通过Design-Expert 13.0软件设计PB试验,PB试验设计及结果见表3,方差分析结果见表4。 通过Design-Expert 13.0软件对表3试验数据进行多元一次回归拟合分析,得到线性回归方程:Y1=270.07-0.88A+0.062B-1.03C+0.493D+5.63C+5.01F-6.88G。
表3 Plackett-Burman试验设计及结果
Table 3 Design and results of Plackett-Burman experiments
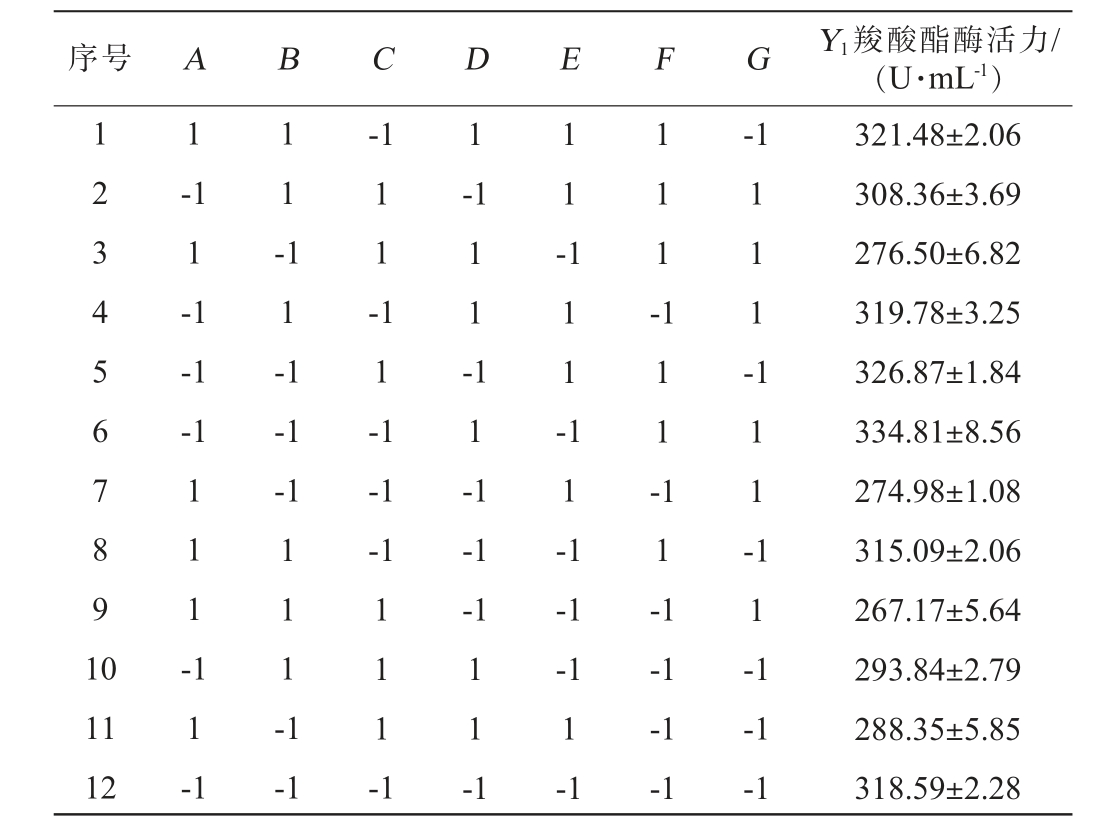
序号 A B C D E F G Y1羧酸酯酶活力/(U·mL-1)1 2 3 4 5 6 7 8 9 1 0 1-1 1-1-1 1-1-1-1 1 1 --1 1 1 -1 1 1 -1 1 -1 1 -1 1 1 -1 1 -1 1 1 1 -1 1 1 -1 1 1 --1-1-1-1 1 1 -1 1 1 1 -1 1 1 -1 1 1 -1 1 1 -1-1-1 1 1 1 -1 1 -1 11 12 1 1-1-1 1 1 1 -1 1 1 -1-1-1 1-1-1-1-1-1-1 321.48±2.06 308.36±3.69 276.50±6.82 319.78±3.25 326.87±1.84 334.81±8.56 274.98±1.08 315.09±2.06 267.17±5.64 293.84±2.79 288.35±5.85 318.59±2.28
表4 Plackett-Burman试验结果方差分析
Table 4 Variance analysis of Plackett-Burman experiment results
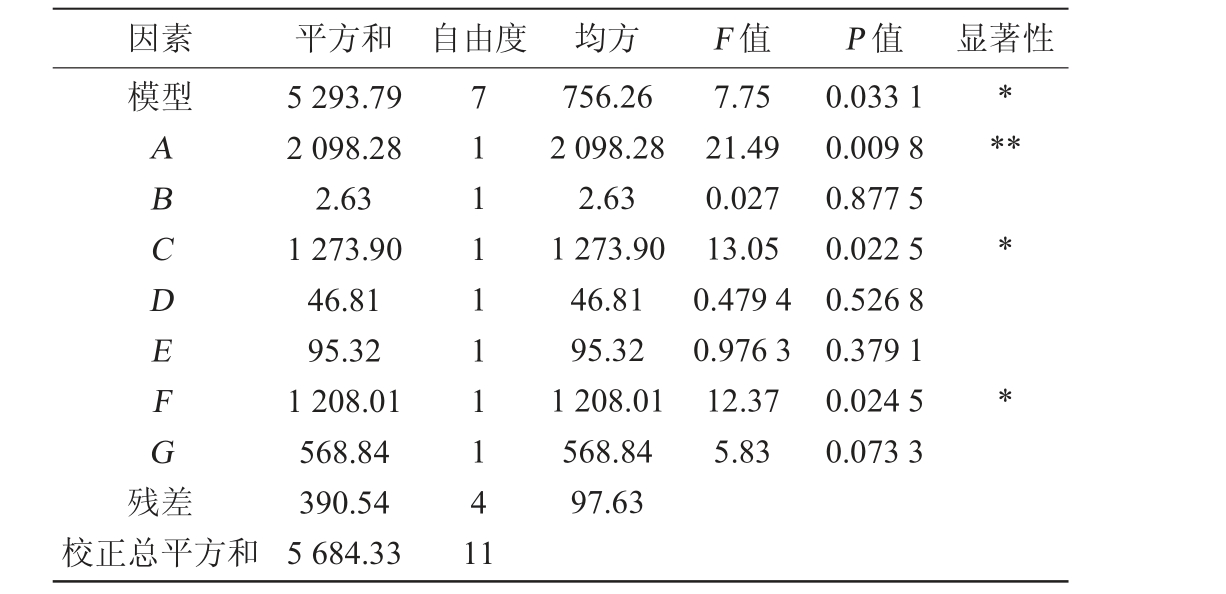
注:“*”表示对结果影响显著(P<0.05);“**”表示对结果影响极显著(P<0.01)。下同。
因素 平方和 自由度 均方 F 值 P 值 显著性模型*ABCDEFG残差756.26 2 098.28 2.63 1 273.90 46.81 95.32 1 208.01 568.84 97.63 7.75 21.49 0.027 13.05 0.479 4 0.976 3 12.37 5.83 0.033 1 0.009 8 0.877 5 0.022 5 0.526 8 0.379 1 0.024 5 0.073 3****校正总平方和5 293.79 2 098.28 2.63 1 273.90 46.81 95.32 1 208.01 568.84 390.54 5 684.33 7111111141 1
由表4可知,模型的F值为7.75(P<0.05),表明该模型具有统计学意义。 回归模型的决定系数R2=0.931 3,表明模型拟合度良好;调整决定系数R2Adj=0.811 1,说明该模型可解释81.11%的响应值变化,具有较好的预测能力。由P值可知,蔗糖添加量(A)对羧酸酯酶活力影响极显著(P<0.01),而MgSO4添加量(C)和接种量(F)对羧酸酯酶活力的影响显著(P<0.05),而其他因素的影响不显著(P>0.05)。因此,选取这3个关键因素进行BB响应面试验。
2.2.3 BB响应面试验设计及结果
基于单因素试验及PB试验结果,固定酵母粉添加量15 g/L、NaCl添加量20 g/L、初始pH 7.5、诱导温度24 ℃,选取对重组酯酶Est40活力影响较大的蔗糖添加量(X1)、MgSO4添加量(X2)和接种量(X3)为考察因素,以重组羧酸酯酶Est40活力(Y2)为响应值,采用Design-Expert 13.0软件设计3因素3水平的BB响应面试验,试验设计及结果见表5,方差分析结果见表6。
表5 Box-Behnken试验设计及结果
Table 5 Design and results of Box-Behnken experiments
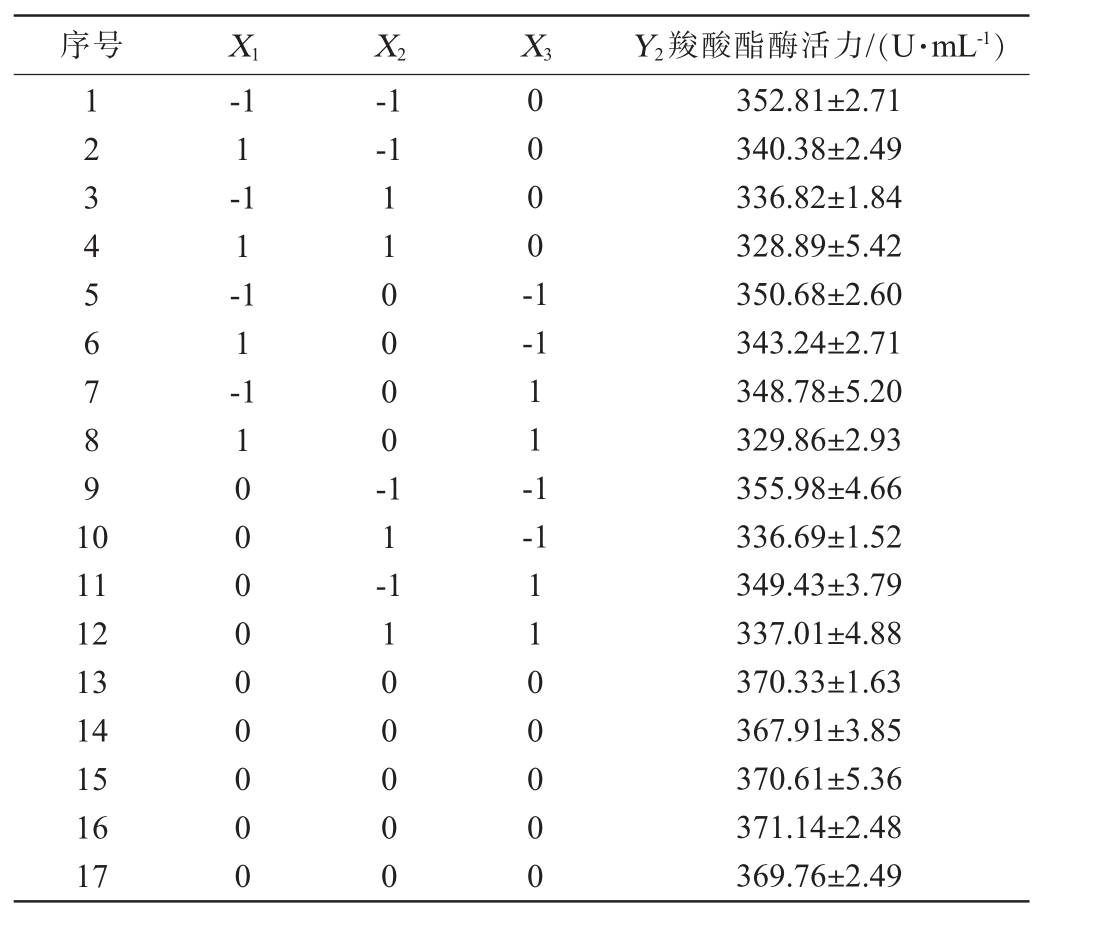
序号 X1 X2 X3 Y2羧酸酯酶活力/(U·mL-1)1234567891 0-11--1-1 11-11-110000-0000-1-1 11-1-1 11 12 13 14 15 16 17 11000000000 11-1100000 1100000 352.81±2.71 340.38±2.49 336.82±1.84 328.89±5.42 350.68±2.60 343.24±2.71 348.78±5.20 329.86±2.93 355.98±4.66 336.69±1.52 349.43±3.79 337.01±4.88 370.33±1.63 367.91±3.85 370.61±5.36 371.14±2.48 369.76±2.49
表6 回归模型方差分析
Table 6 Variance analysis of regression model
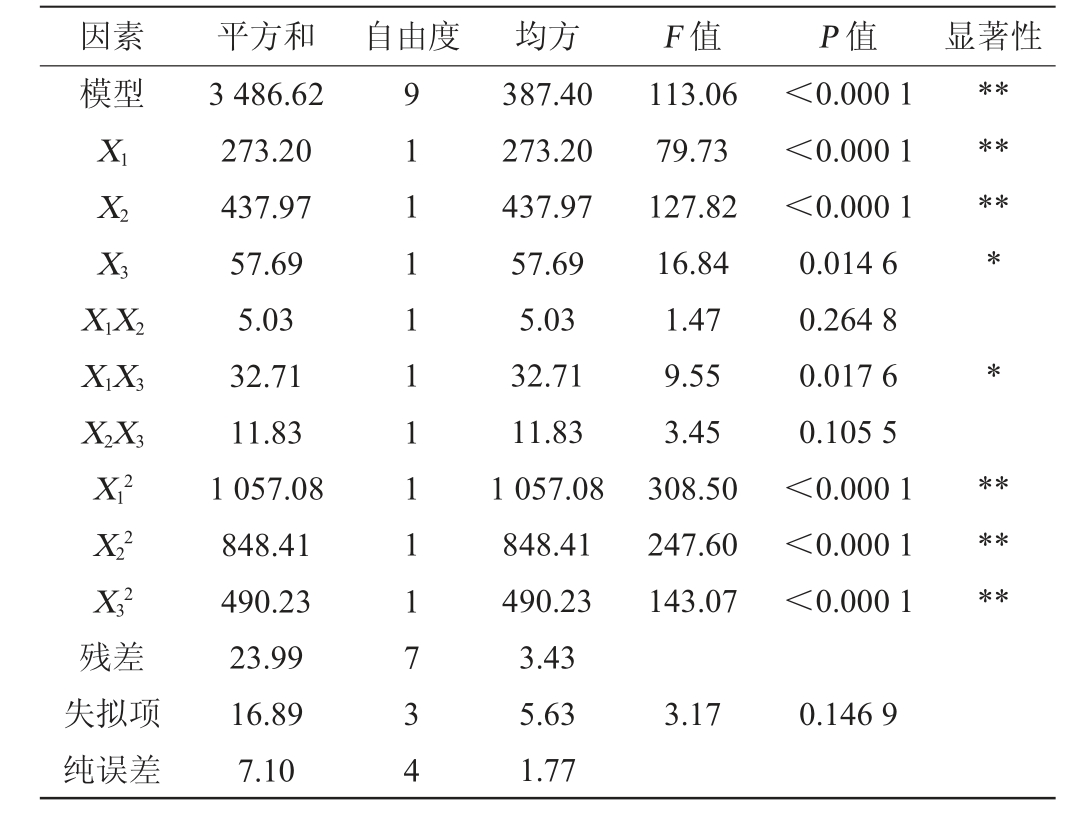
因素 平方和 自由度 均方 F 值 P 值 显著性模型X1 X2 X3 X1X2 X1X3 X2X3 X12 X22 X32残差失拟项纯误差3 486.62 273.20 437.97 57.69 5.03 32.71 11.83 1 057.08 848.41 490.23 23.99 16.89 7.10 9111111111734 387.40 273.20 437.97 57.69 5.03 32.71 11.83 1 057.08 848.41 490.23 3.43 5.63 1.77 113.06 79.73 127.82 16.84 1.47 9.55 3.45 308.50 247.60 143.07<0.000 1<0.000 1<0.000 1 0.014 6 0.264 8 0.017 6 0.105 5<0.000 1<0.000 1<0.000 1******** ******3.17 0.146 9
采用Design-Expert 13.0软件对表5的试验数据进行多元二次回归拟合分析,建立了预测重组工程菌产重组羧酸酯酶活力的多元二次回归模型:Y2=369.76-5.84X1-7.40X2-2.69X3+1.12X1X2-2.86X1X3+1.72X2X3-15.84X12-14.19X22-10.79X32。
由表6可知,该回归模型极显著(P<0.01),失拟项不显著(P>0.05),说明模型建立成功。模型的决定系数R2=0.993 2,表明模型能解释99.32%的响应值变异,拟合效果优异;调整决定系数R2Adj=0.984 4,进一步验证了模型具有良好的预测能力。由P值可知,一次项X1、X2及二次项X12、X22和X32对结果影响极显著(P<0.01);一次项X3及交互项X1X3对结果影响显著(P<0.05),其他项对结果影响不显著(P>0.05);由F值可知,各因素对结果影响的主次顺序为X2>X1>X3。 采用Design-Expert 13.0绘制各因素间交互作用对酯酶活力的影响,结果见图6。由图6可知,响应面均呈凸面,存在最大值,交互项X1X2、X2X3的响应面陡峭,等高线趋于圆形,说明对结果影响较小,而交互项X1X3的响应面平缓,等高线呈椭圆形,说明对结果影响较大,这与方差分析结果一致。
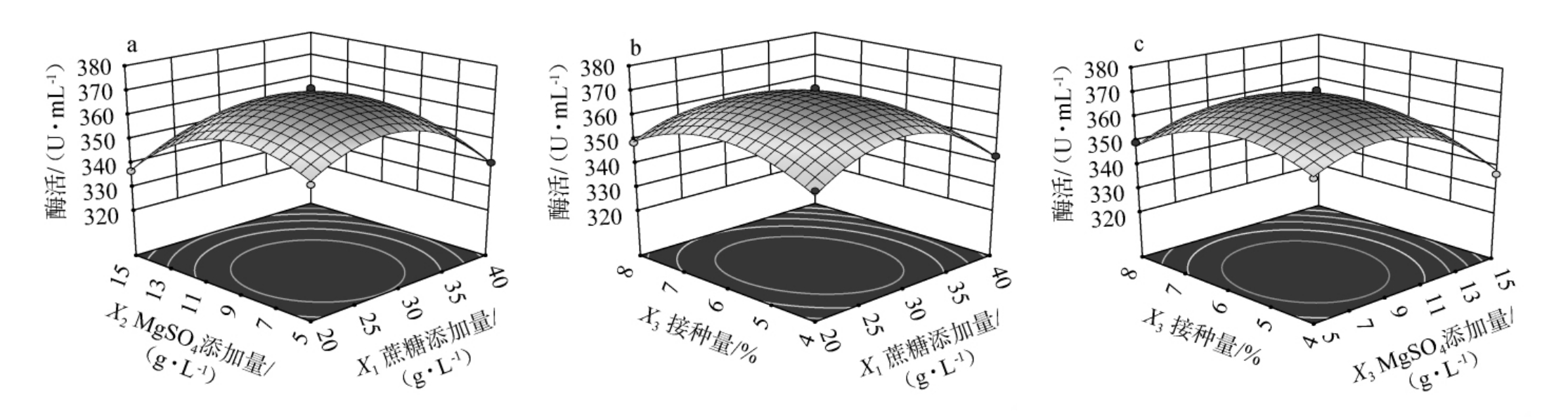
图6 各因素间交互作用对重组工程菌产重组羧酸酯酶Est40活力影响的响应面及等高线
Fig.6 Response surface plots and contour lines of effect of interaction between each factor on the activities of recombinant carboxylesterase Est40 produced by recombinant strain
采用Design-Expert 13.0软件对多元二次回归方程进行最优求解,得到重组工程菌的最佳发酵条件为:蔗糖34.67 g/L、MgSO412.25 g/L、接种量5.69%。为便于实际操作,将最优条件修正为蔗糖35 g/L、MgSO412 g/L,酵母粉15 g/L,NaCl 20 g/L,初始pH 7.5、诱导温度24 ℃及接种量6%,在此条件下羧酸酯酶活力的预测值为377.16 U/mL。在最佳发酵条件下进行3次重复验证试验,结果显示,该工艺条件下羧酸酯酶活力为(378.63±2.76)U/mL,与预测值接近,表明优化模型可信度较高。 通过响应面试验优化后,羧酸酯酶活力较优化前[(273.17±2.27)U/mL]提高38.61%。这一结果验证了响应面优化方法的可靠性。
3 结论
本研究通过基因克隆、重组质粒构建和异源表达技术,成功将盐湖嗜盐细菌Vreelandella sp.CH40的耐盐羧酸酯酶Est40在E.coli Rosetta(DE3)宿主细胞中高效表达。为提升重组羧酸酯酶的生产效率,以重组羧酸酯酶Est40活力为响应值,通过单因素试验、PB试验及BB响应面试验优化得到重组工程菌株产重组羧酸酯酶Est40的最佳发酵条件为蔗糖35 g/L、MgSO4 12 g/L、酵母粉15 g/L、NaCl 20 g/L、初始pH 7.5、诱导温度24 ℃和接种量6%,在此条件下,重组羧酸酯酶Est40活力达(378.63±2.76)U/mL,较优化前提高38.61%。该研究成果为后续重组羧酸酯酶的扩大化生产提供了可靠的技术参数和理论支撑。
[1]BORNSCHEUER U T.Microbial carboxyl esterases:classification,properties and application in biocatalysis[J].FEMS Microbiol Rev,2002,26(1):73-81.
[2]DELGADO-GARCIA M,RODRIGUEZ J A,MATEOS-DIAZ J C,et al.Halophilic archaeal lipases and esterases:Activity,stability,and food applications[J].Enzyme Food Technol,2018(11):243-262.
[3]DAHIYA D,SINGH NIGAM P.Sustainable biosynthesis of esterase enzymes of desired characteristics of catalysis for pharmaceutical and food industry employing specific strains of microorganisms[J].Sustainability,2022,14(14):8673.
[4]吕佳萱,龙启福,朱德锐,等.古菌酯酶的最新进展及应用[J].中国生物工程杂志,2025,45(Z1):147-163.
[5]毕文慧,桂伦,姚健.微生物酯酶的开发及其在食品工业中的应用现状[J].食品与发酵工业,2019,45(17):272-277.
[6]AKRAM F,FATIMA T,SHABBIR I,et al.Abridgement of microbial esterases and their eminent industrial endeavors[J].Mol Biotechnol,2024,67(3):817-833.
[7]MELTON L, SHAHIDI F, VARELIS P, et al.Encyclopedia of food chemistry[M].Netherlands:Elsevier,2019:158-167.
[8]BHATT P,ZHOU X,HUANG Y,et al.Characterization of the role of esterases in the biodegradation of organophosphate,carbamate,and pyrethroid pesticides[J].J Hazard Mater,2021,411:125026.
[9]刘喆,李家霖,白利平.微生物酯酶研究进展[J].微生物学报,2023,63(2):451-464.
[10]叶茂,邓毛程,顾宗珠,等.一种耐盐性酯酶在高盐稀态酱油酿造过程中的应用研究[J].中国调味品,2016,41(11):59-61.
[11]LIU L,YANG H,SHIN H,et al.How to achieve high-level expression of microbial enzymes: strategies and perspectives[J].Bioengineered,2013,4(4):212-223.
[12]KAUR J,KUMAR A,KAUR J.Strategies for optimization of heterologous protein expression in E.coli: Roadblocks and reinforcements[J].Int J Biol Macromol,2018,106:803-822.
[13]POURESMAEIL M, AZIZI-DARGAHLOU S.Factors involved in heterologous expression of proteins in E.coli host[J].Arch Microbiol,2023,205(5):212.
[14]李全发,房金鑫,喻娇,等.灰色链霉菌热碱稳定性酯酶的异源表达和酶学性质[J].微生物学报,2025,65(5):2201-2213.
[15]邓名,杨云娟.酯酶基因Est2的异源表达及酶学性质研究[J].云南师范大学学报(自然科学版),2021,41(2):51-58.
[16]魏振勇,于爽,迟雪梅,等.海洋来源成团泛菌Y-03产低温甾醇酯酶发酵条件优化[J].中国酿造,2025,44(1):219-225.
[17]易明花,任国平,胡琼,等.高产肌醇-1-磷酸合酶大肠杆菌重组工程菌的构建及其产肌醇发酵条件优化[J].中国酿造,2024,43(12):143-148.
[18]XIA S,LIN R,CUI X,et al.The application of orthogonal test method in the parameters optimization of PEMFC under steady working condition[J].Int J Hydrogen Energ,2016,41(26):11380-11390.
[19]BEZERRA M A,SANTELLI R E,OLIVEIRA E P,et al.Response surface methodology(RSM)as a tool for optimization in analytical chemistry[J].Talanta,2008,76(5):965-977.
[20]LV J X, ZHANG L, ZHU D R, et al.Optimization, purification and characterization of an intracellular salt-tolerant esterase Est40 from Vreelandella sp.CH40[J].Arch Microbiol,2025,207(9):191.
[21]MA X,LV J X,MA X R,et al.Isolation optimization and screening of halophilic enzymes and antimicrobial activities of halophilic archaea from the high-altitude, hypersaline Da Qaidam Salt Lake, China[J].J Appl Microbiol,2025,136(1):lxaf002.
[22]MATSUMOTO H, HANIU H, KOMORI N.Determination of protein molecular weights on SDS-PAGE[J].Methods Mol Biol,2019:101-105.
[23]DING J, ZHOU Y, ZHU H, et al.Characterization of EstZY: A new acetylesterase with 7-aminocephalosporanic acid deacetylase activity from Alicyclobacillus tengchongensis[J].Int J Biol Macromol, 2020,148:333-341.
[24]卢培利,杨涵,丁阿强,等.碳源与氮源限制下细菌代谢调节研究进展[J].微生物学报,2023,63(3):946-962.
[25]黄智鹏,薛茹男,王淑艺,等.贝莱斯芽孢杆菌A4的发酵条件优化及生防效果评价[J/OL].微生物学通报,1-15[2025-10-09].https://doi.org/10.13344/j.microbiol.china.250686.
[26]CESAR S, ANJUR-DIETRICH M, YU B, et al.Bacterial evolution in high-osmolarity environments[J].mBio,2020,11(4):e01191-20.
[27]杜丽红,郝亚男,陈宁,等.有机氮源及其在微生物发酵中的应用[J].发酵科技通讯,2019,48(1):1-4.
[28]付文娟,刘志伟,钟鸣,等.间型脉孢菌固体发酵豆渣产蛋白酶条件优化及其酶学特性研究[J].饲料研究,2025,48(4):80-86.
[29]MOUSAVI M,MEHRZAD J,NAJAFI M F,et al.Nitrate and ammonia:Two key nitrogen sources for biomass and phycocyanin production by Arthrospira(Spirulina)platensis[J].J Appl Phycol,2022,34(5):2271-2281.
[30]VOGEL H C,TODARO C M.Fermentation and biochemical engineering handbook[M].Norwich:William Andrew Publishing,2014:37-57.
[31]PILCHOVA I,KLACANOVA K,TATARKOVA Z,et al.The involvement of Mg2+ in regulation of cellular and mitochondrial functions[J].Oxid Med Cell Longev,2017,2017:6797460.
[32]YANG L,XIANG B,ZHAO H,et al.Influence of inorganic and organic salts on the hydration mechanism of montmorillonite based on molecular simulation[J].Sci Rep,2023,13(1):9090.
[33]ARENSE P, BERNAL V, IBORRA J L, et al.Metabolic adaptation of Escherichia coli to long-term exposure to salt stress[J].Process Biochem,2010,45(9):1459-1467.
[34]RESTREPO-PINEDA S, SANCHEZ-PUIG N, PEREZ N O, et al.The pre-induction temperature affects recombinant HuGM-CSF aggregation in thermoinducible Escherichia coli[J].Appl Microbiol Biot,2022,106(8):2883-2902.
[35]KRULWICH T, SACHS G, PADAN E.Molecular aspects of bacterial pH sensing and homeostasis[J].Nat Rev Microbiol, 2011, 9(5): 330-3439.
[36]RATZKE C,GORE J.Modifying and reacting to the environmental pH can drive bacterial interactions[J].PLoS Biol,2018,16(3):e2004248.
[37]MASSON P, LUSHCHEKINA S.Conformational stability and denaturation processes of proteins investigated by electrophoresis under extreme conditions[J].Molecules,2022,27(20):6861.
[38]张文勇,张恒慧,王晓丽.紫外线诱变选育高产豆豉纤溶酶菌株及其产酶条件优化[J].中国酿造,2024,43(3):177-181.