甲壳素脱乙酰酶(chitin deacetylase,CDA)是一种能专一降解甲壳素,水解脱去甲壳素上乙酰基的酶[1-2]。甲壳素主要存在于各类甲壳动物的外壳及真菌细胞壁中,但其难溶于一般的稀酸、稀碱及有机溶剂,难以应用[3-4],而甲壳素经脱乙酰化后的产物壳聚糖易于进行化学修饰且溶解性能好,并具有抗癌、降低胆固醇、降血压等生物活性,广泛应用于轻工业、美容业、农业及生物医学等领域[5-6]。相较于化学法和物理法,利用甲壳素脱乙酰酶制备壳聚糖绿色环保[7-9],是实现虾蟹壳废弃物高值化利用的关键[10]。
鲁氏毛霉(Mucor rouxii)、赖氨酸芽孢杆菌属(Lysinibacillus sp.)等天然菌株已被广泛用于CDA生产,但普遍存在酶活低[11-13]、纯化困难等问题,难以满足工业化需求[14-16]。近年来,基因工程技术的发展为CDA的异源表达提供了新途径,从真菌、细菌及节肢动物中可分离出CDA基因[17-18],并尝试在大肠杆菌、昆虫细胞及毕赤酵母等系统中进行表达[19-22]。其中,毕赤酵母(Pichia pastoris)凭借其高效的蛋白分泌能力和真核修饰优势成为理想宿主,SHRESTHA B等[23]将来源于炭疽菌(Colletotrichum lindemuthianum)的CDA基因在毕赤酵母中表达,获得的重组酶比活力为71.67 U/mg,但产量仅为7.2 mg/L;尽管通过优化载体设计提升了同一基因的表达水平,但产量仍达不到工业化水平[24]。传统诱导型启动子pAOX1依赖甲醇诱导,存在安全隐患且操作复杂,而基于组成型启动子pGAP的表达系统无需诱导剂[25-26],已在葡萄糖氧化酶[27]、血管内皮生长因子等蛋白的高效分泌中展现优势,其产物纯度可达98%以上[28]。然而,现有CDA研究多局限于胞内表达,依赖繁琐的破壁提取工艺,且对酶学特性的系统评估不足,限制了其规模化应用。
本研究从酿酒酵母中克隆到甲壳素脱乙酰酶基因ScCDA1和ScCDA2,并对其进行生物信息学分析,采用同源重组技术将其分别连接至pGAPK载体(含pGAP组成型强启动子),在毕赤酵母GS115中实现异源表达,研究其脱乙酰基酶活性,并采用碱量法测定重组菌株对甲壳素的脱乙酰度,以期为甲壳素脱乙酰酶的工业化发酵生产提供理论依据,为甲壳素脱乙酰酶的应用研究提供技术支撑。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株、载体及引物
酿酒酵母(Saccharomyces cerevisiae)S288C:河北北纳生物科技有限公司;毕赤酵母(Pichia pastoris)GS115:北京酷来搏科技有限公司;表达载体pCAMBIA1301:北京全式金生物技术股份有限公司;酵母表达载体pGAPZB:武汉淼灵生物科技有限公司。基因克隆、载体构建、菌落聚合酶链式反应(polymerase chain reaction,PCR)鉴定所需引物见表1,引物由生工生物工程(上海)股份有限公司合成。
表1 本研究所用引物
Table 1 Primers used in this study

引物名称 序列(5'→3')ScCDA1-F1 ScCDA1-R1 ScCDA2-F1 ScCDA2-R1 Zeo-F Zeo-R Kan-CE-F1 Kan-CE-R1 pGAP-F ATGAAAATTTTCAATACAATAC CTAGTCGTAGCGTTCGA ATGAGAATACAACTAAATACAATTG TTAGGACAAGAATTCTTTTATG CACGTCCGACGGCGGCCCAC GGTTTAGTTCCTCACCTTGTCGT ACAAGGTGAGGAACTAAACCATGGCTAAAATGAGAATATCACCGG GTGGGCCGCCGTCGGACGTGCTAAAACAATTCATCCAGTAAAATATAATATTT GTCCCTATTTCAATCAATTGAA
1.1.2 试剂
高保真酶PrimeSTARHS(Premix):宝日医生物技术(北京)有限公司;限制性内切酶(EcoR Ⅰ、Kpn Ⅰ):美国Thermo Scientific公司;同源重组ClonExpress II One Step Cloning试剂盒、FastPure Gel 脱氧核糖核酸(deoxyribonucleic acid,DNA)Extraction Mini试剂盒:南京诺唯赞生物科技股份有限公司;酵母基因组DNA提取试剂盒:北京索莱宝科技有限公司;EasyPure Plasmid MiniPrep试剂盒、1 kb DNA Ladder、Trans DL 2000 DNA Marker:北京全式金生物技术股份有限公司;10×磷酸盐缓冲液:北京酷来搏科技有限公司;遗传霉素(G418)、卡那霉素(Kanamycin,Kan):上海碧云天生物技术有限公司;免染聚丙烯酰胺凝胶电泳(polyacrylamidegel electrophoresis,PAGE)凝胶快速制备试剂盒(10%)、Omni-EasyTM速溶型蛋白上样缓冲液(变性,还原型,5×)、三色预染蛋白Marker(10~180 kDa):上海雅酶生物医药科技有限公司;非干扰型蛋白质浓度测定试剂盒:生工生物工程(上海)股份有限公司;对硝基乙酰苯胺、对硝基苯胺:上海阿拉丁生化科技股份有限公司;甲壳素:上海麦克林生化科技股份有限公司。本研究所用试剂均为国产分析纯。
1.1.3 培养基
酵母浸出粉胨葡萄糖(yeast extract peptone dextrose,YPD)液体培养基:青岛海博生物技术有限公司;BMGY/BMMY基础培养基:北京酷来搏科技有限公司。
1.2 仪器与设备
MyCycler 梯 度PCR 仪、GTMINI 水 平 电 泳 仪、Mini-PROTEAN Tetra垂直电泳系统:美国Bio-Rad公司;ZF-288凝胶成像系统:上海嘉鹏科技有限公司;Micro 21R微量离心机:美国Thermo Fihser科技公司;ZQZY-98CN全温振荡培养箱:上海知楚仪器有限公司;HH-4双列数显恒温水浴锅:常州国华仪器制造有限公司;Synergy HI多功能酶标仪:美国Bio Tek公司;SW-CJ-2FD超净工作台:苏州安泰空气技术有限公司。
1.3 方法
1.3.1 甲壳素脱乙酰酶基因的克隆及生物信息学分析
根据酿酒酵母S288C全基因组序列和甲壳素脱乙酰酶基因ScCDA1、ScCDA2的编码序列,利用Primer 5.0软件设计特异引物(ScCDA1-F1/ScCDA1-R1和ScCDA2-F1/ScCDA2-R1),以酿酒酵母基因组DNA为模板扩增目的基因。利用BioEdit软件(Version 7.2.6)对ScCDA1和ScCDA2蛋白序列进行多序列比对分析(Clustal W),比较同源性。对得到的甲壳素脱乙酰酶基因进行生物信息学分析:利用Expasy在线网站(https://web.expasy.org/protparam/)对蛋白质的理化性质进行分析,利用DeepTMHMM-1.0软件(https://services.healthtech.dtu.dk/services/DeepTMHMM-1.0/)和SignalP6.0(https://services.healthtech.dtu.dk/services/SignalP-6.0/)在线工具分别对蛋白质进行跨膜结构预测和信号肽分析。此外,利用PRIPRED Workbench(http://bioinf.cs.ucl.ac.uk/psipred/)对蛋白的二级结构进行预测,再利用SWISSMODEL(https://swissmodel.expasy.org/interactive)中 的 同源建模法分析蛋白的三维结构。
1.3.2 重组质粒pGAPK的构建
分别以质粒pGAPZB和pCambia1301为模板,设计引物扩增不包含Zeocin抗性基因的pGAP骨架大片段(Zeo-F/Zeo-R)和Kan 抗 性 基 因(Kan-CE-F1/Kan-CE-R1),利 用FastPure Gel DNA Extraction Mini试剂盒切胶回收线性化载体和抗性基因片段后(方法参见说明书中“凝胶回收方案”),同源重组连接以上两个片段。用EcoRⅠ单酶切pGAPK载体,利用FastPure Gel DNA Extraction Mini试剂盒过柱回收酶切产物(方法参见说明书中“PCR反应液回收方案”),利用同源重组ClonExpress II One Step Cloning试剂盒分别进行ScCDA1、ScCDA2基因和pGAPK的连接,鉴定得到阳性重组质粒。
1.3.3 毕赤酵母的转化与重组蛋白表达
将阳性重组质粒进行Kpn Ⅰ单酶切,采用化转法转化毕赤酵母GS115感受态细胞,取100 μL菌液涂布于含有不同质量浓度(0.5mg/mL、1mg/mL、2 mg/mL、4 mg/mL)G418的YPD平板上,筛选得到高拷贝的重组转化子,并进行PCR验证。
挑取阳性单克隆,接种于5 mL YPD培养基中,28 ℃、250 r/min振荡培养24 h,按1%的接种量接种至25 mL BMGY培养基中扩大培养,28 ℃、250 r/min振荡培养至OD600nm值=2~4,4 500 r/min离心5 min收集细胞,用BMGY培养基重悬细胞至OD600nm值=1.0,28 ℃、250 r/min振荡培养进行摇瓶发酵,分别于0、24 h、48 h、72 h和96 h取样,室温12 000 r/min离心5 min,取上清液,即为粗酶液。
取培养72 h后的发酵上清液进行十二烷基硫酸钠(sodium dodecyl sulfate,SDS)-PAGE电泳检测。采用非干扰型蛋白质浓度测定试剂盒测定粗酶液中的蛋白质含量,实验方法参见试剂盒说明书。
1.3.4 甲壳素脱乙酰酶酶活测定
精确称取0.01 g对硝基苯胺,加水溶解并定容于100 mL容量瓶中,混匀后进行梯度稀释至质量浓度为4mg/L、8mg/L、12 mg/L、16 mg/L和20 mg/L,以蒸馏水作空白参照,测定各稀释液在波长400 nm处的吸光度值(OD400nm值)。以对硝基苯胺质量浓度(x)为横坐标,以OD400nm值(y)为纵坐标绘制标准曲线,获得标准曲线回归方程y=0.021 9x+0.000 9,相关系数R2为0.999 9。
采用对硝基乙酰苯胺为底物,测定重组蛋白的脱乙酰基酶活性。取200 mg/L的对硝基乙酰苯胺溶液1 mL和0.05 mol/L的Tris-HCl缓冲液(pH=8.0)3 mL,50 ℃水浴保温3 min后,加入1.3.3中粗酶液1 mL,50 ℃水浴反应15 min,沸水浴终止酶促反应,加水定容至10 mL,混匀,3000r/min离心10min,测定上清液在波长400nm处的吸光度值(OD400nm值)。
CDA酶活单位的定义:在上述反应条件下每小时产生1μg对硝基苯胺所需要的酶量定义为1个酶活力单位(U/mL)。计算公式如下:
式中:ΔOD400 nm值为酶解液样品与空白组在波长400 nm条件下的吸光度值的差异;T 为酶促反应时间,h;k为标准曲线的线性系数。
比酶活的定义:在上述反应条件下,单位质量(mg)蛋白质所具有的酶活性单位数为比酶活(U/mg)。比酶活计算公式如下:
1.3.5 脱乙酰度的测定
分别制备酵母菌pGAPK-ScCDA1/GS115和pGAPKScCDA2/GS115的种子液,按1%的接种量分别接种至100mL BMGY培养基中(每瓶培养基中添加3 g甲壳素),取一瓶未接种的培养基作为对照,28 ℃、250 r/min培养3 d。发酵结束后,将发酵液在8 000 r/min离心15 min,除去上清液,并用去离子水反复洗涤沉淀,备用。
采用碱量法测定脱乙酰度。分别取上述制备的烘干至恒定质量的底部沉淀0.2 g,置于250mL锥形瓶中,加入30 mL 0.1 mol/L的盐酸标准滴定溶液,在20~25 ℃下搅拌至试样溶解后,加入2滴甲基橙-苯胺蓝指示液,用0.1 mol/L的NaOH标准滴定溶液滴定,当溶液颜色由紫红色变为蓝绿色时,停止滴定。读取所耗NaOH标准滴定溶液体积数值,计算脱乙酰度,其计算公式如下:
式中:c2为盐酸标准滴定溶液的浓度,mol/L;V2为加入盐酸标准滴定溶液的体积,mL;c0为氢氧化钠标准滴定溶液的浓度,mol/L;V0为滴定用氢氧化钠标准滴定溶液的体积,mL;16为氨基的摩尔质量,g/mol;m2为试样质量,g;0.099 4为理论氨基含量(16/161)。
2 结果与分析
2.1 甲壳素脱乙酰酶基因的克隆与生物信息学分析
ScCDA1和ScCDA2基因扩增电泳图及其蛋白序列的同源性分析见图1。
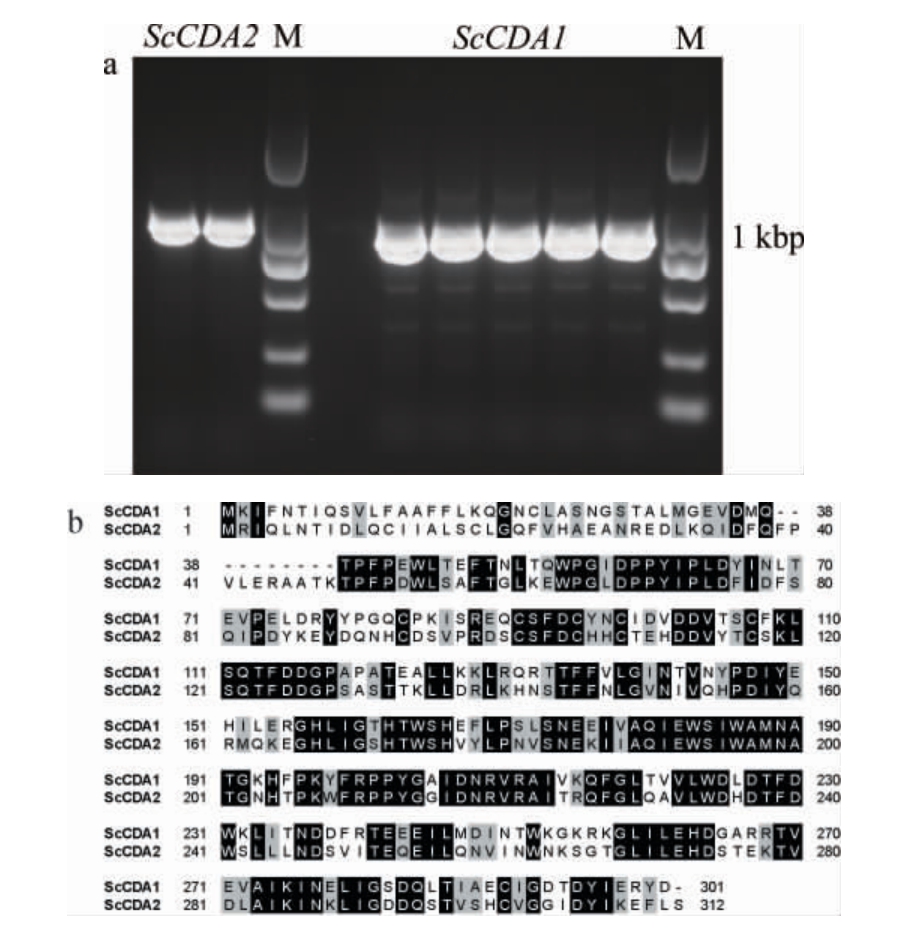
图1 ScCDA1和ScCDA2基因扩增电泳图(a)及其蛋白序列的同源性分析(b)
Fig.1 Amplification electropherogram of ScCDA1 and ScCDA2 genes(a) and homology analysis of their protein sequences (b)
M为Trans DL 2000 DNA Marker。
由图1a可知,ScCDA1和ScCDA2目的基因碱基长度分别为906 bp和939 bp,扩增条带单一且条带位置与目的基因大小相符。由图1b可知,两个蛋白同源性为51%。
一级序列分析结果表明,ScCDA1和ScCDA2结构简单,均不含有内含子。ScCDA1和ScCDA2蛋白质的理化性质分析结果表明,ScCDA1蛋白由301个氨基酸组成,分子质量为34.64 kDa,不稳定系数为32.03<40,属于稳定蛋白;亲水性平均系数(GRAVY)为-0.246<0,属于亲水性蛋白。ScCDA2蛋白由312个氨基酸组成,分子质量为35.69 kDa,不稳定系数为32.74<40,属于稳定蛋白;亲水性平均系数(GRAVY)为-0.365<0,属于亲水性蛋白。蛋白质跨膜结构预测结果表明,ScCDA1和ScCDA2蛋白均没有跨膜区,不存在跨膜的可能。信号肽分析结果表明,ScCDA1和ScCDA2N端序列Sec/SPI信号肽概率值分别为0和0.0005,说明ScCDA1和ScCDA2蛋白均属于非分泌蛋白,不存在信号肽序列。
ScCDA1和ScCDA2蛋白的二级结构预测分析结果见图2。由图2可知,ScCDA1和ScCDA2蛋白序列中含有α螺旋(Helix)、β折叠(Strand)和大量的无规则卷曲(Coil)。
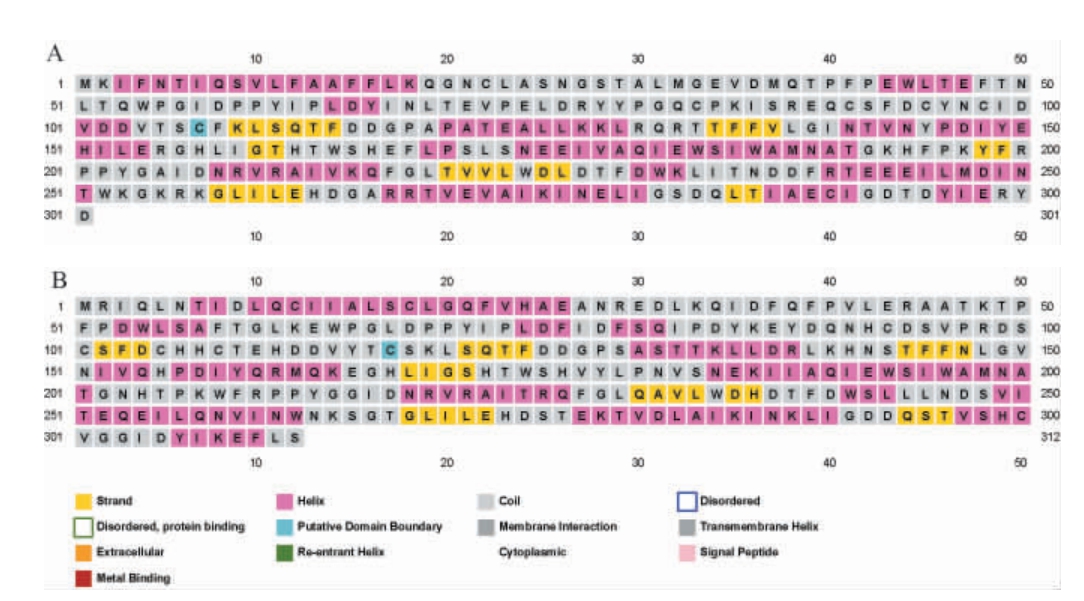
图2 ScCDA1(A)和ScCDA2(B)蛋白的二级结构预测分析
Fig.2 Prediction analysis of secondary structure of ScCDA1 (A)and ScCDA2 (B)proteins
ScCDA1和ScCDA2蛋白的三维结构预测分析结果见图3。
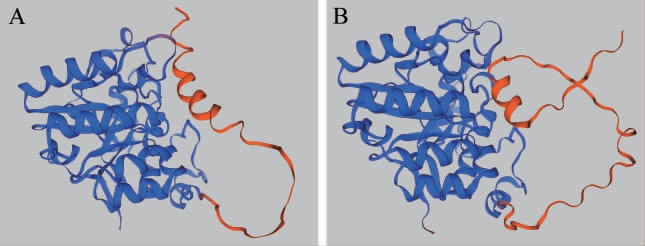
图3 ScCDA1(A)和ScCDA2(B)蛋白的三维结构预测分析
Fig.3 Prediction analysis of three-dimensional structure of ScCDA1 (A)and ScCDA2 (B)proteins
由图3可知,模板A0A7I9FQ65与ScCDA1序列的一致度为99%,全球性模型质量估测(global model quality estimation,GMQE)值为0.89;模板A0A6C1DWW9_SACPS与ScCDA2序列完全一致,GMQE值为0.87。其中,GMQE值越接近1则建模质量越好,表示模型越可靠。
2.2 重组表达载体的构建
基因扩增及单菌落PCR扩增产物电泳图见图4。由图4A可知,以质粒pGAPZB为模板,设计引物扩增不包含Zeocin抗性基因的pGAP质粒骨架大片段,得到碱基长度为2 507 bp的线性化载体。由图4B可知,以质粒pCambia1301为模板,扩增得到长度为816 bp的Kan抗性基因。由图4C可知,通过同源重组将Kan片段与pGAP载体进行连接,经菌落PCR鉴定和测序后,得到筛选标记为卡那霉素(G418)的毕赤酵母表达载体pGAPK。由图4D可知,pGAPK载体为3 401 bp,用EcoRⅠ单酶切载体后,电泳结果符合。由图4E可知,利用同源重组试剂盒分别将ScCDA1、ScCDA2基因和pGAPK连接,挑选转化子进行菌落PCR扩增,在952 bp(ScCDA1-pGAPK)和985 bp(ScCDA2-pGAPK)处有明显条带,表明成功得到阳性转化子。
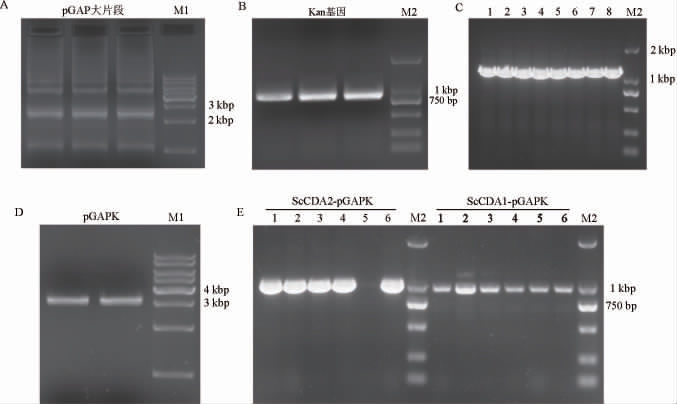
图4 基因扩增及单菌落聚合酶链式反应扩增产物电泳图
Fig.4 Electrophoretogram of gene amplification and single colony polymerase chain reaction amplification products
A为pGAP大片段扩增电泳图;B为Kan抗性基因扩增电泳图;C为pGAPK载体构建的菌落PCR扩增产物电泳图;D为pGAPK单酶切电泳图;E:ScCDA1-pGAPK和ScCDA2-pGAPK载体构建的菌落PCR扩增产物电泳图;M1为1 kb DNA Ladder;M2为Trans 2k DNA Marker。
2.3 重组酵母菌株的筛选与蛋白表达
将鉴定正确的重组质粒ScCDA1-pGAPK和ScCDA2-pGAPK分别线性化后转化毕赤酵母GS115感受态细胞,将菌液涂布到含有不同浓度G418的YPD平板上,30 ℃恒温培养至长出转化子,挑取高浓度G418(4 mg/mL)平板上的单克隆进行菌落PCR扩增,结果见图5。由图5可知,条带大小与预计大小一致(952 bp和985 bp),最终筛选得到pGAPKScCDA1/GS115和pGAPK-ScCDA2/GS115高拷贝重组阳性转化子。
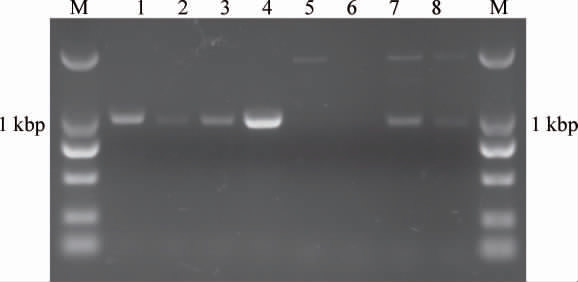
图5 重组酵母转化子的聚合酶链式反应扩增产物电泳图
Fig.5 Electrophoretogram of polymerase chain reaction amplification products of recombinant yeast transformant
M为Trans DL 2000 DNA Marker;1~4为ScCDA2-pGAPK重组转化子;5~8为ScCDA1-pGAPK重组转化子。
将重组菌株发酵表达后的上清液进行SDS-PAGE分析,结果见图6。
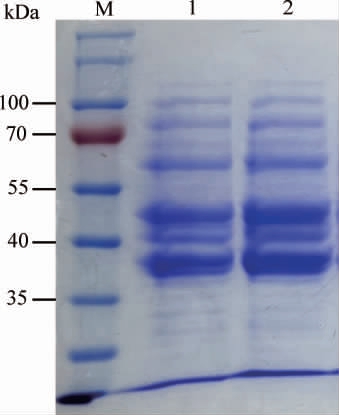
图6 甲壳素脱乙酰酶重组蛋白的十二烷基硫酸钠-聚丙烯酰胺凝胶电泳图
Fig.6 Sodium dodecyl sulfate-polyacrylamide gel electrophoresis of recombinant chitin deacetylase protein
M为三色预染蛋白分子质量标准(10~180 kDa);1为ScCDA1蛋白上清液;2为ScCDA2蛋白上清液。
由图6可知,重组子在<40kDa处有明显条带,与预期得到带有标签的目的蛋白分子质量一致(36.7 kDa和37.7kDa),表明重组ScCDA1和ScCDA1蛋白在毕赤酵母中成功表达。据报道,利用毕赤酵母表达系统,孙晓彤等[29]成功表达了重组甜菜夜蛾几丁质脱乙酰酶SeCDA1蛋白和构巢曲霉(Aspergillus nidulans)几丁质脱乙酰酶(AnCDA)蛋白。
2.4 培养时间对甲壳素脱乙酰酶酶活的影响
根据PCR鉴定结果,选取1株ScCDA1和1株ScCDA2重组酵母菌株进行摇瓶发酵培养,选取培养至24 h、48 h、72 h和96 h表达的重组蛋白进行酶活测定。采用对硝基乙酰苯胺为底物,测定重组CDA蛋白的脱乙酰基酶活性,分别以0 h的酶液为对照,各重组菌株中表达的脱乙酰基酶活见图7。由图7可知,随发酵时间在0~72 h范围内的延长,酶的活性逐渐升高;发酵时间为72 h,酶活达到最高值,分别为18.69 U/mL和18.14 U/mL,比酶活分别为17.18 U/mg和17.41 U/mg,高于天然菌株的产酶活性[14,16]和异源表达的重组酶活性[30],此后酶活开始下降且不再增加。这可能是由于发酵时间过长,菌体衰老甚至死亡,不利于酶的产生。酿酒酵母ScCDA1和ScCDA2基因编码蛋白的同源性并不高(51%),它们在结构上的区别在于ScCDA2是由一小段原结构域(prodomain,Cda1p)和成熟区(Cda2p)组成,Cda2p更活跃,执行主要的脱乙酰化功能,而Cda1p有助于精细调控[31]。MARTINOU A等[32]研究发现,在大肠杆菌中表达ScCDA2的完整序列,得到的酶无活性,本研究中2个CDA异源重组表达时脱乙酰酶酶活接近,这是由于酵母等真核生物表达系统可以对Cda1p区进行切割,从而表达有活性的酶蛋白,也表明ScCDA和ScCDA2在甲壳素脱乙酰功能上可能存在冗余现象[33]。因此,最佳培养时间为72 h。
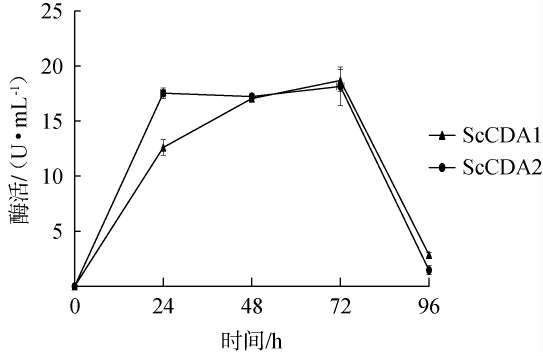
图7 培养时间对脱乙酰基酶活的影响
Fig.7 Effect of culture time on deacetylase activity
2.5 重组菌株对甲壳素脱乙酰度的测定
分别将pGAPK-ScCDA1/GS115 和pGAPK-ScCDA2/GS115重组菌株接种至添加有甲壳素的发酵培养基中培养3 d,测定甲壳素脱乙酰度,结果表明,重组菌株均具有脱乙酰基的能力,甲壳素脱乙酰度分别为59.20%和53.55%,极显著高于对照组(P<0.01)。从自然界中筛选的赖氨酸芽孢杆菌菌株对甲壳素的脱乙酰度最高为10.646%[14],AnCDA和ScCDA2经过原核和真核表达后,对甲壳素的脱乙酰活性也仅为0.5%和20.33%[12],本研究构建的重组菌株对底物具有更高的水解活性,能得到性能更好高品质的壳聚糖产物。
3 结论
本研究成功克隆了酿酒酵母S.cerevisiae的甲壳素脱乙酰酶基因ScCDA1和ScCDA2,生物信息学分析显示ScCDA1和ScCDA2结构简单,其编码蛋白均为亲水性蛋白且不含跨膜区和信号肽,序列比对结果显示两个蛋白同源性为51%。利用基因同源重组,成功将ScCDA1和ScCDA2基因分别连接到改造后的含有组成型强启动子pGAP的表达载体pGAPK上,并整合至毕赤酵母GS115中,构建了无需甲醇诱导且成本更低的重组表达系统(抗性标记优化为npt II),通过引入组氨酸标签(histidine-tag,His-Tag)融合蛋白设计为后续蛋白纯化及功能解析奠定基础。利用G418高浓度(4 mg/mL)平板成功筛选出高拷贝的pGAPK-ScCDA1/GS115和pGAPK-ScCDA2/GS115重组子。重组子发酵表达72 h后的发酵上清液中检测到目的蛋白相应条带,并且脱乙酰酶酶活分别达到18.69 U/mL和18.14 U/mL,对底物甲壳素的脱乙酰度达到50%以上。本研究在毕赤酵母中成功表达了酿酒酵母中的2个甲壳素脱乙酰酶基因,克服了跨物种异源表达障碍,为甲壳素脱乙酰酶的应用研究提供了依据。
[1]ROMAN D L,ROMAN M,SLETTA H,et al.Assessment of the properties of chitin deacetylases showing different enzymatic action patterns[J].J Mol Graph Model,2019,88:41-48.
[2]TSIGOS I,MARTINOU A,KAFETZOPOULOS D,et al.Chitin deacetylases:new,versatile tools in biotechnology[J].Trends Biotechnol,2000,18(7):305-312.
[3]CRINI G.Historical review on chitin and chitosan biopolymers[J].Environ Chem Lett,2019,17:1623-1643.
[4]ZOU P,YANG X,WANG J,et al.Advances in characterisation and biological activities of chitosan and chitosan oligosaccharides[J].Food Chem,2016,190:1174-1181.
[5]WANG W,MENG Q,LI Q,et al.Chitosan derivatives and their application in biomedicine[J].Int J Mol Sci,2020,21(2):487.
[6]RISEH R S,VAZVANI M G,KENNEDY J F.The application of chitosan as a carrier for fertilizer:A review[J].Int J Biol Macromol,2023,252:126483.
[7]GRÉGORIO C, LICHTFOUSE E.Sustainable Agriculture Reviews 36:Chitin and Chitosan:Applications in Food,Agriculture,Pharmacy,Medicine and Wastewater Treatment[M].Switzerland:Cham:Springer International Publishing,2019:45-79.
[8]LIN S, QIN Z, CHEN Q, et al.Efficient Immobilization of bacterial GH family 46 chitosanase by carbohydrate-binding module fusion for the controllable preparation of chitooligosaccharides[J].J Agric Food Chem,2019,67(24):6847-6855.
[9]PILLAI C K S,PAUL W,SHARMA C P.Chitin and chitosan polymers:Chemistry,solubility and fiber formation[J].Prog Polym Sci,2009,34(7):641-678.
[10]王丹,吴反修.中国渔业统计年鉴[M].北京:中国农业出版社,2022:89-94.
[11]KAFETZOPOULOS D, BOURIOTIS M V.Bioconversion of chitin to chitosan: purification and characterization of chitin deacetylase from Mucor rouxii[J].Proc Natl Acad Sci USA,1993,90(7):2564-2568.
[12]LIU Z, GAY L M, TUVENG T R, et al.Structure and function of a broad-specificity chitin deacetylase from Aspergillus nidulans FGSC A4[J].Sci Rep,2017,7(1):1746.
[13]GAO X D,KATSUMOTO T,ONODERA K.Purification and characterization of chitin deacetylase from Absidia coerulea[J].J Biochem,1995,117(2):257-263.
[14]孟欣,艾国峰,姜国涛,等.产甲壳素脱乙酰酶赖氨酸芽孢杆菌的筛选及产酶初探[J].食品工业科技,2022,43(24):139-144.
[15]郭依依,吴峥,马天颖,等.蜡状芽胞杆菌促进红球菌发酵产甲壳素脱乙酰酶[J].食品与发酵工业,2021,47(23):158-163.
[16]王晓玲,万芳芳,刘高强.湘江河岸土壤中高产甲壳素脱乙酰酶菌株的筛选及鉴定[J].微生物学通报,2016,43(5):1019-1026.
[17]ARAKI Y,ITO E.A pathway of chitosan formation in Mucor rouxii:Enzymatic deacetylation of chitin[J].Biochem Biophys Res Commun,1974,56(3):669-675.
[18]SONG L,CHEN Y,AN X,et al.Chitin deacetylase 2 is essential for molting and survival of Tetranychus urticae[J].Pestic Biochem Physiol,2021,179:104962.
[19]JIANG Z,NIU T,LV X,et al.Secretory expression fine-tuning and directed evolution of diacetylchitobiose deacetylase by Bacillus subtilis[J].Appl Environ Microbiol,2019,85(17):e01076-19.
[20]KARBALAEI M,REZAEE S A,FARSIANI H.Pichia pastoris:A highly successful expression system for optimal synthesis of heterologous proteins[J].J Cell Physiol,2020,235(9):5867-5881.
[21]LI X, CHU J, DIAO P, et al.Identification and expression profiles of chitin deacetylase family genes in the Chinese mitten crab, Eriocheir sinensis[J].Aquac Res,2021,52(7):15166.
[22]危蓉萍,杨东航,卜莹莹,等.蜜蜂螺原体几丁质脱乙酰酶基因的克隆、表达及酶学性质[J].南京农业大学学报,2016,39(3):417-424.
[23]SHRESTHA B, BLONDEAU K, STEVENS W F, et al.Expression of chitin deacetylase from Colletotrichum lindemuthianum in Pichia pastoris:purification and characterization[J].Protein Expr Purif,2004,38(2):196-204.
[24]KANG L,CHEN X,ZHAI C,et al.Synthesis and high expression of chitin deacetylase from Colletotrichum lindemuthianum in Pichia pastoris GS115[J].J Microbiol Biotechnol,2012,22(9):1202-1207.
[25]WATERHAM H R,DIGAN M E,KOUTZ P J,et al.Isolation of the Pichia pastoris glyceraldehyde-3-phosphate dehydrogenase gene and regulation and use of its promoter[J].Gene,1997,186(1):37-44.
[26]曹东艳,柳倩,贺晓云,等.源于GAP启动子的毕赤酵母组成型表达系统的研究进展[J].食品安全质量检测学报,2013(4):1217-1221.
[27]石义超,王凤忠,江均平,等.毕赤酵母组成型高效表达启动子的筛选鉴定[J].食品工业科技,2018,39(13):110-116.
[28]周伟杰,吴凤梅,姚冬生,等.通过毕赤酵母表达系统获得高纯度的重组人血管内皮生长因子(rhVEGF165)[J].生物工程学报,2021,37(11):4083-4094.
[29]孙晓彤,赵丹,闫晓平,等.甜菜夜蛾几丁质脱乙酰酶1(SeCDA1)在毕赤酵母中的表达与活性测定[J].中国生物防治学报,2015,31(4):586-591.
[30]张巧,李永成.Rhizopus stolonifer几丁质脱乙酰酶的异源表达及其与几丁质酶的协同作用研究[J].食品与发酵工业,2024,50(2):177-183.
[31]MARTINOU A,KOUTSIOULIS D,BOURIOTIS V.Expression,purification,and characterization of a cobalt-activated chitin deacetylase(Cda2p)from Saccharomyces cerevisiae[J].Protein Expr Purif,2002,24(1):111-116.
[32]MARTINOU A, KOUTSIOULIS D, BOURIOTIS V.Cloning and expression of a chitin deacetylase gene(CDA2)from Saccharomyces cerevisiae in Escherichia coli:Purification and characterization of the cobaltdependent recombinant enzyme[J].Enzyme Microb Technol,2003,32(6):757-763.
[33]CHRISTODOULIDOU A,BOURIOTIS V, THIREOS G.Two sporulation-specific chitin deacetylase-encoding genes are required for the ascospore wall rigidity of Saccharomyces cerevisiae[J].J Biol Chem,1996,271(49):31420-31425.