虾因其丰富的营养和鲜美的风味而成为全球消费最广泛的水产品之一。由于生虾极易腐败,为延长其保质期,收获后必须立即进行加工[1]。虾酱是一种具有营养价值的传统发酵食品,通常以毛虾、虾头或虾肉为原料[3],采用盐发酵进行加工,使生虾在室温下“日晒夜露”完成自然发酵[2-3]。在发酵过程中,微生物代谢将虾体内的大分子碳水化合物、蛋白质和脂肪酸分解为鲜味肽、游离氨基酸和短链脂肪酸等代谢产物,赋予虾酱独特的营养价值和感官特性[4-5]。此外,长期“日晒夜露”发酵有助于加速虾酱成熟,并保持其品质稳定。然而,虾酱的品质受原料种类、发酵工艺和生态环境等因素影响,同时与发酵过程中微生物群落的组成密切相关[6]。
虾酱中微生物群落结构组成不仅决定发酵产品的品质[8]、质地和风味[9],还可能影响其安全性。班雨函等[10]研究表明,在低盐发酵虾酱过程中,四联球菌属(Tetragenococcus)的丰度与氨基酸态氮、挥发性盐基氮和丙二醛含量均呈正相关,而乳酸杆菌属(Lactobacillus)则与丙二醛和生物胺含量呈负相关。王才政[11]研究发现协同接种嗜盐四联球菌、浮游盐扁平菌和植物乳杆菌可提高虾酱的氨基酸态氮含量,并降低挥发性氨基氮和生物胺含量。然而,不同产地的虾酱生产多依赖自然发酵,导致虾酱品质特征具有明显的地域差异。因此,研究不同地区虾酱细菌群落结构对产品品质的提升具有重要意义。
本研究以环渤海地区不同产地的自然发酵虾酱为对象,采用常规检测方法分析其理化指标(水分含量、盐含量、pH、氨基酸态氮、挥发性盐基氮和丙二醛),采用高通量测序技术分析其细菌群落结构,基于检测结果进行主坐标分析(principal co-ordinates analysis,PCoA)、层次聚类分析(hierarchical cluster analysis,HCA),采用线性判别分析效大小(linear discriminant analysis effect size,LEfSe)对差异菌群进行分析,并对细菌群落结构与理化指标进行冗余分析(redundancy analysis,RDA)及相关性分析。以期为不同产地虾酱细菌群落和理化品质的差异提供理论依据,为虾酱标准化工业生产提供数据支撑。
1 材料与方法
1.1 材料与试剂
1.1.1 原料
虾酱:虾酱于2024年3~4月采集自辽宁大连、河北秦皇岛、河北唐山、山东烟台等4个产地,所用原料品种为毛虾,采用“日晒夜露”传统工艺酿造。每个产地采集4份不同家庭自制的虾酱,其样品信息见表1。
表1 虾酱样品信息
Table 1 Information of shrimp paste samples
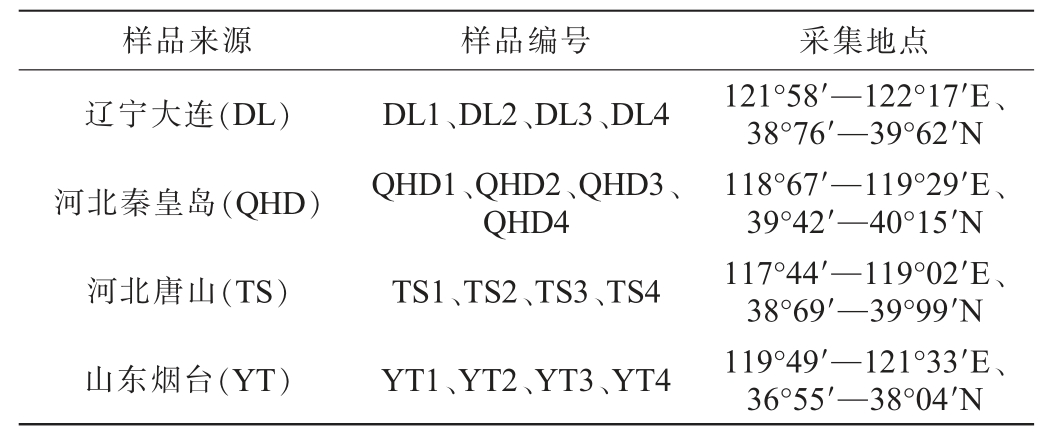
样品来源 样品编号 采集地点辽宁大连(DL)河北秦皇岛(QHD)河北唐山(TS)山东烟台(YT)DL1、DL2、DL3、DL4 QHD1、QHD2、QHD3、QHD4 TS1、TS2、TS3、TS4 YT1、YT2、YT3、YT4 121°58′—122°17′E、38°76′—39°62′N 118°67′—119°29′E、39°42′—40°15′N 117°44′—119°02′E、38°69′—39°99′N 119°49′—121°33′E、36°55′—38°04′N
1.1.2 试剂
硝酸银、亚铁氰化钾、乙酸锌、氢氧化钠、甲醛、酚酞、邻苯二甲酸氢钾、三氯乙酸、硼酸、硫代巴比妥酸、乙二胺四乙酸二钠(均为分析纯):国药集团化学试剂有限公司;FavorPerp Soil 脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒:台湾惠晶生物科技股份有限公司;PyrobestTM DNA聚合酶:北京宝日医生物技术有限公司。
脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒:台湾惠晶生物科技股份有限公司;PyrobestTM DNA聚合酶:北京宝日医生物技术有限公司。
1.2 仪器与设备
L124-1SCN型分析天平:德国赛多利斯公司;Orion Star A211型pH计:美国赛默飞公司;DHG-9145A型电热鼓风干燥箱、DKZ-2B型恒温振荡水槽:上海一恒科学仪器有限公司;UV-5200型紫外可见分光光度计、B-600型超微量分光光度计:上海元析仪器有限公司;DYCP-32B型琼脂糖水平电泳仪:北京六一仪器厂;1600R型凝胶图像分析系统:上海天能生命科学有限公司;Miseq PE300型高通量测序仪:美国Illumina公司。
1.3 方法
1.3.1 理化指标测定
水分含量:参照国标GB 5009.3—2016《食品安全国家标准 食品中水分的测定》;pH:参照国标GB 5009.237—2016《食品安全国家标准食品pH值的测定》;盐含量:参照国标GB 5009.44—2016《食品安全国家标准食品中氯化物的测定》;氨基酸态氮:参照国标GB 5009.235—2016《食品安全国家标准食品中氨基酸态氮的测定》;挥发性盐基氮:参照国标GB 5009.228—2016《食品安全国家标准 食品中挥发性盐基氮的测定》;丙二醛:参照国标GB 5009.181—2016《食品安全国家标准食品中丙二醛的测定》[4]。
1.3.2 DNA提取、高通量测序及序列分析
DNA提取:称取0.5 g样品,按照FavorPerp Soil  DNA提取试剂盒说明书提取不同产地虾酱样品的微生物总DNA。采用超微量分光光度计测定样品DNA的浓度和纯度,并结合2%琼脂糖凝胶电泳检测其完整性[4]。
DNA提取试剂盒说明书提取不同产地虾酱样品的微生物总DNA。采用超微量分光光度计测定样品DNA的浓度和纯度,并结合2%琼脂糖凝胶电泳检测其完整性[4]。
PCR扩增:应用细菌16S rRNA V3-V4扩增引物(338F:5'-ACTCCTACGGGAGGCAGCA-3'和806R:5'-GGACTACHVGGGTWTCTAAT-3')对样品进行聚合酶链式反应(polymerasechainreaction,PCR)扩增[4]。PCR扩增体系(20μL):5 μL 2×Gflex PCR缓冲液,1 μL上游引物(5 pmol/μL)和下游引物(5 pmol/μL),1 μL DNA聚合酶,1 μL模板DNA,并加双蒸水(ddH2O)补足[12]。PCR扩增条件为:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火20 s,72 ℃延伸30 s,30次循环;72 ℃再延伸10 min[13]。使用2%琼脂糖凝胶电泳检测PCR扩增产物,切胶回收后,委托广州迈景基因医学科技有限公司利用Illumina Miseq PE300平台进行高通量测序。
使用QIIME2[14]软件分析测序数据,根据原始序列的条形码和碱基得分对样品进行解复用和质量过滤,获得优质序列,随后,通过DADA2插件切除测序引物,拼接双端序列,去除嵌合体,得到扩增子序列变体(amplicon sequence variants,ASVs),基于Silva 138细菌数据库对ASVs进行比对,并以80%置信度进行分类学注释,获得每个ASV的注释信息[4]。
1.3.3 统计分析
采用GraphPad Prism 10软件绘图,并使用SPSS 21.0软件进行单因素方差分析(analysis of variance,ANOVA);采用布雷柯蒂斯距离(Bray-Curtis Distance)计算样品间差异,并通过置换多元方差分析(permutational multivariate analysis of variance,ADONIS)进行统计检验;线性判别分析效应大小(LEfSe)用于发现和解释高维度数据中生物标识差异,线性判别分析(linear discriminant analysis,LDA)值越大,表明该生物标识在区分不同组别中的贡献越大,根据LDA值>2.0,P<0.05筛选显著差异微生物。
2 结果与分析
2.1 不同产地虾酱理化指标的差异分析
不同产地虾酱的理化指标检测结果见图1。由图1A可知,不同产地虾酱的水分含量范围为62%~70%,其中DL和TS地区的虾酱水分含量分别为69.18%、66.99%,显著高于QHD和YT地区(P<0.05)。盐是虾酱发酵过程中重要的防腐剂和调味剂,可有效抑制有害菌生长,延缓腐败变质[9]。由图1B可知,QHD、DL、YT及TS地区虾酱的盐含量分别为12.72%、10.05%、8.59%和6.49%,差异均显著(P<0.05),表明不同产地虾酱的加工工艺存在差异。由图1C可知,DL、QHD、TS和YT地区虾酱的pH值分别为7.68、7.99、7.08和7.36,各地区之间差异显著(P<0.05),可能与脂质氧化过程有关。脂质氧化可促进蛋白质氧化水解,生成游离氨基酸和脂肪酸,进而增加虾酱酸度,降低pH值[3]。氨基酸态氮是衡量虾酱品质的重要指标[4]。由图1D可知,各产地虾酱的氨基酸态氮含量均高于1.2 g/100 g(参照GB/T 18186—2025《酱油质量通则》特级酱油),其中YT、DL、QHD和TS地区虾酱的氨基酸态氮含量分别为1.38 g/100 g、1.34 g/100 g、1.33 g/100 g和1.25 g/100 g,DL及QHD样品氨基酸态氮含量差异不显著(P>0.05)。微生物分泌的蛋白酶可将蛋白质等含氮化合物水解为氨基酸,从而提高虾酱的氨基酸态氮含量[4]。此外,氨基酸与碳水化合物发生美拉德反应,生成褐色色素及风味物质,促进虾酱成熟[15]。挥发性盐基氮和丙二醛是衡量虾酱新鲜度的重要指标,主要受腐败菌代谢及内源性酶活性的影响[4-5]。由图1E可知,各产地虾酱的挥发性盐基氮含量为180~280 mg/100 g,均低于450mg/100g[4],其中TS地区虾酱的挥发性盐基氮含量最高,可达261.09 mg/100g,显著高于其他地区(P<0.05)。由图1F可知,YT和TS地区虾酱的丙二醛含量分别为1.53 mg/kg和1.38 mg/kg,显著高于DL和QHD地区(P<0.05)。挥发性盐基氮主要来源于蛋白质分解和氨基酸脱氨作用,而丙二醛则由脂质氧化产生,其含量受原料新鲜度、发酵工艺及微生物活性影响[16]。过高的挥发性盐基氮和丙二醛含量不仅降低虾酱的营养价值和感官品质,还可能带来食品安全风险[17]。因此,不同产地虾酱的理化指标存在明显区别。

图1 不同产地虾酱的理化指标检测结果
Fig.1 Detection results of physicochemical indexes of shrimp paste from different producing areas
不同小写字母表示差异显著(P<0.05)。
2.2 不同产地虾酱细菌群落序列数目及Alpha多样性分析
不同产地虾酱细菌群落的序列数量和Alpha多样性分析结果见表2。由表2可知,各地区虾酱的优质序列数目为72 561~82 306条,且各地区之间无显著差异(P>0.05)。此外,各地区虾酱微生物群落覆盖率均>99%,表明测序深度足以全面反映虾酱的物种多样性,测序数据可靠,可用于后续分析[13]。基于Chao1指数、Shannon指数和Simpson指数对虾酱微生物的Alpha多样性进行综合评估,Chao1指数反映物种丰富度,Shannon指数和Simpson指数用于反映物种多样性;指数越高,说明其物种多样性和丰富度越高,微生物种类越多[18]。TS和YT地区虾酱的Shannon指数分别为4.93和4.24,Simpson指数分别为0.86、0.73,显著高于DL和QHD地区(P<0.05)。表明TS和YT地区虾酱的物种多样性较高。此外,TS地区虾酱的Chao1指数可达375,显著高于其他地区(P<0.05),进一步表明TS地区虾酱的物种丰富度较高。盐含量是影响传统发酵食品微生物多样性的关键因素之一。一般而言,盐含量越高,物种丰富度和多样性越低,能够适应高渗透压环境的微生物种类相对减少[9,19]。此外,高盐发酵环境不仅抑制有害菌的生长,还限制酿造微生物的繁殖速率,减缓发酵进程[9]。综上,不同产地虾酱的微生物Alpha多样性存在明显区别。
表2 不同产地虾酱细菌群落的序列数目和Alpha多样性分析结果
Table 2 Sequences number and Alpha diversity analysis results of bacterial communities in shrimp paste from different producing areas
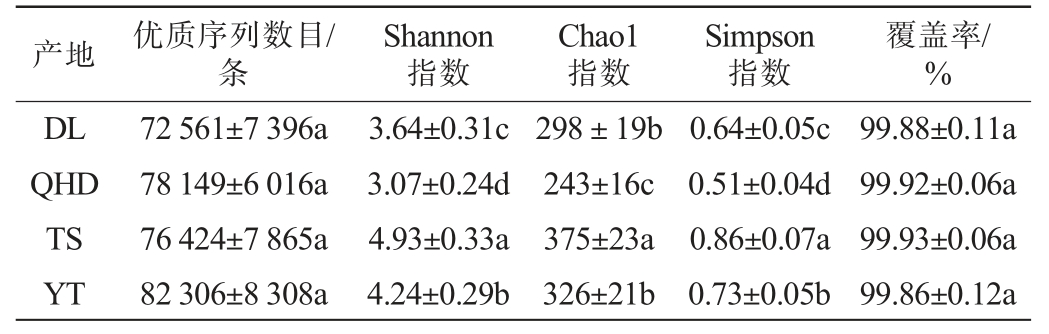
注:同列不同小写字母表示差异显著(P<0.05)。
产地 优质序列数目/条Shannon指数Chao1指数Simpson指数覆盖率/%DL QHD TS YT 72 561±7 396a 78 149±6 016a 76 424±7 865a 82 306±8 308a 3.64±0.31c 3.07±0.24d 4.93±0.33a 4.24±0.29b 298±19b 243±16c 375±23a 326±21b 0.64±0.05c 0.51±0.04d 0.86±0.07a 0.73±0.05b 99.88±0.11a 99.92±0.06a 99.93±0.06a 99.86±0.12a
2.3 不同产地虾酱微生物差异分析
2.3.1 ASV分析
不同产地虾酱中细菌ASVs的韦恩图见图2。由图2可知,不同产地虾酱共有121个ASV,分别占DL、QHD、TS和YT地区虾酱ASV总数的40.6%、49.79%、32.27%和37.12%,进一步说明TS地区虾酱的微生物多样性最高,可能由于其盐含量较低,使多种微生物共存并繁殖,从而形成多种优势菌群。QHD地区虾酱独有的ASV为10个,少于其他地区,表明QHD地区缺乏其他地区所含的大量微生物种类。气候环境、光照强度及发酵工艺均对虾酱微生物多样性产生重要影响[2]。因此,不同产地虾酱测序的ASV数量存在差异。
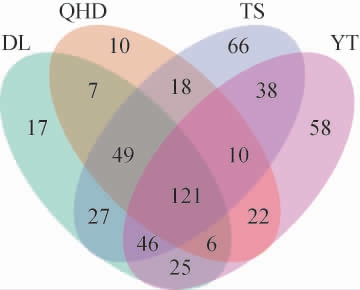
图2 不同产地虾酱中细菌群落扩增子序列变异体的韦恩图
Fig.2 Venn diagram of amplicon sequence variants of bacterial communities in shrimp paste from different producing areas
2.3.2 不同产地虾酱细菌群落结构分析
基于门水平不同产地虾酱细菌群落结构分析结果见图3。由图3可知,环渤海地区不同产地的虾酱共检测出7个优势细菌门(相对丰度>1%),分别为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、螺旋体门(Spirochaetes)、拟杆菌门(Bacteroidetes)、盐厌氧菌门(Halanaerobiaeota)、弯曲杆菌门(Epsilonbacteraeota)和放线菌门(Actinobacteria),不同产地的虾酱中主要优势细菌门为厚壁菌门、变形菌门和拟杆菌门。该结果与段泽瑞等[9]研究结论一致。在DL、QHD、TS和YT地区的虾酱中,厚壁菌门的平均相对丰度分别为72.51%、90.41%、55.76%和36.94%;变形菌门的平均相对丰度分别为9.52%、3.86%、14.11%和18.58%;拟杆菌门的平均相对丰度分别为9.97%、1.12%、14.05%和14.83%。YT地区虾酱的盐厌氧菌门相对丰度最高,可达22.34%,而QHD和TS地区分别为3.86%和7.11%,DL地区则几乎检测不到。此外,放线菌门仅存在于DL和TS地区的虾酱中,相对丰度分别为4.18%和5.28%,而螺旋体门仅在YT地区的虾酱检测到,其相对丰度为4.71%。综上,不同产地虾酱的优势细菌门组成及丰度存在差异。
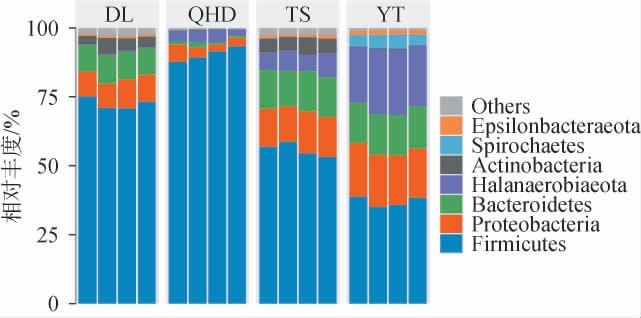
图3 基于门水平不同产地虾酱细菌群落结构分析结果
Fig.3 Analysis results of bacterial community structure of shrimp paste from different producing areas based on phylum level
Others表示相对丰度<0.5%的菌门。下同。
由图4可知,不同产地的16份虾酱共检测到60个细菌属(15个优势细菌属),其中相对丰度较高的有四联球菌属(Tetragenococcus)、乳酸杆菌属(Lactobacillus)、盐厌氧菌属(Halanaerobium)、芽孢杆菌属(Bacillus)、埃希氏-志贺氏菌属(Escherichia-Shigella)、肠球菌属(Enterococcus)和鳞球菌属(Sphaerochaeta)。在DL、QHD、TS和YT地区虾酱中,四联球菌属的平均相对丰度分别为50.22%、84.15%、9.64%和15.12%,其分布差异可能与虾酱酿造工艺有关。四联球菌属是传统发酵食品中的主要微生物,对产品的品质和风味形成起关键作用[21]。四联球菌属能够在高盐发酵环境中生长繁殖,分解蛋白质生成氨基酸,同时抑制生物胺的形成,从而改善虾酱的品质、风味和安全性[22]。该结果与图1趋势一致。乳酸杆菌属在DL、QHD、TS和YT地区虾酱中的平均相对丰度分别为6.83%、0.14%、25.99%和10.29%,而盐厌氧菌属的平均相对丰度分别为0.39%、3.86%、7.11%和22.34%。乳酸杆菌属在低盐发酵环境中快速生长,占据生态位,并通过产生乳酸和乙酸等有机酸,稳定虾酱的pH,抑制腐败菌生长,从而提升虾酱的风味和保质期[4,23]。盐厌氧菌属广泛存在于多种发酵海产品中,能够分解有机物、调节pH值并产生风味物质,这些功能共同促进发酵产品的成熟[4,24]。此外,芽孢杆菌属在YT地区虾酱中的平均相对丰度最高,可达5.07%,而在DL、QHD和TS地区虾酱中仅为2.19%、0.76%和3.47%。芽孢杆菌属具有较强的环境适应能力,能够在较宽的温度和pH范围内生长,并分泌多种蛋白酶、碳水化合物酶、酯酶及其他代谢产物,从而影响虾酱的品质和微生物群落组成[4,25]。芽孢杆菌属与盐厌氧菌属形成互利共生关系,后者利用前者分泌的酶将虾中的蛋白质和碳水化合物降解为小分子肽和糖,作为碳、氮源供自身生长繁殖,并产生乙酸、丁酸及CO2,形成无氧且酸性的发酵环境,进一步促进芽孢杆菌属的增殖[26]。埃希氏-志贺氏菌属在DL、QHD、TS和YT地区虾酱的平均相对丰度分别为0.99%、0.69%、0.86%和7.34%,该细菌属主要源自虾的肠道,其丰度差异可能受不同产地水体环境的影响。此外,TS地区虾酱中肠球菌属的相对丰度为2.68%,高于其他地区,而鳞球菌属仅存在与YT地区虾酱中。综上,不同产地虾酱的细菌群落结构存在显著差异,这种差异与原料来源、气候环境及酿造工艺密切相关。
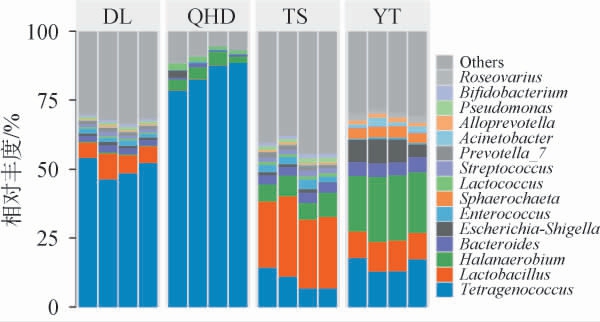
图4 基于属水平不同产地虾酱细菌群落结构分析结果
Fig.4 Analysis results of bacterial community structure of shrimp paste from different producing areas based on genus level
2.4 主坐标分析和层次聚类分析
不同产地虾酱细菌群落结构的主坐标分析和层次聚类分析见图5。由图5A可知,PCoA的前两轴共同可解释不同产地虾酱细菌群落方差总变异的86.24%,其中PCoA第一轴解释了总变异的47.55%,PCoA第二轴解释了总变异的38.69%。不同产地虾酱在PCoA坐标轴上分布于不同象限,表明其群落组成存在显著的地理差异。此外,QHD地区的虾酱位于坐标轴的第一象限,而DL、TS和YT地区的虾酱分别位于坐标轴的第二、三和四象限,表明不同产地虾酱的细菌群落组成存在显著差异(R2=0.942,P<0.05)。由图5B可知,同一地区的虾酱样本聚集在一起,且其细菌群落组成的内部差异小于地区间差异,表明地理环境是影响虾酱细菌群落组成的主要因素,该结果与MA Z Y等[20]研究相一致。因此,不同产地虾酱细菌群落组成差异显著。
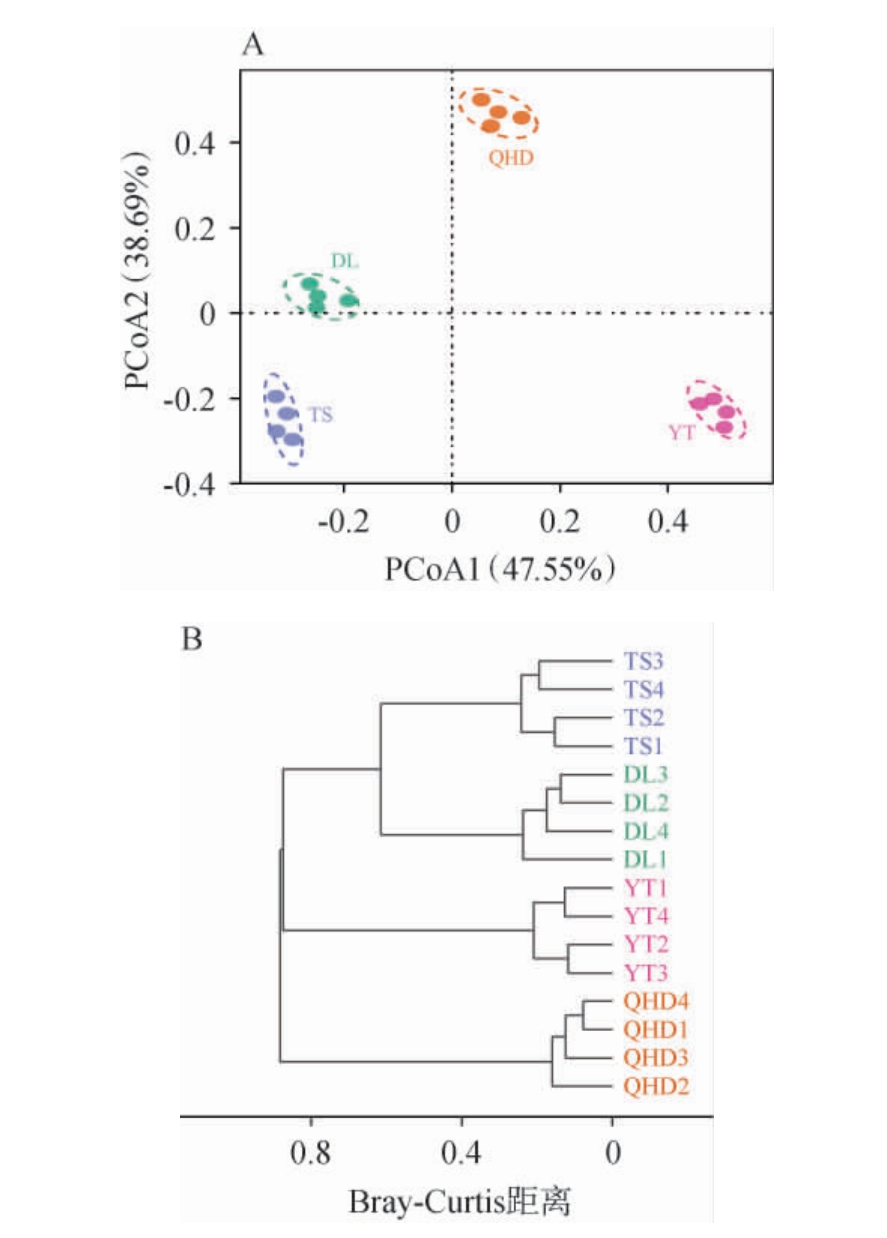
图5 不同产地虾酱细菌群落结构的主坐标分析(A)和层次聚类分析(B)
Fig.5 Principal coordinate analysis (A) and hierarchical cluster analysis (B) of bacterial community structure of shrimp paste from different producing areas
2.5 不同产地虾酱细菌群落结构线性判别分析效应大小分析
采用LEfSe分析法进一步鉴定不同产地虾酱细菌群落的差异性类群。基于属水平不同产地虾酱细菌群落结构的差异见图6。由图6可知,根据线性判别分析(LDA)值>2及方差分析P值<0.05,不同产地虾酱间显著差异细菌类群有一定区别。其中,DL地区虾酱仅在拟杆菌目(Bacteroidales)、Frankiales和疣微菌目(Verrucomicrobiales)的相对丰度上显著高于其他三个地区,而芽孢杆菌纲(Bacilli)、乳杆菌目(Lactobacillales)、弧 菌 目(Vibrionales)和 厚 壁 菌 门 在QHD地区虾酱中显著富集。此外,YT地区虾酱的差异类群最多,共鉴定出21个差异细菌类群,包括芽孢杆菌目(Bacillales)、弯曲菌目(Campylobacterales)、丹毒丝菌目(Erysipelotrichales)、盐厌氧菌目(Halanaerobiales)、红细菌目(Rhodobacterales)、交替单胞菌目(Alteromonadales)、肠杆菌目(Enterobacteriales)、巴斯德氏菌目(Pasteurellales)、螺旋体目(Spirochaetales)等,这些微生物易富集于虾肠道,与当地水体环境密切相关。而双歧杆菌目(Bifidobacteriales)、Microtrichales、梭菌目(Clostridiales)、硒单胞菌门(Selenomonadales)、梭杆菌目(Fusobacteriales)、根瘤菌目(Rhizobiales)、β-变形杆菌目(Betaproteobacteriales)、假单胞菌目(Pseudomonadales)则在TS地区虾酱中显著富集,这可能与该地区采用的低盐发酵工艺有关。低盐发酵条件下,多种微生物竞争利用原料中的碳氮源生长繁殖,并逐步形成不同生态位,从而共同促进虾酱成熟[27]。因此,不同产地的水体环境与酿造工艺对虾酱细菌群落组成具有特异性选择作用,从而导致细菌类群差异显著。
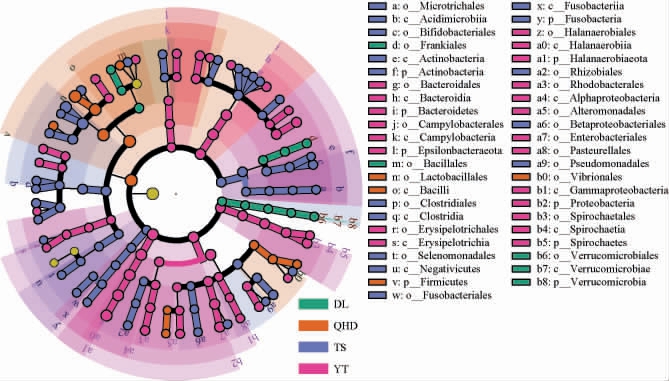
图6 不同产地虾酱细菌群落的线性判别分析效应大小分析结果
Fig.6 Linear discriminant analysis effect size analysis results of bacterial communities in shrimp paste from different producing areas
2.6 不同产地虾酱细菌群落与理化特性的相关性分析
冗余分析是一种用于提取和归纳响应变量变化的方法,并通过解释变量分析其影响因素,主要用于探讨群落物种组成受环境变量的约束关系[27]。不同产地虾酱细菌群落与理化指标的冗余分析见图7。
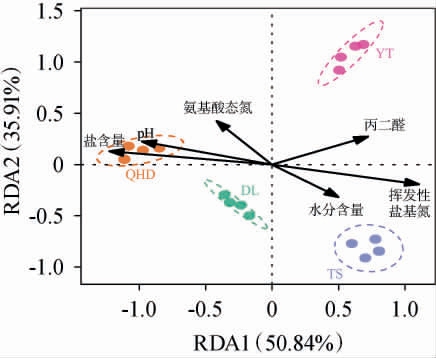
图7 不同产地虾酱细菌群落与理化指标的冗余分析
Fig.7 Redundancy analysis of bacterial communities and physicochemical indexes of shrimp paste from different producing areas
由图7可知,RDA1、RDA2分别解释了50.84%和35.91%的群落变异。其中,QHD地区的细菌群落与盐含量、pH和氨基酸态氮呈正相关,YT地区仅与丙二醛含量呈正相关,而TS地区则主要与水分含量和挥发性盐基氮含量呈正相关。此外,基于条件限制分析,进一步探讨不同理化指标对各产地虾酱细菌群落的影响,结果表明盐含量的影响最为明显,可解释25.11%的群落变异,其次为挥发性盐基氮含量(21.13%)和pH值(18.89%)。孙筱[19]研究发现,盐含量是影响自制香肠微生物多样性和群落结构的最显著因素,这一结果与本研究一致,进一步表明盐含量是塑造虾酱细菌群落结构的关键理化指标。因此,不同产地虾酱的细菌群落受多种理化指标的共同调控,其中盐含量的影响最为显著。
采用斯皮尔曼分析不同产地虾酱优势细菌属(相对丰度>1%)与理化指标之间的相关性,结果见图8。
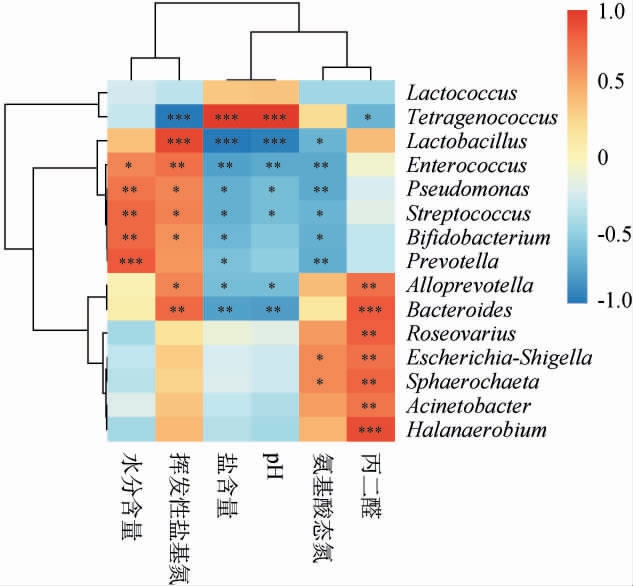
图8 不同产地虾酱优势细菌属与理化指标的相关性分析
Fig.8 Correlation analysis between dominant bacterial genera and physicochemical indexes of shrimp paste from different producing areas
“*”表示相关性显著(P<0.05);“**”表示相关性极显著(P<0.01);“***”表示相关性高度显著(P<0.001)。
由图8可知,四联球菌属与盐含量和pH值呈高度显著正相关(P<0.001),而与丙二醛和挥发性盐基氮分别呈显著、高度显著负相关(P<0.05、P<0.001)。这一结果与LI W Y等[28]的研究结果相反,其发现在低盐虾酱的酿造过程中四联球菌属与挥发性盐基氮和丙二醛呈正相关。这种差异可能源于不同产地虾酱的酿造工艺和发酵环境差异,从而导致其理化特征及微生物群落结构的显著不同。乳酸杆菌属、肠球菌属和芽孢杆菌属分别与挥发性盐基氮呈高度显著、极显著正相关(P<0.001、P<0.01),而假单胞菌属(Pseudomonas)、链球菌属(Streptococcus)、双歧杆菌属(Bifidobacterium)及拟普雷沃菌属(Alloprevotella)呈显著正相关(P<0.05),这些菌属与盐含量和pH值呈负相关。此外,乳酸杆菌属、链球菌属、双歧杆菌属(Bifidobacterium)及普雷沃菌属(Prevotella)与氨基酸态氮呈显著负相关(P<0.05),肠球菌属、假单胞菌属与氨基酸态氮呈极显著负相关(P<0.01);芽孢杆菌属、盐厌氧菌属(Halanaerobium)与丙二醛呈高度显著正相关(P<0.001),拟普雷沃菌属(Alloprevotella)、玫瑰变色菌属(Roseovarius)、埃希氏-志贺氏菌属、鳞球菌属、不动杆菌属(Acinetobacter)与丙二醛呈极显著正相关(P<0.01或P<0.001)。因此,不同细菌属与理化指标的互作关系共同影响不同产地虾酱的细菌群落组成及其品质特征。因此,不同细菌属与理化指标的互作关系共同影响不同产地虾酱的细菌群落组成及其品质特征。
3 结论
本研究以环渤海地区不同产地虾酱为研究对象,通过理化分析技术与高通量测序技术对其理化特性及细菌群落结构进行分析。结果表明,不同产地虾酱的水分含量、盐含量、pH值、氨基酸态氮、挥发性盐基氮及丙二醛含量等理化指标均存在明显差异。此外,不同产地虾酱的Chao1指数、Shannon指数和Simpson指数存在显著差异(P<0.05)。各产地虾酱共有ASV数量为121个,而DL、QHD、TS和YT地区的特有ASV数量分别为17个、10个、66个和58个。不同产地的虾酱中共检测出7个优势细菌门(相对丰度>1%),其主要优势细菌门为厚壁菌门、变形菌门和拟杆菌门;共检测出15个优势细菌属,其主要优势细菌属为四联球菌属、乳酸杆菌属、盐厌氧菌属和芽孢杆菌属。不同产地的虾酱细菌菌群相对丰度存在明显差异,可能由原料来源、气候环境及酿造工艺等因素导致。PCoA及HCA均能区分不同产地虾酱;LEfSe表明,不同产地虾酱之间显著差异细菌类群不同(P<0.05);RDA表明,盐含量是影响不同产地虾酱细菌群落的主要理化指标;相关性分析表明,优势细菌属与理化指标之间存在明显相关性。本研究系统比较了不同产地虾酱的品质特性、细菌群落多样性及结构组成差异,为筛选功能性微生物以优化虾酱发酵过程及改进发酵工艺提供数据支持与科学依据。
[1]WANG S Y, HU M Y, ZHAO L, et al.Changes in lipid profiles and volatile compounds of shrimp(Penaeus vannamei)submitted to different cooking methods[J].Int J Food Sci Technol,2022,57(7):4234-4244.
[2]HU M Y,ZHAO L,SUN H H,et al.Recent advances on shrimp paste:key flavor components and biochemical formation pathway, biogenic amine formation,microbial functions,and innovative process strategies[J].Trends Food Sci Tech,2024,152:104694.
[3]GAO R C,XUE J N,SHI T,et al.Effects of'bask in sunlight and dewed at night'on the formation of fermented flavor in shrimp paste after maturation[J].Food Chem,2024,452:139546.
[4]刘荣,任子钰,李银塔.虾酱发酵过程中原核微生物群落及内源环境因素的互作研究[J].食品与发酵工业,2025,51(7):135-142.
[5]GAO R C,LIU H J, LI Y, et al.Correlation between dominant bacterial community and non-volatile organic compounds during the fermentation of shrimp sauces[J].Food Science and Human Wellness,2023,12(1):233-241.
[6]LI X,WANG Y,SUN Y Q,et al.Nutritional evaluation,flavor characteristics and microbial community of shrimp paste made from different materials and variance analysis[J].Food Chem Adv,2023,2:100268.
[7]胡梓晴,刘晓艳,白卫东,等.高通量测序技术在白酒微生物多样性中的研究进展[J].中国酿造,2023,42(5):15-21.
[8]LI Y,LENG W J,XUE J N,et al.A multi-omics-based investigation into the flavor formation mechanisms during the fermentation of traditional Chinese shrimp paste[J].Food Res Int,2023,166:112585.
[9]段泽瑞,田梅兰,刘杨柳,等.食盐添加量对虾酱理化性质和细菌多样性的影响[J].食品科学,2024,45(15):85-93.
[10]班雨函,王利文,杨兵兵,等.商品化低盐虾酱发酵过程中的微生物群落演替和品质变化[J].食品科学,2022,43(24):182-191.
[11]王才政.江苏麻虾酱发酵途径解析及低盐快速发酵菌株的筛选[D].镇江:江苏大学,2023.
[12]安香玉,郝建雄,王成祥,等.基于高通量测序技术老面酵头微生物菌群多样性分析[J].中国酿造,2024,43(10):35-41.
[13]唐艳平,高秀兵,倪婕,等.白酒发酵副产物黄水原核微生物组成及理化特性研究[J].中国酿造,2024,43(3):72-78.
[14]BOLYEN E,RIDEOUT J R,DILLON M R,et al.Reproducible,interactive,scalable and extensible microbiome data science using QIIME 2[J].Nat Biotechnol,2019,37:852-857.
[15]FAN Y, YIN L A, XUE Y, et al.Analyzing the flavor compounds in Chinese traditional fermented shrimp pastes by HS-SPME-GC/MS and electronic nose[J].J Ocean U China J,2017,16:311-318.
[16]刘佳乐,郑兰萍,刘颖,等.不同产地虾酱代谢物组的比较[J].食品科学,2025,46(4):147-153.
[17]彭盼盼,赵成,赵竟翔.低盐虾酱发酵过程中理化性质及微生物多样性的变化[J].中国调味品,2024,49(11):75-78.
[18]班雨函,杨兵兵,马爱进,等.不同香辛料对低盐虾酱中微生物群落和理化性质的影响[J].食品科学,2023,44(8):143-151.
[19]孙筱.盐浓度对自制香肠理化因子及原核微生物群落多样性的影响[J].中国酿造,2021,40(9):145-149.
[20]MA Z Y,JIAO S,ZHENG K K,et al.Multiple spatial scales of bacterial and fungal structural and functional traits affect carbon mineralization[J].Mol Ecol,2024,33(3):e17235.
[21]SANG X,MA X X,ZHANG Y N,et al.Assessment of the distribution and safety of Tetragenococcus muriaticus for potential application in the preparation of Chinese grasshopper sub shrimp paste[J].Front Microbiol,2021,12:628838.
[22]KIM K H,LEE S H,CHUN B H,et al.Tetragenococcus halophilus MJ4 as a starter culture for repressing biogenic amine(cadaverine)formation during saeu-jeot(salted shrimp)fermentation[J].Food Microbiol,2019,82:465-473.
[23]HELMI H,ASTUTI D I,PUTRI S P,et al.Dynamic changes in the bacterial community and metabolic profile during fermentation of low-salt shrimp paste(terasi)[J].Metabolites,2022,12(2):118.
[24]BELLEGGIA L,OSIMANI A.Fermented fish and fermented fish-based products,an ever-growing source of microbial diversity:A literature review[J].Food Res Int,2023,172:113112.
[25]檀茜倩,王丹,麻冰玉,等.芽孢杆菌发酵对虾酱品质和菌群的影响[J].食品工业科技,2024,45(21):103-110.
[26]HUA X H, YAN T T, LIU S Y, et al.Insights into the correlation between bacterial community succession and dynamics of biogenic amines during three types of sufu fermentation[J].Food Control, 2025, 168:110870.
[27]YANG B B,LIU Y L,SANG Y X,et al.Bacterial diversity and physicochemical properties of low-salt shrimp paste fermented at different temperatures[J].LWT-Food Sci Technol,2023,187:115277.
[28]LI W Y, LU H Q, HE Z H, et al.Quality characteristics and bacterial community of a Chinese salt-fermented shrimp paste[J].LWT-Food Sci Technol,2021,136:110358.