Analysis of esterification properties of esterase from Thermoascus aurantiacus QH-1 by molecular docking
橙色嗜热子囊菌(Thermoascus aurantiacus)是一类嗜热真菌,一般出现在温度较高的自然环境中,如堆肥、温泉等,其能通过分泌纤维素酶、木聚糖酶和淀粉酶水解纤维素、木聚糖和淀粉等大分子物质获得营养物质,在医药、食品领域备受研究者们的广泛关注[1-4]。酒曲一般以大麦、小麦和豌豆为原料,生料制曲,开放培养,在其制备过程中有大量的微生物生长代谢产生热量,导致曲坯的温度较高[5-6]。采用高通量技术分析酒曲微生物多样性时,常发现有大量的嗜热子囊菌(Thermoascus)存在,如陕西白水地区的10个清香大曲样品中均检测到嗜热子囊菌,比例最高达到25.57%[7-9];宝丰酒业的红心曲中Thermoascus占比最大,为53.52%[10];鹿头黄酒酿造用高温黑曲中Thermoascus的相对丰度为22.21%[11];徐坊中高温大曲中Thermoascus的相对丰度为33.27%[12]。红心曲是清香白酒酿造的一个重要曲种,而且红心曲酿造的酒醇甜且水果香突出,其香味物质主要来自红心曲红心部位[13]。酯类物质是白酒的主要香气成分[14],其主要由醇类与有机酸在酯酶作用下合成[15]。近年来,T. aurantiacus被认为是红心曲红心部位的关键微生物[16],但红心曲来源的T.aurantiacus是否具有酯化性能并合成酯类物质尚不清楚。
分子对接技术是在计算机软件辅助下,根据蛋白质分子的空间结构来预测生物大分子与小分子配体之间的相互作用、生物大分子的活性位点与小分子的亲和能力[17],近年来被广泛应用于探讨酶与底物之间的亲和力及酶的作用机制,华承伟等[18]利用分子对接技术研究发现,拟青霉(Paecilomyces varioti)的β-1,3(4)-葡聚糖酶对β-1,3(4)-葡聚糖的结合力高于β-1,3-葡聚糖;张杰等[19]利用分子对接技术对高温大曲来源贝莱斯芽胞杆菌(Bacillus velezensis)Pb-1的羧酸酯酶催化位点及底物进行研究,发现Phe21A是其羧酸酯酶具有催化活性的主要氨基酸,并预测该酶能水解三丁酸甘油酯为丁酸和甘油;陈胤熹等[20]利用分子对接研究家蚕碳酸酐酶与乙酸对硝基苯酯对接时发现,范德华力在酶与底物结合中占主导地位,家蚕碳酸酐酶和底物相互作用的区域为138L~150V和209L~217C;潘虹等[21]通过分子对接得到β-葡萄糖苷酶转化人参皂苷Rb1中参与底物识别及结合的关键区域和位点。但目前未见对T.aurantiacus酯酶催化性能的分析。
本研究以清香白酒酿造用的红心曲曲心部位分离得到的6株T.aurantiacus为研究对象,通过测定酯酶活力从中筛选高产酯酶的菌株,在基因组测序的基础上,利用分子对接技术分析其底物特异性及活性位点,并通过固态培养物与不同脂肪酸的酯化反应进行验证,明确T.aurantiacus的酯化特性及酯酶的分子基础,进一步为T.aurantiacus及红心曲在清香白酒酿造中的作用提供理论依据。
1.1.1 原料与菌株
T.aurantiacus菌株QH-1、QH-3、QH-4、FH-5、FH-6、FH-7:分离自清香白酒酿造用的红心曲曲心部位,保存于本实验室;大麦:河南信阳;豌豆:山东菏泽。
1.1.2 试剂
三丁酸甘油酯、聚乙烯醇、乙酸、乳酸、正丁酸、正己酸、庚酸、辛酸、癸酸、月桂酸、肉豆蔻酸(均为分析纯):上海麦克林生化科技有限公司;磷酸二氢钾(分析纯):天津市北辰方正试剂有限公司;磷酸氢二钠(分析纯):天津市永大化学试剂有限公司;蛋白胨(生物试剂):北京奥博星生物技术有限责任公司;MgSO4(分析纯):天津市科密欧化学试剂开发中心;维生素B1(vitamin B1,VB1)(分析纯):广州硕谱生物科技有限公司;NaOH(分析纯):洛阳市化学试剂厂;TruSeqTM脱氧核糖核酸(deoxyribonucleic acid,DNA)Sample Prep Kits:美国Illumina公司;其他试剂均为国产分析纯或生化试剂。
1.1.3 培养基
马铃薯葡萄糖培养基:青岛高科技工业园海博生物技术有限公司。
种子培养基[22]:马铃薯葡萄糖培养基26 g,蛋白胨18 g,KH2PO4 3 g,MgSO4 15 g,VB1 0.05 g,蒸馏水1 000 mL,121 ℃高压蒸汽灭菌20 min。
发酵培养基[22]:大麦与豌豆粉碎过筛按照质量比65∶35混匀,粗粉与细粉的比例为6∶4(粗粉为过30目筛,细粉为过60目筛),按原料质量添加38%的蒸馏水,搅拌均匀后分装,121 ℃高压蒸汽灭菌20 min。
GI80DS高压灭菌锅:致微(厦门)仪器有限公司;ZHJHC1112B双人超净工作台、ZXMP-R1230恒温恒湿培养箱:上海智城分析仪器制造有限公司;D37520低速冷冻离心机:赛默飞世尔科技(中国)有限公司。
1.3.1 高产酯酶Thermoascus aurantiacus菌株的筛选
将6株T.aurantiacus菌株分别接种于种子培养基,于45 ℃、180 r/min条件下活化培养3 d后,再按10%的接种量将活化液接种于发酵培养基中,45 ℃条件下静置培养10 d后得到固体培养物,测定酯酶活力,筛选高产酯酶的T.aurantiacus菌株。
1.3.2 酯酶活力的测定
参照文献[23],将含酶的固体培养物用少量pH为7.5的0.025 mol/L磷酸盐缓冲溶液(phosphate buffer saline,PBS)溶解,并用玻璃棒研磨后,4 ℃、4 000×g条件下离心15 min,收集上清液,即粗酶液。取4 mL乳化液(取90 mL聚乙烯醇溶液加入10 mL三丁酸甘油酯,充分摇匀)和5 mL PBS(0.025 mol/L,pH 7.5)于锥形瓶中,放入40 ℃水浴锅中,预热5~10 min,加入1 mL粗酶液,反应15 min,加入15 mL体积分数为95%的乙醇,滴2~3滴酚酞指试剂,用0.05 mol/L NaOH溶液滴定,记消耗的NaOH溶液体积V1,不加酶液的为对照V2,计算酯酶活力,其计算公式如下:
酯酶活力单位定义:1 g含酶培养物于40 ℃、pH 7.5的条件下,水解三丁酸甘油酯每分钟生成1 μmol的脂肪酸定义为1个酯酶活力单位(U/g)。
1.3.3 生物信息学方法
(1)基因组测序及酯酶基因预测
采用TruSeqTM DNA Sample Prep Kit提取筛选菌株的基因组DNA,委托南京派森诺公司进行全基因组测序,采用全基因组鸟枪法(whole genome shotgun,WGS)策略构建不同插入片段的文库,利用第二代测序技术绘制基因组框架图,采用Swiss-Prot数据库进行基因注释,筛选酯酶基因。
(2)理化性质及生物学特征的预测
将预测到的酯酶基因翻译后得到编码的氨基酸序列,使用ExPASy ProtParam tool(https://web.expasy.org/protparam/)进行等电点(isoelectric point,pI)、分子质量、亲水性等理化性质的预测。利用TMHMM Server 2.0(http://www.cbsdtu.dk/services/TMHMM/)预测酯酶氨基酸序列跨膜区域;运用SingalP 4.1Server(SignalP 4.1-DTU Health Tech-Bioinformatic Services)对酯酶氨基酸序列的信号肽进行预测。
(3)同源建模
利用SWISS-MODEL 同源建模(https://swissmodel.expasy.org/)进行蛋白质模型的预测,将通过模型质量评估的蛋白质模型以pdb格式保存。根据PubChem分子数据库提供的信息,使用ChemDraw Professional 17.0(https://www.chemdraw.com.cn/)绘制底物结构,并将底物的化学结构转化为pdb格式的三维结构。
(4)分子对接
使用Pymol 2.6.0对保存的蛋白质模型进行处理,小分子利用ChemDraw 17.0软件进行能量最小化。利用软件Auto dock4.2.6对蛋白质及底物进行分子对接,预测酯酶蛋白与不同脂肪酸结合的结合能[24]。采用蛋白质-配体相互作用分析器(protein-ligand interaction profiler,PLIP)分析酯酶与底物结合的作用力类型及关键氨基酸[25]。
1.3.4 验证试验
将筛选菌株按照1.3.1固态培养后,参照轻工行业标准QB/T 4257—2011《酿酒大曲通用分析方法》[26],测定对不同底物(乙酸、乳酸、丁酸、己酸、辛酸、癸酸、月桂酸、肉豆蔻酸)的酯化力,分析其酯酶的底物特异性。酯化力单位定义:指每50 g曲在35 ℃,经过7 d催化乙酸(或其他酸)和乙醇合成乙酸乙酯(或其他乙酯)的毫克数为一个单位,符号为U,以mg/50 g·7 d表示。
1.3.5 数据处理与统计分析每个试验均重复3次,结果使用SPSS 25.0进行统计学分析,采用Origin 2018作图。
6株T.aurantiacus的酯酶活力见图1。由图1可知,6株T.aurantiacus菌株的酯酶活力为9.40~16.10 U/g,其中,菌株QH-1的酯酶活力最高,为16.10 U/g,菌株FH-5的酯酶活力最低。故选择T.aurantiacus QH-1为高产酯酶的菌株。
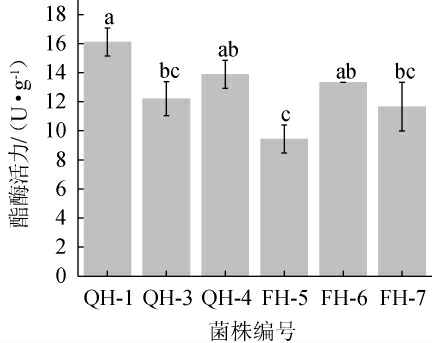
图1 6株橙色嗜热子囊菌菌株的酯酶活力测定结果
Fig.1 Determination results of esterase activities of 6 strains of Thermoascus aurantiacus
不同小写字母表示差异显著(P<0.05)。下同。
T.aurantiacus QH-1基因组框架图已完成,其在美国国家生物技术信息中心(National Center of Biotechnology Information,NCBI)BioProject数据库中的登录号为PRJNA1018823。T.aurantiacus QH-1基因组框架图中包含4 577 565 600 bp碱基,鸟嘌呤(guanine,G)胞嘧啶(cytosine,C)含量为50.52%,共7 216个基因。从T.aurantiacus QH-1基因组中共筛选到12个酯酶基因,对其编码的酯酶分子质量、氨基酸数、pI等基本理化性质、跨膜区域及信号肽进行预测,结果见表1。由表1可知,12个酯酶分子质量相差较大,最大的酯酶含有1 035个氨基酸,分子质量为112 230.48 u;最小的酯酶只含有92个氨基酸,分子质量为9 731.07 u;其余酯酶的氨基酸数在200~600个。12个酯酶中,pI<7的有7个,属于酸性蛋白;pI>7的有5个,属于碱性蛋白。酯酶蛋白稳定性预测中,不稳定系数<40的稳定蛋白有3个,即酯酶scaffold 149.t3、scaffold 198.t7和scaffold 360.t9;不稳定系数接近40的也有3个,即酯酶scaffold 247.t1、scaffold 64.t20和scaffold 7.t5。根据疏水性平均值分析,12个酯酶的疏水性平均值为-0.488~-0.014,均为负值,表明这些酯酶都有一定的亲水性。信号肽与跨膜区预测中,酯酶scaffold 149.t3、scaffold 15.t37和scaffold 360.t9蛋白有信号肽并且没有跨膜螺旋区,推测为分泌蛋白;酯酶scaffold 220.t2和scaffold 12.t30蛋白没有信号肽,只有跨膜螺旋区,推测蛋白质位置在膜上;酯酶scaffold 156.t16、scaffold 247.t1、scaffold142.t18、scaffold 64.t20、scaffold 7.t5和scaffold 323.t6蛋白既没有信号肽,也没有跨膜区,推测为胞内蛋白。
表1 橙色嗜热子囊菌QH-1所产酯酶的理化性质、跨膜区域及信号肽预测结果
Table 1 Results of physicochemical properties, transmembrane region and signal peptide prediction of the esterases produced by Thermoascus aurantiacus QH-1
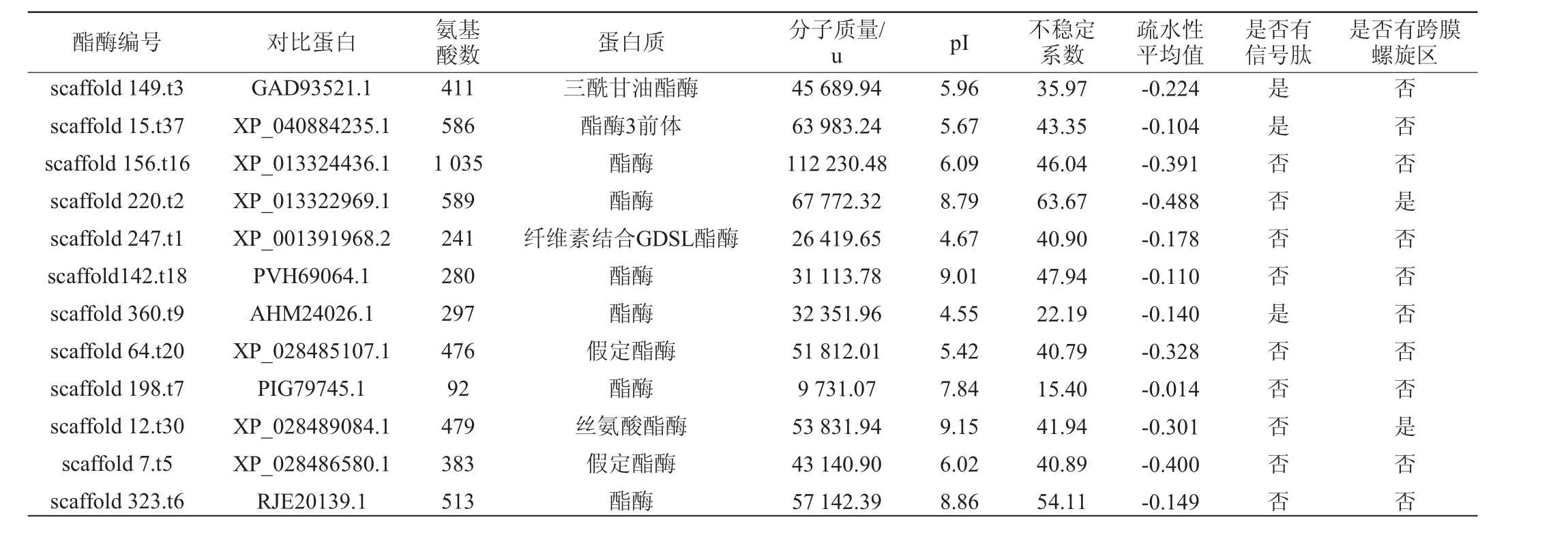
酯酶编号 对比蛋白 氨基酸数 蛋白质 分子质量/u pI 不稳定系数scaffold 149.t3 scaffold 15.t37 scaffold 156.t16 scaffold 220.t2 scaffold 247.t1 scaffold142.t18 scaffold 360.t9 scaffold 64.t20 scaffold 198.t7 scaffold 12.t30 scaffold 7.t5 scaffold 323.t6 GAD93521.1 XP_040884235.1 XP_013324436.1 XP_013322969.1 XP_001391968.2 PVH69064.1 AHM24026.1 XP_028485107.1 PIG79745.1 XP_028489084.1 XP_028486580.1 RJE20139.1 411 586 1 035 589 241 280 297 476 92 479 383 513三酰甘油酯酶酯酶3前体酯酶酯酶纤维素结合GDSL酯酶酯酶酯酶假定酯酶酯酶丝氨酸酯酶假定酯酶酯酶45 689.94 63 983.24 112 230.48 67 772.32 26 419.65 31 113.78 32 351.96 51 812.01 9 731.07 53 831.94 43 140.90 57 142.39 5.96 5.67 6.09 8.79 4.67 9.01 4.55 5.42 7.84 9.15 6.02 8.86 35.97 43.35 46.04 63.67 40.90 47.94 22.19 40.79 15.40 41.94 40.89 54.11疏水性平均值-0.224-0.104-0.391-0.488-0.178-0.110-0.140-0.328-0.014-0.301-0.400-0.149是否有信号肽是是否否否否是否否否否否是否有跨膜螺旋区否否否是否否否否否是否否
利用SWISS-MODEL对稳定系数小于和接近40的6个酯酶蛋白进行同源建模。首先利用这6个酯酶的氨基酸序列在ExPDB蛋白数据库搜索,筛选蛋白序列一致性>30%的模板用于构建模型[27]。并采用拉式构象图(Ramachandran Plot)和全球模型质量评估(global model quality estimation,GMQE)对模型的质量进行评价。6个酯酶的建模及评价结果见表2及图2。
表2 橙色嗜热子囊菌QH-1所产酯酶模型的构建及质量评价
Table 2 Modeling and quality evaluation of esterases produced by Thermoascus aurantiacus QH-1
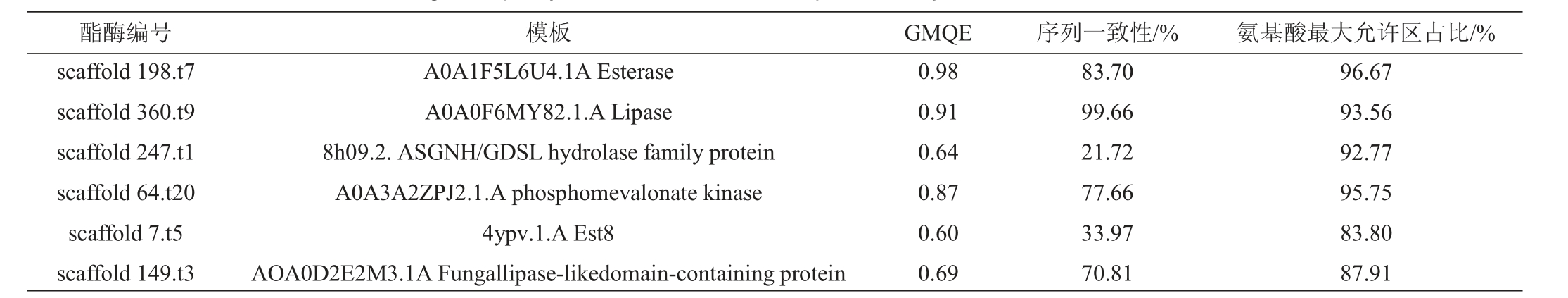
酯酶编号 模板 GMQE 序列一致性/% 氨基酸最大允许区占比/%scaffold 198.t7 scaffold 360.t9 scaffold 247.t1 scaffold 64.t20 scaffold 7.t5 scaffold 149.t3 A0A1F5L6U4.1A Esterase A0A0F6MY82.1.A Lipase 8h09.2.ASGNH/GDSL hydrolase family protein A0A3A2ZPJ2.1.A phosphomevalonate kinase 4ypv.1.A Est8 AOA0D2E2M3.1A Fungallipase-likedomain-containing protein 0.98 0.91 0.64 0.87 0.60 0.69 83.70 99.66 21.72 77.66 33.97 70.81 96.67 93.56 92.77 95.75 83.80 87.91
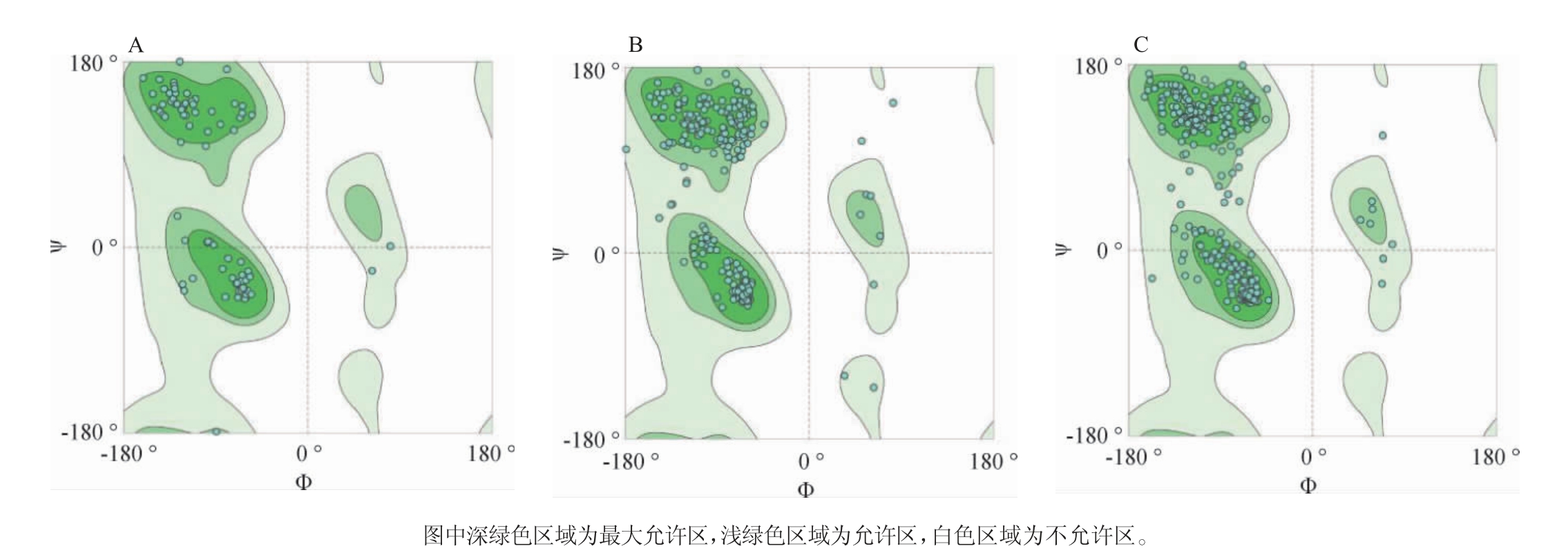
图2 橙色嗜热子囊菌QH-1所产酯酶scaffold198.t7(A)、scaffold 360.t9(B)及scaffold 64.t20(C)的拉式构象图
Fig.2 Ramachandran plot of esterases scaffold198.t7 (A), scaffold 360.t9 (B) and scaffold 64.t20 (C) produced by Thermoascus aurantiacus QH-1
由表2可知,6个酯酶的模板分别是A0A1-F5L6U4.1A、A0A0F6MY82.1.A、8h09.2.ASGNA、A0A3A2ZPJ2.1、4ypv.1.A、AOA0D2E2M3.1A。与模板序列一致性最高的是酯酶scaffold360.t9,高达99.66%;其次是酯酶scaffold 198.t7,一致性为83.70%,而酯酶scaffold 247.t1与模板序列的一致性最低,仅为21.72%。使用Ramachandran Plot对蛋白质模型的合理性进行判断时,主要观察氨基酸分布在哪个区域,允许区和最大允许区>90%的模型一般被认为符合立体化学的规则[28]。采用GMQE评估时,可信度范围为0~1,值越大表明质量越好[29]。由表2及图2可知,酯酶scaffold 198.t7、scaffold360.t9和scaffold 64.t20的模型GMQE接近1,分别为0.98、0.91、0.87,并且落在允许区及最大允许区的氨基酸>90%。综合序列一致性、拉式构象图及GMQE对酯酶模型的评估,可信度较高的蛋白模型依次是酯酶scaffold 198.t7、scaffold 360.t9、scaffold 64.t20。
中国白酒中主要的酯类物质是乙酯类,除乙酸乙酯还可以由酰基转移酶作用合成,其他乙酯物质主要由乙醇与相应的脂肪酸酯化而成[30]。利用Auto dock 4.2.6软件对T. aurantiacus QH-1的3个酯酶(scaffold 198.t7、scaffold 360.t9、scaffold 64.t20)与脂肪酸进行半柔性对接,分子对接结果见表3。由表3可知,3个酯酶与脂肪酸对接的结合能均<0,代表复合物能自发结合。Scaffold 198.t7与脂肪酸的对接结合能,除了乳酸以外,随着有机酸碳链的延长明显降低,最低为-5.25kcal/mol,表现出较强的亲和力;scaffold360.t9与脂肪酸的对接结合能分布在-2.98~-4.11 kcal/mol,与有机酸碳链长短没有明显规律;scaffold 64.t20与脂肪酸对接结合能范围是-2.80~-5.18 kcal/mol,结合能最低的有机酸是辛酸。结果表明,3个酯酶均能与不同脂肪酸起作用生成相应的酯,表明该酯酶的底物特异性较低,这一特性与红曲霉(Monascus)的酯酶较为相似[31-32]。
表3 橙色嗜热子囊菌QH-1的3个酯酶与脂肪酸的分子对接结合能
Table 3 Molecular docking binding energy of 3 esterases from Thermoascus aurantiacus QH-1 and fatty acids
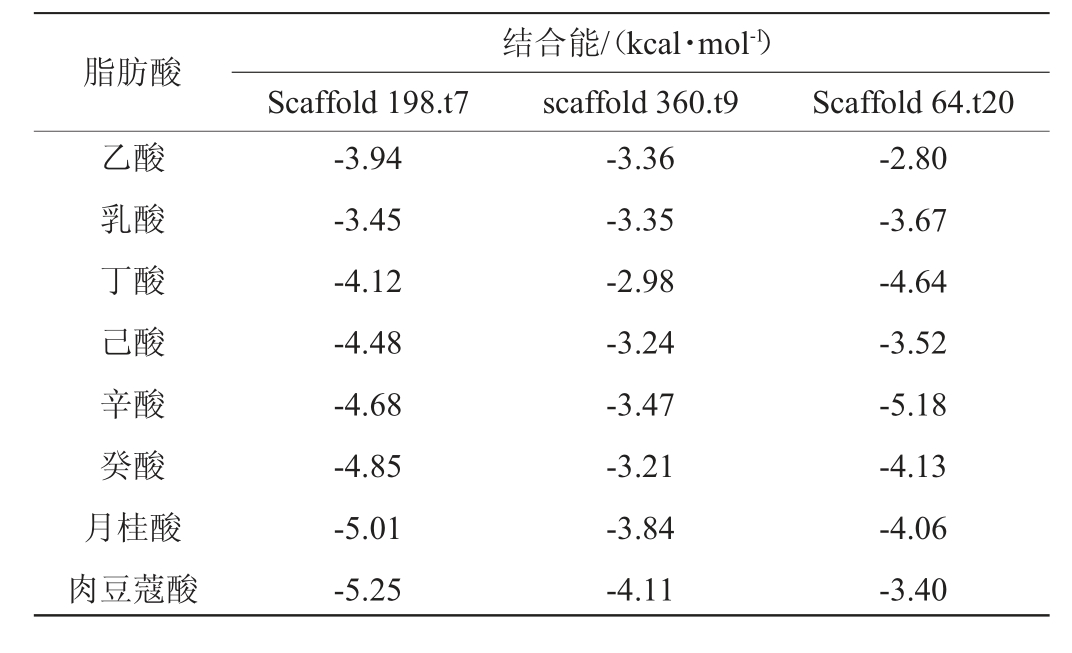
脂肪酸乙酸乳酸丁酸己酸辛酸癸酸月桂酸肉豆蔻酸结合能/(kcal·mol-1)Scaffold 198.t7 scaffold 360.t9 Scaffold 64.t20-3.94-3.45-4.12-4.48-4.68-4.85-5.01-5.25-3.36-3.35-2.98-3.24-3.47-3.21-3.84-4.11-2.80-3.67-4.64-3.52-5.18-4.13-4.06-3.40
为了了解T.aurantiacus QH-1所产酯酶的底物特异性,将T.aurantiacus QH-1的固体培养物与不同碳链的脂肪酸酯化后,检测其酯化力,结果见图3。由图3可知,T.aurantiacus QH-1固体培养物对乙酸、乳酸、丁酸、己酸、辛酸、癸酸、月桂酸、肉豆蔻酸均能酯化,但不同脂肪酸的利用率差别较大,对底物辛酸的利用率最高,酯化力达到152 mg/50 g·7 d,对癸酸的酯化力也较高,为148 mg/50 g·7 d,显著高于其他脂肪酸(P<0.05),对其余脂肪酸的酯化力依次为己酸、丁酸和乙酸;酯化力最低的是月桂酸和肉豆蔻酸。结合3个酯酶蛋白质的分子对接结合能变化趋势,可初步判断酯酶scaffold 64.t20应该起主要酯化作用。
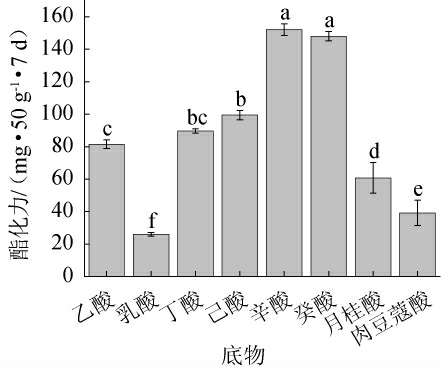
图3 橙色嗜热子囊菌QH-1所产酯酶的底物特异性
Fig.3 Substrate specificity of esterases from Thermoascus aurantiacus QH-1
使用PILP分析酯酶scaffold 64.t20与不同脂肪酸的结合部位及作用力类型,结果见表4。
表4 酯酶scaffold 64.t20与底物结合作用力预测
Table 4 Prediction of binding force between esterase scaffold64.t20 and substrate
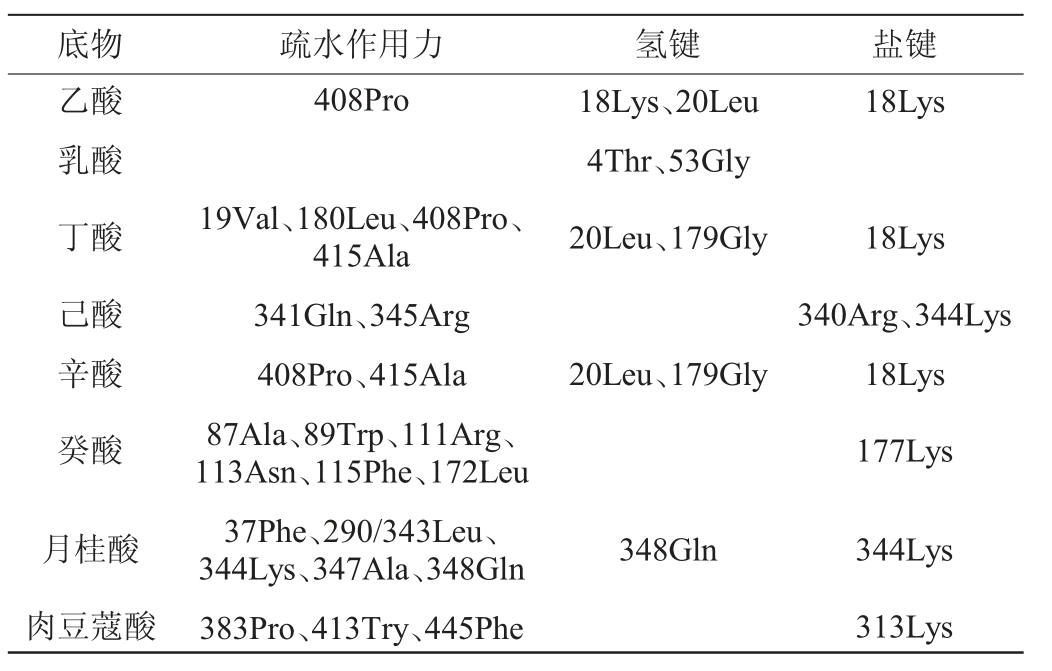
底物 疏水作用力 氢键 盐键乙酸乳酸丁酸己酸辛酸癸酸408Pro 18Lys、20Leu 4Thr、53Gly 20Leu、179Gly 18Lys 19Val、180Leu、408Pro、415Ala 341Gln、345Arg 408Pro、415Ala 87Ala、89Trp、111Arg、113Asn、115Phe、172Leu 37Phe、290/343Leu、344Lys、347Ala、348Gln 383Pro、413Try、445Phe 20Leu、179Gly 18Lys 340Arg、344Lys 18Lys 177Lys月桂酸肉豆蔻酸348Gln 344Lys 313Lys
由表4可知,酯酶scaffold 64.t20与脂肪酸结合的作用力主要有疏水作用力、氢键与盐键三种类型。酯酶scaffold 64.t20与不同底物涉及的作用力不完全一样,如与乳酸结合时只有氢键,与己酸、癸酸和肉豆蔻酸结合时有疏水作用力和盐键,与乙酸、丁酸、辛酸和月桂酸结合时有3种类型作用力。酯酶Scaffold 64.t20与脂肪酸形成疏水作用力时活跃氨基酸有408脯氨酸(proline,Pro)和415丙氨酸(alanine,Ala);与脂肪酸形成氢链时活跃的氨基酸有20亮氨酸(leucine,Leu)和179甘氨酸(glycine,Gly);与脂肪酸形成盐键时主要是赖氨酸(lysine,Lys),只是位点不一样,有第18位、344位、177位和313位的Lys。另外87位Ala、89位色氨酸(tryptophan,Trp)、111位精氨酸(arginine,Arg)、113位天冬氨酸(asparagine,Asn)、115位苯丙氨酸(phenylalanine,Phe)、172位Leu与癸酸主要形成疏水作用力。同时还发现酯酶scaffold 64.t20与丁酸和辛酸结合时,除了丁酸与19缬氨酸(valine,Val)和180Leu形成疏水作用力,其他的作用力及位点完全一致。
T.aurantiacus QH-1所产酯酶底物特异性验证实验中,辛酸为底物时酯化力最高;分子对接预测中,酯酶scaffold 64.t20与辛酸结合能最低,表明两者最容易结合。因此,预测T.aurantiacus QH-1所产酯酶scaffold 64.t20与辛酸对接的模式,结果见图4。由图4可知,酯酶scaffold 64.t20的氨基酸20Leu和179Gly以氢键与辛酸相互结合,氨基酸408Pro和415Ala与辛酸形成疏水相互作用力,氨基酸18Lys与辛酸以盐桥与辛酸相互结合。
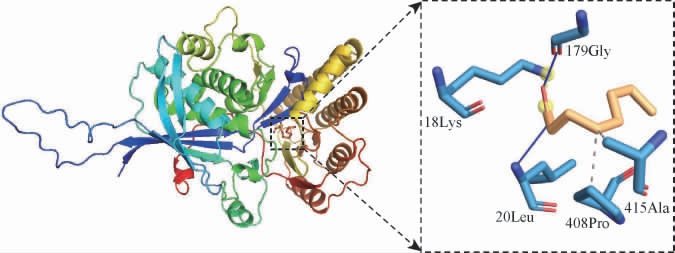
图4 橙色嗜热子囊菌QH-1所产酯酶scaffold 64.t20与辛酸的结合位点图
Fig.4 Binding sites of the esterase scaffold64.t20 produced by Thermoascus aurantiacus QH-1 and octanoic acid
蓝色实线代表氢键结合,灰色虚线代表疏水作用力,黄色虚线代表盐键。
本研究旨在了解Thermoascus aurantiacus的酯化性能,从分离自清香白酒酿造用的红心曲曲心部位的6株T.aurantiacus菌株中筛选得一株酯酶活力较高的菌株QH-1,酯酶活力为16.10 U/g。根据基因组注释筛选出12个酯酶基因,根据酯酶分子稳定性及同源建模评价,选出3个酯酶分子scaffold 198.t7、scaffold 360.t9和scaffold 64.t20。利用Auto dock 4.2.6软件与脂肪酸(乙酸、乳酸、丁酸、己酸、辛酸、葵酸、月桂酸和肉豆蔻酸)进行半柔性分子对接,预测到分子对接的自由能均为负值,表明T.aurantiacus QH-1能将乙醇与脂肪酸合成相应的乙酯,也说明T.aurantiacus QH-1所产酯酶的底物专一性低。其中,酯酶scaffold 64.t20分子对接的辛酸自由能最低,表明亲和力最好。在T.aurantiacus QH-1固态培养物与不同脂肪酸酯化反应中,检测到所有脂肪酸相应的乙酯,当底物为辛酸时,酯化力最高(152mg/50g·7d),与分子对接结果吻合,推测scaffold 64.t20有可能表达量高,是T.aurantiacus QH-1的主要酯酶。进一步分析酯酶scaffold 64.t20与底物结合的作用力,发现其酯化机制主是通过疏水作用力、氢键和盐键三种类型,疏水作用力整体随碳链延长涉及的氨基酸数量增加,氢键主要涉及两个氨基酸,与丁酸、辛酸结合的位点完全一样,盐键主要是赖氨酸,有18位和344位等。本研究结合模型预测和实验分析,表明T.aurantiacus QH-1基因组中的酯酶基因能够顺利表达合成酯类物质,证实了T.aurantiacus在酿造食品中一项重要功能。关于其酯酶的合成条件及酶学性质等还需要进一步研究,才能有效应用于酿造食品生产中。
[1]赵春青,李多川.嗜热真菌的分类研究概况[J].菌物学报,2007(4):601-606.
[2]李晓丽,涂涛,姚斌,等.嗜热子囊菌JCM12803来源的双功能木聚糖/纤维素酶[J].生物工程学报,2018,34(12):1996-2006.
[3]庞宗文,郭法谋,徐勇,等.高产木聚糖酶嗜热子囊菌固体发酵条件与酶学性质[J].食品工业科技,2012,33(3):170-173,236.
[4]杨雄振,郭玉杰,涂涛,等.嗜热子囊菌Thermoascus crustaceus JCM12803来源的低温α-淀粉酶功能验证及其适冷机制分析[J].微生物学报,2018,58(12):2161-2173.
[5]邓皖玉,王会,赵荣寿,等.高温大曲中产酱味霉菌的分离及研究[J].酿酒科技,2015(3):9-11.
[6]卢中明,张祥,叶光斌.不同生产月份中高温成品大曲理化指标、挥发性风味物质分析[J].食品与发酵工业,2024,50(19):315-320.
[7]吴树坤,谢军,卫春会,等.四川不同地区浓香型大曲微生物群落结构比较[J].食品科学,2019,40(14):144-152.
[8]杨少勇,黎婷玉,蔡文超,等.襄阳地区高温大曲和中高温大曲真菌多样性解析[J].中国酿造,2021,40(5):76-80.
[9]陈佩,翟彩宁,刘莹,等.白水地区清香型大曲理化指标及真菌多样性分析[J].食品与发酵工业,2024,50(10):197-204.
[10]赵志军,郜燕燕,刘延波,等.采用高通量测序技术分析宝丰清香型白酒大曲的真菌多样性[J].现代牧业,2022,6(2):45-52.
[11]柯悦,邓子文,姜在明,等.鹿头黄酒酿造用高温大曲理化指标和真菌类群的相关性[J].中国酿造,2023,42(9):91-95.
[12]侯强川,王文航,徐媛媛,等.徐坊中高温大曲发酵过程中理化品质、风味及真菌群落结构变化规律研究[J].食品工业科技,2024,45(15):133-141.
[13]李佐华.红心曲的生产技术[J].酿酒科技,1985(3):12-15.
[14]肖冬光.白酒酿造过程中酯类物质形成机理探讨[J].酿酒科技,2022(9):17-24.
[15]宋川,刘双平,敖宗华,等.白酒酿造微生物酯化酶的研究进展[J].酿酒,2024,51(2):34-40.
[16]TANG Y,ZHAO W,CUI M,et al.Identification and functional prediction of the key fungus of Hongxinqu for Light-flavor Baijiu brewing[J].LWT-Food Sci Technol,2024,195:194115605.
[17]BROOIJMANS N,KUNTZ I.Molecular recognition and docking algorithms[J].Annu Rev Bioph Biom,2003,32(1):335-373.
[18]华承伟,师玉忠,于江傲,等.拟青霉β-1,3(4)-葡聚糖酶同源模建、分子对接与分子动力学模拟[J].计算机与应用化学,2013,30(4):370-374.
[19]张杰,侯珺淇,代振玉,等.一株产羧酸酯酶菌的鉴定及酯酶特征的研究[J].食品工业科技,2021,42(19):126-134.
[20]陈胤熹,黎菁菁,罗佳伟,等.利用同源建模、分子模拟和分子对接技术分析家蚕中碳酸酐酶的结构和催化机制[J].现代食品科技,2022,38(1):159-164.
[21]潘虹,姚向钰,洪一楠,等.β-葡萄糖苷酶J384W位点突变的生物信息学及分子对接[J].西安工程大学学报,2024,38(3):61-67.
[22]唐艺婧,来欢欢,赵微,等.红心曲来源Thermoascus aurantiacus QH-1的生长代谢特征[J].食品工业科技,2023,44(22):117-124.
[23]张秀红,刘秋林,孔健,等.一株产酯化酶细菌的筛选[J].酿酒科技,2012(6):56-58.
[24]PAGADALA S,SYED K,TUSZYNSKI J.Software for molecular docking:A review[J].Biophys Rev,2017,9(2):91-102.
[25]SALENTIN S,SCHREIBER S,HAUPT J,et al.PLIP:Fully automated protein-ligand interaction profiler[J].Nucleic Acids Res,2015,43(W1):W443-W447.
[26]中华人民共和国工业和信息化部.QB/T 4257—2011 酿酒大曲通用分析方法[S].北京:中国标准出版社,2011.
[27]RAVIKUMAR A,RAMAKRISHNA C,SRINIVASAN N,et al.Stereochemical assessment of outliers in protein structures using bond geometryspecific Ramachandran steric-maps[J].Structure, 2019, 27(12): 1875-1884.
[28]BERTONI M,KIEFER F,BIASINI M,et al.Modeling protein quaternary structure of homo- and hetero-oligomers beyond binary interactions by homology[J].Sci Rep,2017,7(1):10480.
[29]MARCO B,STEFAN B,ANDREW W,et al.SWISS-MODEL:Modelling protein tertiary and quaternary structure using evolutionary information[J].Nucleic Acids Res,2014,42(Web Server issue):W252-258.
[30]丁力杰,唐云,赵亚雄,等.白酒中的重要酯类物质及主要来源研究现状[J].酿酒科技,2024(3):91-100.