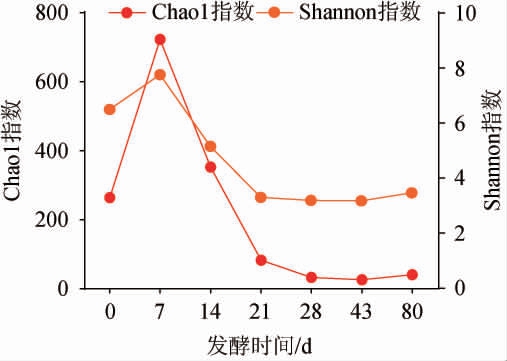
图1 酒醅发酵过程中细菌菌群Chao1指数和Shannon指数的变化
Fig.1 Changes in Chao1 index and Shannon index of bacterial communities of fermented grains during fermentation process
Changes of bacterial community and its metabolic characteristic in fermentation process of strong-flavor Baijiu fermented grains based on PacBio sequencing
浓香型白酒依靠传统固态发酵工艺形成其独特的风味[1-3]。酒醅是其中重要的发酵载体,各种微生物在酒醅中交互作用产生白酒香味物质,影响酒体质量[4-6]。因此,解析浓香型白酒酿造过程的微生物群落及其功能变化对深入了解白酒发酵机理和指导酿造工艺的优化改良具有重要意义[7]。
近年来,16S rRNA基因高通量测序技术被广泛应用于酿酒微生物研究,极大地推动了人们对白酒酿造微生态的认识水平[8-14]。然而以往研究主要针对16S rRNA基因的V3~V4高变区进行测序[8-14],由于V3~V4高变区基因序列较短(约400 bp),对微生物种水平群落结构识别的精确程度尚有不足[15-16]。相对而言,16S rRNA全长测序技术针对V1~V9高变区进行测序,测序长度达1 500 bp,可实现对微生物种水平的准确鉴定[17]。目前,基于16S rRNA基因的第三代全长测序技术PacBio已被运用到土壤[18]、肠道[19]等样品的微生物群落研究,取得了良好的应用效果。
为进一步揭示浓香型白酒酿造微生态系统,本研究采用第三代全长测序技术PacBio技术解析酒醅微生物在酿造过程中的群落及其代谢特性变化,并对细菌菌群与挥发性风味物质间的相关性进行分析,以期为相关研究的深入提供依据。
1.1.1 酒醅样品
酒醅样品采集自四川省泸州市某浓香型白酒酒厂,采样时间点为0、7 d、14 d、21 d、28 d、43 d、80 d。每个时间点采集的样品来自于3个窖池,具体采样方法参见文献[14]。
1.1.2 试剂
2×Hieff Robust聚合酶链式反应(polymerase chain reaction,PCR)MasterMix、Hieff NGSTM脱氧核糖核酸(deoxyribonucleic acid,DNA)Selection Beads:宝生物工程(大连)有限公司;Fast DNATM SPIN Kit for Soil:美国MP Biomedicals公司。其他试剂均为国产分析纯。
XPR205D5/AC电子分析天平:瑞士Mettler Toledo公司;PB10 pH仪:德国Sartorius公司;MiniAmp PCR仪:美国ABI公司;PacBio Sequel II测序平台:上海派森诺生物科技股份有限公司。
1.3.1 酒醅中细菌菌群的高通量测序分析
采用Fast DNATM SPIN Kit for Soil提取酒醅样品微生物菌群宏基因组DNA。以其为模板,采用引物27F(5'-AGAGTTTGATCMTGGCTCAG-3')和1492R(5'-ACCTTGTTACGACTT-3')PCR扩增16S rRNA V1~V9基因序列。PCR扩增体系及程序参照文献[20]进行。采用PacBio Sequel II测序平台对PCR扩增产物进行高通量测序。测序文库的构建及测序由上海派森诺生物科技股份有限公司完成。
1.3.2 高通量测序结果分析
组装策略参照文献[21],采用QIIME2对16S rRNA扩增子序列进行优化处理[22],将相似度>97%的序列归为一个扩增子序列变异体(amplicon sequence variants,ASV),将每个ASV的代表序列在数据库Silva 132进行分类学比对[23],在各分类水平下统计每个样品的群落组成。采用QIIME2计算超1(Chao1)指数和香农(Shannon)指数,表征细菌菌群的Alpha多样性,采用主坐标分析(principal coordinate analysis,PCoA)表征细菌菌群Beta多样性[24]。16S rRNA基因的功能预测分析由PICRUSt2.0分析软件完成[25],参考数据库为京都基因与基因组百科全书(Kyoto Encyclopedia ofGenes and Genomes,KEGG)。
1.3.3 统计分析
采用IBMSPSSStatistics20进行统计分析,利用Spearman相关系数揭示酒醅挥发物质含量[14]与微生物群落之间的关系,利用Graphpad Prism8.0.2进行绘图。
2.1.1 Alpha多样性分析
采用PacBio测序技术从酒醅样品细菌菌群中共获得225 444条高质量有效序列。样品有效序列的覆盖率为99.9%~100%,表明测序结果能代表样品的真实情况。酒醅样品细菌菌群的Alpha多样性指数见图1。Chao1指数和Shannon指数分别描述菌群物种的丰富度与多样性,Chao1指数和Shannon指数值越大,说明群落多样性越高[26]。由图1可知,酒醅发酵过程中,细菌菌群的Chao1指数和Shannon指数均随着发酵时间的延长呈先升高后下降再趋于稳定的趋势,且均在发酵第7天达到最大值,分别为722和7.8,说明细菌菌群多样性在第7天达到峰值,经过发酵后菌群物种多样性降低。

图1 酒醅发酵过程中细菌菌群Chao1指数和Shannon指数的变化
Fig.1 Changes in Chao1 index and Shannon index of bacterial communities of fermented grains during fermentation process
2.1.2 Beta多样性分析
基于细菌ASV水平对酒醅样品进行PCoA,结果见图2。由图2可知,PCo1和PCo2对酒醅样品中细菌菌群差异的方差贡献率分别为61.5%和10.6%,累计方差贡献率为72.1%,说明该结果包含了72.1%的原始信息,结果可信。在PCo1上,发酵0 d和7 d的酒醅样品聚在一起,发酵21~80 d的酒醅样品聚在一起,发酵14 d的酒醅样品位于两者之间。结果表明,细菌群落在酿酒过程经历了明显的变化,根据PCoA结果可将此过程分为变化期(0~14 d)和稳定期(14~80 d)。在变化期,酒醅样品中细菌菌群的多样性先增长后衰亡,并在稳定期维持在较低水平,这可能与酿酒环境的驯化作用有关,表明大部分不适应于酿酒环境的物种被淘汰。
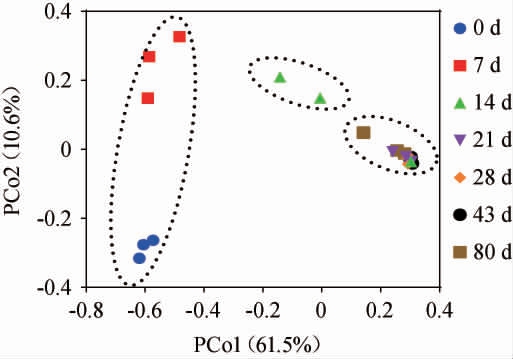
图2 基于细菌ASV水平酒醅样品的主坐标分析结果
Fig.2 Principal coordinates analysis results of fermented grains samples based on bacterial ASV levels
从酒醅样品中共注释到6个优势细菌门(相对丰度≥1.00%)、4个优势细菌纲和12个优势细菌种,其在酒醅发酵过程中的变化见图3。
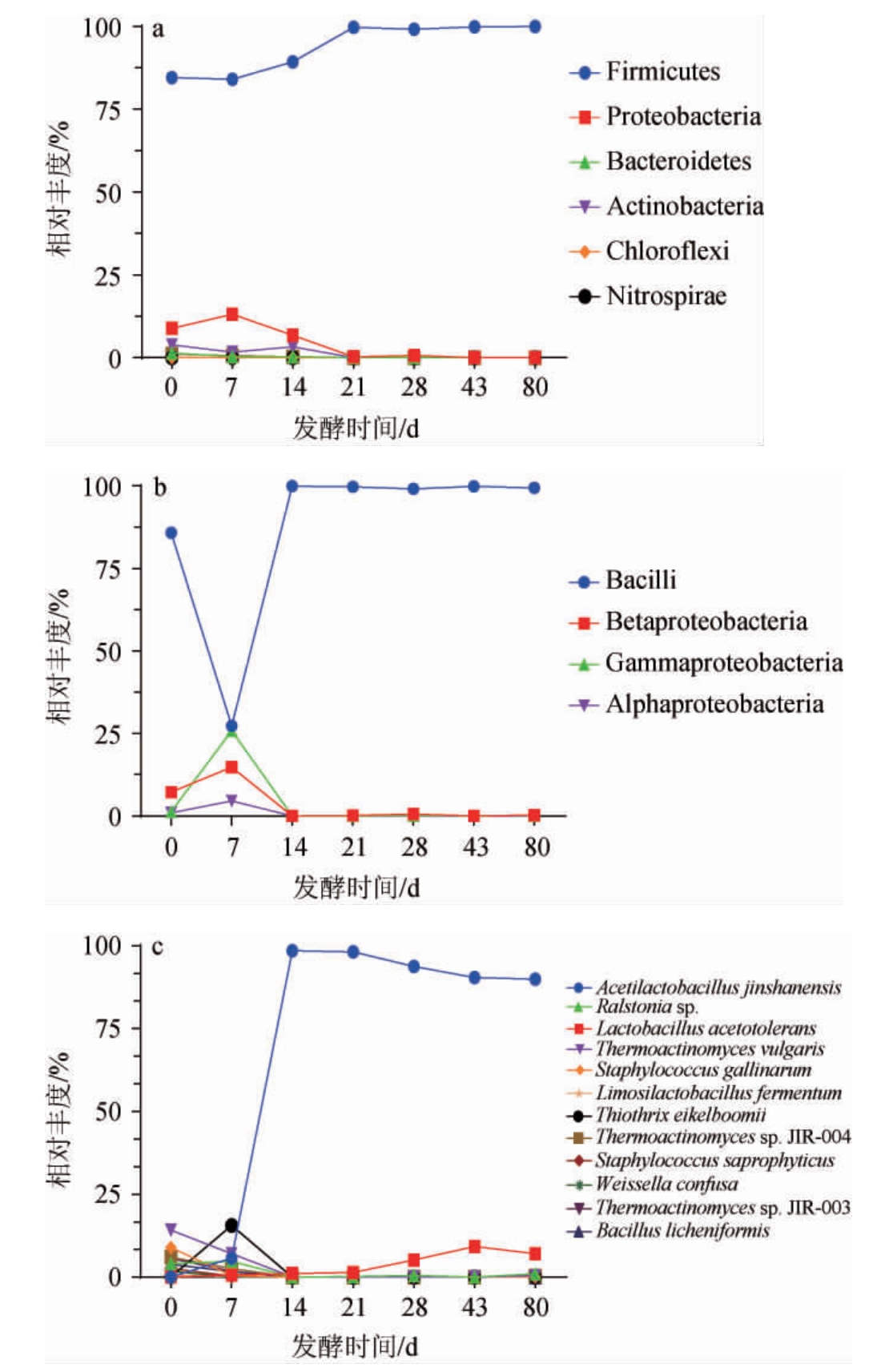
图3 基于门(a)、纲(b)和种(c)水平酒醅发酵过程中优势细菌群落的变化
Fig.3 Changes in dominant bacterial communities of fermented grains during fermentation process based on phylum (a),class (b) and species (c) levels
由图3可知,在门水平上,厚壁菌门(Firmicutes)在整个发酵过程中均为绝对优势类群,其相对丰度在发酵过程中逐渐增加(84.6%~99.9%)。变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、绿弯菌门(Chloroflexi)、硝化螺旋菌门(Nitrospirae)在变化期为优势细菌门,但它们的相对丰度在稳定期逐渐减少。在纲水平上,芽孢杆菌纲(Bacilli)在变化期的波动较大,相对丰度先降低再上升,随后在稳定期保持绝对优势(84.04%~99.91%),其他优势细菌纲包括β-变形菌纲(Betaproteobacteria)、γ-变形菌纲(Gammaproteobacteria)、α-变形菌纲(Alphaproteobacteria)的变化与芽孢杆菌纲相反,在稳定期逐渐衰亡。在种水平上,第一优势细菌为金山醋酸乳杆菌(Acetilactobacillus jinshanensis)(60.72%),其次是高温放线菌(Thermoactinomyces vulgaris)(5.78%)、嗜酸乳杆菌(Lactobacillus acetotolerans)(4.74%)、地衣芽孢杆菌(Bacillus licheniformis)(1.72%)、鸡葡萄球菌(Staphylococcus gallinarum)(1.62%)、丝硫菌(Thiothrix eikelboomii)(1.60%)、嗜热放线菌(Thermoactinomyces sp.)JIR-004(1.60%)、嗜热放线菌(Thermoactinomyces sp.)JIR-003(1.58%)、腐生葡萄球菌(Staphylococcus saprophyticus)(1.55%)、发酵乳杆菌(Limosilactobacillus fermentum)(1.48%)、融合魏斯氏乳酸菌(Weissella confusa)(1.46%)、未命名罗尔斯通氏菌(Ral stonia sp.)(1.26%)。其中,金山醋酸乳杆菌在发酵0~7 d相对丰度较低,7~14 d相对丰度大幅提高,从5.8%迅速上升到98.52%,并在此后保持稳定。嗜酸酸乳杆菌、未命名的罗尔斯通氏菌等其他物种整体呈现减少趋势,并在稳定期保持相对丰度较低状态。
前期分析了浓香型白酒酿造过程中挥发性风味物质含量的变化,将其中11种主要挥发性风味物质[14],与本研究12种优势细菌种的相对丰度进行相关性分析,结果见图4。
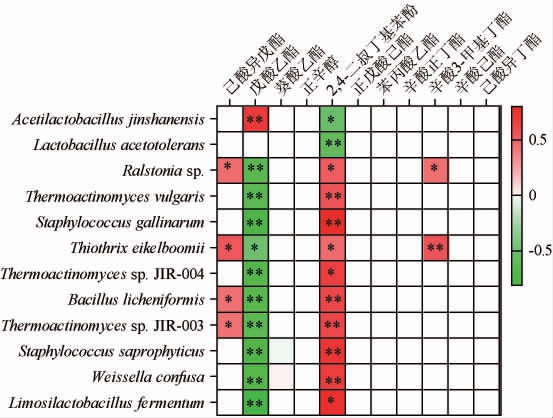
图4 酒醅样品中优势细菌种与挥发性风味物质间的相关性分析结果
Fig.4 Correlation analysis results between dominant bacterial species and volatile flavor compounds in fermented grains samples
“*”表示显著相关(P<0.05),“**”表示极显著相关(P<0.01)。
由图4可知,白酒酿造过程中的微生物与挥发性风味物质之间具有一定的相关性,其中,与未命名的罗尔斯通氏菌(Ralstonia sp.)、丝状菌(Thiothrix eikelboomii)相关的挥发性风味物质较多,有己酸异戊酯、戊酸乙酯、2,4-二叔丁基苯酚、辛酸3-甲基丁酯。另一方面发现,2,4-二叔丁基苯酚和戊酸乙酯几乎与所有优势细菌种存在显著相关性(P<0.05),表明酿造过程风味物质的产生依赖多种微生物的参与。
浓香型白酒的主体香味物质包括己酸乙酯、乳酸乙酯、丁酸乙酯、乙酸乙酯,前期工作初步整合了与它们合成相关的代谢通路[14],基于此,采用PICRUSt2.0分析软件对酒醅样品中优势细菌种代谢通路相对丰度的变化进行分析,结果见图5。
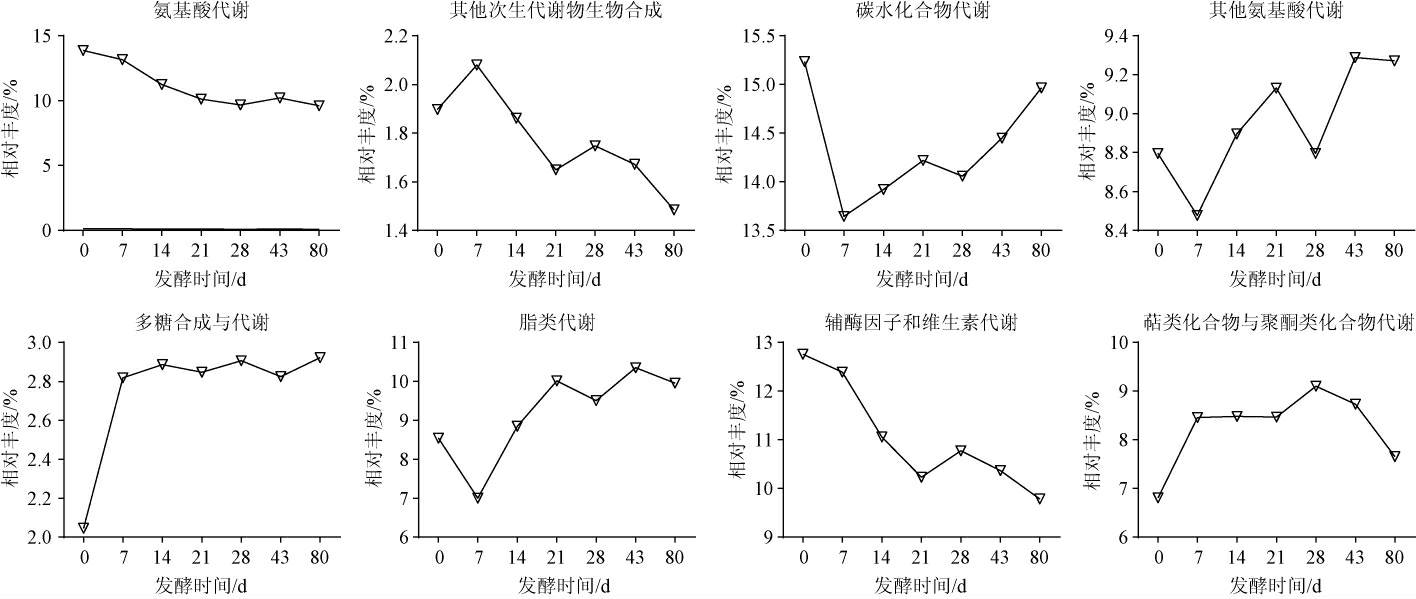
图5 浓香型白酒发酵过程中优势细菌种代谢特性变化分析结果
Fig.5 Analysis results of metabolic characteristic changes of dominant bacteria species of strong-flavor Baijiu during fermentation process
由图5可知,在酿造过程中,氨基酸代谢、其他次生代谢产物的生物合成、辅酶因子和维生素的代谢呈下降趋势;脂类代谢、其他氨基酸代谢、碳水化合物代谢在发酵0~7 d下降,随后整体呈升高趋势;甘氨酸的生物合成和在发酵0~7 d呈上升趋势,之后保持稳定;萜类和聚酮类化合物的代谢在发酵0~7 d上升但在发酵28 d后下降。结果表明,随微生物群落结构的演替,与白酒生产相关的代谢功能也发生了复杂的变化。
16S rRNA基因是微生物检测和鉴定最常用的区域,基因碱基长度约1 500 bp,包括9个可变区(V1~V9),对不同可变区进行测序会造成研究结果的差异[27]。目前,传统常用的测序只针对某个可变区进行,最常检测的区域为V3~V4区,这种对微生物的检测鉴定,能在属水平提供较为详细的信息,同时也具有一定的局限性,不能在种及种以下水平鉴定出微生物菌群。在功能预测方面由于PacBio测序结合了一代Sanger测序的读长优势和Illumina测序的高通量优势,具有覆盖度广、通量高、预测信息全等优势[12]。本研究使用16S rRNA基因全长测序技术PacBio分析浓香型白酒酿造过程中酒醅细菌群落及其功能的动态变化。在门水平上,共注释到6个优势细菌门,该结果与邓杰等[28-29]在窖泥中通过V3~V4测序得到的结果相比较,发现内厚壁菌门、放线菌门、拟杆菌门均为优势菌门,说明酿酒环境的微生物群落具有相似性。芽孢杆菌纲为酒醅样品中的优势细菌纲,这与陈慧颖等[30]通过V3~V4测序揭示的酒醅微生物群落结构一致,但芽孢杆菌纲在本研究中发酵14~80 d期间相对丰度高达99.91%,与陈慧颖等[30]报道的结果(69%)具有较大差异。除测序技术不同外,此差异还可能与酒醅样品来源不同、DNA提取质量不同等多种因素有关[31]。本研究发现金山醋酸乳杆菌是酒醅中最丰富的微生物,在发酵14d后相对丰度>90%,这与以往报道的乳杆菌是最优势物种不同[32]。金山醋酸乳杆菌是乳杆菌科(Lactobacillaceae)的新物种,其模式菌株HSLZ-75T分离自镇江香醋醋醅,与乳杆菌属的16S rRNA基因序列高度相似[33]。金山醋酸乳杆菌与乳杆菌在本研究中均被检出在一定程度上体现了16S rRNA全长基因测序技术PacBio在种水平研究微生物群落的优越性。
基于16S rRNA基因全长测序技术PacBio解析浓香型白酒酿造过程的细菌群落结构,在酿造过程中,细菌群落及其代谢特性发生明显的变化演替,可分为变化期(0~14 d)和稳定期(14~80 d)两个阶段。同时从酒醅样品共检测到6个优势细菌门(相对丰度≥1.00%)(厚壁菌门、变形菌门、放线菌门、拟杆菌门、绿弯菌门、硝化螺旋菌门)、4个优势细菌纲(芽孢杆菌纲、β-变形菌纲、γ-变形菌纲、α-变形菌纲)和12个优势细菌种(金山醋酸乳杆菌、高温放线菌、嗜酸乳杆菌、地衣芽孢杆菌、鸡葡萄球菌、丝硫菌、嗜热放线菌JIR-004、嗜热放线菌JIR-003、腐生葡萄球菌、发酵乳杆菌、融合魏斯氏乳酸菌、未命名的罗尔斯通氏菌),其中,金山醋酸杆菌在整个酿造过程占据主导地位。通过相关性分析发现,白酒酿造过程中的微生物与挥发性风味物质之间具有一定的相关性,其中,未命名的罗尔斯通氏菌和丝状菌具有明显相关性的挥发性风味物质较多,有己酸异戊酯、戊酸乙酯、2,4-二叔丁基苯酚、辛酸3-甲基丁酯等。另一方面,2,4-二叔丁基苯酚和戊酸乙酯几乎与所有优势细菌种存在显著相关性(P<0.05)。通过功能预测发现,随微生物群落结构在酿造过程中的演替,与白酒生产相关的代谢功能也发生了复杂的变化,与白酒酿造相关的代谢通路中甘氨酸的生物合成和代谢、脂质代谢、核苷酸代谢的相对丰度在发酵过程中提高,但氨基酸代谢、其他次生代谢产物生物合成、辅因子和维生素代谢呈下降趋势。本研究结果可为深入认识此白酒发酵机制提供理论依据。
[1]范锦琳,超易,王诗宇,等.基于高通量测序技术解析菌草酒发酵过程中细菌菌群变化[J].中国酿造,2023,42(8):52-57.
[2]张大凤,李可,刘森.中国浓香型白酒窖池糟醅中微生物群落演替分析[J].食品科学,2012,33(15):183-187.
[3]范锦琳,张丽丽,张煜隆,等.菌草酒酿造工艺优化及其品质分析[J].福建农林大学学报(自然科学版),2023,52(1):97-105.
[4]李梦婷,许德富,敖宗华,等.整粒高粱投料浓香型白酒糟醅超长发酵及回酒蒸馏研究[J].中国酿造,2022,41(6):117.
[5]罗明有,超刘,松王,等.不同窖龄糟醅理化指标和细菌区系差异研究[J].食品科技,2022,49(9):68-73.
[6]禄凌飞,沈小娟,蕾郑,等.接种堆积成熟糟醅对酱香型酒堆积异常堆子的影响[J].中国酿造,2023,42(8):47-51.
[7]郑佳,杨康卓,张建敏,等.浓香型白酒窖泥香气成分的空间分布规律[J].食品与发酵工业,2022,46(1):11-15.
[8]张奶英,唐清兰,徐姿静.高通量测序技术分析剑南春白酒母糟真菌群落多样性特征[J].酿酒,2022,49(6):110-113.
[9]罗爱国,郗鑫瑞,郑同庆,等.基于Illumina MiSeq高通量测序分析清香型白酒酒糟微生物群落多样性[J].中国酿造,2022,41(1):98-102.
[10]于文娟,许玲,杨庆振,等.高通量测序技术解析扳倒井浓香型白酒窖泥原核微生物群落结构[J].酿酒,2021,48(3):81-85.
[11]杨旭,马歌丽,王光路,等.高通量测序解析白酒中高温大曲细菌和真菌群落结构[J].中国酿造,2020,39(6):119-123.
[12]韩国强,孙协平,吴鹏飞,等.基于高通量测序分析复配小曲白酒发酵过程中微生物群落结构及多样性[J].食品科学,2021,42(18):80-85.
[13]LIU M K,TANG Y M,LIU C Y,et al.Variation in microbiological heterogeneity in Chinese strong-flavor Baijiu fermentation for four representative varieties of sorghum[J].Int J Food Microbiol,2023,397:110212.
[14]LIU M K,TANG Y M,GUO X J,et al.Structural and functional changes in prokaryotic communities in artificial pit mud during Chinese Baijiu production[J].mSystems,2020,5(2):e00829-19.
[15]李耀门.基于MinION 16S rDNA全长扩增子测序的畜禽病原菌快速检测方法的建立及初步应用[D].扬州:扬州大学,2023.
[16]MIZRAHI-MAN O,DAVENPORT E R,GILAD Y.Taxonomic classification of bacterial 16S rRNA genes using short sequencing reads:Evaluation of effective study designs[J].PloS One,2013,8(1):e53608.
[17]MYER P R, KIM M, FREETLY H C, et al.Evaluation of 16S rRNA amplicon sequencing using two next-generation sequencing technologies for phylogenetic analysis of the rumen bacterial community in steers[J].J Microbiol Meth,2016,127:132-140.
[18]NAM Y D,LEE S Y,LIM S I.Microbial community analysis of Korean soybean pastes by next-generation sequencing[J].Int J Food Microbiol,2012,155(1-2):36-42.
[19]WANG Z,LIU H,WANG F,et al.A refined view of airway microbiome in chronic obstructive pulmonary disease at species and strain-levels[J].Front Microbiol,2020,11:1758.
[20]DONG W,ZENG Y,CUI Y,et al.Unraveling the composition and succession of microbial community and its relationship to flavor substances during Xin-flavor Baijiu brewing[J].Int J Food Microbiol,2022,372:109679.
[21]LIU M K,TIAN X H,LIU C Y,et al.Microbiologic surveys for Baijiu fermentation are affected by experimental design[J].Int J Food Microbiol,2024,413:110588.
[22]BOKULICH N A, KAEHLER B D, RIDEOUT J R, et al.Optimizing taxonomic classification of marker-gene amplicon sequences with QIIME 2's q2-feature-classifier plugin[J].Microbiome,2018,6(1):90.
[23]QUAST C,PRUESSE E,YILMAZ P,et al.The SILVA ribosomal RNA gene database project:Improved data processing and web-based tools[J].Nucleic Acids Res,2012,41(D1):D590-D596.
[24]SCHLOSS P D,WESTCOTT S L,RYABIN T,et al.Introducing mothur:Open-source, platform-independent, community-supported software for describing and comparing microbial communities[J].Appl Environ Microbiol,2009,75(23):7537-75441.
[25]DOUGLAS G M, MAFFEI V J, ZANEVELD J R, et al.PICRUSt2 for prediction of metagenome functions[J].Nat Biotechnol, 2020, 38(6):685-688.
[26]翟磊,于学健,冯慧军,等.宜宾产区浓香型白酒酿造生境中细菌的群落结构[J].食品与发酵工业,2020,46(2):18-24.
[27]王艳丽,张会敏,孟雅静,等.基于16S rDNA V4与V3-V4可变区高通量测序对浓香型白酒窖泥菌群组成的比较分析[J].现代食品科技,2020,36(6):147-154.
[28]邓杰,黄治国,卫春会,等.基于高通量测序的浓香型白酒窖池细菌群落结构分析[J].现代食品科技,2015,31(7):50-55.
[29]叶光斌,罗惠波,杨晓东,等.基于免培养法研究泸州地区浓香型白酒窖泥原核微生物群落结构[J].食品科学,2013,34(17):176-181.
[30]陈慧颖,李慧星,许彬,等.高通量测序研究浓香型酒酷的细菌菌群多样性及共变性[J].工业微生物,2021,51(4):35-43.
[31]MTHETHWA N P,AMOAH I D,REDDY P,et al.A review on application of next-generation sequencing methods for profiling of protozoan parasites in water: Current methodologies, challenges, and perspectives[J].J Microbiol Meth,2021,187:106269.
[32]ZHENG J, WU C, HUANG J, et al.Spatial distribution of bacterial communities and related biochemical properties in Luzhou flavor liquor fermented grains[J].J Food Sci,2014,79(12):M2491-M2498.
[33]孙佳.金山醋酸乳杆菌比较基因组分析与酿醋功能评价[D].无锡:江南大学,2021.