Effect of NaCl on papain activity with molecular dynamics simulation
木瓜蛋白酶是一种从木瓜中提取的水解酶,也称为木瓜酶或木瓜蛋白,主要存在于木瓜的果实和未成熟的种子中。木瓜蛋白酶为巯基水解酶,活性中心含半胱氨酸,具有耐热性能强、稳定性高、天然卫生安全等特点[1]。其为一种天然的食品添加剂,作为肉用调理粉的核心成分广泛应用于食品行业,在肉制品嫩化、改善肉制品风味以及制备调味料的原料等方面起到了重要作用。木瓜蛋白酶的活性与许多因素有关,如温度[3]、酶蛋白所处体系溶液环境[4-5]、无机离子[6-7]以及压力的大小[8-9]等。NaCl作为影响肉制品品质的重要因素之一[10],可以提升肉制品的嫩度以及多汁性,有效地抑制腐败微生物的生长代谢,延长肉制品的保存期限[11-13]。NaCl对于蛋白酶活性及结构的影响的相关研究也有很多,如保护酶[14]、毛霉酶[15]以及抗氧化酶[16]等。由于各类酶蛋白结构以及活性中心的不同,食盐对于它们活性的影响并不一致,在0~6%的加盐量下均促进毛霉酶系降解大豆蛋白[14];而抗氧化酶随着NaCl浓度的升高,活性呈现先促进后抑制的趋势,在120 mmol/L起出现抑制作用[16]。
近年来,许多技术都可用于研究蛋白质结构,如量热技术[17]、核磁共振以及X射线晶体学等,但是这类技术有一定局限性,仅能测得蛋白质的静态结构,而蛋白质多处于动态变化。若需测得蛋白质的动态结构,这涉及分子、原子以及离子多个层面。分子模拟技术现已成为有效手段之一,许多学者在研究酶结构时均采用了此技术,如氧化三甲基胺脱甲基酶[18]、细胞壁降解酶[19]以及α-葡萄糖苷酶[20]等。随着相关研究的不断深入,分子模拟技术得到了发展,特别是在结构化蛋白质的天然结构与构象变化、固有无序蛋白质的动态结构及其结合底物的动力学过程等方面取得了显著成果,但从分子层面探究木瓜蛋白酶活性变化规律的研究较少,因此有必要对其进行研究。
鉴于木瓜蛋白酶和NaCl均常运用于食品体系,该研究以不添加NaCl作为对照组,以不同NaCl浓度(0.25 mol/L、0.50 mol/L、0.75 mol/L)对木瓜蛋白酶进行处理,并测定在各浓度盐溶液中酶活性,再进行150 ns的分子模拟,进一步探究两者相互作用后木瓜蛋白酶的结构变化机制,为促进NaCl和木瓜蛋白酶共存于食品加工中的进一步应用奠定了一定的理论和实践基础。
木瓜蛋白酶(酶活>200 U/mg)、十二水合磷酸氢二钠、干酪素、三氯乙酸、L-酪氨酸:上海麦克林生化科技股份有限公司;L-半胱氨酸盐酸盐:北京博奥拓达科技有限公司;乙二胺四乙酸二钠:天津市天力化学试剂有限公司;盐酸:湖南汇虹试剂有限公司;氢氧化钠:国药集团化学试剂有限公司;氯化钠:天津市福晨化学试剂厂。所有试剂均为分析纯。
TP-1901紫外分光光度计:北京普析通用仪器有限责任公司;F98荧光分光光度计:上海棱光技术有限公司。
1.3.1 样品处理
酶缓冲溶液的配制:称取0.528 g L-半胱氨酸盐酸盐、0.223 6 g乙二胺四乙酸二钠,用磷酸氢二钠溶液(0.05 mol/L)溶解(共加入约90 mL),用1 mol/L的盐酸溶液调pH至6.0,并用纯水定容至100 mL。
待测酶溶液(0.5 mg/L)配制:准确取1.25 g木瓜蛋白酶溶于酶缓冲溶液(5 mL),静置20 min后定容至100 mL,再吸取1 mL至25 mL容量瓶,加酶缓冲液定容[21]。
1.3.2 木瓜蛋白酶的活性测定
准备4支干净试管,向对照组试管中加入1 mL蒸馏水,在3个NaCl浓度处理组试管中分别加入1 mL三种不同(0.25 mol/L、0.50 mol/L、0.75 mol/L)的NaCl溶液,并在4支试管中各加入5 mL待测酶溶液,放置在40 ℃水浴锅中水浴10 min后,吸取试管中溶液1 mL,加入5 mL酪蛋白溶液,振荡使之充分混合后封口放回40 ℃水浴锅中水浴10 min,再加入5 mL三氯乙酸溶液,立即摇匀,过滤,取滤液,用紫外分光光度计于波长280 nm处测定吸光度值,以纯水作空白[21]。木瓜蛋白酶活力测定公式如下:
式中:A1为酶溶液测试滤液的吸光度值;A2为酶空白测试滤液的吸光度值;A3为酪氨酸标准液的吸光度值;n为待测酶样品的稀释倍数;m为待测酶样品的质量,g。
1.3.3 分子动力学模拟
先在RCSB(https://www.rcsb.org/)网站上下载木瓜蛋白酶结构(ID号:1ppn),再用PyMOL软件手动去除结晶水,以得到纯蛋白。模拟采用Gromacs(2019.6)软件,选用GROMOS54a7力场、SPC水模型,将木瓜蛋白酶放置于立方体水盒子中并设置蛋白酶离盒子边缘最短距离为1 nm,通过添加不同数量的Na+和Cl-使体系达到预设的NaCl浓度并使整个体系保持电中性。使用最速下降法将体系能量最小化,先在NVT和NPT系统下分别进行400 ps的平衡,然后进行150 ns的分子动力学模拟,模拟步长为2 fs,每10 ps贮存1次数据,每个处理平行模拟3次。
模拟结束后,使用Gromacs软件相应的分析指令分析不同NaCl浓度处理时木瓜蛋白酶的叠加后骨架原子距离坐标的均方根偏差(root mean square deviation,RMSD)、蛋白质残基的距离均方根波动(root mean square fluctuation,RMSF)、回旋半径(radius of gyration,Rg)、氢键数量、可及表面积、二级结构的变化。其中,RMSF取150ns的数据,每0.1 ns提取一次,共1 500个数据点,以分析整个模拟过程中的蛋白结构变化;RMSD、氢键数量、蛋白二级结构、可及表面积、回旋半径则只选取了结构较稳定的100~150 ns进行分析计算,共500个数据点。
1.3.4 木瓜蛋白酶与底物对接分析
用ROSETTA(模拟蛋白质与底物对接)软件模拟不同条件下酶与蛋白结合位点的差异,取两组酶活有较大差异的样本,提取势能最低的结构进行对接。再采用I-TASSER在线服务器构建β-酪蛋白结构,使用ROSETTA3.7软件进行分子对接。共进行10次独立对接,每次对接产生一个对接打分。用PyMOL软件作出木瓜蛋白酶与底物对接结构图。
1.3.5 木瓜蛋白酶内源性荧光光谱的测定
将对照组及加入不同浓度(0.25 mol/L、0.50 mol/L、0.75 mol/L)NaCl溶液的木瓜蛋白酶溶液分别放入1.0 cm石英池中进行荧光光谱分析。荧光光谱条件为激发波长280 nm,扫描波长范围250~320 nm,激发和发射狭缝宽度5 nm。
1.3.6 数据处理
所有测量均进行3次重复,采用WPS Office软件对数据进行计算和整理,数据取平均值加减标准差;分子模拟部分,利用Origin 2021作图分析。
NaCl溶液对木瓜蛋白酶活性的影响结果见图1。由图1可知,三种浓度的NaCl(0.25 mol/L、0.50 mol/L、0.75 mol/L)溶液较对照组均会抑制木瓜蛋白酶活性,其抑制效果随着浓度的升高而增大,与对照组相比,NaCl浓度0.25 mol/L、0.50 mol/L、0.75 mol/L组活性分别下降13.86%、23.94%、31.73%。宁校平等[22]对β-葡萄糖苷酶的研究结果发现,随着体系NaCl溶液的浓度上升至1.21 mol/L时,残余酶活逐渐降低;何立超[23]的研究结论与本研究存在差异,认为NaCl对于酶活性具有双重作用,在NaCl添加量0.516 mol/L以下时,激活了脂肪氧合酶,但随着NaCl浓度继续升高,酶活性降低。这可能是因为各酶的结构和活性中心不同,从而导致不同浓度NaCl溶液对于酶蛋白活性的影响有所不同。
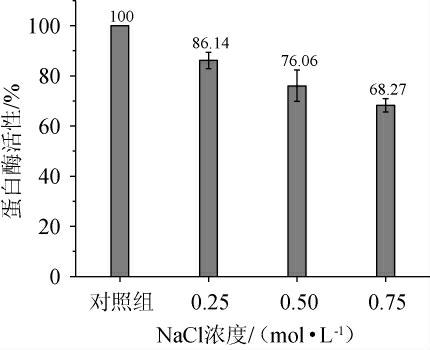
图1 NaCl溶液浓度对木瓜蛋白酶活性的影响
Fig.1 Effects of NaCl solution concentration on papain activities
蛋白质原子的均方根偏差(RMSD)通常是衡量蛋白质结构在模拟前后的结构偏差[31],一般用叠加起来的蛋白质骨架原子的距离来进行表示及比较的[24],是评价研究体系是否稳定的重要指标。不同NaCl浓度对木瓜蛋白酶骨架原子坐标距离的均方根偏差(RMSD)影响结果见图2。
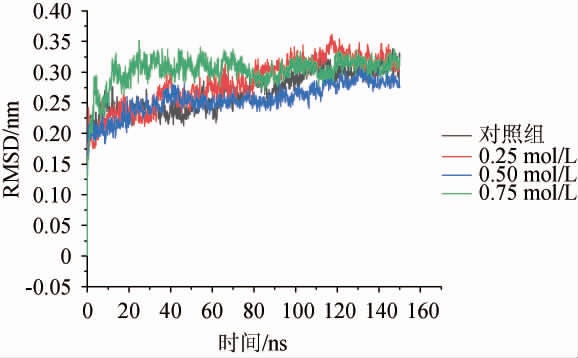
图2 不同浓度NaCl对酶蛋白均方根偏差的影响
Fig.2 Effects of different NaCl concentration on root mean square deviation of enzyme protein
由图2可知,因100 ns后体系趋于稳定,故选取100 ns后的数据进行分析,对照组及三种NaCl浓度(0.25 mol/L、0.50mol/L、0.75mol/L)下体系的RMSD均值分别为0.3047nm、0.323 7 nm、0.280 6 nm、0.310 5 nm。总体上来说,对照组及三种NaCl浓度组体系下的RMSD在整个模拟过程中较为平稳,波动较小,且各组之间均值差异较小。邵冬青[25]认为低浓度的盐溶液对大肠杆菌二氢酯酰胺酶结构影响不大,随着离子数目的增加,酶蛋白结构稳定性可能会随之减弱,这与本研究基本一致。因此,在NaCl浓度不高于0.75 mol/L条件下,木瓜蛋白酶与低浓度NaCl结合后,酶蛋白结构的RMSD变化不明显。
蛋白质残基的距离均方根波动(RMSF)可体现蛋白质中各氨基酸残基的波动情况[26],反映了一定时间内各氨基酸残基各原子坐标与平均位置坐标的波动情况。不同浓度NaCl处理对酶蛋白RMSF的影响结果见图3。
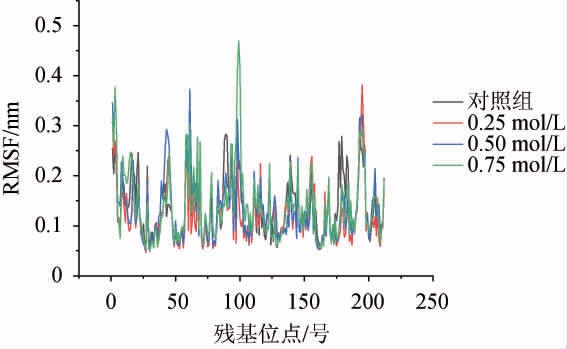
图3 不同浓度NaCl处理对酶蛋白距离均方根波动的影响
Fig.3 Effects of different NaCl concentration on root mean square fluctuation of enzyme protein
由图3可知,在对照组及三种不同浓度NaCl(0.25 mol/L、0.50 mol/L、0.75 mol/L)处理时,木瓜蛋白酶残基距离的均方根波动分别为0.123 4 nm、0.122 4 nm、0.136 0 nm、0.137 7 nm。可以看出,0.50 mol/L、0.75 mol/L两浓度组的RMSF值略高于对照组,且0.75 mol/L的NaCl溶液浓度下,几乎所有残基的RMSF值均较对照组出现了明显波动,推测出此浓度下木瓜蛋白酶存在柔性较大的不稳定区域[27]。在0.50 mol/L和0.75 mol/L这两组浓度下,木瓜蛋白酶活性中心的三个氨基酸残基(25号、158号、159号位的残基)发生明显波动,这说明较高盐浓度可能会使酶的活性中心结构稳定性下降,这与前面酶活结果一致。可以看出,木瓜蛋白酶在较高NaCl浓度条件下,各残基波动较大,活性中心结构较不稳定。
回旋半径(Rg)越小说明蛋白结构越致密,反之结构越疏松,回旋半径测量可以用于蛋白质大小表征,并衡量蛋白质变性过程的分子尺度变化[28],不同浓度NaCl处理对酶蛋白回旋半径的影响结果见图4。
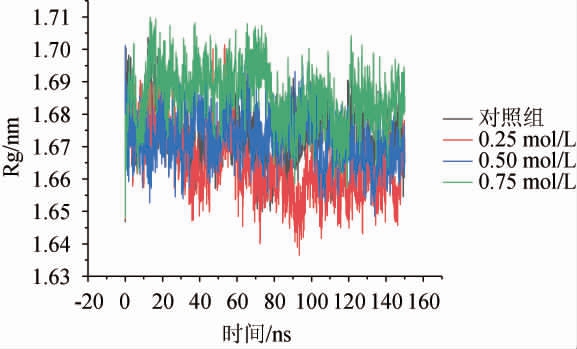
图4 不同浓度NaCl处理对酶蛋白回旋半径的影响
Fig.4 Effects of different NaCl concentration on circumflex radius of enzyme protein gyration
由图4可知,各浓度组分回旋半径基本在100 ns后趋于稳定,计算出模拟后100 ns的平均回旋半径,对照组及三种不同浓度NaCl(0.25 mol/L、0.50 mol/L、0.75 mol/L)处理时,酶蛋白平均回旋半径分别为1.672 nm、1.668 nm、1.670 nm、1.684 nm。这与邵冬青[25]在研究各种盐类对于大肠杆菌二氢酯胺脱氢酶半径的影响得出的结论一致,回转半径的最大值出现在0.8 mol/L附近。可以看出,各体系回旋半径差距不大,0.75 mol/L时相对最大,此时蛋白结构膨胀,表明主体结构变得较不稳定,这与前文酶活变化趋势基本相符。
氢键是一种常见的分子间作用力,在维持生物大分子的空间结构和生物活性方面具有重要意义。不同浓度NaCl处理对酶蛋白氢键数量的影响结果见图5。
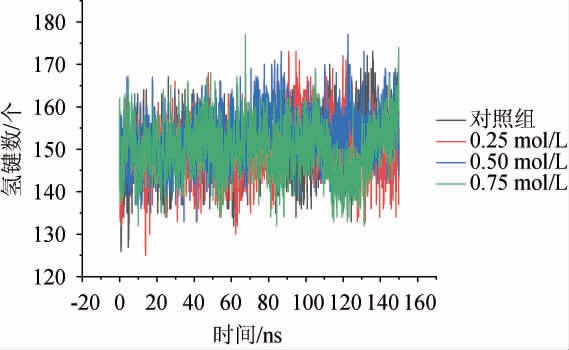
图5 不同浓度NaCl处理对酶蛋白氢键数量的影响
Fig.5 Effects of different NaCl concentration on hydrogen bonds number of enzyme proteins
由图5可知,随着NaCl溶液浓度的升高,蛋白质间氢键的数目降低,可能导致酶蛋白结构部分被破坏,从而降低蛋白酶的活性[30]。以模拟100 ns后进行计算,对照组及三种不同NaCl浓度(0、0.25 mol/L、0.50 mol/L、0.75 mol/L)处理时蛋白质平均氢键个数为151.8、151.14、154.61、149.94。陈立德[29]研究NaCl对肌原纤维蛋白影响时,发现NaCl浓度<0.50 mol/L时,对肌原蛋白氢键成促进作用,这可能是0.50 mol/L浓度时氢键个数升高的原因。0.75 mol/L时较对照组的氢键减少两个,说明该浓度的盐溶液对氢键具有破坏性,这与杨丹等[30]在实验中得出的结论一致,其认为高浓度氯盐对氢键破坏能力增强,这可能是该浓度下酶活显著降低的原因之一。因此,高浓度组的蛋白质间氢键的数目降低,可能导致酶蛋白结构部分被破坏,从而降低蛋白酶的活性[31]。
不同浓度NaCl处理对酶蛋白溶剂可及表面积的影响见表1。由表1可知,随着NaCl浓度的升高,木瓜蛋白酶总可及表面积、疏水表面积出现先增大后减小的趋势,亲水表面积并无明显规律变化。在0.75 mol/L时,酶的疏水和亲水表面积同时减小。前期疏水性的增加可能是NaCl溶液的存在破坏了蛋白分子外部亲水作用与内部疏水作用的平衡[32],而后期可能是过高添加量的NaCl抑制了蛋白质内部的疏水和二硫键共价作用,或者是因蛋白氧化加深导致内部分子产生交联,形成聚合物导致对蛋白酶敏感性降低[33]。在本研究中,在四种不同浓度NaCl添加量下,亲水表面积均大于疏水表面积,表明木瓜蛋白酶在不同浓度NaCl处理下均为水溶的。因此,亲水性食品体系中NaCl的存在不会影响木瓜蛋白酶。
表1 不同浓度NaCl处理对酶蛋白溶剂可及表面积的影响
Table 1 Effect of different NaCl concentration on accessible surface area of enzyme protein solvents
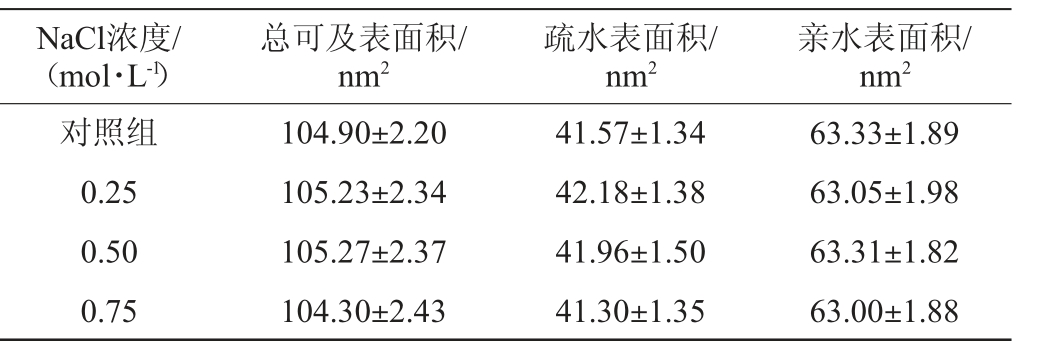
注:数据为100~150 ns模拟中500个数据的平均值及标准差。下同。
NaCl浓度/(mol·L-1)总可及表面积/nm2疏水表面积/nm2亲水表面积/nm2对照组0.25 0.50 0.75 104.90±2.20 105.23±2.34 105.27±2.37 104.30±2.43 41.57±1.34 42.18±1.38 41.96±1.50 41.30±1.35 63.33±1.89 63.05±1.98 63.31±1.82 63.00±1.88
对照组及三种不同浓度NaCl处理对木瓜蛋白酶二级结构的影响见表2。由表2可知,随着NaCl浓度的升高,α-螺旋先增加后减少、β-折叠则是先减小后增加。陆益钡等[33]认为不同浓度的NaCl溶液使得丝氨酸蛋白酶的α-螺旋结构减少,β-折叠以及无规则卷曲结构增多;楼宵玮等[34]也报道肌原纤维蛋白酶在0.75 mol/L的NaCl浓度下α-螺旋减少显著,这可能是因为0.75 mol/L浓度下蛋白酶氢键被破坏,而氢键是维持α-螺旋的重要因素,进而导致α-螺旋数目减少。金承昊等[35]研究发现,α-螺旋结构数量占比会影响蛋白质体系稳定性,α-螺旋占比越小,蛋白质体系稳定性越弱,进而可能影响酶活性。无规则卷曲随着NaCl浓度的升高而升高,说明酶的构象随着浓度增加变得越来越无序[36],这从结构变化上证实了NaCl的存在降低了酶的活性,且高浓度组表现得尤为明显。
表2 不同浓度NaCl处理对酶蛋白二级结构的影响
Table 2 Effects of different NaCl concentrations on the secondary structure of enzyme proteins
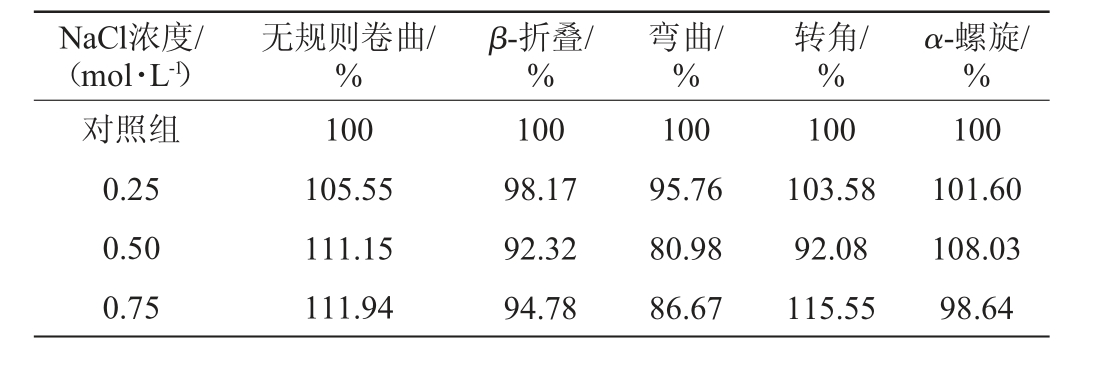
NaCl浓度/(mol·L-1)无规则卷曲/%β-折叠/%弯曲/%转角/%α-螺旋/%对照组0.25 0.50 0.75 100 105.55 111.15 111.94 100 98.17 92.32 94.78 100 95.76 80.98 86.67 100 103.58 92.08 115.55 100 101.60 108.03 98.64
分子模拟结束后,以对照组与0.75 mol/L NaCl浓度两组为例,使用ROSETTA3.7软件各进行10次独立分子对接。其中,对照组10次分子对接打分平均值为-245.205,0.75 mol/L下的10次分子对接打分平均值为-232.019。结果表明,对照组最低能量结构与β-酪蛋白对接,其打分明显低于0.75 mol/L组,说明此时木瓜蛋白酶结构与β-酪蛋白结合的更为紧密,催化活性更高。这与酶活及分子模拟分析结果一致。
木瓜蛋白酶与β-酪蛋白对接后得分最低的表面结构见图6,其红色部位为木瓜蛋白酶活性中心的三个氨基酸(Cys25、Asp158、His59)。由图6a可以看出,对照组蛋白结合部位无缝隙,结合较为紧密均匀,且结合部位含有苯丙氨酸(66号位)和赖氨酸(62号位),这两种氨基酸均为木瓜蛋白酶可特异性裂解的氨基酸肽链;由图6b可知,在NaCl浓度为0.75 mol/L时,蛋白结合部位则较为松散,空隙也较大,这与前面打分结果一致,也佐证了在0.75 mol/L时,木瓜蛋白酶活性降低。
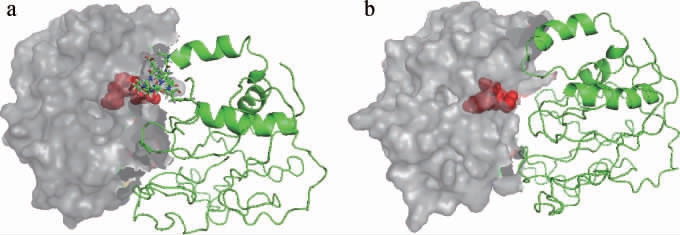
图6 不同浓度NaCl下木瓜蛋白酶与β-酪蛋白对接结构
Fig.6 Docking structure of papain and β-casein under different NaCl concentrations
a.对照组;b.0.75 mol/L。
为考察木瓜蛋白酶对NaCl溶液的敏感性,测量了对照组及使用三种不同浓度NaCl溶液处理后的木瓜蛋白酶的荧光强度,结果见图7。由图7可知,木瓜蛋白酶的荧光强度随着NaCl浓度的升高而降低[37],表明两者结合成了复合物,引发了木瓜蛋白酶产生内源性猝灭[38],说明木瓜蛋白酶与NaCl发生了结合。随着NaCl浓度的升高,荧光光谱发生轻微蓝移现象[39],蛋白酶最大吸收波长逐渐减小,说明此时蛋白酶芳香族氨基酸侧链逐渐暴露于分子外部,发生变性[38]。
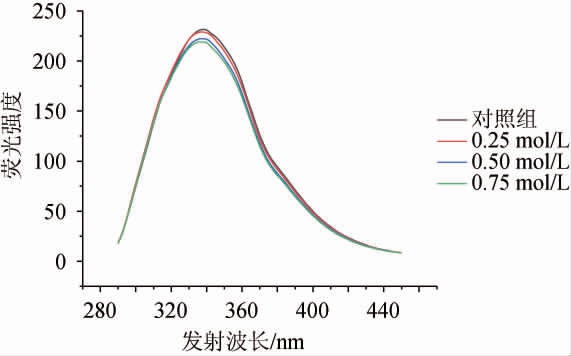
图7 不同浓度NaCl对木瓜蛋白酶荧光光谱的影响
Fig.7 Effect of different NaCl concentrations on fluorescence spectrum of papain
将酶活实验数据与分子动力学模拟及荧光分析结果相结合分析发现,NaCl浓度的升高引起的酶活降低,与该条件下蛋白酶各结构发生变化有关。主要表现为,随着NaCl浓度的升高,木瓜蛋白酶活性中心的三个氨基酸残基出现较大波动,且0.75 mol/L时蛋白酶回旋半径相对最大,此时蛋白结构膨胀,这表明高浓度组下的木瓜蛋白酶主体结构变得较不稳定。其次,高浓度NaCl下木瓜蛋白酶酶结构的破坏,也是影响其酶活性的原因,例如高浓度组下蛋白酶的总可及表面积以及疏水表面积减小、分子间氢键出现断裂,间接导致α-螺旋数目减少占比减小[35],无规则卷曲占比增加,蛋白质体系稳定性变弱[36]。
本研究考察不同浓度的NaCl(0、0.25 mol/L、0.50 mol/L、0.75 mol/L)对木瓜蛋白酶活性及分子结构的影响。结果表明,NaCl对木瓜蛋白酶有一定的抑制作用,且效果随着浓度的升高而增大,0.25 mol/L、0.50 mol/L、0.75 mol/L NaCl浓度组活性分别下降13.86%、23.94%、31.73%。分子模拟结果显示,0.50 mol/L和0.75 mol/L时蛋白酶活性中心的三个氨基酸残基有较大波动。另外,三个浓度组活性下降还与该条件下蛋白酶结构的变化有关,如氢键的破坏、疏水表面积的减小、α-螺旋的减少以及无规则卷曲增多等二级结构的变化等。对照组较0.75 mol/LNaCl浓度处理组相比,木瓜蛋白酶活性中心与β-酪蛋白结合得更为紧密,且蛋白内部结构更致密,更利于蛋白酶的催化作用。本研究同时利用实验手段和分子模拟技术,探讨了四种不同浓度NaCl添加量对木瓜蛋白酶活性的影响,为促进NaCl和木瓜蛋白酶共存于食品加工中的进一步应用及提高相关产品品质奠定了一定的理论和实践基础。
[1]曾颖,朱新儒,余垒,等.响应面法优化离子液体双水相萃取木瓜蛋白酶[J].食品研究与开发,2018,39(20):29-34.
[2]白玲燕.木瓜蛋白酶处理对牛肉品质影响的研究[J].食品安全导刊,2021(26):122-124.
[3]LI L,CHEN H J,LV X,et al.Effects of papain concentration,coagulation temperature,and coagulation time on the properties of model soft cheese during ripening[J].LWT-Food Sci Technol,2022,161:113404.
[4]吴胤霆,王洋,郑贵中,等.NaCl、糖类和木瓜蛋白酶对碱诱导蛋清凝胶性的影响[J].现代食品科技,2022,38(1):256-263.
[5]余垒.PEG/无机盐/离子液体、离子液体/无机盐双水相分离纯化木瓜蛋白酶的研究[D].海口:海南大学,2019.
[6]蔡西玲,曾虹燕,蔡联辉,等.Hg2+对木瓜蛋白酶的修饰作用及其动力学[J].中南大学学报(自然科学版),2013,44(10):3991-3997.
[7]刘学英,王帆,何东生,等.有机汞化合物对木瓜蛋白酶的失活机理研究[J].包装学报,2019,11(4):32-38.
[8]刘平,胡志和,吴子健,等.超高压对木瓜蛋白酶构象及酶活力的影响[J].食品科学,2015,36(23):23-27.
[9]孙树远,陈琪,何静,等.木瓜蛋白酶与超高压技术嫩化驼肉的研究[J].中国食品学报,2023,23(4):239-250.
[10]芮李彤,李海静,张婷婷,等.食盐对肉制品品质形成的作用及减盐技术研究进展[J].肉类研究,2022,36(7):61-67.
[11]王栋,张琦,陈玉峰,等.干腌肉制品低盐加工技术及其减盐机制研究进展[J].食品科学,2022,43(7):222-231.
[12]INGUGLIA E S,ZHANG Z,TIW ARII B K,et al.Salt reduction strategies in processed meat products-A review[J].Trends Food Sci Tech,2017,59:70-78.
[13]RHEE K S, ZIPRIN Y A.Pro-oxidative effects of NaCl in microbial growth-controlled and uncontrolled beef and chicken[J].Meat Sci,2001,57(1):105-112.
[14]杨俊鸾.NaCl胁迫对银杏叶绿素和保护酶活性的影响[J].山东林业科技,2019,49(3):49-52.
[15]王亚芬,舒青青,夏延斌.毛霉酶系降解大豆蛋白和多糖的影响因素研究[J].农产品加工(学刊),2013(15):7-9.
[16]李妍,张超,王士岭,等.NaCl胁迫对荞麦生长及抗氧化酶活性的影响[J].现代农业科技,2021(1):9-11.
[17]黄业传,张喜才,曾奕秀,等.高压处理对木瓜蛋白酶活性的影响及分子动力学模拟[J].食品工业科技,2023,44(12):102-107.
[18]曾祥权,曹瑞,余美宏,等.基于分子模拟技术的不同聚合度多酚与氧化三甲胺脱甲基酶的相互作用机制研究[J].食品与发酵工业,2024,50(11):85-97.
[19]LIU Y,QU W J,LIU Y X,et al.Assessment of cell wall degrading enzymes by molecular docking and dynamics simulations:Effects of novel infrared treatment[J].Int J Biol Macromol,2024,258(1):128825.
[20]SHARMA P,SHARMA V,MATHPAL S,et al.Identification of alphaglucosidase enzyme inhibitors from phytochemicals via integrated deep learning,molecular docking,molecular dynamics simulation,and MMPBSA analysis[J].S Afr J Bot,2024,167:48-61.
[21]张兴灿,陈朝银,李汝荣.木瓜蛋白酶的活力检测标准研究[J].食品工业科技,2011,32(10):435-437.
[22]宁校平,陈文清.硫酸铵盐析法提高β-葡萄糖苷酶活探究实验[J].四川化工,2016,19(2):17-19.
[23]何立超.鸭肉中脂肪氧合酶酶学特性及脂质氧化调控研究[D].南京:南京农业大学,2012.
[24]BABALOLA B A,AKINWANDE A I,OTUNBA A A,et al.Therapeutic benefits of Carica papaya:A review on its pharmacological activities and characterization of papain[J].Arab J Chem,2024,17(1):105369.
[25]邵冬青.温度与盐溶液对蛋白质结构稳定性的影响[D].杭州:中国计量大学,2022.
[26]ZHANG L,WANG P,YANG Z Y,et al.Molecular dynamics simulation exploration of the interaction between curcumin and myosin combined with the results of spectroscopy techniques[J].Food Hydrocolloid,2020,101(6):105455.
[27]钱辉,陆兆新,张充,等.鱼腥藻脂肪氧合酶热稳定性提高的分子动力学模拟[J].食品科学,2017,38(2):1-6.
[28]白丽燕,陈春琴,林东强,等.蛋白质水力学半径的测定及用于蛋白质变性过程的实时监控[J].化工学报,2008(6):1485-1489.
[29]陈立德.肌原纤维蛋白凝胶作用力影响因素的研究[D].重庆:西南大学,2010.
[30]杨丹,徐文艺.常见氯盐溶液中阳离子对水分子氢键影响的拉曼光谱研究[J].光谱学与光谱分析,2009,29(10):2694-2698.
[31]刘杭鑫.分子动力学模拟研究水合蛋白质的结构和动力学特征[D].南京:南京师范大学,2021.
[32]薛思雯,邹玉峰,杨慧娟,等.氯化钠浓度对僵直前高压处理兔肉中肌球蛋白凝胶保水性及其胶凝过程中分子特性和结构变化的影响[J].食品工业科技,2017,38(3):97-101.
[33]陆益钡,吕春霞,廖慧琦,等.NaCl对添加丝氨酸蛋白酶的肌原纤维蛋白凝胶特性的影响[J].食品与发酵工业,2021,47(2):78-86.
[34]楼宵玮,蒋娅婷,潘道东,等.氯化钠对肌原纤维蛋白与风味物质相互作用的影响[J].食品科学,2017,38(5):74-79.
[35]刘忠义,王璋,许时婴,等.草鱼肠道胰蛋白酶同工酶GT-B的结构信息[J].浙江大学学报(农业与生命科学版),2008(3):266-272.
[36]金承昊,冯宙,姜舟婷,等.α螺旋结构影响蛋白质稳定性的模拟研究[J].中国计量大学学报,2023,34(2):265-270.
[37]GAO W W,ZHANG F X,ZHANG G X,et al.Key factors affecting the activity and stability of enzymes in ionic liquids and novel applications in biocatalysis[J].Biochem Eng J,2015,99:67-84.
[38]田康永.[Cnmim]+、[CnPy]+类离子液体与木瓜蛋白酶相互作用的分子对接和荧光光谱研究[D].海口:海南大学,2020.
[39]韩永光,寿先苗,李晓飞.光谱法研究胞磷胆碱钠与牛血清白蛋白的相互作用[J].化学试剂,2019,41(4):402-405.