盐酸菜细菌菌群多样性及其与风味特征的关联分析
张振东1,罗习云1,罗春露1,马秀秀1,郭 壮2,3,杨 勇1,王玉荣2,3*
(1.黔南民族师范学院 生物科学与农学院,贵州 都匀 558000;2.湖北文理学院 湖北省食品配料工程技术研究中心,湖北 襄阳 441053;3.湖北文理学院 乳酸菌生物技术与工程襄阳市重点实验室,湖北 襄阳 441053)
摘 要:该研究采用高通量测序技术,电子鼻及电子舌分别对盐酸菜细菌菌群多样性及风味特征进行分析,并采用皮尔森相关系数对两者之间的相关性进行分析。结果表明,从盐酸菜中共注释到10个细菌门和335个细菌属,优势细菌门(平均相对丰度≥1%)为厚壁菌门(Firmicutes)(70.05%)、变形菌门(Proteobacteria)(25.16%)和放线菌门(Actinoacteria)(3.22%),优势细菌属为乳酸杆菌属(Lactobacillus)(15.72%)、促生乳杆菌属(Levilactobacillus)(12.70%)、泛菌属(Pantoea)(7.36%)等21个菌属。在气味指标上,不同来源盐酸菜的氮氧化合物、有机硫化物和萜类化合物、烷类和醇类化合物的差异较大;在滋味品质上,酸味和咸味的差异较大。相关性分析发现,促生乳杆菌属可能对盐酸菜的气味品质影响较大,而沙雷氏菌属(Serratia)可能起到增加盐酸菜鲜味的回味的作用。该研究为盐酸菜的发酵工艺改进以及品质控制提供了数据支撑。
关键词:盐酸菜;细菌菌群多样性;电子鼻;电子舌;风味品质;相关性
中图分类号:TS261.1
文章编号:0254-5071(2025)01-0119-05
doi: 10.11882/j.issn.0254-5071.2025.01.018
引文格式:张振东,罗习云,罗春露,等.盐酸菜细菌菌群多样性及其与风味特征的关联分析[J].中国酿造,2025,44(1):119-123.
收稿日期:2024-04-18
修回日期:2024-06-16
基金项目:黔南民族师范学院支持引进高层次人才研究专项(qnsyrc2022011)
作者简介:张振东(1985-),男,副教授,博士,研究方向为食品生物技术。
*通讯作者:王玉荣(1993-),女,讲师,博士,研究方向为食品生物技术。
Correlation analysis of bacterial flora diversity and flavor characterstic in Yansuancai
ZHANG Zhendong1,LUO Xiyun1,LUO Chunlu1,MA Xiuxiu1,GUO Zhuang2,3,YANG Yong1,WANG Yurong2,3*
(1.School of Biological Science and Agriculture,Qiannan Normal University for Nationalities,Duyun 558000,China;2.Hubei Provincial Engineering and Technology Research Center for Food Ingredients,Hubei University of Arts and Science,Xiangyang 441053,China;3.Xiangyang Lactic Acid Bacteria Biotechnology and Engineering Key Laboratory,Hubei University of Arts and Science,Xiangyang 441053,China)
Abstract:In this study, the bacterial flora diversity and flavor characteristics of Yansuancai (a traditional fermented vegetable) were analyzed using high-throughput sequencing technology,electronic nose and tongue,respectively,and the correlation between them was analyzed by Pearson correlation coefficient.The results showed that a total of 10 bacterial phyla and 335 bacterial genera were annotated,and the dominant bacterial phyla(mean relative abundance≥1%) were Firmicutes(70.05%), Proteobacteria (25.16%) and Actinoacteria (3.22%), and the dominant genera were 21 genera such as Lactobacillus(15.72%),Levilactobacillus(12.70%),Pantoea(7.36%).In terms of odor indexes,there were great differences in nitrogen oxides,organic sulfides and terpenoids,alkanes and alcohols from different sources of Yansuancai.In the taste quality, the difference in sour taste and salty taste was great.Correlation analysis results showed that the Levilactobacillus might have a great influence on the odor quality of Yansauncai, while Serratia might play a role in increasing the umami aftertaste of Yansauncai vegetable.This study provided data support for the improvement of fermentation technology and quality control of Yansuancai.
Key words:Yansauncai;bacterial flora diversity;electronic nose;electronic tongue;flavor quality;correlation
贵州省独山县的盐酸菜,最初叫“坛酸”,是贵州省南部地区布依族、水族和苗族等的一种特产发酵蔬菜[1]。盐酸菜所用的主要原料为十字花科芥菜型的青菜,辅以贵州“三名椒”之首的独山辣椒,配以食盐、大蒜、干辣椒、糯米甜酒、冰糖、白糖及食用白酒等,加上少量的纯碱入罐密封发酵制成[1-2],产品咸、酸、甜、辣俱全,色美清香、味鲜适口,深受消费者的欢迎[3-4]。可以直接作为小菜食用,也可以与其他蔬菜和肉品一起烹饪做菜。
由于微生物的生长代谢会使发酵食品具有特殊的风味,鉴别并利用提高发酵食品品质的微生物对人类的食品工业具有重要意义[5]。现阶段对于发酵食品中微生物的研究主要通过传统培养方法,即通过样品富集、菌株分离纯化及形态观察、生理生化试验结合16S rRNA等分子标记的分子生物学方法等步骤来确定分离物的分类,该方法受限于培养条件和培养基等因素,无法对相当一部分微生物进行分析[6]。第二代高通量测序技术因具有成本低,通量高,可以快速产生大量数据,能检测食品中的低丰度微生物,迅速成为食品、医学、环境等领域的常用技术,用来分析微生物多样性[7]。风味是食品的重要特征,决定了食品在市场上的可接受程度。电子鼻和电子舌作为一种新出现的仿生技术,其对样品的滋味与风味具有整体的选择性,不仅很好的解决了各类呈味物质因相互作用而难以分辨的情况,还避免了许多人为因素和实验操作带来的误差影响,在食品品质的评价方面具有无可比拟的优势[8-9]。由于盐酸菜通过自然发酵方式制作,品质不稳定[10],因此,建立盐酸菜中核心微生物与盐酸菜风味品质的关系,对于传统发酵食品质量的控制具有重要意义。
本研究采用电子鼻、电子舌技术对采集自贵州省独山县的盐酸菜样品的风味进行测定,通过Illumina MiSeq测序平台分析其细菌菌群多样性,并通过多元统计学方法对盐酸菜的菌群与风味进行关联分析,以期为盐酸菜中有价值的微生物菌群进行筛选与分离及提高盐酸菜的品质提供数据支撑及理论参考。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
盐酸菜样品:采集自贵州省黔南布依族苗族自治州独山县农贸市场,首先将样品迅速装入无菌瓶,放入低温样品箱,随后带回实验室,使用液氮冷冻后放入-70 ℃冰箱冷藏备用。采集的盐酸菜样品要求颜色正常,无异味。盐酸菜样品编号为YS1~YS11,不同编号表明样品采集自不同的商家或农户。
1.1.2 试剂
引物338F(5'-barcodeACTCCTACGGGAGGCAGCA-3')和806R(5'-GGACTACHVGGGTWTCTAAT-3'):武汉天一辉远有限公司;脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphates,dNTPs)Mix、10×聚合酶链式反应(polymerase chain reaction,PCR)buffer、FastPfu Fly脱氧核糖核酸(deoxyribonucleic acid,DNA)聚合酶(5 U/μL):北京全式金生物技术有限公司;QIAGEN DNeasy maricon Food Kit DNA基因组提取试剂盒:德国QIAGEN公司;电子鼻测定用阳离子溶液、阴离子溶液、内溶液、参比溶液:日本INSENT公司。其他试剂均为国产分析纯或生化试剂。
1.2 仪器与设备
PEN3电子鼻:德国Airsense公司;SA-402B电子舌:日本Insent公司;Illumina MiSeq高通量测序平台:美国Illumina公司;LC-LX-HRF250E高速离心机:上海力辰仪器科技有限公司;UVPCDS8000凝胶成像分析系统:美国Protein Simple公司;ND-2000C微量紫外分光光度计:美国NanoDrop公司。
1.3 方法
1.3.1 盐酸菜风味特征分析
采用仿生学仪器电子舌和电子鼻分别对盐酸菜的滋味指标和气味指标进行测定[11-12]。电子舌配备了酸味、苦味、涩味、咸味、鲜味和甜味传感器,以及三种回味:后味A(涩味的回味)、后味B(苦味的回味)和浓郁度(鲜味的回味)传感器。而电子鼻配备的传感器包括W1C、W1S、W1W、W2S、W2W、W3C、W3S、W5C、W5S和W6S[12]。
1.3.2 宏基因组DNA提取、PCR扩增及高通量测序
采用QIAGEN DNeasy maricon Food Kit DNA提取试剂盒提取盐酸菜样品的总基因组DNA,以其为模板,采用引物338F和806R PCR扩增细菌16S rRNA V3~V4区基因片段,PCR扩增体系及参数参照GUO Z等[13]的方法进行。采用1%琼脂糖凝胶电泳对PCR扩增产物进行检测,将检验合格的PCR扩增产物送往上海美吉生物医药科技有限公司,基于Illumina HiSeq平台进行高通量测序(双末端测序)。
1.3.3 生物学信息分析
将测序平台产生的原始序列使用QIIME 1.9.0平台进行生物信息学的分析[14]。首先根据引物中的序列标签拆分样品,然后去掉低质量的序列及接头。然后基于重叠区域合并双末端序列[15],去除嵌合体后,使用VSEARCH v2.14.1[16]按照97%的相似度划分操作分类单元(operational taxonomic unit,OTU),去掉嵌合体序列[17],使用RDP v18数据库[18]进行注释。最后使用QIIME v1.9.0平台内置脚本基于OTU对盐酸菜样品细菌菌群进行α和β多样性分析。
1.3.4 数据处理
使用R包自带的函数corr.test进行Pearson相关性分析,使用R包中的ggpubr v0.6.0和ggplot2 v3.5.0对数据进行可视化统计[19]。
2 结果与分析
2.1 盐酸菜样品中细菌菌群高通量测序结果及多样性分析
2.1.1 盐酸菜中细菌群落高通量测序结果及α多样性分析
盐酸菜样品中细菌菌群高通量测序结果及α多样性分析结果见表1。由表1可知,不同盐酸菜样品细菌菌群的高通量测序结果共产生267 191条有效序列,4 646个OTU,每个样本的平均有效序列数为24290条,平均OTU数为1245个。通过与物种RDP v18数据库对比,共注释到9个细菌门、20个细菌纲、53个细菌目、133个细菌科和335个细菌属。质控后,基于9 010的测序深度数据计算香浓(Shannon)指数和超1(Chao1)指数。Shannon指数常用来评价整体的物种多样性,而Chao1指数常用来标示物种丰富度[20]。由表1亦可知,盐酸菜样品YS01的Shannon指数值最高(6.86),而样品YS02的Shannon指数值最低(3.53),说明盐酸菜样品YS01的物种多样性最高,而样品YS02的物种多样性最低。盐酸菜样品YS07的Chao1指数值最高(1979.53),而样品YS02的Chao1指数值最低(861.02),说明盐酸菜样品YS07的物种丰富度最高,而样品YS02的物种丰富度最低。综上,不论是从α多样性来看,还是从OTU数来看,取自不同农家制作的盐酸菜样品中的细菌菌群多样性差别较大,分析原因可能是不同盐酸菜样品尽管采取了类似的制作过程及方法,但是制作的原材料使用量、原料品质、制作环境、添加的辣椒中的辣椒素含量等原因存在差异所致[21-22]。
表1 不同盐酸菜样品细菌菌群高通量测序结果及α多样性分析结果
Table 1 High-thoughput sequencing results and alpha diversity analysis results of bacterial flora in different Yansauncai samples
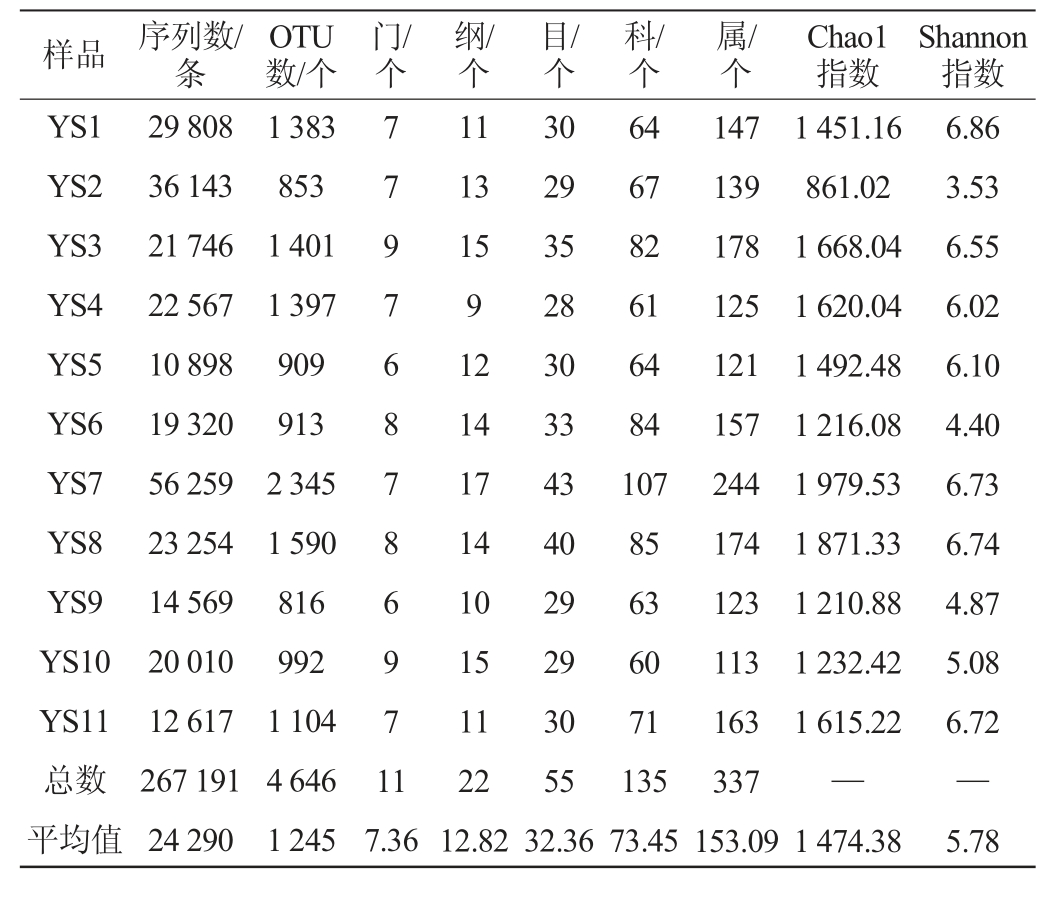
样品 序列数/条OTU数/个门/个纲/个目/个科/个属/个Chao1指数Shannon指数YS1 YS2 YS3 YS4 YS5 YS6 YS7 YS8 YS9 YS10 YS11总数平均值29 808 36 143 21 746 22 567 10 898 19 320 56 259 23 254 14 569 20 010 12 617 267 191 24 290 1 383 853 1 401 1 397 909 913 2 345 1 590 816 992 1 104 4 646 1 245 7 7 9 7 6 8 7 8 6 9 7 1 1 7.36 11 13 15 9 12 14 17 14 10 15 11 22 12.82 30 29 35 28 30 33 43 40 29 29 30 55 32.36 64 67 82 61 64 84 107 85 63 60 71 135 73.45 147 139 178 125 121 157 244 174 123 113 163 337 153.09 1 451.16 861.02 1 668.04 1 620.04 1 492.48 1 216.08 1 979.53 1 871.33 1 210.88 1 232.42 1 615.22—1 474.38 6.86 3.53 6.55 6.02 6.10 4.40 6.73 6.74 4.87 5.08 6.72—5.78
2.1.2 盐酸菜样品中细菌群落结构分析
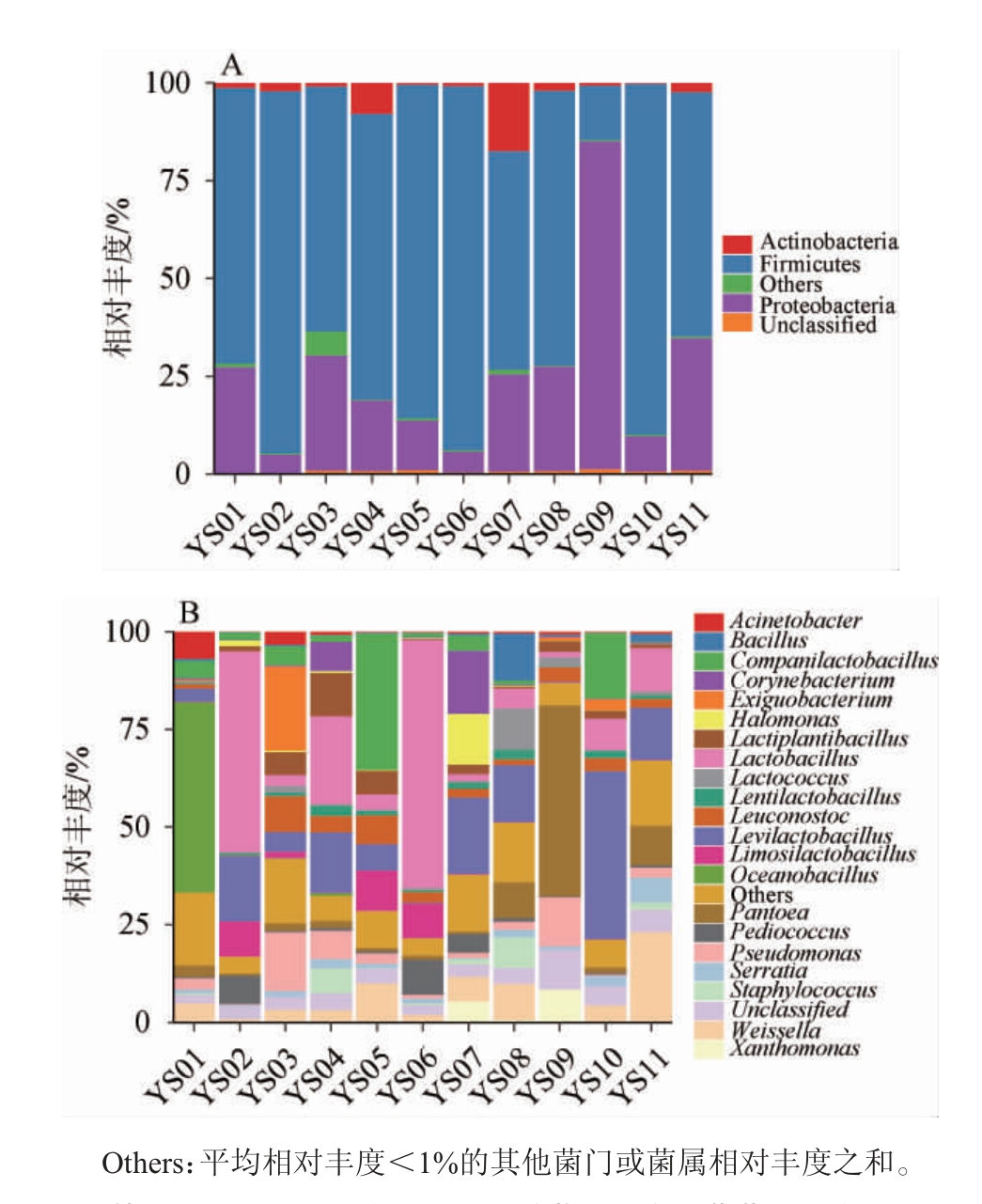
基于门和属水平不同盐酸菜样品中细菌菌群结构见图1。由图1A可知,在门水平上,盐酸菜样品中的优势细菌门(平均相对丰度≥1%)为厚壁菌门(Firmicutes)(70.05%)、变形菌门(Proteobacteria)(25.16%)和放线菌门(Actinoacteria)(3.22%)。
在属水平上,盐酸菜样品中的优势细菌属(平均相对丰度≥1%)有21个,包括乳酸杆菌属(Lactobacillus)(15.72%)、促生乳杆菌属(Levilactobacillus)(12.70%)、泛菌属(Pantoea)(7.36%)、伴生乳杆菌属(Companilactobacillus)(6.59%)、魏斯氏菌属(Weissella)(6.07%)、海洋芽孢杆菌属(Oceanobacillus)(4.59%)、假单胞菌属(Pseudomonas)(4.48%)、明串珠菌属(Leuconostoc)(3.40%)、乳植杆菌属(Lactiplantibacillus)(3.02%)、粘液乳杆菌属(Limosilactobacillus)(2.73%)、微小杆菌属(Exiguobacterium)(2.38%)、棒状杆菌属(Corynebacterium)(2.23%)、片球菌属(Pediococcus)(2.15%)、葡萄球菌属(Staphylococcus)(1.80%)、沙雷氏菌属(Serratia)(1.67%)、乳球菌属(Lactococcus)(1.64%)、芽孢杆菌属(Bacillus)(1.51%)、卤单胞菌属(Halomonas)(1.40%)、黄单胞菌属(Xanthomonas)(1.34%)、不动杆菌属(Acinetobacter)(1.20%)和迟缓乳杆菌属(Lentilactobacillus)(1.10%),其中,乳酸菌的平均相对丰度达到55.12%。盐酸菜在发酵方式上,属于半固态到固态发酵,但菌群结构与泡菜等发酵食品类似,以乳酸菌为优势菌。
2.2 基于仿生设备盐酸菜样品风味特征的分析
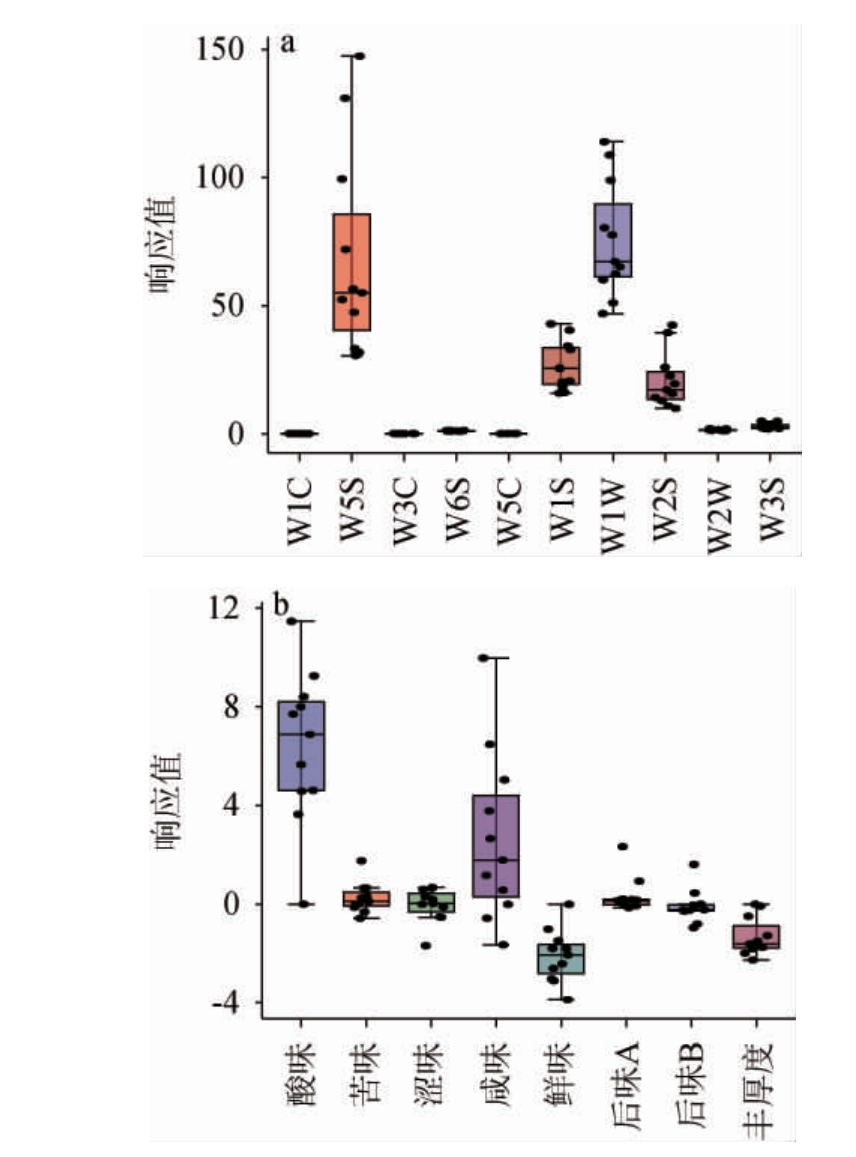
使用电子鼻和电子舌分别对盐酸菜的气味和滋味特征进行数字化的评价,结果见图2。由图2a可知,不同盐酸菜样品的气味特征具有较大差异。电子鼻传感器W1C(对芳香类物质灵敏)、W3C(对芳香类物质灵敏)、W6S(对氢气有选择性)、W5C(对烷烃,芳香类物质灵敏)对不同盐酸菜样品气味的响应值的极差值最小,W2W(对有机硫化物灵敏)、W3S(对烷烃灵敏)两个传感器对不同盐酸菜样品气味的响应值的极差值较大,传感器W5S(对氮氧化合物灵敏)、传感器W1W(对有机硫化物、萜类物质灵敏)、W1S(对烷类物质灵敏)和W2S(对醇类物质灵敏)对不同盐酸菜样品气味的响应差异最大。结果表明,采集的盐酸菜样品间的差异主要在于氮氧化合物、有机硫化物、萜类、烷类物质和醇类化合物。
由图2b可知,盐酸菜样品的滋味差异主要体现在苦味、涩味、鲜味、咸味、酸味和丰厚度上,尤其是酸味和咸味。研究表明,食盐能显著影响发酵食品的微生物菌群结构与风味[22]。推测可能的原因是在盐酸菜的制作过程中,添加的食盐量差异较大,因此咸味差异较大,而食盐含量又影响了盐酸菜中乳酸菌菌群的组成,由于不同乳酸菌发酵产酸能力不一,最终造成盐酸菜的酸味组内差异较大[23]。
2.3 盐酸菜样品细菌菌群与风味指标的相关性分析
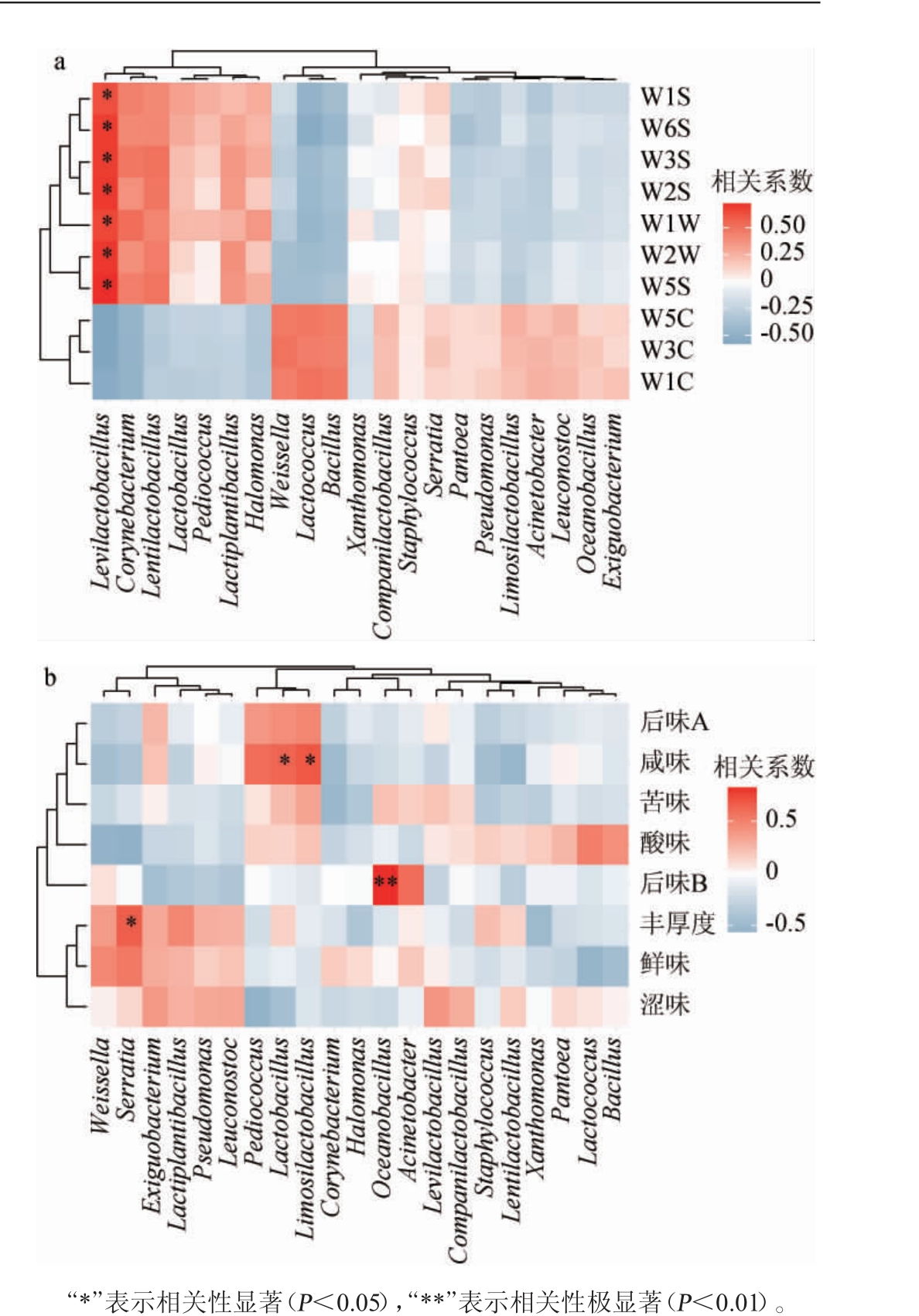
采用皮尔森相关系数对盐酸菜样品中的优势细菌属与电子鼻及电子舌的各响应值进行关联分析,结果见图3。
由图3a可知,盐酸菜样品中促生乳杆菌(Levilactobacillus)的相对丰度与WS型传感器W1S、W2S、W3S、W5S、W6S,以及WW型传感器W1W和W2W的响应值呈现显著正相关(P<0.05),即盐酸菜样品体系中的促生乳酸杆菌属相对丰度越高,样品中的有机硫化物、萜类物质、氮氧化合物、烷类物质、醇类物质等物质越多,反之则越少。其他优势细菌属与电子鼻的传感器响应值无显著相关性(P>0.05)。促生乳杆菌属是乳酸菌的重要成员之一,呈革兰氏阳性,杆状,过氧化氢酶阴性,异型乳酸发酵,一般能在发酵蔬菜和酸面团中检测到,于2020年乳杆菌属(Lactobacillus)中分出来成为单独的一个属[24]。有研究显示,该属是鲊广椒和虾酱等自然发酵制品的核心微生物属,该属与鲊广椒的特征性风味呈正相关,而与其缺陷型风味呈负相关[25]。该属的代表性物种是短促生乳杆菌(Levilactobacillus brevis),一些属于该类群的菌株能合成大量的γ-氨基丁酸,对人类胃肠环境的适应性好,以及对人类胃肠的健康和免疫有益[28]。也有研究发现,短促生乳杆菌能改善食品的气味[26]。尽管短乳杆菌对盐酸菜风味的作用尚未得到证实,结合已报道的研究能表明,盐酸菜中富集的促生乳杆菌属仍然对盐酸菜是有益的。
由图3b可知,沙雷氏菌属的相对丰度与样品的丰厚度,即鲜味的回味呈显著正相关(P<0.05),即盐酸菜样品体系内的沙雷氏菌属细菌相对丰度越高,盐酸菜样品鲜味的回味的就越重。同时,海洋芽孢杆菌属的相对丰度与样品的后味B,即苦味的回味呈极显著正相关(P<0.01)。另外,乳酸杆菌属和粘液乳酸杆菌属的相对丰度与样品的咸味呈显著正相关(P<0.05),即样品体系内的的乳酸杆菌属和粘液乳酸杆菌属的相对丰度越高,盐酸菜咸味越重。除此以外,还有许多优势菌属与盐酸菜的滋味指标呈现出一定的相关性,但均不显著(P>0.05)。未得到与盐酸菜酸味具有显著相关性的细菌菌群。酸味和鲜味是盐酸菜的特征性风味,而涩味和苦味是盐酸菜的缺陷型风味[27-28]。后味B是苦味的回味。结果显示,海洋芽孢杆菌属可能起到增强盐酸菜苦味的回味的作用,而沙雷氏菌属则可能起到增强盐酸菜鲜味的回味的作用。有研究发现,一些肽和氨基酸都呈苦味[27],海洋芽孢杆菌属是大曲中的常见菌,能产生蛋白酶、淀粉酶、酯酶等[29],因此具有分解蛋白质产生肽和氨基酸的能力,可能是其与盐酸菜苦味呈正相关的原因。尽管研究显示,沙雷氏菌与盐酸菜鲜味的回味呈显著正相关,可能起到增强盐酸菜鲜味回味的作用,然而该菌属于条件致病菌[30],因此需要尽可能的降低该属菌的含量。
3 结论
本研究结合高通量测序方法和电子鼻、电子舌技术,分别对盐酸菜样品的细菌菌群多样性和风味特征进行分析,并建立了盐酸菜微生物菌群与风味品质的关系。结果表明,盐酸菜中细菌主要由厚壁菌门(70.05%),变形菌门(25.16%)与放线菌门(3.22%)构成,其中厚壁菌门在盐酸菜中占绝对优势。优势细菌属包括乳酸杆菌属(15.72%)、促生乳杆菌属(12.70%)、泛菌属(7.36%)等21个细菌属,其中,乳酸菌的相对丰度>50%,可能是盐酸菜中的功能菌群,相关性分析结果证实了这一点。不同来源盐酸菜的氮氧化合物、有机硫化物和萜类化合物、烷类和醇类化合物的差异较大,促生乳杆菌对盐酸菜的这些风味指标具有促进作用。另外,不同来源盐酸菜的酸味和咸味的差异最大,与盐酸菜咸味相关的菌属有乳酸杆菌属和粘液乳杆菌属。此外,海洋芽孢杆菌属可能起到增加盐酸菜苦味的作用,而沙雷氏菌属能起到增加盐酸菜鲜味的回味的作用。本研究结果为盐酸菜的发酵工艺改进以及品质控制提供了数据支撑。
参考文献:
[1]许存宾,孔超华,黎成霞,等.贵州特色“独山三酸”的研究进展[J].中国调味品,2020,45(6):120-122.
[2]曾承露,李锋,黄德娜.盐酸菜中降胆固醇、亚硝酸盐乳酸菌筛选及功能特性研究[J].中国酿造,2017,36(7):37-41.
[3]袁睿.贵州盐酸菜的饮食人类学研究[D].贵阳:贵州民族大学,2023.
[4]陈梦玉,林平,林茂,等.辐照对盐酸菜感官品质的影响[J].贵州农业科学,2015,43(9):196-199.
[5]杨泞溶,余佳龙,宋家俊.发酵食品中微生物的功能特性[J].中国食品工业,2023(11):89-90.
[6]曹荣,张井,孟辉辉,等.高通量测序与传统纯培养方法在牡蛎微生物群落分析中的应用对比[J].食品科学,2016,37(24):137-141.
[7]HU T S, CHITNIS N, MONOS D, et al.Next-generation sequencing technologies:An overview[J].Hum Immunol,2021,82(11):801-811.
[8]牟心泰,杜险峰.电子鼻与电子舌在食品行业的应用[J].现代食品,2020(5):118-119.
[9]ZABADAJ M,UFNALSKA I,CHREPTOWICZ K,et al.Performance of hybrid electronic tongue and HPLC coupled with chemometric analysis for the monitoring of yeast biotransformation[J].Chemometr Intell Lab,2017,167:69-77.
[10]莫龙玲,张远林,何书成,等.盐酸菜产品开发现状及展望[J].贵州农机化,2019(4):22-24.
[11]WANG Y R,HOU Q C,GUO Z,et al.Bacterial community structure in acidic gruel from different regions and its influence on quality[J].Food Res Int,141:110130.
[12]LIU X T,WANG X L,CHENG Y W,et al.Variations in volatile organic compounds in Zhenyuan Daocai samples at different storage durations evaluated using E-nose,E-tongue,gas chromatography,and spectrometry[J].LWT-Food Sci Technol,2023,173:114186.
[13]GUO Z, WANG Y R, XIANG F S, et al.Bacterial diversity in pickled cowpea(Vigna unguiculata[Linn.]Walp)as determined by Illumina MiSeq sequencing and culture dependent methods[J].Curr Microbial, 2021,78(4):1286-1297.
[14]CAPORASO J G,KUCZYNSKI J,STOMBAUGH J,et al.QIIME allows analysis of high-throughput community sequencing data[J].Nat Methods,2010,7(5):335-336.
[15]MAGOCˇT, SALZBERG S L.FLASH: fast length adjustment of short reads to improve genome assemblies[J].Bioinformatics,2011,27(21):2957-2963.
[16]EDGAR R C.Search and clustering orders of magnitude faster than BLAST[J].Bioinformatics,2010,26(19):2460-2461.
[17]EDGAR R C, HAAS B J, CLEMENTE J C, et al.UCHIME improves sensitivity and speed of chimera detection[J].Bioinformatics,2011,27(16):2194-2200.
[18]COLE J R,CHAI B,FARRIS R J,et al.The ribosomal database project(RDP-II):Introducing my RDP space and quality controlled public data[J].Nucleic Acids Res,2007,35:169-172.
[19]赵恒,陈富安,赵慧君,等.茨河腐乳细菌与真菌多样性及其功能关联分析[J].食品与发酵工业,2022,48(10):212-218.
[20]WANG Q Q,PAN H,CHEN X,et al.Species identification and spatial diversity patterns of the Giant Panda National Park (GPNP) in Ya'an,Sichuan,China[J].Glob Ecol Conserv,2024,51(19):e02938.
[21]JONES N L,SHABIB S,SHERMAN P M.Capsaicin as an inhibitor of the growth of the gastric pathogen Helicobacter pylori[J].FEMS Microbial Lett,1997,146(2):223-227.
[22]刘鑫,王家旺,隋雨萌,等.中国传统发酵蔬菜中微生物多样性及其对品质形成影响研究进展[J].食品科学,2024,45(10):290-297.
[23]BANGAR S P, SURI S, TRIF M, et al.Organic acids production from lactic acid bacteria:A preservation approach[J].Food Biosci,2022,46:101615.
[24]ZHENG J S,WITTOUCH S,SALVETTI E,et al.A taxonomic note on the genus Lactobacillus: Description of 23 novel genera, emended description of the genus Lactobacillus Beijerinck 1901,and union of Lactobacillaceae and Leuconostocaceae[J].Int J Syst Evol Microbial,2020,70(4):2782-2858.
[25]LI Q,WANG Y W,TIAN Y Y,et al.Dynamic analysis of microbial community,flavor components of low-salt fermented red pepper sauce and exploring the key flavours formation[J].Food Biosci,2023,56:103423.
[26]ALFANO A, PERILLO F, FUSCO A, et al. Lactobacillus brevis CD2:fermentation strategies and extracellular metabolites characterization[J].Probiotics Antimicro,2020,12(4):1542-1554.
[27]CHU X Y,ZHU W S,LI X,et al.Bitter flavors and bitter compounds in foods:identification,perception,and reduction techniques[J].Food Res Int,2024,183:114234.
[28]SAFFARIONPOU,S.Off-flavors in pulses and grain legumes and processing approaches for controlling flavor-plant protein interaction: application prospects in plant-based alternative foods[J].Food Bioprocess Technol,2024,17:1141-1182.
[29]WANG L M,CHENG Y X,HU X X,et al.Analysis of bacterial diversity and functional differences of Jiang-flavored Daqu produced in different seasons[J].Front Nutr,2023,9:1078132.
[30]MAHLEN S D.Serratia infections:from military experiments to current practice[J].Clin Microbiol Rev,2011,24(4):755-791.