米酒是一种以稻米为主要原料,尤其是糯米,拌入酒曲发酵而成的低度酒精饮料[1]。在我国多个地区已经形成了许多具有地域特色的知名米酒,如孝感米酒、恩施米酒、桂林米酒、山兰米酒、海门米酒、客家米酒等,而在贵州则以黔南布依族苗族自治州和黔东南苗族侗族自治州的民族特色米酒为主,其是采用当地的糯米为原料,不同民族按照各自独特的酿造工艺生产的蒸馏型、发酵型米酒,按照NY/T 1885—2017《绿色食品米酒》的标准可将此类米酒定义为清汁型米酒(去除酒糟的米酒),两个特色产区的米酒酿造饮用文化体系极其丰富,现有苗家米酒、侗家米酒、三都县水族米酒、布依族黑糯米酒等具有代表性的米酒产品[2],其风味独特、米香协调、酸甜适口、酒体醇和,本土居民把它当作待客上品,除了直接饮用外,还是家常烹饪用于去腥、增香的首选[3-4]。米酒中富含多种氨基酸、有机酸、维生素和矿物质等营养成分[5-6],具有饭后开胃、活气养血、滋阴补肾、抗疲劳等功能,深受消费者青睐[7]。
贵州特色米酒的发酵多以民间自制酒曲为糖化发酵剂,酒曲的生产主要采用传统工艺进行,在适宜的条件下人工接种种曲后培养,通过广泛富集并保留种曲或环境中的有益微生物,获得具有糖化力高和出酒率高的酒曲[8],有研究表明,非无菌环境下自然发酵的传统酒曲具有极其丰富的微生物多样性,可以赋予产品独特的风味,但是以家庭自制、小作坊等开放型方式生产的酒曲,其微生物功能性差、安全性低,不能保障米酒产品的品质[9-11]。米酒的发酵是多种细菌、霉菌、酵母协同作用的过程,酒曲中的微生物种类、数量及菌群相互作用对米酒的风味品质至关重要[12],因此,认识米酒曲中的微生物群落结构十分必要。
目前,贵州特色米酒的标准化、规模化生产程度较低,同时关于黔南州、黔东南州米酒曲微生物的相关研究报道较少。因此,本研究采用Illumina NovaSeq高通量测序技术分析黔南州、黔东南州米酒曲的微生物多样性,寻找6个米酒曲的共同优势菌属,并通过线性判别分析效应大小(linear discriminant analysis effect size,LEfSe)对不同米酒曲中的差异菌群进行分析,为筛选酒曲中的共性和存在差异的功能微生物并制作强化酒曲,以及米酒发酵工艺的控制提供基础依据。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
米酒曲:采自黔南布依族苗族自治州(长顺县GL酒坊,编号QN1;三都县民间酒曲,编号QN2;都匀市QYS酒曲坊,编号QN3)和黔东南苗族侗族自治州(贵州省PF酒曲有限公司,编号QDN1;丹寨县SY酒坊,编号QDN2;凯里市FY酒曲厂,编号QDN3),采集的样品置于室温、避光干燥储存。
1.1.2 试剂
溴化十六烷基三甲铵(cetyltrimethyl ammonium bromide,CTAB)试剂:北京诺博莱德科技有限公司;PhusionR High-Fidelity聚合酶链式反应(polymerase chain reaction,PCR)Master Mix with GC Buffer(M0531S)、PhusionRHigh-Fidelity脱氧核糖核酸(deoxyribonucleic acid,DNA)polymerase(M0530S):美国New England Biolabs公司;2%琼脂糖凝胶:西班牙biowest公司;DNA纯化回收试剂盒:天根生化科技(北京)有限公司。
1.2 仪器与设备
DYCP-32C型琼脂糖水平电泳仪:北京市六一仪器厂;Covaris S2 System超声波破碎仪:美国Massachusetts公司;Qubit 2.0型量子比特荧光定量仪:美国Thermo Scientific公司;Agilent 5400生物分析仪:安捷伦科技有限公司;T100型PCR扩增仪:美国Bio-Rad公司;Nova seq 6000测序仪:美国Illuimina公司。
1.3 方法
1.3.1 DNA提取、PCR扩增及Illumina NovaSeq测序
采用CTAB法对样本基因组DNA进行提取[13],利用2%琼脂糖凝胶电泳检测DNA的纯度。
取适量的DNA为模板,利用引物515F(5'-GTGCCAGCMGCCGCGGTAA-3')和806R(5'-GGACTACCAGGGTATCTAAT-3')对细菌的16S rDNA V4可变区基因序列进行PCR扩增;利用ITS5-1737F(5'-GGAAGTAAAAGTCGTAACAAGG-3')和ITS2-2043R(5'-GCTGCGTTCTTCATCGATGC-3')对真菌ITS1 rDNA-5F可变区基因序列进行PCR扩增。PCR扩增程序为:98 ℃预变性1 min,30个循环(98 ℃变性10 s,50 ℃退火30 s,72 ℃延伸30 s),最后72 ℃延伸5 min。PCR扩增体系:Phusion Master Mix(2×)15 μL,正反向引物(1 μmol/μL)各1 μL,DNA模板10 μL,双蒸水(ddH2O)补足至30 μL。PCR扩增产物经2%琼脂糖凝胶电泳检测合格后,用DNA纯化回收试剂盒进行回收。
使用NEB NextR Ultra DNA Library Prep Kit建库试剂盒进行文库构建,将构建好的文库使用Aglient 5400生物分析仪进行检测和实时荧光定量PCR(real-time fluorescence quantitative polymerase chain reaction,RT-fqPCR)定量;待文库合格后,使用Illumina NovaSeq 6000平台进行高通量测序和生物信息学分析。
1.3.2 生物信息学分析与数据处理
参照CALLAHAN B J等[14]的方法对原始测序数据进行质控、修剪、去噪、拼接、去除嵌合体后得到有效序列。采用QIIME2分析平台将有效序列按照99%相似度构建操作分类单元(operational taxonomic units,OTUs),采用featuretable插件剔除含有嵌合体的OTUs[15];利用core-diversity插件计算Alpha多样性指数。使用Origin 2018软件绘制堆叠柱状图,在微科盟在线分析平台绘制稀疏曲线、韦恩图并进行LEfSe分析。
2 结果与分析
2.1 贵州特色米酒产区米酒曲微生物菌群α多样性分析
通过高通量测序从6个米酒曲样品中获得的细菌和真菌原始序列数分别为439 098条、507 778条,经过质控、修剪、去噪、拼接、去嵌合体后获得376 030条细菌16S rDNA片段有效序列和451 370条真菌ITS1 rDNA片段有效序列。从样品中随机抽取一定测序量与OUTs数目构建α多样性稀释性曲线,反映测序数据量的合理性和样品中的物种丰富程度,结果见图1。由图1可知,随着测序量的增加,6个米酒曲样品的细菌和真菌OUTs数稀释曲线均先急剧上升,达到一定测序数后接近饱和,说明该测序深度足以代表样品中的微生物多样性。

图1 不同米酒曲样品细菌(A)和真菌(B)OTUs数稀释性曲线
Fig.1 OTUs number dilution curves of bacteria (A) and fungi (B) of different rice wine Jiuqu
α多样性指数可以评估某个样本的物种丰富度(Faith's Phylogenetic Diversity(PFD)指数、Chao1指数)和多样性(Shannon指数)[16-18],以及样本检出率(覆盖率)[19-20]。黔南州、黔东南州中不同米酒曲样品细菌和真菌菌群α多样性指数见表1。由表1可知,6个米酒曲样品微生物的覆盖率均>0.99,说明从样品中检出物种的概率高,测序结果可以较好地反映米酒曲的物种丰富度和多样性;黔南州3个米酒曲样品中样品QN1的细菌菌群丰富度和多样性最高,真菌菌群丰富度和多样性最高的分别是样品QN3、QN2;黔东南州3个米酒曲样品中QDN1的细菌菌群丰富度和多样性最高,样品QDN3的真菌菌群丰富度和多样性最高。
表1 不同米酒曲样品微生物菌群α多样性分析结果
Table 1 Results of α-diversity analysis of different rice wine Jiuqu
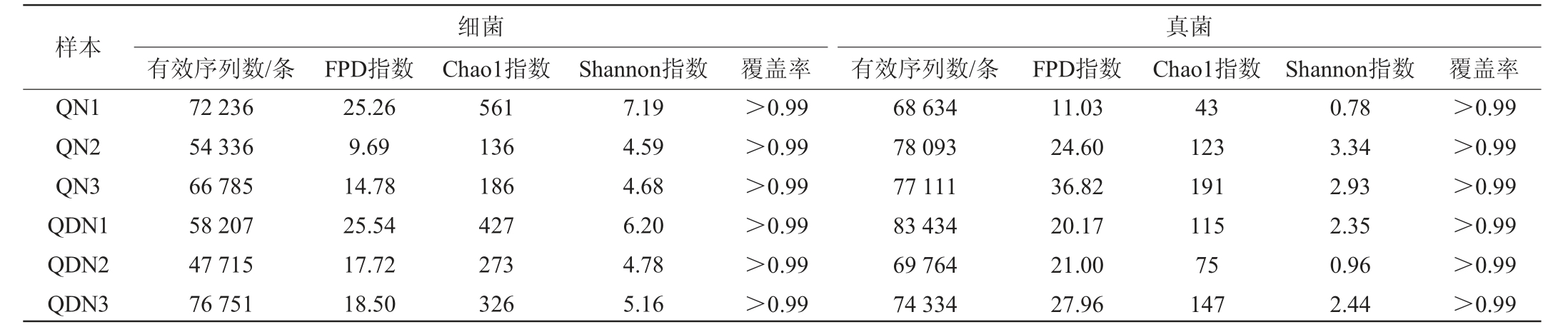
样本QN1 QN2 QN3 QDN1 QDN2 QDN3细菌有效序列数/条 FPD指数 Chao1指数 Shannon指数72 236 54 336 66 785 58 207 47 715 76 751 25.26 9.69 14.78 25.54 17.72 18.50 561 136 186 427 273 326 7.19 4.59 4.68 6.20 4.78 5.16覆盖率>0.99>0.99>0.99>0.99>0.99>0.99真菌有效序列数/条 FPD指数 Chao1指数 Shannon指数 覆盖率68 634 78 093 77 111 83 434 69 764 74 334 11.03 24.60 36.82 20.17 21.00 27.96 43 123 191 115 75 147 0.78 3.34 2.93 2.35 0.96 2.44>0.99>0.99>0.99>0.99>0.99>0.99
为了进一步掌握黔南州和黔东南州米酒曲样品中共有或特有微生物的物种丰富度差异,基于OUTs数目构建细菌和真菌韦恩图,结果见图2。
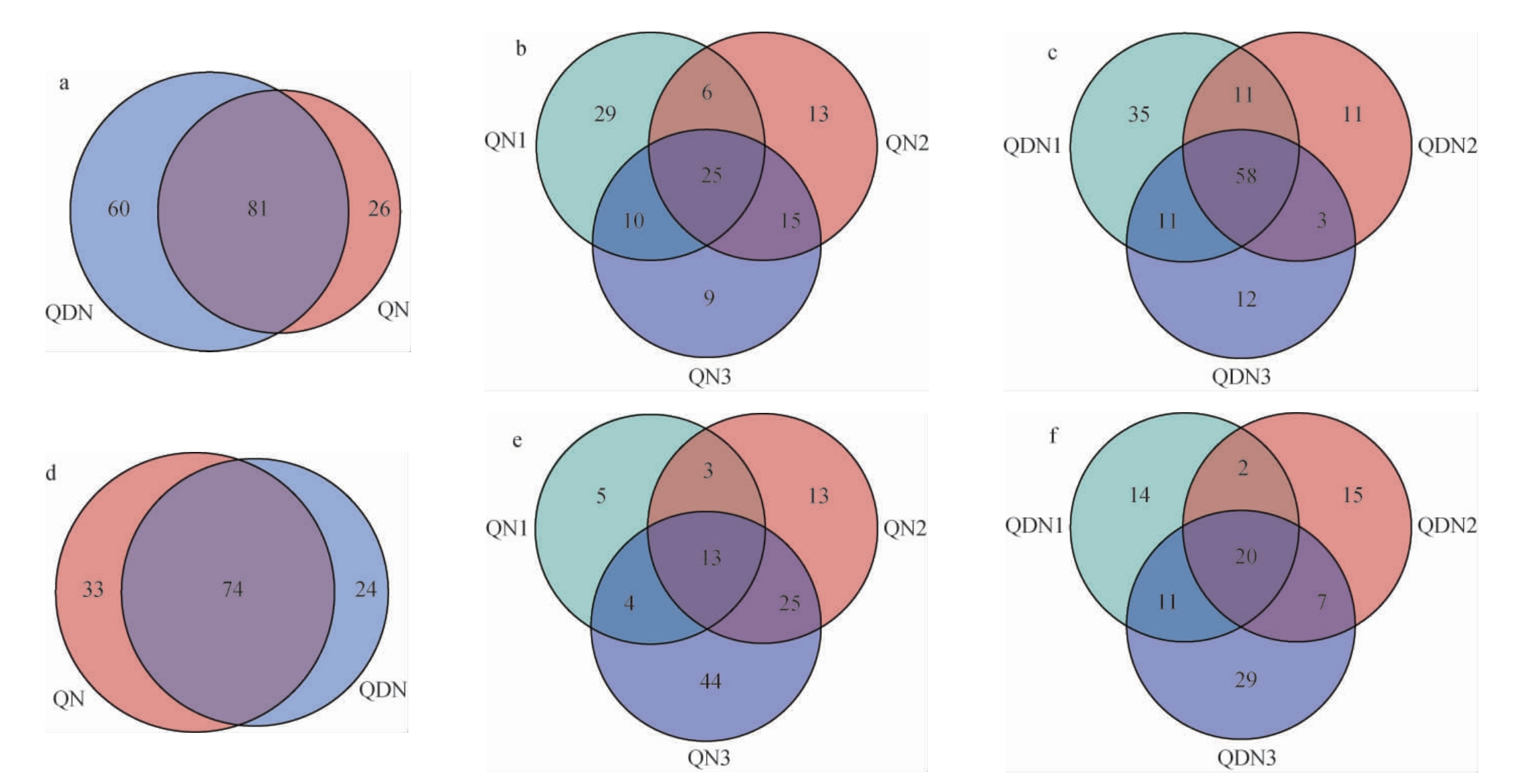
图2 基于OTUs数细菌(a、b、c)及真菌(d、e、f)菌群韦恩图
Fig.2 Venn diagram of bacterial (a, b, c) and fungal (d, e, f) flora based on OTUs numbers
由图2a可知,黔东南州与黔南州米酒曲样品的共有细菌OUTs数有81个,特有细菌OUTs数分别为60个和26个,由此说明黔东南州米酒曲样品细菌的丰富度更高,与α多样性指数分析结果一致。由图2b和2c可知,3种黔南州米酒曲样品的共有细菌OUTs数有25个,样品QN1、QN2、QN3特有细菌OUTs数分别为29个、13个、9个,说明米酒曲样品QN1的细菌菌群丰富度较高,而3种黔东南州米酒曲样品的共有细菌OUTs数有58个,样品QDN1、QDN2、QDN3特有细菌OUTs数分别为35个、11个、12个,说明米酒曲QDN1的细菌菌群丰富度较高。
同样,由图2d可知,黔南州和黔东南州米酒曲样品的共有真菌OUTs数有74个,特有真菌OUTs数分别为33个和24个,说明黔南州米酒曲样品真菌菌群丰富度较高,与α多样性指数分析结果一致。由图2e可知,3种黔南州米酒曲样品的共有真菌OUTs数有13个,样品QN1、QN2、QN3的特有真菌OUTs数分别为5个、13个、44个,表明米酒曲样品QN3的真菌菌群丰富度较高。由图2f可知,3种黔东南州米酒曲样品的共有真菌OUTs数为20个,样品QDN1、QDN2、QDN3的特有真菌OUTs数分别为14个、15个、29个,表明米酒曲样品QDN3的真菌菌群丰富度较高。
2.2 贵州特色米酒产区米酒曲微生物群落结构组成分析
2.2.1 细菌群落结构分析
从6个米酒曲样品中共注释到18个细菌门和21个细菌属,细菌群落结构见图3。由图3a可知,在门水平上,黔南州、黔东南州6个米酒曲样品中主要的细菌门为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria),这也是恩施米酒发酵过程中的优势细菌门[21]。6个米酒曲样品的共有优势细菌门(相对丰度>1%)为厚壁菌门、变形菌门和拟杆菌门,放线菌门仅是米酒曲样品QN2(2.28%)、QN3(5.06%)、QDN1(4.62%)的共有优势细菌门,该细菌门也是绍兴黄酒发酵初期(0~5 d)的优势细菌门(87%)[22]。有研究表明,隶属于变形菌门的运动发酵单胞菌(Zymomonas mobilis)在米酒发酵过程中的酒化效率更高[23]。姜丽[24]研究发现,黑糯米酒发酵过程中的优势细菌门为厚壁菌门和变形菌门,发酵前期(0~6 d)主要以变形菌门为主,该细菌门在样品QN1中的相对丰度为3.2%,而在其他5个米酒曲样品中的相对丰度均>34%,最高可达61.67%(样品QDN1),而黑糯米酒发酵后期(11~24 d)逐渐被厚壁菌门取代,该细菌门在6个米酒曲样品中的相对丰度均>25%,最高可达72.28%(样品QDN2)。
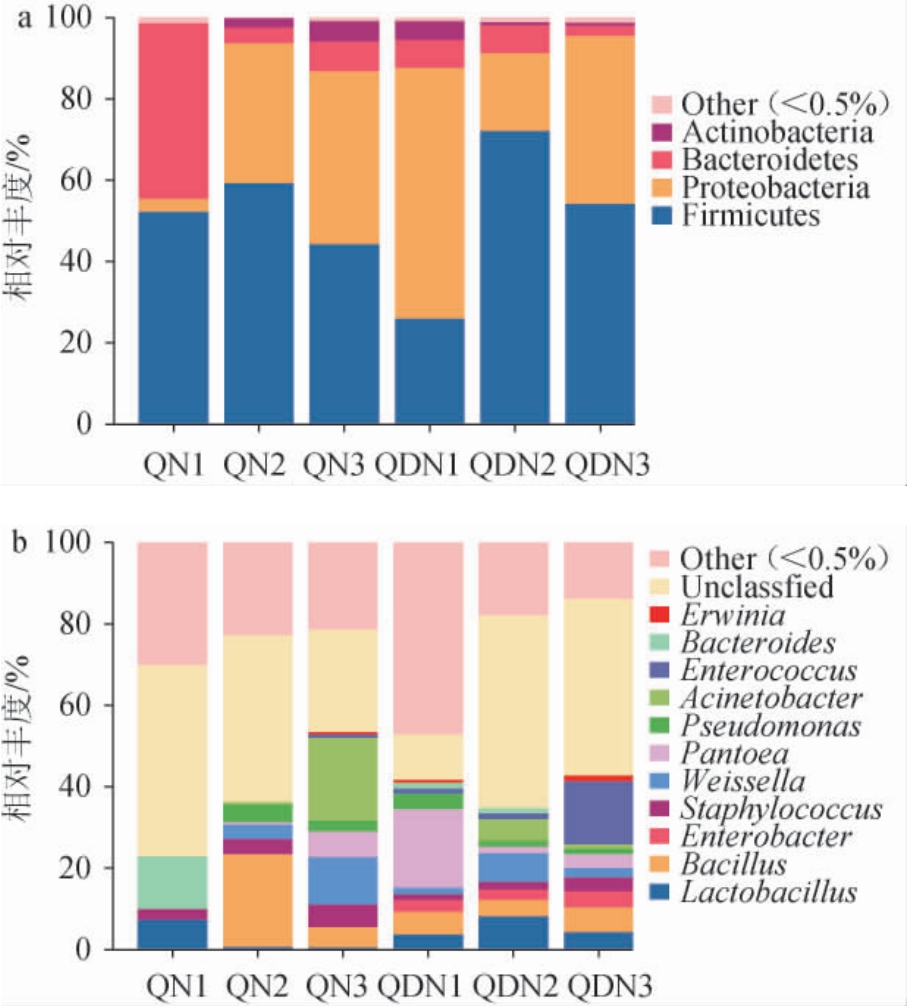
图3 基于门(a)和属(b)水平不同米酒曲样品的细菌群落结构
Fig.3 Bacterial community structure of different rice wine Jiuqu at phylum (a) and genus (b) level
Other:相对丰度<0.5%的其他菌门或菌属。下同。
由图3b可知,在属水平上,黔南州、黔东南州6个米酒曲样品中主要的细菌属为乳杆菌属(Lactobacillus)、芽孢杆菌属(Bacillus)、肠杆菌属(Enterobacter)、葡萄球菌属(Staphylococcus)、魏斯氏菌属(Weissella)、泛菌属(Pantoea)、假单胞菌属(Pseudomonas)、不动杆菌属(Acinetobacter)、肠球菌属(Enterococcus)、拟杆菌属(Bacteroides)、欧文氏菌属(Erwinia)。6个米酒曲样品的共有优势细菌属(相对丰度>1%)为葡萄球菌属,该菌属是红曲黄酒发酵前期的优势微生物[25];各米酒曲样品还含有的其他优势细菌属具有差异,米酒曲样品QN1的优势细菌属还有乳杆菌属(7.54%)和拟杆菌属(13.01%),除样品QN1外,其他5个米酒曲样品的共有优势细菌属为芽孢杆菌属、魏斯氏菌属、假单胞菌属,其中,芽孢杆菌属是米酒曲及米酒发酵过程中的重要细菌,能够降解谷物中的蛋白质和淀粉质,促进米酒中丁酸乙酯、己酸乙酯等风味化合物的产生[26],乳杆菌属、魏斯氏菌属等乳酸菌类群的主要功能是发酵碳水化合物代谢产生乳酸、乙酸、苹果酸、琥珀酸等有机酸,通过产酸降低米酒中的苦味,并为酯类化合物的生成提供前体物质[27],魏斯氏菌属与米酒的后味、涩味和苦味呈显著负相关关系[28],通过控制发酵过程中该细菌属的相对含量可能会改善米酒的品质。肠杆菌属(江米酒发酵前期的优势微生物[29])和肠球菌属仅存在于黔东南州3个米酒曲样品中,欧文氏菌属仅是样品QDN3的特有优势细菌属,相对丰度为1.44%。
2.2.2 真菌群落结构分析
从6个米酒曲样品中共注释到14个真菌门和22个真菌属,真菌群落结构见图4。由图4a可知,在门水平上,黔南州、黔东南州6个米酒曲样品中主要分布的真菌门是毛霉菌门(Mucoromycota)、子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、被孢霉门(Mortierellomycota)。6个米酒曲样品的共有优势真菌门(相对丰度>1%)为毛霉菌门和子囊菌门,这与杨甜甜等[30]的研究结果一致,其中,子囊菌门是贵州黑糯米酒、印度酒酿造过程中的主要真菌门,对传统酒曲、发酵米酒的品质和风味起关键作用[31-32];担子菌门仅为样品QN2(32.75%)和QN3(1.49%)的共有优势真菌门,相关研究表明,该真菌门只在黑糯米酒发酵初始阶段被检测到[24];被孢霉门仅为样品QN3(3.62%)和QDN3(2.08%)的共有优势真菌门,被孢霉门可以改善酒的后味,如隶属于被孢霉门的菌种在发酵过程中主要起糖化作用,厌氧环境下具有酒化作用,可以将糖进一步转化为乙醇[33],此外,一些青霉属(Penicillium)、曲霉属(Aspergillus)、根霉属(Rhizopus)还具有产蛋白酶、纤维素酶、酒化酶、单宁酶等作用,从而促进氨基酸、醇类、酯类等风味物质的生成,增加产品的风味品质[34]。
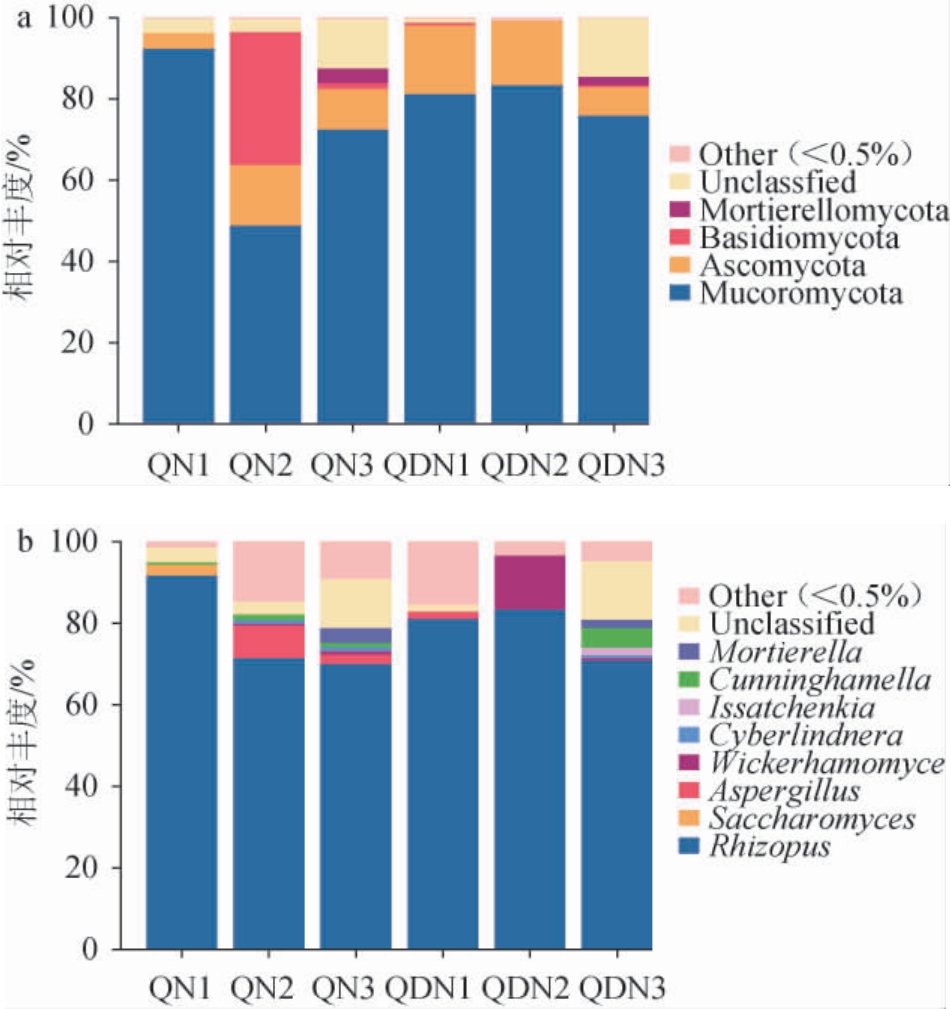
图4 基于门(a)和属(b)水平不同米酒曲样品的真菌群落结构
Fig.4 Fungal community structure of different rice wine Jiuqu at phylum (a) and genus (b) level
由图4b可知,在属水平上,黔南州、黔东南州6个米酒曲样品中主要的真菌属为根霉属(Rhizopus)、酵母菌属(Saccharomyces)、曲霉属(Aspergillus)、威克汉姆酵母属(Wickerhamomyce)、塞伯林德纳氏酵母属(Cyberlindnera)、伊萨酵母属(Issatchenkia)、刺孢小克银汉霉属(Cunninghamella)、被孢霉属(Mortierella)。所有米酒曲样品的共有优势真菌属(相对丰度>1%)为根霉属,相对丰度均>70%,最高可达91.82%(样品QN1),这也是南宁地区、湖北孝感地区和海南山兰米酒曲的优势真菌[11,30],根霉属具有较强的糖化力,可将淀粉转化为可发酵性糖,增加米酒的甜度,还可以将糖转化为乙醇,以及产生蛋白酶、酒化酶、脂肪酶等酶系[35],进而促进柠檬酸、乳酸和琥珀酸等有机酸的生成,其在发酵过程中的相对丰度与发酵体系的酸度、还原糖含量、乳酸含量呈正相关[36],因此,可以通过改变发酵过程中的pH值、还原糖等条件实现该菌属的定向代谢调控。样品QN1的特有优势真菌属仅有酵母菌属(2.59%),酵母菌属是产酒功能菌株,为酯类化合物的生成提供前体物质,在酿造过程中具有较强的发酵能力[37];样品QN3的特有优势真菌属是塞伯林德纳氏酵母属(1.01%),样品QDN2的特有优势真菌属为威克汉姆酵母属(13.22%),非酿酒酵母属(如威克汉姆酵母属)伴随发酵过程可以产生多种酶(糖苷酶、蛋白酶、酯酶),以及其他风味化合物(酸类、高级醇、醛类、酮类、酯类和萜烯类化合物)[38-39],威克汉姆酵母属是红曲糯米酒发酵最后阶段的重要微生物,是主要挥发性化合物的核心功能群,但该属在发酵过程中因为自身氧化和弱发酵代谢,使其对发酵速率、乙醇含量和温度的升高较敏感,耐受性差[40]。样品QDN3的特有优势真菌属是伊萨酵母属(1.84%)。
2.3 贵州特色米酒产区米酒曲中差异菌群分析
LEfSe方法可以探究分组间差异菌群及其丰度,通常结合非参数检验和线性判别分析(linear discriminant analysis,LDA)进行分析[41]。进一步对贵州特色米酒产区米酒曲样品的细菌菌群和真菌菌群进行LEfSe分析,结果分别见图5和图6。物种分支进化图从内至外表示界、门、纲、目、科、属、种的分类级别,黄色节点表示在两组中无显著差异的物种,蓝色和红色的节点分别表示黔南州、黔东南州酒曲中丰度相对较高且具有显著差异的物种,节点大小与相对丰度呈正比;柱状图的长度代表相应物种对组间差异的贡献大小,其长度越长,对组间差异的贡献越大。
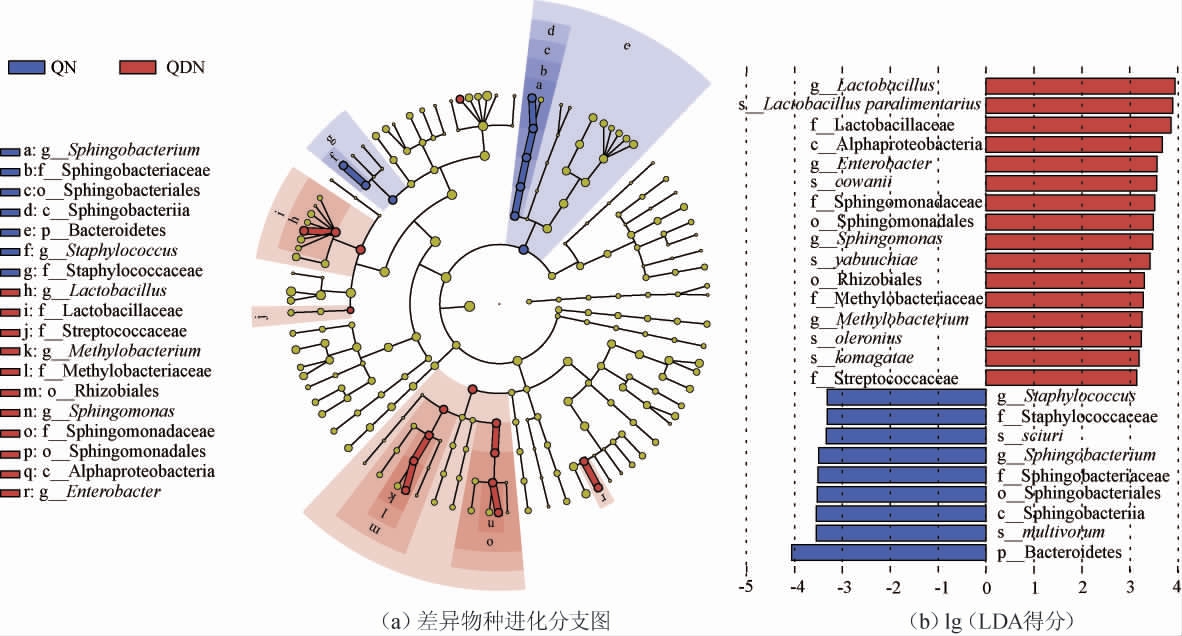
图5 不同米酒曲样品细菌群落性判别分析效应大小分析结果
Fig.5 Linear discriminant analysis effect size analysis results of bacterial community of different rice wine Jiuqu
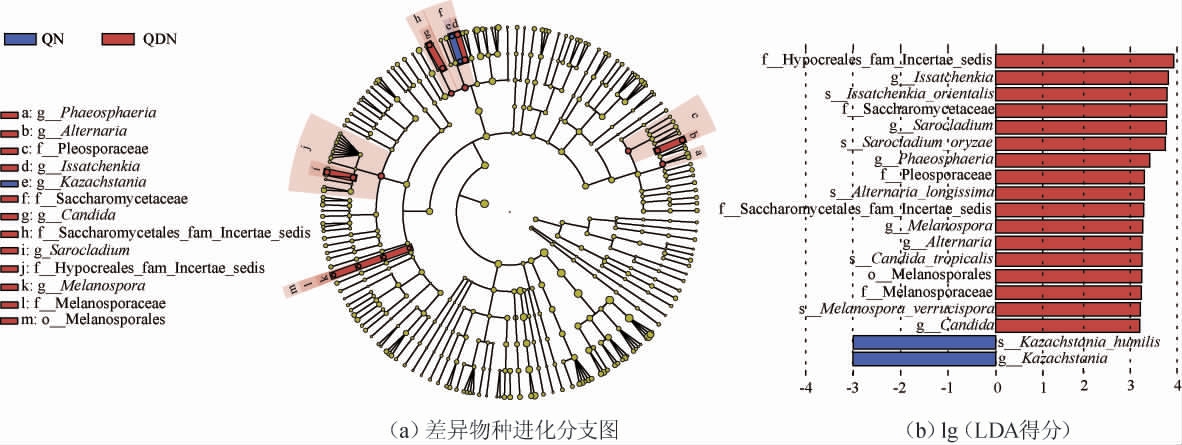
图6 不同米酒曲样品真菌群落线性判别分析效应大小分析结果
Fig.6 Linear discriminant analysis effect size analysis results of fungal community of different rice wine Jiuqu
由图5可知,从所有米酒曲样品中共检测到1门、2纲、3目、6科、6属、7种的差异细菌类群(LDA值>3,P<0.05),隶属于拟杆菌门、厚壁菌门和变形菌门。在门或属水平上,黔南州米酒曲样品中,拟杆菌门是对分组差异贡献最大的菌群(LDA值4.06,P<0.05),葡萄球菌属作为6个米酒曲的共有优势细菌属,也富集在该地区米酒曲的差异细菌类群中,这是由于其在该地区3个米酒曲样本中的相对丰度较高所致;而在黔东南州米酒曲样品中,乳杆菌属是对分组差异贡献最大的菌(LDA值3.94,P<0.05)。
由图6可知,从所有米酒曲样品中共检测到1目、5科、7属、6种的差异真菌类群(LAD值>3,P<0.05),均隶属于子囊菌门。在门或属水平上,黔南州米酒曲样品中哈萨克斯坦酵母属的差异贡献最大(LDA值3.00,P<0.05);黔东南州米酒曲样品中,伊萨酵母属(LDA值3.64,P<0.05)对分组间的差异贡献最大(LDA值3.75,P<0.05)。
由此可见,在门或属水平上,拟杆菌门、哈萨克斯坦酵母属、乳杆菌属和伊萨酵母属是引起黔南州和黔东南州米酒曲样品细菌和真菌类群差异最主要的菌群。
3 结论
采用Illumina NovaSeq高通量测序技术对贵州特色米酒产区6个米酒曲的微生物菌群多样性进行分析,结果表明,从6个米酒曲样品中共注释到18个细菌门和21个细菌属,其中,共有优势细菌门是厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes),共有优势细菌属为葡萄球菌属(Staphylococcus)。从6个米酒曲样品中共注释到14个真菌门和22个真菌属,共有优势细菌门为毛霉菌门(Mucoromycota)和子囊菌门(Ascomycota),共有优势细菌属为根霉属(Rhizopus)。通过LEfSe分析对比黔南州米酒曲与黔东南州米酒曲的差异微生物,结果发现,引起黔南州、黔东南州6个米酒曲存在差异的微生物是隶属于拟杆菌门、厚壁菌门、变形菌门的差异细菌类群(包含1门、2纲、3目、6科、6属、7种),以及隶属于子囊菌门的差异真菌类群(包含1目、5科、7属、6种),黔南州米酒曲样品中对分组间差异贡献最大的是拟杆菌门和哈萨克斯坦酵母属(Kazachstania),黔东南州米酒曲样品中对分组间差异贡献最大的是乳杆菌属(Lactobacillus)和伊萨酵母属(Issatchenkia)。该研究结果将为筛选酒曲中的共性或者存在差异的功能微生物并制作强化酒曲,以及发酵工艺的控制提供参考,对促进贵州特色米酒产业持续健康发展具有重要意义。
[1]张洋洋,张伟国,徐建中,等.米酒液态发酵过程中理化指标和风味物质变化规律[J].东北农业大学学报,2020,51(12):79-89.
[2]贤娟.苗族传统饮食文化族内与族际传播模式研究——以贵州苗族为例[J].中国民族博览,2018(2):1-3.
[3]杨梅,邓彦睿,宋涵冰,等.贵州少数民族酒歌在饮食文化中的功能与价值研究[J].牡丹,2023(20):120-122.
[4]王雨.米酒风味的嗅觉-味觉跨模态表征方法研究[D].镇江:江苏大学,2022.
[5]温承坤,陈孝,王奕芳,等.米酒功能性成分研究进展[J].中国酿造,2019,38(12):5-8.
[6]苏钰亭,赵思明.甜米酒加工技术与品质特征研究进展[J].中国酿造,2014,33(4):27-30.
[7]陈欲云,边名鸿,杨跃寰.米酒对小鼠免疫功能的影响[J].中国酿造,2013,32(7):53-54.
[8]相飞.甜酒曲中微生物群落结构及辣蓼甜酒曲的制曲工艺研究[D].上海:上海海洋大学,2015.
[9]王文平,熊英梅,陈赛浙,等.不同地区米酒曲细菌多样性解析及表型预测[J].食品工业科技,2021,42(20):134-139.
[10]宁亚丽,吴跃,何嫱,等.基于高通量测序技术分析朝鲜族传统米酒及其酒曲中微生物群落多样性[J].食品科学,2019,40(16):107-114.
[11]杨春敏,黄建初,查丽艳.海南山兰米酒酒曲微生物多样性分析[J].食品工程,2019(4):44-48.
[12]龚小会,张东亚,谢玲,等.米酒微生物与其风味品质形成关系研究进展[J].食品科学,2024,45(16):358-366.
[13]YUAN B C, YU D G, HU A, et al.Effects of green manure intercropping on soil nutrient content and bacterial community structure in litchi orchards in China[J].Front Environ Sci,2023,10:1059800.
[14]CALLAHAN B J, MCMURDIE P J, ROSEN M J, et al.DADA2:High-resolution sample inference from Illumina amplicon data[J]. Nat Methods,2016,13(7):581-583.
[15]BOKULICH N A, KAEHLER B D, RIDEOUT J R, et al.Optimizing taxonomic classification of marker-gene amplicon sequences with QIIME 2's q2-feature-classifier plugin[J].Microbiome,2018,6(1):90-107.
[16]WANG Y,SHENG H F,HE Y,et al.Comparison of the levels of bacterial diversity in freshwater,intertidal wetland,and marine sediments by using millions of illumina tags[J]. Appl Environ Microbiol, 2012, 78(23):8264-8271.
[17]The Human Microbiome Project Consortium.Structure,function and diversity of the healthy human microbiome[J]. Nature, 2012, 486(7402):207-214.
[18]WU X Y,JING R X,CHEN W H,et al.High-throughput sequencing of the microbial diversity of roasted-sesame-like flavored Daqu with different characteristics[J].3 Biotech,2020,10(11):502-513.
[19]李静心,王艳丽,何宏魁,等.基于高通量测序技术解析高温大曲和中高温大曲的真菌群落结构[J].食品与发酵工业,2018,44(12):52-59.
[20]武俊男,刘昱辛,周雪,等.基于Illumina MiSeq测序平台分析长期不同施肥处理对黑土真菌群落的影响[J].微生物学报,2018,58(9):1658-1671.
[21]向凡舒,折米娜,何萌,等.基于DGGE和Illumina MiSeq技术解析恩施地区米酒细菌多样性[J].食品与发酵工业,2019,45(14):41-46.
[22]XIE G F,WANG L,GAO Q K,et al.Microbial community structure in fermentation process of Shaoxing rice wine by Illumina-based metagenomic sequencing[J].J Sci Food Agr,2013,93(12):3121-3125.
[23]YANG S H, TSCHAPLINSKI T J, ENGLE N L, et al.Transcriptomic and metabolomic profiling of Zymomonas mobilis during aerobic and anaerobic fermentations[J].BMC Genomics,2009,10(1):34-50.
[24]姜丽.黑糯米酒发酵过程中微生物多样性及风味品质研究[D].贵阳:贵州大学,2020.
[25]洪家丽,李秋艺,潘雨阳,等.红曲黄酒传统酿造过程挥发性风味组分及微生物菌群多样性分析[J].食品科学,2019,40(12):137-144.
[26]苏钰亭.甜米酒的风味形成及特征研究[D].武汉:华中农业大学,2015.
[27]KANDLER O.Carbohydrate metabolism in lactic acid bacteria[J].Antonie Van Leeuwenhoek,1983,49:209-224.
[28]向凡舒,蔡文超,郭壮,等.大竹米酒滋味品质与细菌类群的关联性分析[J].食品科学,2023,44(18):231-238.
[29]李昊颖.基于组学的传统米酒发酵糖化过程相关酶研究[D].无锡:江南大学,2022.
[30]杨甜甜,杨少勇,侯强川,等.基于高通量测序技术分析比较南宁和孝感地区米酒曲中真菌多样性[J].中国酿造,2021,40(9):110-114.
[31]LIANG Z C,LIN X Z,HE Z G,et al.Comparison of microbial communities and amino acid metabolites in different traditional fermentation starters used during the fermentation of Hong Qu glutinous rice wine[J].Food Res Int,2020,136:109329.
[32]ANUPMA A, TAMANG J P.Diversity of filamentous fungi isolated from some amylase and alcohol-producing starters of India[J]. Front Microbiol,2020,11:905.
[33]王春晓,唐佳代,吴鑫颖,等.酿酒小曲中功能微生物的研究进展[J].食品科学,2019,40(17):309-316.
[34]孙剑秋,刘雯雯,臧威,等.酱香型白酒酒醅中霉菌群落组成与功能酶活性[J].中国食品学报,2013,13(8):239-247.
[35]邓开野,林佩芳,黎尔纳.甜酒曲中产糖化酶根霉菌株紫外诱变选育[J].食品科学,2012,33(7):204-208.
[36]KANG J M,ZHENG X W,YANG X,et al.Contrasting summer versus winter dynamic microbial communities and their environmental driving factors in the solid-state saccharification process of Fuyu-flavor Baijiu[J].Food Res Int,2022,154:111008.
[37]HIRST M B,RICHTER C L.Review of aroma formation through metabolic pathways of Saccharomyces cerevisiae in beverage fermentations[J]. Am J Enol Viticult,2016,67(4):361-370.
[38]WANG X S,DU H,ZHANG Y,et al.Environmental microbiota drives microbial succession and metabolic profiles during Chinese liquor fermentation[J].Appl Environ Microbiol,2018,84(4):1-35.
[39]WANG H Y,XU Y.Microbial succession and metabolite changes during the fermentation of Chinese light aroma-style liquor[J].J I Brewing,2019,125(1):162-170.
[40]HUANG Z R,HONG J L,XU J X,et al.Exploring core functional microbiota responsible for the production of volatile flavor during the traditional brewing of Wuyi Hong Qu glutinous rice wine[J].Food Microbiol,2018,76:487-496.
[41]赵锐环,陈乾睿,谷大海,等.不同贮藏年份禄丰香醋细菌多样性分析[J].食品科学,2024,45(10):118-125.