东方酸瓜是海南地区具有传统特色的瓜果类发酵食品,可直接食用也可作为烹煮菜肴的调味料,在海南发酵食品产业中占有重要地位,深受本地居民和外地游客的喜爱。东方酸瓜以小西瓜、葫芦瓜、哈密瓜等果蔬为原料,直接依赖原料及环境中的微生物进行开放式发酵生产[1]。整个发酵过程都存在丰富的微生物群系,通过微生物代谢产生的酶、氨基酸和维生素等健康物质,使东方酸瓜具有促进肠道健康,辅助降血脂和提高机体免疫力等作用[2];发酵产生的有机酸、乙醇和细菌素等抑制性代谢产物,可以有效延长东方酸瓜的食品保质期,同时在微生物的作用下,东方酸瓜产生了独特的风味、色泽和口感,极大地提升了其感官质量[3-4]。
传统发酵果蔬食品主要依赖细菌、酵母和霉菌等多种微生物“多微共酵”的发酵方式生产,参与发酵的微生物菌群通过动态演替共同发挥作用,发酵果蔬食品中乳杆菌(Lactobacillus)、乳球菌(Lactococcus)、链球菌等细菌的种类、含量以及其产生的酶等物质可以改变发酵果蔬食品的色泽和质量,乳酸积累可使发酵环境中pH降低从而抑制腐败菌的生长,对产品质量的提高起到重要作用[5-7]。发酵果蔬食品中酵母类真菌产生的酸碱、代谢糖类物质产生的芳香醇类和芳香醛类物质,也可使发酵果蔬呈现一定的特殊风味。BUYTAERS F E等[8]在传统发酵酒曲和豆豉类食品中分离得到了西弗汉逊氏酵母(Hansenula severus)、异常汉逊氏酵母(Hansenula anomala)等10多种真菌类微生物,并且根据风味成分对其进行相关性分析,结果发现,微生物可以加速蛋白质的降解,加速风味物质的形成,抑制有害菌生长繁殖。WU R N等[9]在传统酸菜中发现汉逊德巴利酵母(Debaryomyces hansenii)能将葡萄糖转化为D-阿拉伯糖醇和乙酸乙酯,从而影响发酵果蔬食品的风味。此外,高玉荣等[10]研究发现,使用鲁氏毛霉(Mucor roxianus)发酵得到的豆豉不仅具有自身特有的香气,而且具有浓郁的醇香、酱香和酯香。除影响发酵蔬菜食品的风味外,某些真菌的存在也是造成发酵蔬菜腐败变质的主要原因。LIU L等[11]研究发现,毕赤酵母(Pichia)在发酵后期迅速生长,是造成发酵泡菜腐败的主要微生物。吴锦兰等[12]在对袋装酸笋的研究中发现,枝状枝孢霉(Cladosporium cladosporioides)和烟曲霉(Aspergillus fumigatus)是导致酸笋腐败变质的主要原因。这类真菌的存在不仅造成营养成分的大量流失,也会增加发酵蔬菜食品的食用风险。因此,研究东方酸瓜中真菌群落分布,对稳定东方酸瓜发酵的质量,提高东方酸瓜品质、新菌种资源的开发以及食品安全性评价具有重要意义[13]。
目前,关于发酵食品如发酵果蔬[14-16]、发酵豆食品[17-19]、酸鱼[20]等微生物群落研究大多关注于细菌群落多样性的研究,对发酵果蔬类食品的真菌菌群多样性研究较少。本研究采用高通量测序技术对3种不同原料制作的东方酸瓜真菌菌群多样性进行分析,揭示其真菌群落结构的多样性以及不同原料腌制东方酸瓜样品间的差异性,为进一步研究东方酸瓜真菌菌落结构对其质量、风味以及食用安全性的影响提供理论依据,也为东方酸瓜发酵菌种资源开发和发酵工艺的全面优化奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
选取原料为海南省东方市的未成熟的小西瓜、葫芦瓜和哈密瓜,采集的同一品种要求大小相近,新鲜度、成熟度一致,无机械损伤,置于无菌袋中。
1.1.2 试剂
DNeasy Power Soil Kit脱氧核糖核酸(deoxyribonucleic acid,DNA)抽提试剂盒、琼脂糖(分析纯):北京QIAGEN公司;DNA Marker、DNA聚合酶:大连Takara公司;真菌ITS区引物:上海生工生物科技有限公司。
1.2 仪器与设备
Centrifuge 5804R型高速冷冻离心机、BioPhotometerD30型核酸蛋白分析仪:德国Eppendorf公司;HYC-610型海尔医用冷藏冰箱:青岛海尔特种电器有限公司;MS3Basic型涡旋混匀仪:德国IKA公司;BSA6202S型电子天平(精度0.01 g):赛多利斯科学仪器有限公司;琼脂糖凝胶电泳仪:美国Bio-Rad公司;NovaSeq6000二代测序仪:美国Illumina公司;GeneAmp R 9700 型聚合酶链式反应(polymerase chain reaction,PCR)仪:美国ABI公司。
1.3 方法
1.3.1 东方酸瓜的制备
挑选优质的西瓜、葫芦瓜、哈密瓜,清洗切分后,采用海南东方市本地传统方法进行腌制,加入相同质量的食用盐,装罐压实,放置在25~30 ℃条件下密封腌制发酵4 d,得到东方酸瓜,编号分别为XG、HLG、HMG。
1.3.2 DNA提取及PCR扩增
无菌条件下从每罐东方酸瓜上、中、下3个部位取发酵液共50 mL,重复取样三次。将发酵液在4 ℃条件下10 000 r/min离心5 min,去上清,取沉淀物。采用DNeasy Power Soil Kit DNA抽提试剂盒提取东方酸瓜微生物菌群基因组总DNA,采用BioPhotometerD30核酸蛋白分析仪检测DNA浓度和纯度。
以提取基因组DNA为模板,采用引物ITS1F(5'CTTGGTCATTTAGAGGAAGTAA-3')和ITS2R(5'-GCTGCGTTCTTCATCGATGC-3')进行PCR扩增。PCR扩增体系:5×Fast Pfu Buffer缓冲液4 μL,2.5 mmol/L脱氧核糖核苷三磷酸(deoxyribonucleotide triphosphates,dNTPs)2 μL,上游引物(5 μmol/L)0.8 μL,下游引物(5 μmol/L)0.8 μL,DNA聚合酶0.4 μL,模板DNA 10 ng,双蒸水(ddH2O)补足至20 μL。每个样本3个重复[21]。PCR扩增程序:98 ℃预变性2 min;98 ℃变性15 s,55 ℃退火30 s,72 ℃延伸30 s,循环35次;最后72 ℃延伸5 min。PCR扩增产物经2%琼脂糖凝胶电泳检测,4 ℃保存[22],将每个样本3个重复的PCR产物混合。
1.3.3 高通量测序
将PCR扩增产物送至上海美吉生物医药科技有限公司采用Illumina Nextseq 2000平台进行高通量测序,使用Fastp(version 0.19.6)软件中滑动窗口法对原始测序序列进行质控,使用Flash(version 1.2.11)软件进行拼接。使用QIIME(version 1.9.1)软件去除paired end序列中含有N碱基的序列,得到clean tags序列。使用UCHIME(version 2.4.2)软件去除clean tags中的嵌合体,最终得到高质量tags。
1.3.4 数据处理与分析
采用Uparse(version 7.0.1090)软件对质控得到的优质序列按照97%的相似度进行操作分类单元(operational taxonomic unit,OTU)聚类,并选取每个OTU中丰度最大的序列作为该OTU的代表序列。采用RDP classifier(version 2.13)分类算法将OTU代表序列与Unite真菌ITS数据库进行比对,使用Funguild(version 1.0)软件进行注释。使用QIIME软件计算超1(Chao1)指数、香农(Shannon)指数、辛普森(Simpson)指数和样本覆盖率,进行Alpha多样性分析。通过PERMANOVA算法的主坐标分析(principal coordinate analysis,PCoA)进行Beta多样性分析。通过ANOVA统计算法,计算不同分组间的差异物种。
2 结果与分析
2.1 高通量测序结果分析
通过Illumina Nextseq 2000高通量测序从3种东方酸瓜共9个样品中得到736 396条序列,碱基长度主要分布在115~523 bp,平均碱基长度为227 bp,共划分到432个操作分类单元(OTUs)。XG、HML、HLG样品中所获得的平均有效序列数分别为70 576条、72 078条、82 243条。
稀释曲线是采用对测序序列进行随机抽样的方法,以抽到的序列数与多样性指数做曲线,主要用来判断测序深度是否合理。当曲线趋向平坦时,说明测序数据量合理,反之则表明继续测序还可能产生较多新的OTU,测序深度不够[23]。因此,由各抽取的序列数与OTU数构建稀释曲线,结果见图1。
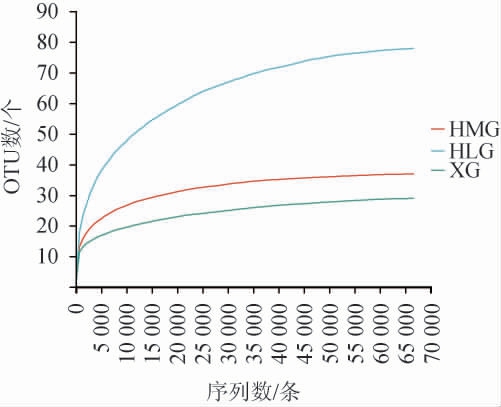
图1 不同原料腌制东方酸瓜样品中真菌菌群稀释曲线
Fig.1 Dilution curves of fungal community in Dongfang pickled melon samples with different raw materials
由图1可知,东方酸瓜样品中的OTU数量随序列数的增加而逐渐增加,最终趋于平缓,达到平台期,结果表明本次分析的测序数量合理,能够较为全面地反应样品的真菌菌群多样性情况,满足实验需求。
Venn图用于统计多个样本中所共有和独有的OTU数目,可直观地表现样本的OTU数目组成相关性及重叠情况[24]。为了解不同原料腌制东方酸瓜的OTU数目,基于真菌OTUs绘制Venn图,结果见图2。由图2可知,3种东方酸瓜样品拥有共同的真菌OTU平均数为22个,HLG样品的真菌OTU平均数为71个,XG样品的真菌OTU平均数为73个,HMG样品的真菌OTU平均数为66个。结果表明,3种不同原料腌制的东方酸瓜存在一定的相似性,而XG样品的真菌多样性最为丰富,HLG次之,HMG样品的真菌多样性最差。
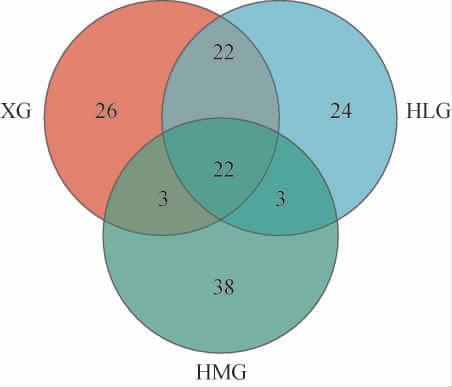
图2 不同原料腌制东方酸瓜样品中真菌OTUs Venn图
Fig.2 OTUs Venn diagram of fungal community in Dongfang pickled melon samples with different raw materials
2.2 不同原料腌制东方酸瓜样品的α-多样性分析
α-多样性是对一个特定区域或者生态系统内评价微生物群落丰富性和多样性的指标[25]。Coverage指数反映群落覆盖度,其数值越高,则样本中序列被测出的概率越高;Chao1指数,反映样品群落的丰富度,其数值越高说明群落物种的丰富度越高;Shannon指数和Simpson指数则反映样品群落的多样性,其中Shannon指数包括物种丰富度和物种在各种间个体分配的均匀度,当物种的丰富度和均匀度都高时,Shannon指数值才最大,而Simpson指数却恰恰相反,其数值越高,说明群落多样性越低[26]。不同原料腌制东方酸瓜样品真菌菌群的α-多样性分析结果见表1。
表1 不同原料腌制东方酸瓜样品真菌群落α-多样性分析结果
Table 1 α-diversity analysis results of fungal community in Dongfang pickled melons with different raw materials
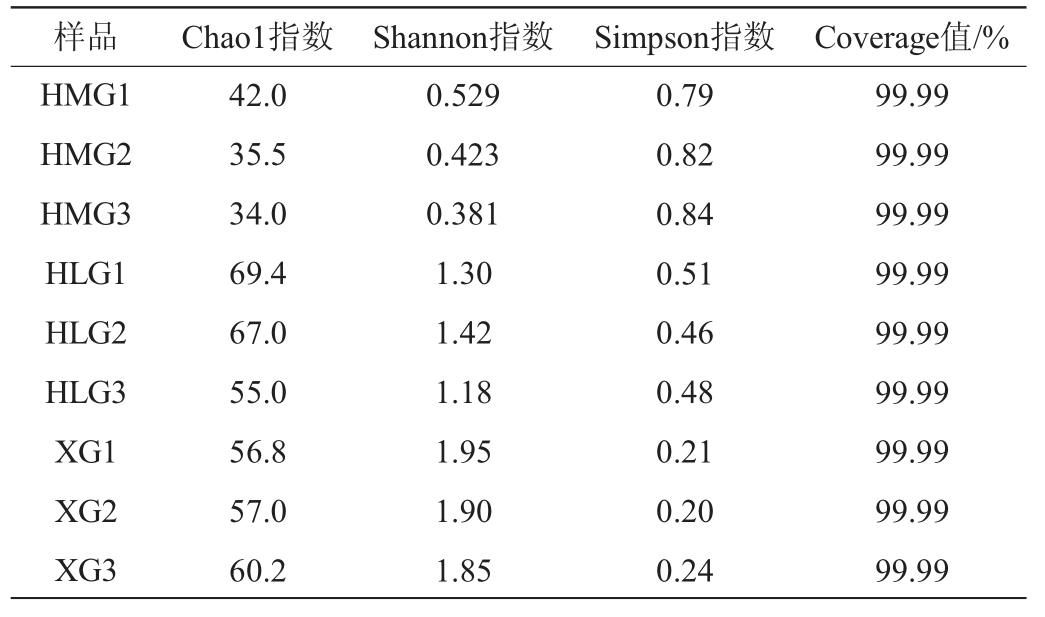
样品 Chao1指数 Shannon指数 Simpson指数 Coverage值/%HMG1 HMG2 HMG3 HLG1 HLG2 HLG3 XG1 XG2 XG3 42.0 35.5 34.0 69.4 67.0 55.0 56.8 57.0 60.2 0.529 0.423 0.381 1.30 1.42 1.18 1.95 1.90 1.85 0.79 0.82 0.84 0.51 0.46 0.48 0.21 0.20 0.24 99.99 99.99 99.99 99.99 99.99 99.99 99.99 99.99 99.99
由表1可知,不同原料腌制的东方酸瓜样品的Coverage值均>99%,表明样本的测序结果能真实反映样本中真菌菌群情况,可以对数据进行后续分析。HLG样品的Chao1指数最大,HMG样品的Chao1指数最小,说明这3种原料腌制的东方酸瓜中,以HLG样品中真菌群落最丰富,以HMG样品则最少。XG样品的Shannon最大,其次为HLG样品,HMG样品最小;Simpson指数的数值与之相反,说明这3种原料腌制的东方酸瓜样品中,以XG样品中真菌群落多样性最高,HLG样品中最低。
丰度等级曲线可以用来展示样品的物种丰富度和均匀度,曲线在横轴上跨越的范围越大,物种丰富度越高;样本曲线的延伸终点位置为该样本的物种数量,如果曲线越平滑下降,表明样本的物种多样性越高,而曲线快速陡然下降表明样本中的优势菌群所占的比例高,多样性较低[27]。为了反映3种不同东方酸瓜样品的物种丰富程度和均匀程度,绘制不同东方酸瓜样品的丰度等级曲线见图3。由图3可知,HLG样品的物种丰富度最大,物种组成的均匀度也较高,这与α-多样性分析结果一致。
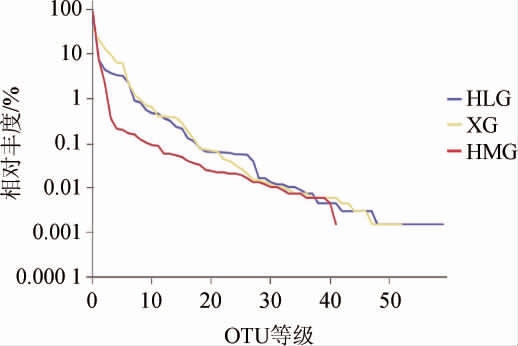
图3 不同原料腌制东方酸瓜样品真菌群落的丰度等级曲线
Fig.3 Rank-abundance curves of fungal community in Dongfang pickled melon samples with different raw materials
2.3 不同原料腌制东方酸瓜样品的真菌群落组成
从不同原料腌制东方酸瓜样品中共注释到的真菌隶属于3门、14纲、26目、47科、68属。基于门和属水平对不同原料腌制东方酸瓜样品中的真菌群落结构进行分析,结果见图4。
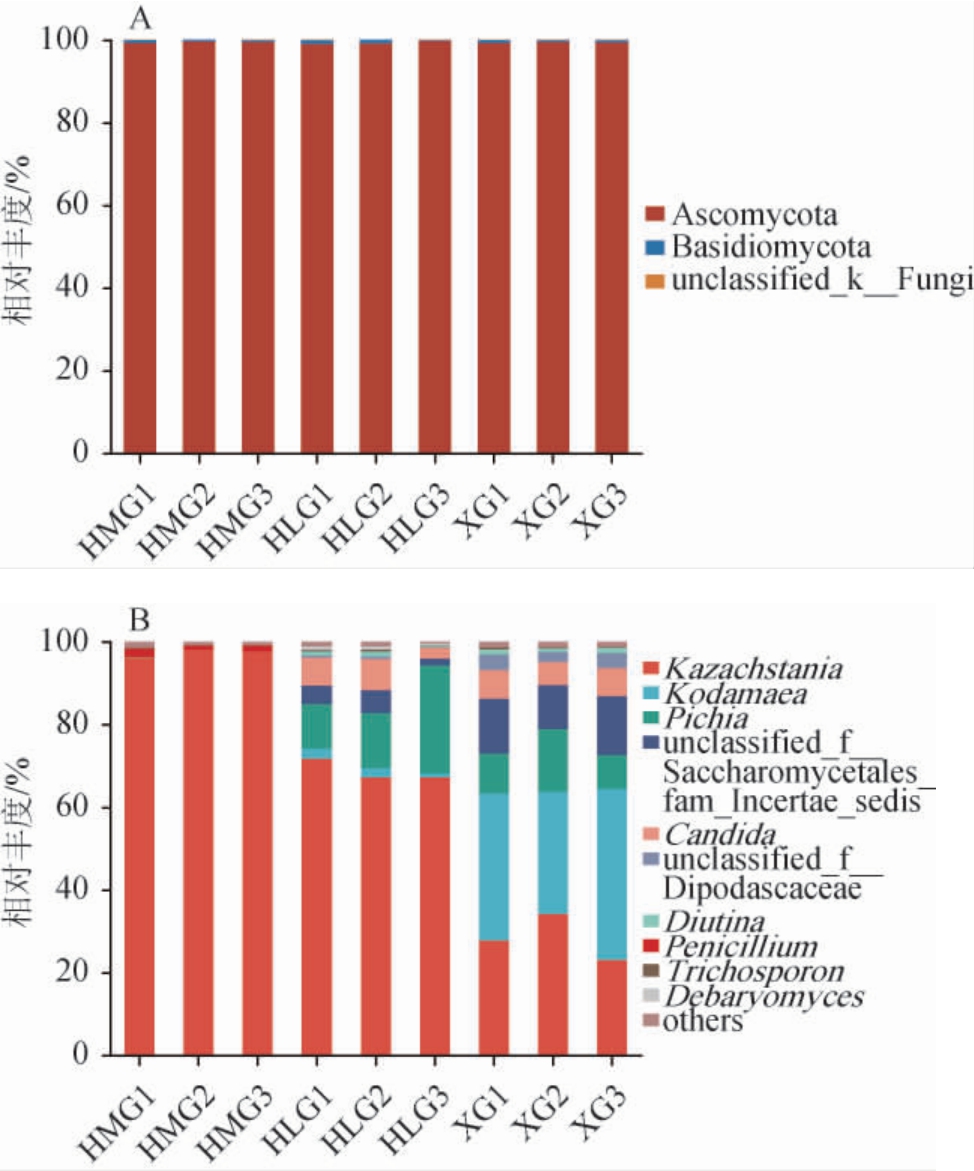
图4 基于门(A)和属(B)水平不同原料腌制东方酸瓜样品中真菌菌群结构的比较
Fig.4 Comparison of fungal community structure in Dongfang pickled melon samples with different raw materials based on phylum (A)and genus (B) levels
others:平均相对丰度排名10之后的其他真菌属。
由图4A可知,从3种东方酸瓜样品中共注释到3个真菌门,分别为子囊菌门(Ascomycota)、担子菌门(Basidiomycota)和unclassified_k_Fungi,不同原料腌制东方酸瓜样品的真菌菌群结构相似,优势真菌门(相对丰度>1%)均为子囊菌门(Ascomycota),相对丰度均>90%,担子菌门的相对丰度仅为0.2%~0.8%。这与唐丽等[28-29]在发酵萝卜、大头菜等发酵果蔬中的研究结果一致。
由图4B可知,从3种东方酸瓜样品中共注释到68个真菌属,主要包括柯达酵母属(Kodamaea)、哈萨克斯坦酵母属(Kazachstania)、毕赤酵母属(Pichia)、unclassified_f_Saccharomycetales_fam_Incertae_sedis、假丝酵母属(Candida)、Diutina、青霉属(Penicillium)、毛孢子菌属(Trichosporon)、德巴利酵母属(Debaryomyces)、unclassified_o__Saccharomycetales等。不同原料腌制的东方酸瓜样品在属水平上的分布差异较大,XG样品中的优势真菌属为Kodamaea(35.6%)、Kazachstania(27.7%)、unclassified_f_Sac charomycetales_fam_Incertae_sedis(13.2%)和Pichia(9.6%)、Candida(6.98%)、unclassified_f__Dipodascaceae(3.74%)。HLG样品中优势真菌属为Kazachstania(71.76%)、Pichia(10.80%)、Candida(6.86%)、unclassified_f_Saccharomycetales_fam_Incertae_sedis(4.42%)、Kodamaea(2.41%)。HMG样品中的优势真菌属为Kazachstania(95.9%)、Penicillium(2.19%)。
在3种不同原料腌制的东方酸瓜中存在较多的是Pichia、Kazachstania、Kodamaea和Debaryomyces。Pichia和Debaryomyces是发酵制品中最常见的酵母菌,能够产生β-葡萄糖苷酶活性,促进果蔬中风味和香气化合物等的非挥发性前体分解,释放挥发性化合物,从而改善东方酸瓜的风味品质[30-32]。Kazachstania广泛分布于酸面团[33]、泡菜[34]、青贮饲料[35]等植物乳酸发酵过程,同化乳酸,并水解葡萄糖醛糖苷作为异型发酵乳酸菌的代谢底物,促进其利用果糖产乙酸,Kazachstania turicensis还可通过调节肠道皮肤轴改善特应性皮炎[36],呈现特殊益生作用,具有潜在商业应用前景。奥默柯达酵母(Kodamaea ohmeri)常在自然发酵的腌肉、泡菜、奶酪等中分离到,是这些发酵食品的正常菌群[37-38],对产品质量有一定贡献,且具有能够降解鱼露中组胺、酪胺的特性[39]。虽然在东方酸瓜自然发酵过程中,有益微生物的大量生长在一定程度上能够抑制有害微生物滋生,但是由于东方酸瓜现目前由当地手工作坊自行生产,在原料、制作工艺以及环境卫生等方面存在差异,仍可能存在致病微生物残存的风险。本研究从东方酸瓜样品XG、HLG中均检出Candida,有研究表明,热带假丝酵母(Candida tropicalis)是一种条件致病菌,在食品生产中通常会引起食品腐败变质,能导致血液系统恶性肿瘤患者和免疫功能低下个体的感染发病率升高,甚至有死亡的风险[40],严重威胁着消费者的健康和东方酸瓜产业的发展。除以上已知真菌菌属外,东方酸瓜中还存有一些未知功能的真菌菌落,本研究有助于对不同原料腌制的东方酸瓜真菌多样性的认知,后续可在此基础上进一步分析这些未知真菌的具体代谢途径和生理生化反应来探究其对发酵东方酸瓜的风味和品质的影响,可为筛选发酵东方酸瓜优良酵母菌提供指导意义。
2.4 不同原料腌制东方酸瓜样品真菌菌群的Beta多样性分析
为进一步揭示不同原料腌制东方酸瓜样品真菌菌群相似度,采用nuifrac距离,基于真菌OTUs对不同原料腌制东方酸瓜样品进行主坐标分析,结果见图5。由图5可知,PCoA 1的方差贡献率为62.09%,PCoA 2的方差贡献率为34.79%,两个主坐标成分的累计方差贡献率为96.88%,说明结果包含了96.88%的原始信息,结果可信。不同原料腌制东方酸瓜样品分布在不同的象限,说明3种原料腌制的东方酸瓜样品真菌菌群组成在OTU水平上存在一定的差异性。
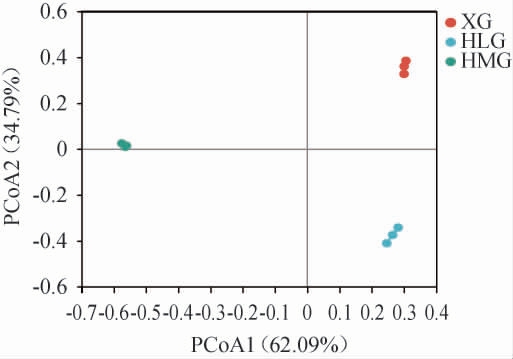
图5 基于OTU水平不同原料腌制东方酸瓜样品的主坐标分析结果
Fig.5 Principal coordinate analysis results of Dongfang pickled melon samples with different raw materials based on OTU level
2.5 不同原料腌制东方酸瓜样品真菌菌群差异性分析
为了更加直观的了解3种东方酸瓜样品真菌菌群的差异,利用单因素方差分析对3种不同原料腌制东方酸瓜间真菌属的相对丰度进行差异显著性检验分析,结果见图6。由图6可知,在属水平上,在3种不同原料腌制东方酸瓜样品中,Kazachstania的相对丰度差异极显著(P<0.001),Debaryomyces、Kodamaea、Candida、unclassified_f_Saccharomycetales_fam_Incertae_sedis的相对丰度均存在较显著差异(P<0.01),Pichia、unclassified_f_Dipodascaceae、接合囊酵母属(Zygoascus)、枝孢属(Cladosporium)、Cutaneotrichosporon的相对丰度均存在显著差异(P<0.05)。自然发酵的东方酸瓜微生物群落通常与生产工艺、发酵环境、发酵时间、原料有较大的关系,由于本次研究的东方酸瓜都是由同一人员使用相同的制作方法在统一的环境下发酵相同时间腌制而成,因此造成此次真菌菌群差异的原因可能是哈密瓜、葫芦瓜、西瓜自身所含有的蛋白质、糖分、脂肪、纤维素、碳水化合物以及水分的组成不同导致。
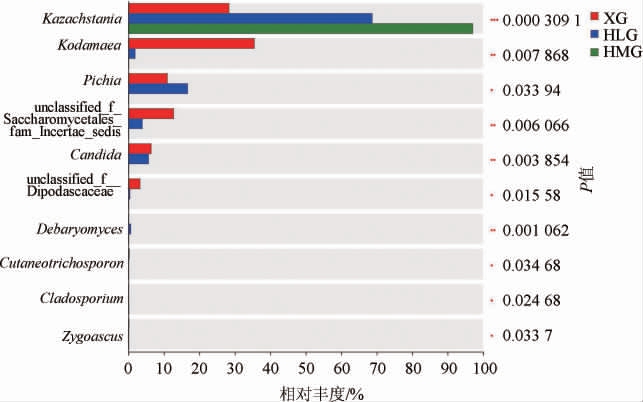
图6 基于属水平不同原料腌制东方酸瓜样品的细菌菌群差异分析
Fig.6 Analysis of differences in bacterial community of Dongfang pickled melon samples with different raw materials based on genus level
“***”表示差异极显著(P<0.001),“**”表示差异较显著(P<0.01),“*”表示差异显著(P<0.05)。
3 结论
本研究以3种不同原料(西瓜、葫芦瓜、哈密瓜)腌制的东方酸瓜样品(XG、HLG、HMG)为研究对象,采用高通量测序技术解析其真菌群落结构,结果表明,HLG样品真菌群落丰富度最高,XG样品真菌群落多样性最高。在门水平上,3种东方酸瓜样品的真菌菌群结构相似,均以Ascomycota为优势菌门(相对丰度>1%);在属水平,真菌菌群结构差异较大,HLG和HMG样品中Kazachstania的相对丰度最高,分别为71.76%、95.9%,XG样品则以Kodamaea的相对丰度(35.6%)最高,其次为Kazachstania(27.7%)。Pichia、Candida、Kodamaea主要存在于HLG和XG样品中,Penicillium仅存在于HMG样品中。在属水平上菌群差异性分析结果 显 示,Kazachstania、Debaryomyces、Kodamaea、Candida等10个真菌属在3种不同原料腌制东方酸瓜样品中都存在显著差异(P<0.05)。在后续研究中,可通过解析东方酸瓜发酵过程中微生物群落的变化及筛选优势菌株来确保东方酸瓜生产的稳定性,还可进一步对东方酸瓜中风味物质进行研究,了解微生物多样性与风味物质的关系,以此来提升东方酸瓜品质,加快工业化生产进程。
[1]任聪,杜海,徐岩.中国传统发酵食品微生物组研究进展[J].微生物学报,2017,57(6):885-898.
[2]燕平梅,魏爱丽.蔬菜半固态发酵的探究[J].中国调味品,2020,45(11):34-37,50.
[3]WASTYK H C,FRAGIADAKIS G K,PERELMAN D,et al.Gut-microbiota-targeted diets modulate human immune status[J]. Cell, 2021, 184(16):4137-4153.
[4]姚粟,王鹏辉,白飞荣,等.中国传统发酵食品用微生物菌种名单研究(第二版)[J].食品与发酵工业,2022,48(1):272-285.
[5]毛丙永,殷瑞敏,赵楠,等.四川老卤泡菜基本理化指标及特征菌群分离鉴定[J].食品与发酵工业,2018,44(11):22-27.
[6]何鹏晖,厍晓,钱杨,等.发酵蔬菜中腐败微生物及其防控的研究进展[J].食品工业科技,2017,38(11):374-378.
[7]叶陵,李勇,王蓉蓉,等.我国传统发酵蔬菜中乳酸菌群落结构多样性的研究进展[J].食品科学,2018,39(15):306-311.
[8]BUYTAERS F E,FRAITURE M A,BERBERS B,et al.A shotgun metagenomics approach to detect and characterize unauthorized genetically modified microorganisms in microbial fermentation products[J]. Food Chem: Mol Sci,2021,2:100023.
[9]WU R N,YU M L,LIU X Y,et al.Changes in flavour and microbial diversity during natural fermentation of suan-cai,a traditional food made in Northeast China[J].Int J Food Microbiol,2015,211:23-31.
[10]高玉荣,刘晓燕.多菌种低盐分段发酵生产豆豉工艺[J].农业工程学报,2010,26(S1):369-373.
[11]LIU L,SHE X,QIAN Y,et al.Effect of different fermenting containers on the deterioration of Sichuan pickle[J].Food Sci Technol,2019,111:829-836.
[12]吴锦兰,李静怡,白云,等.袋装酸笋中优势腐败霉菌的分离与鉴定[J].农产品加工,2023(6):70-72,76.
[13]姚尚杰,金垚,周荣清,等.传统发酵食品中微生物间相互作用及应用[J].生物产业技术,2019,7(4):48-50.
[14]LIANG H P, CHEN H Y, ZHANG W X, et al.Investigation on microbial diversity of industrial Zhacai paocai during fermentation using highthroughput sequencing and their functional characterization[J]. LWTFood Sci Technol,2018,91:460-466.
[15]石晶红.红腌菜发酵过程中细菌多样性动态分析[J].食品工业科技,2022,43(22):173-180.
[16]鲍伟,韩姣姣,张旨轩,等.基于高通量测序技术对浙江传统发酵蔬菜微生物多样性的解析[J].食品科学,2021,42(6):178-185.
[17]李薇,罗沈斌,邱泽瑞,等.永川毛霉型豆豉传统发酵过程中微生物群落结构及动态演替规律[J].食品与发酵工业,2020,46(23):60-67.
[18]CHEN Y H, LIU X W, HUANG J L, et al.Microbial diversity and chemical analysis of Shuidouchi,traditional Chinese fermented soybean[J].Food Res Int,2019,116:1289-1297.
[19]XU D D,WANG P,ZHANG X,et al.High-throughput sequencing approach to characterize dynamic changes of the fungal and bacterial communities during the production of sufu,a traditional Chinese fermented soybean food[J].Food Microbiol,2020,86:103340.
[20]孙海鑫,陈力,石文琪,等.自然发酵酸鱼中微生物群落多样性与理化性质分析[J].食品研究与开发,2023,44(12):187-195.
[21]邹妍,陈中爱,董楠,等.贵州腌鱼中微生物群落的多样性研究[J].食品工业科技,2021,42(5):119-125.
[22]MUKHERJEE P K, CHANDRA J, RETUERTO M, et al.Oral mycobiome analysis of HIV-infected patients: identification of Pichia as an antagonist of opportunistic fungi[J].PLoS Pathog,2014,10:e1003996.
[23]郭倩倩,卢彪.晴隆酸菜发酵过程中微生物菌群多样性动态分析[J].中国酿造,2020,39(8):86-91.
[24]赵德庆,于平,乔飞.基于高通量测序分析海南粗榧树皮和叶片中微生物多样性[J].山东农业科学,2021,53(8):54-59.
[25]于佳琦,夏亚男,乔晓宏,等.锡林郭勒牧区鲜马奶风味物质与微生物多样性分析[J].中国乳品工业,2021,49(5):8-14.
[26]陶康,吴凌伟,金晓芳,等.基于高通量基因测序分析腐乳微生物多样性[J].食品科学,2021,42(8):143-149.
[27]于其可,王赛,王盼,等.不同葡萄表皮真菌菌群的多样性分析[J].中国酿造,2022,41(7):122-127.
[28]唐丽,魏雯丽,赵雅娇,等.工业萝卜泡菜发酵过程中理化特性及真菌群落多样性分析[J].食品与发酵工业,2022,48(6):25-31.
[29]吴进菊,曾瑞萍,张俊毅,等.高通量测序分析大头菜发酵过程中真菌的多样性[J].食品科学,2020,41(10):75-80.
[30]陈怡,刘洋,蒋立文,等.基于高通量测序分析不同浏阳豆豉中真菌的多样性[J].中国酿造,2020,39(11):66-70.
[31]JOLLY N P,VARELA C,PRETORIUS I S.Not your ordinary yeast:non-Saccharomyces yeasts in wine production uncovered[J]. FEMS Yeast Res,2014,14(2):215-237.
[32]王俊钢,李宇辉,刘成江,等.新疆哈萨克族传统风干肉中真菌多样性分析[J].食品与发酵工业,2021,47(1):35-42.
[33]MARILÙ D,MATTIA Q,GIOVANNI R,et al.Evaluation of microbial consortia and chemical changes in spontaneous maize bran fermentation[J].AMB Express,2017,7(1):205.
[34]SUZUKI A, MURAOKA N, NAKAMURA M, et al.Identification of undesirable white-colony-forming yeasts appeared on the surface of Japanese kimchi[J].Biosci Biotech Bioch,2018,82(2):1-9.
[35]SANTOS M C, GOLT C, JOERGER R D, et al.Identification of the major yeasts isolated from high moisture corn and corn silages in the United States using genetic and biochemical methods[J]. J Dairy Sci,2017,100(2):1151-1160.
[36]KIM JH,KIM K,KANJANASUNTREE R,et al.Kazachstania turicensis CAU Y1706 ameliorates atopic dermatitis by regulation of the gutskin axis[J].J Dairy Sci,2019,102(4):2854-2862.
[37]EL-SHAROUD W M,BELLOCH C,PERIS D,et al.Molecular identification of yeasts associated with traditional egyptian dairy products[J].J Food Sci,2009,74(7):M341-M346.
[38]BORELLI B M,FERREIRA E G,LACERDA I C A,et al.Yeast populations associated with the artisanal cheese produced in the region of Serra da Canastra,Brazil[J].World J Microb Biot,2006,22:1115-1119.
[39]王挥,付湘晋,吴伟,等.1株奥默柯达酵母对鱼露中生物胺的降解特性[J].中国食品学报,2014,14(8):137-141.
[40]YU H,LIN Z X,XIANG W L,et al.Antifungal activity and mechanism of D-limonene against foodborne opportunistic pathogen Candida tropicalisl[J].LWT-Food Sci Technol,2022,159:113144.