源头减量、资源化利用和无害化处置是“无废城市”绿色发展理念的内涵所在。工业生产过程中的污水处理关乎城市水体环境和公共卫生,必须满足排放标准方能进入自然水体。物理与生物相结合已成为工业废水处理的方法之一,运行稳定的生物处理工序对水质净化尤为重要。因此,了解生物处理过程中微生物菌群多样性及其活动规律对调控废水处理效果具有重要意义[1]。
明胶作为重要的工业产品多以动物骨、皮为原料制得,生产过程会消耗大量工业用水,每生产1 t明胶平均消耗1 000~1 500 t水,有的甚至高达2 000 t;因此明胶生产会产生大量废水,每处理1 t骨原料约产生300 m3废水[2-3]。明胶废水的总氮(total nitrogen,TN)和氨氮(ammonia nitrogen,NH4+-N)含量分别为140~300 mg/L和70 mg/L左右,大约存在1 000~2 500 mg/L的钙离子[4],4 600~5 600 mg/L的氯离子[5]。高钙、高磷、高碱、高蛋白质是明胶废水的主要特性,如何有效处理、达标排放是明胶生产企业面临的重要问题[6-7]。明胶废水因碱度很高,含有较高浓度的钙、蛋白质和脂肪等有机物[8-9],实际处理中多采用厌氧-好氧两步法[10]。厌氧-好氧处理过程中,厌氧和好氧反应器均开放运行。缺氧条件下,通过兼性菌水解可使大分子难降解有机物分解成较易降解的小分子有机物,非溶解性有机物分解成溶解性有机物,改善了废水的可生化性,为后续好氧处理创造有利条件。在好氧过程中,利用好氧菌的同化和异化作用对有机物进行降解,同时好氧池通过机械搅拌增加气液界面,加强了氧气融入[10-11]。因此,微生物菌群是明胶加工废水的净化主体,生物系统的处理效果和水质变化与微生物群落结构及其功能紧密相关[12]。
近年来,基于宏基因组的高通量测序技术因其通量高、测序周期短、定量准确等优点备受关注,该技术能全面揭示环境中的功能微生物,还能克服传统分离方法获得少量微生物信息的局限性[13],已被广泛用于污泥、废水等环境微生物菌群分析[14-15]。VIEIRA A M S等[16]研究发现,明胶废水中的兼性厌氧菌多属于肠杆菌科(Enterobacteriaceae),如大肠杆菌(Escherichia coli)、弗格森埃希菌(Escherichia fergusonii)等。乳酸菌(Lactobacillus)、芽孢杆菌(Bacillus)和李斯特菌(Listeria)能有效降解利用废水中蛋白质、明胶、蔗糖和乳糖等污染物[17]。丛毛单胞菌(Comamonas)、陶厄氏菌(Thauera)和氢噬胞菌(Hydrogenophaga)是去除废水中油脂成分的重要菌属[18]。淀粉芽孢杆菌(Bacillus amyloliquefaciens)和地衣芽孢杆菌(Bacillus licheniformis)也能提高废水油脂的去除率[19]。然而,有关明胶生产废水处理过程中的微生物群落研究还鲜有报道,有必要对其进行研究。
鉴于此,本研究采用高通量测序技术对明胶加工废水处理工艺中的关键微生物群落结构进行分析,并结合废水水质净化结果构建微生物菌群与水体环境因子之间的关系,探讨关键微生物菌群的功能作用,以期为明胶废水处理工艺的运行调控奠定理论基础。
1 材料与方法
1.1 材料与试剂
明胶加工废水样品:取自甘肃阿敏生物清真明胶有限公司污水处理站的初沉池、好氧池、二沉池和排放口,分别命名为初沉水(water solution,WS)、好氧水(suspended substance,SS)、二沉泥(wastage nanofiltration,WN)和排放水(blowoff standard,BS)。
聚丙烯酰胺:湖北净迪科技有限公司;硫酸(分析纯):青岛海洋化工厂;The Power Soil脱氧核糖核酸(deoxyribonucleic acid,DNA)Isolation Kit:成都福际生物技术有限公司;Buffer缓冲溶液:北京索莱宝科技有限公司;Q5高保真DNA聚合酶:天津益元利康生物科技有限公司;二甲基亚砜:武汉艾美捷科技有限公司;琼脂糖(生物试剂):北京擎科生物科技有限公司;脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP):大连宝生物工程有限公司。
1.2 仪器与设备
UB-7 pH计:北京丹佛仪器有限公司;5B-6C多参数水质测定仪:苏州市莱顿科学仪器有限公司;K9840 半自动凯氏定氮仪:山东海能科学仪器有限公司;BWB-FL多元素火焰光度计:上海磐麦科技有限公司;DYY-11电泳仪:北京市六一仪器厂;MG 96聚合酶链式反应(polymerase chain reaction,PCR)仪:杭州朗基科学仪器有限公司;TG16-W微型台式高速离心机:湖南湘仪离心机仪器有限公司。
1.3 方法
1.3.1 样品采集
2019年3月,在企业污水处理站进行了样品采集,每个取样点采集4个样品(水温<12 ℃),4个取样点供采集16个样品,采样点布置在各处理单元的出水口,样品保存方法参考HJ 493—2009《水质样品的保存和管理技术规定》[20],在0~4 ℃条件下保存运输,48 h内完成水质检测分析。
1.3.2 明胶加工废水的处理工艺流程
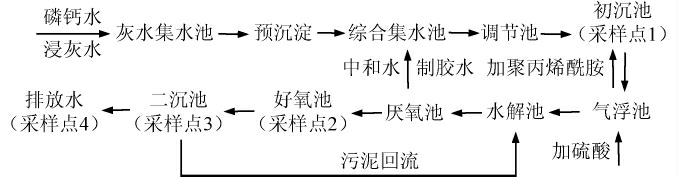
废水注入灰水集水池,经预沉池沉淀部分悬浮物后进入综合集水池,经管道依次流经调节池和初沉池,加入聚丙烯酰胺进行悬浮颗粒的凝聚和澄清,然后注入气浮池,再依次通过水解池和厌氧池,接着注入有泵搅拌的好氧池使有机物降解较为彻底;最后在二沉池中进行再次沉淀后形成排放水,其中部分污泥从二沉池中回流至水解池。
1.3.3 分析检测
废水样中理化指标分析:pH值采用pH计测定;化学需氧量(chemical oxygen demand,COD)和氨氮(NH4+-N):采用COD-氨氮双参数测定仪测定;总氮(TN)采用K9840自动凯氏定氮仪测定;钙离子(Ca2+)采用原子吸收分光光度计测定(测定条件:波长422.7 nm,狭缝宽0.7 nm,电流10 mA,火焰类型 空气-乙炔,燃气流量2.0 L/min,助燃气流量15.0 L/min)[4]。
DNA提取:按照ThePowerSoilDNAIsolationKit说明书提取4个样品中的微生物基因组DNA。提取的DNA用乙醇沉淀法进行提纯。DNA浓度保证OD260nm/OD280nm和OD260nm/OD230 nm的值分别大于1.8和2.0。取2 μL DNA溶液,用1.2%琼脂糖凝胶电泳检测抽提的基因组DNA完整性,然后将符合要求的DNA置于-80 ℃保存备用。
聚合酶链式反应扩增:针对细菌16SrRNA V3~V4区进行PCR扩增(正向引物:5'-ACTCCTACGGGAGGCAGCA-3';反向引物:5'-GGACTACHVGGGTWTCTAAT-3')。所有PCR扩增均在50 μL标准反应体系中进行,含有10 μL Buffer,0.2μLQ5高保真DNA聚合酶,10μL二甲基亚砜,1μLdNTP,10 μm引物和60 ng基因组DNA。PCR扩增条件为:95 ℃预变性5 min,然后在95 ℃变性1 min,50 ℃退火1 min和72 ℃延伸1 min,循环15次,最后在72 ℃延伸7 min。将第一步所得的PCR产物纯化后,于40 μL反应中进行第二轮PCR扩增,包含20 μL 2×Phusion High-Fidelity PCR Master Mix(高保真PCR混合物),8 μL双蒸H2O,10 μm引物和10 μL第一步PCR产物。PCR扩增条件为:98 ℃条件下预变性30 s;然后在98 ℃变性10 s,65 ℃退火30 s和72 ℃延伸30 s,循环10次;在72 ℃下延伸5 min。最后通过Quant-iTTM dsDNA HS试剂定量。
PCR产物分析:扩增产物经1.8%的琼脂糖凝胶电泳(电压120 V,电泳40 min)进行检测,凝胶成像仪观察电泳结果,切胶并回收。通过Illumina Hiseq 2500平台进行细菌16S rRNA基因的高通量分析。
废水样中的微生物菌落分析:微生物多样性是基于Illumina HiSeq测序平台,利用双末端测序(Paired-End)的方法,构建小片段文库进行测序。通过对碱基序列(Reads)拼接过滤,操作分类单元(operationaltaxonomicunits,OTUs)聚类,并进行物种注释及相对丰度分析,可以揭示样品的物种构成;进一步进行Alpha多样性分析,可以挖掘样品之间的差异。
1.3.4 数据分析
采用SPSS 26.0软件对污水水质进行单因素方差分析。运用Excel 2016软件统计细菌群落数据并绘制柱状图。利用mothur(Version 1.33.3)进行Alpha多样性分析,包括物种丰富度统计(Chao1指数、ACE指数)、物种多样性统计(Shannon指数、Simpson指数)、稀释曲线和Venn图绘制。应用R软件绘制主成分分析(principal component analysis,PCA),同时运行相似度分析对环境因素和细菌群落结构进行分析。
2 结果与分析
2.1 废水水质分析以及细菌群落与水质相关性
明胶加工废水4个取样点的水质分析结果如表1所示。在微生物生长的范围内,pH值的变化给微生物代谢活动带来极大影响,包括碳和能源(蛋白质和脂肪)的利用,以及底物降解效率;pH 6.0~7.0时有利于乙酸、丙酸、丁酸和氢气的产生[21],从而使好氧水(pH 6.59)和排放水(pH 6.74)的pH值处于较低状态[10]。由表1可知,二沉泥的各项指标显著高于其他样品(除氨氮外)(P<0.05),这是因为明胶生产过程中使用大量石灰乳,导致废水中pH和Ca2+浓度较高,进入生化系统后与产生的CO2结合生成碳酸钙沉淀,造成污泥中有机成分升高(化学需氧量(chemical oxygen demand,COD)=166.15 mg/L)[22]。排放水中的总氮(total nitrogen,TN)、氨氮(ammoniacal nitrogen,NH4+-N)及化学需氧量显著低于其他样品(P<0.05),这是因为好氧池中的微生物菌群大量繁殖,使得有机物被分解和代谢所致[5]。水质分析结果表明,该废水处理工艺运行稳定,能使排放水达到GB 18918—2002《城镇污水处理厂污染物排放标准》的一级A排放指标。
表1 四个取样点的废水水质分析
Table 1 Analysis on wastewater quality of four samples points
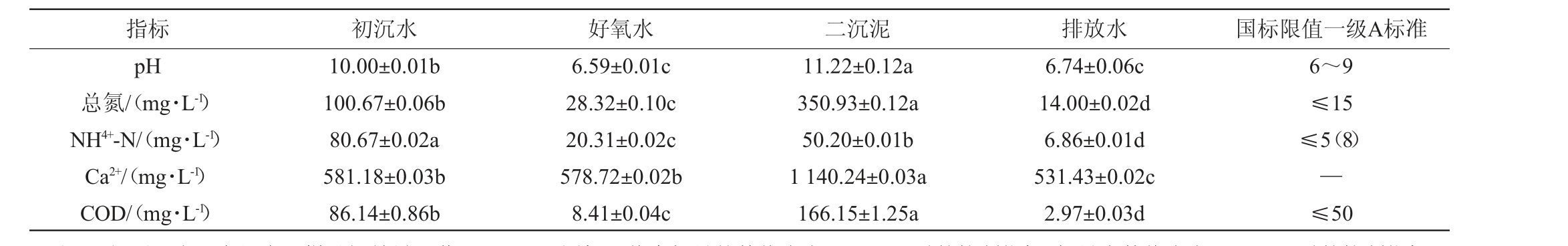
注:同行不同小写字母表示样品间差异显著(P<0.05);国标限值中括号外数值为水温>12 ℃时的控制指标,括号内数值为水温<12 ℃时的控制指标。
指标 初沉水 好氧水 二沉泥 排放水 国标限值一级A标准pH总氮/(mg·L-1)NH4+-N/(mg·L-1)Ca2+/(mg·L-1)COD/(mg·L-1)10.00±0.01b 100.67±0.06b 80.67±0.02a 581.18±0.03b 86.14±0.86b 6.59±0.01c 28.32±0.10c 20.31±0.02c 578.72±0.02b 8.41±0.04c 11.22±0.12a 350.93±0.12a 50.20±0.01b 1 140.24±0.03a 166.15±1.25a 6.74±0.06c 14.00±0.02d 6.86±0.01d 531.43±0.02c 2.97±0.03d 6~9≤15≤5(8)—≤50
细菌群落与水质环境因子的相关性分析结果见图1。由图1可知,嗜碱菌属(Alkalibacterium)与TN浓度呈极显著正相关(P<0.01),与COD浓度呈显著正相关(P<0.05)。这类微生物既能分解污泥,又能适应碱性环境[23],同时嗜碱菌属对高硝态氮、高盐分环境也有较强适应性,在处理高浓度硝态氮废水方面起重要作用[24]。另一方面,莱茵海默氏菌(Rheinheimera)与Ca2+浓度、NH4+-N浓度和pH值呈显著正相关(P<0.05),能产生大量蛋白酶和脂肪酶,很好地降解水体中有机氮物质[25]。陈兆进等[25]在丹江口水库枯水期的水样中也发现莱茵海默氏菌属与NH4+-N含量呈显著正相关(P<0.05),与该文结论一致。另外,Uncultured bacterium c Candidatus Moranbacteria与TN含量呈显著负相关(P<0.05),可能是水体中的脱氮微生物[26],Uncultured bacterium p Saccharibacteria与COD呈显著负相关(P<0.05),能够抑制COD浓度的上升,这与阴泽阳[27]的研究结果一致。
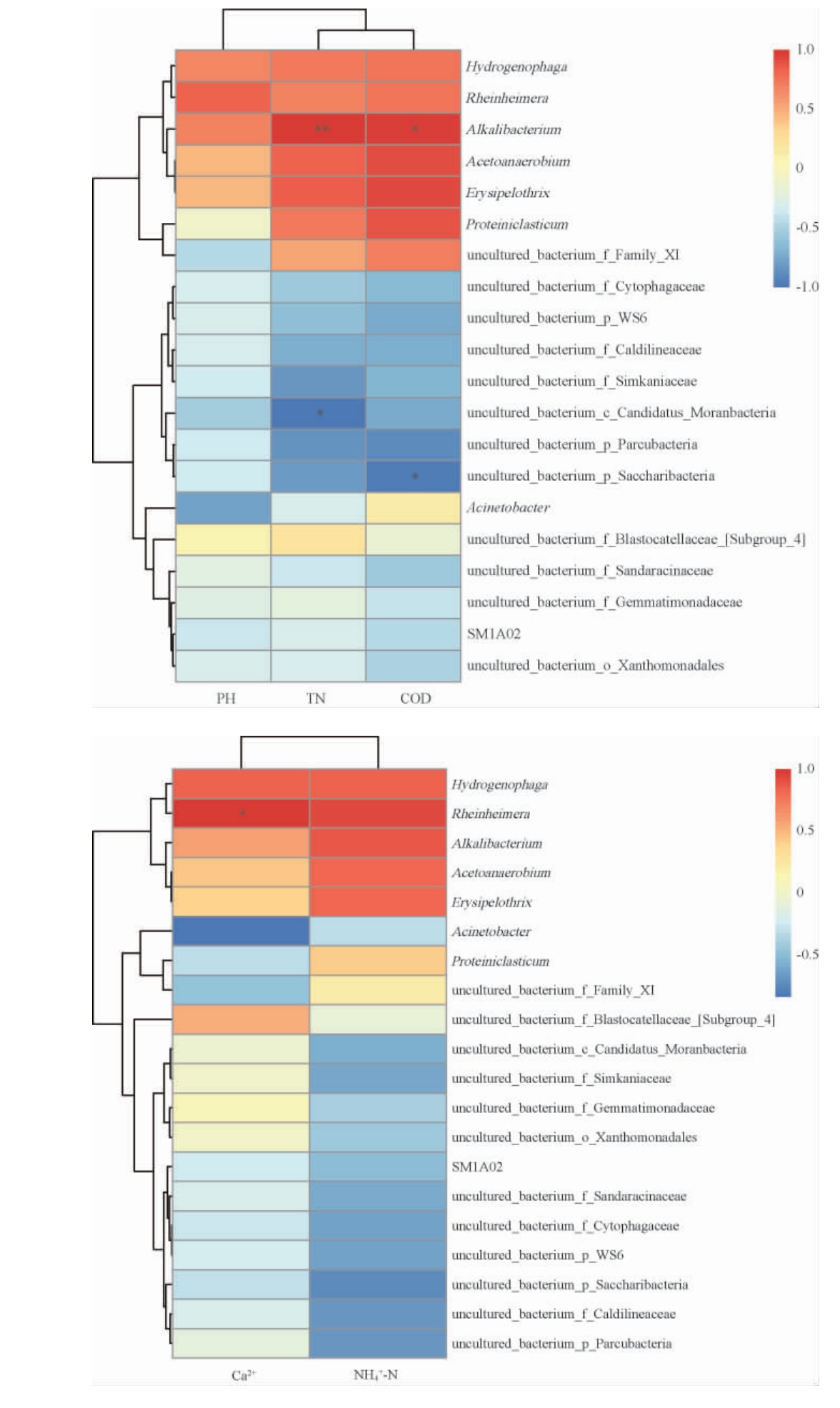
图1 细菌群落与水质环境因子的相关性分析
Fig.1 Correlation analysis between bacterial community and environmental factors of water quality
“*”表示显著相关(P<0.05);“**”表示极显著相关(P<0.01)。
2.2 四个取样点废水的稀释曲线及Venn图分析
四个废水取样点的稀释曲线见图2。由图2可知,二沉泥(WN)、初沉水(WS)和排放水(BS)三个样品的测序条数超过40 000时稀释曲线趋于平缓,好氧水(SS)样品测序条数超过30 000时稀释曲线趋于平缓,说明该测序数据量合理,且随着测序条数的增加几乎不产生新的OTU[28-29]。四个样品的OTU数量大小依次为二沉泥(WN)>排放水(BS)>排放水(BS)>初沉水(WS)。这可能是因为,一方面,在好氧池中好氧菌的同化和异化作用使有机物大部分降解,有利于微生物生长;另一方面,二沉泥经过沉淀营养成分更丰富,微生物生长繁殖适应性强,故OTU数量最高;而初沉水因其处于高pH环境,大部分微生物不适宜生存,导致其OTU数量较低[10]。
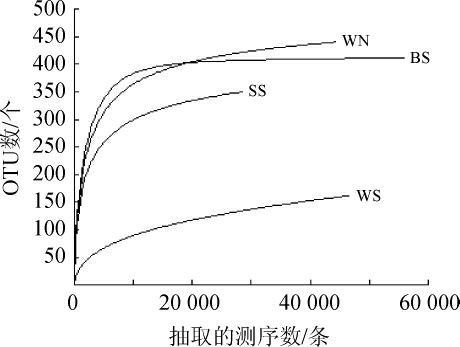
图2 四个取样点废水取样点基于操作分类单元数量的稀释曲线
Fig.2 Dilution curve of wastewater from four sampling points based on operational taxonomic unit quantity
Venn图也能展示样品之间共有和特有的OTU数目,直观表现出样品间OTU的重合情况[30],结果见图3。由图3可知,WS、SS、WN、BS中独有的OTU数目分别是8、2、15、101。其中,以好氧水和排放水两个样本相距最远,差异性最大。初沉水、好氧水、二沉泥和排放水4个样品共有OTU数为74。初沉水和好氧水共有OTU数为93,说明由初沉池到好氧水的过程中,随着有机物的降解,微生物菌群细菌群落结构差异更相似。好氧水和二沉泥共有的OTU数为328,二沉泥和排放水共有OTU数为285,说明好氧水与二沉泥、二沉泥和排放水的微生物细菌群落结构相似性较大,这可能与废水处理工序的衔接性密切相关。初沉水、好氧水和排放水共有75个OTU,说明初沉水、好氧水和排放水的微生物菌落结构相似性较小,因为各废水处理池的微环境给微生物菌群提供的生存条件存在较大差异,从而使得初沉水、好氧水和排放水的菌落结构发生改变,特别是功能菌群的增殖[31]。
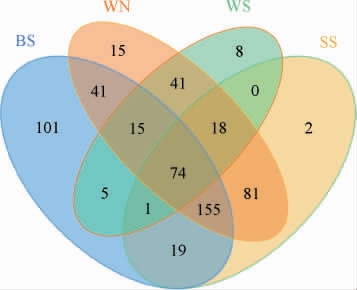
图3 四个取样点废水基于操作分类单元数量的Venn图
Fig.3 Venn diagram of wastewater from four sampling points based on operational taxonomic unit quantity
2.3 四个取样点废水微生物菌群的Alpha多样性分析
Alpha多样性指数是用于评价微生物丰富度和群落多样性的综合指标[28],四个取样点废水的Alpha多样性分析结果见表2。由表2可知,4个样品的覆盖率均在0.99以上,说明测序结果对明胶加工废水样品的微生物覆盖率高,测序结果可满足菌群多样性分析需求[32]。分析Shannon和Simpson指数的变化趋势可知,初沉水、好氧水、排放水和二沉泥中的微生物多样性依次呈增加趋势。刘志伟等[33]在研究生活污水污染物的去除过程中也发现,细菌物种数和群落多样性越丰富,COD和氨氮的去除效果越好。
表2 四个取样点废水的Alpha多样性分析
Table 2 Alpha diversity analysis of wastewater from four sampling points
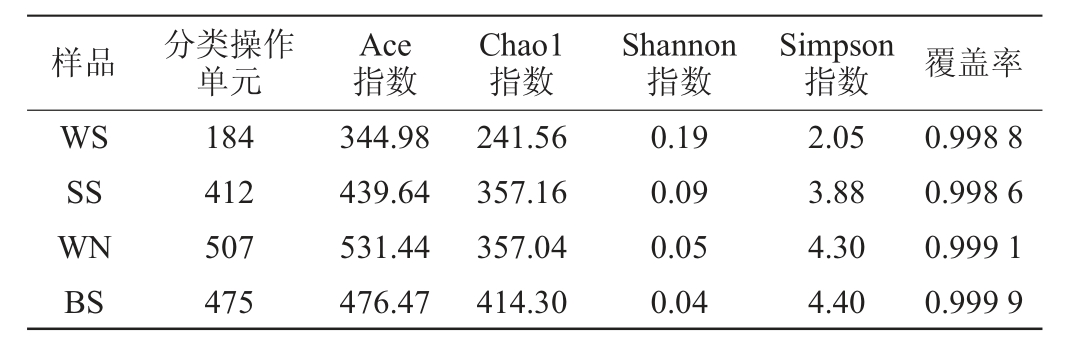
样品 分类操作单元Ace指数Chao1指数Shannon指数Simpson指数 覆盖率WS SS WN BS 184 412 507 475 344.98 439.64 531.44 476.47 241.56 357.16 357.04 414.30 0.19 0.09 0.05 0.04 2.05 3.88 4.30 4.40 0.998 8 0.998 6 0.999 1 0.999 9
2.4 四个取样点废水中的微生物菌群结构分析
2.4.1 门水平细菌群落分析
门水平细菌菌落组成见图4。由图4可知,初沉水的门水平优势菌群为变形菌门(Proteobacteria)(69.62%)和厚壁菌门(Firmicutes)(29.77%)。好氧水中以uncultured bacterium WS6(34.48%)和变形菌门(24.85%)为主。钟宇等[34]在长沙第二污水处理厂的剩余活性污泥中也鉴定出了uncultured bacterium WS6,并发现它属于丝状菌,且大多专性或兼性厌氧。二沉泥中以unculturedbacteriumWS6(22.75%)和变形菌门(22.88%)为主,螺旋体菌门(Saccharibacteria)(8.34%)次之。排放水的门水平优势菌为螺旋体菌门(8.89%)和变形菌门(16.49%)。厚壁菌门是糖类降解菌革兰阳性菌,可以降解蛋白质[35]。可见,随着废水处理工序的进行,厚壁菌门和变形菌门的相对丰度逐渐下降,而螺旋体菌门和uncultured bacterium WS6的相对丰度呈现增加趋势。变形菌门在整个处理过程中始终保持较高的相对丰度(>16%),有利于有机物和营养物的去除[36],这与SUN Y M等[37]研究结果一致。
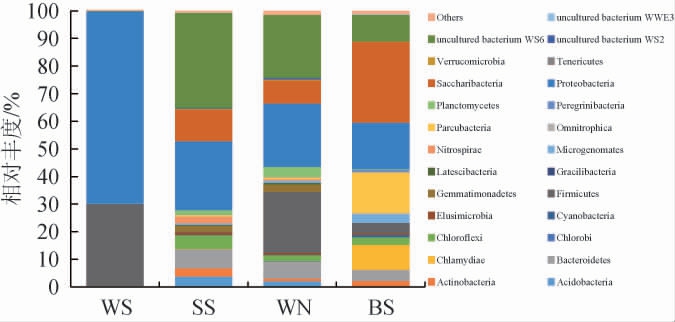
图4 基于门水平四个取样点废水细菌菌落组成
Fig.4 Compositions of bacterial community of wastewater from four sampling points based on phylum level
2.4.2 科水平细菌群落分析
科水平细菌菌落组成结果见图5。由图5可知,在科水平上,初沉水中以着色菌科(Chromatiaceae)(31.35%)和丛毛单胞菌科(Comamonadaceae)(37.59%)为主,着色菌科是光合细菌的一种,可迅速利用低分子有机物[38],能减少氨氮、硝态氮、硫化物和其他有毒物质含量,净化水质[39],已有研究表明,着色菌科在光照厌氧条件下,进行光能异养生长,从而处理有机废水[40]。好氧水和二沉泥中的优势菌群均为uncultured bacterium p Saccharibacteria(11.53%和8.34%),排放水中的优势菌群为uncultured bacterium p Saccharibacteria(28.89%),说明uncultured bacterium p Saccharibacteria一定具有分解葡萄糖的作用,进一步说明发生了糖酵解[27]。丛毛单胞菌科、着色菌科和消化链球菌科的相对丰度随废水处理工序的进行呈下降趋势,uncultured bacterium p Saccharibacteria相对丰度呈增加趋势,而uncultured bacterium p Saccharibacteria在废水处理过程中始终保持较高相对丰度(>8%,除初沉水外)。在废水处理体系中,丛毛单胞菌科可以降解利用蛋白质,降低总氮含量[41];韦荣球菌科细菌能很好利用短链脂肪酸,同时也可利用异丁酸、异戊酸和乙酸等小分子酸[42]。
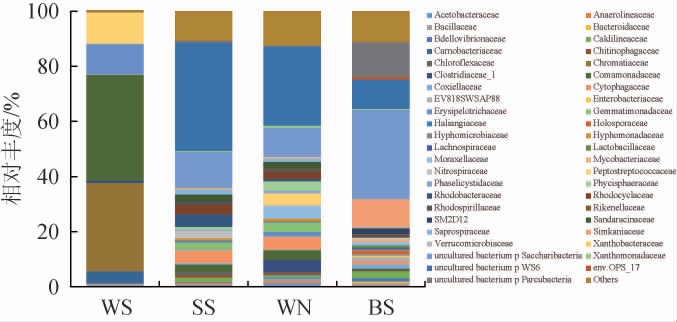
图5 基于科水平四个取样点废水细菌菌落组成
Fig.5 Compositions of bacterial community of wastewater from four sampling points based on family level
2.4.3 属水平细菌群落分析
属水平细菌菌落组成结果见图6。由图6可知,在属水平上,初沉水中以噬氢菌属(Hydrogenophaga)和莱茵海默氏菌属(Rheinheimera)为主,二者相对丰度分别为37.57%和31.35%,噬氢菌属对废水中的氮、磷均有很好去除效果,是典型的废水处理过程中的好氧微生物[43]。好氧水和二沉泥的优势菌属均为uncultured bacterium p WS6(34.48%,22.76%),属于丝状菌属,大多是专性或间性厌氧菌、或氧亲和系数较低的菌[34]。二沉泥样品中还有少量厌氧醋菌属(Acetoanaerobium),这类微生物是具有产酸作用的主要功能优势菌,能充分利用剩余污泥中的溶解性有机物产生挥发性有机酸[44]。排放水中的优势菌群以uncultured bacterium p Saccharibacteria(28.89%)为主,这可能是本研究中的特征微生物。
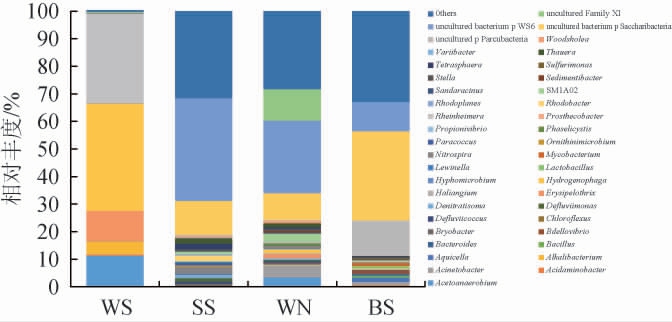
图6 基于属水平四个取样点废水细菌菌落组成
Fig.6 Compositions of bacterial community of wastewater from four sampling points based on genus level
另一方面,噬氢菌属、莱茵海默氏菌属和厌氧醋菌属的相对丰度均随着废水处理工序的进行而呈显著下降趋势,uncultured bacterium p Saccharibacteria的相对丰度呈上升趋势。Uncultured bacterium p Saccharibacteria和莱茵海默氏菌属在蛋白酶、脂肪酶的产生和有机氮的降解中发挥着重要的作用[45]。纪树兰等[46]在城市污水好氧颗粒污泥中也发现了大量的uncultured bacterium(42.7%),这些未知菌群在废水中的功能作用有待进一步研究。
2.5 四个取样点废水的群落结果差异分析
PCA是一种分析和简化数据集的技术[47],两个样品距离越近,则表示这两个样品的组成越相似[48]。对四个取样点废水的细菌群落差异性进行分析,结果见图7。
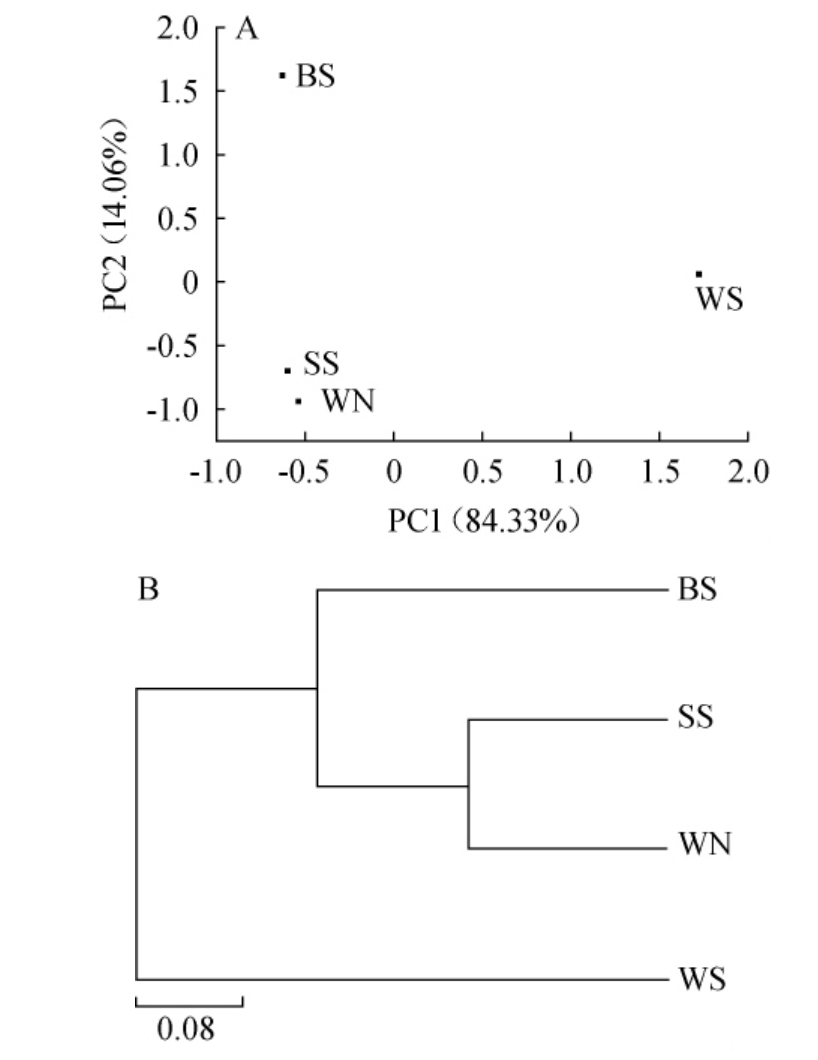
图7 四个取样点废水的细菌群落结构主成分分析(A)及非加权平均分析(B)
Fig.7 Principal component analysis (A) and unweighted average analysis (B) of bacterial community structure in wastewater from four sampling points
由图7A可知,好氧水和二沉泥样品的相对距离很近,说明好氧水和二沉泥的细菌群落相似性最大。初沉水、好氧水、排放水三者之间的相对距离较远,说明细菌群落相似性较低,这可能是微生物群落对不同环境因素的差异化响应结果,且主成分分析法能将这四个取样点的废水进行区分。由图7B可知,非加权配对平均(unweightedpair-group method with arithmetic means,UPGMA)分析表明,细菌群落结构的变化是随着处理工序的进行而逐步发生改变的,UPGMA结果显示,WN和SS先聚为一类,后又与BS聚为一类,WS单独为一类,表明好氧水和二沉泥的细菌群落结构相似性较高,与PCA结果一致。
3 结论
明胶加工废水处理系统含有丰富多样的细菌群落,且群落结构因废水处理工序不同而存在差异。在门水平,初沉水的优势菌门为变形菌门和厚壁菌门,好氧水和二沉泥的为uncultured bacterium WS6和变形菌门,排放水的为螺旋体菌门和变形菌门;在属水平,初沉水的优势菌属为噬氢菌属,好氧水和二沉泥的优势菌属为uncultured bacterium p WS6,排放水优势菌属为uncultured bacterium p Saccharibacteria,且好氧水和二沉泥样品之间的微生物菌群组成相似度较高。废水中的细菌群落与水质环境因子之间存在显著相关性(P<0.01),其中莱茵海默氏菌与废水中Ca2+和氨氮浓度、pH值呈显著正相关(P<0.05),嗜碱菌属与总氮浓度呈极显著正相关(P<0.01),与COD浓度呈显著正相关(P<0.05)。这些微生物功能菌群的存在得益于明胶加工废水提供的合理生存条件,也对水质净化处理效果起到了良好功能作用,进而达到污水排放标准。然而,还有一些微生物菌群尚未能探明,有待进一步的研究分析。
[1]CHEN Y,LAN S,WANG L,et al.A review:Driving factors and regulation strategies of microbial community structure and dynamics in wastewater treatment systems[J].Chemosphere,2017,174:173-182.
[2]SILVANO C,FREITAS P S L,REZENDE R,et al.Application effect of different rates of wastewater from gelatin production in the chemical attributes of the soil[J].Engenharia Agrícola,2018,38(4):606-615.
[3]KRUTHIKA L N, KARTHIKA S, RAJU B G, et al.Efficacy of electrocoagulation and electrooxidation for the purification of wastewater generated from gelatin production plant[J].J Environ Chem Eng,2013,1(3):183-188.
[4]唐多利.明胶废水中耐钙微生物的筛选、鉴定及其钙吸附研究[D].兰州:兰州理工大学,2020.
[5]石杨.白湿革废料制备明胶的污水处理方法及成本核算研究[J].中国皮革,2021,50(9):120-124.
[6]DETTMER A,CAVALLI É,AYUB A M,et al.Environmentally friendly hide unhairing:enzymatic hide processing for the replacement of sodium sulfide and delimig[J].J Clean Product,2013,47(5):11-18.
[7]ARTURI S T, SEIJAS J C, BIANCHI L G.A comparative study on the treatment of gelatin production plant wastewater using electrocoagulation and chemical coagulation[J].Heliyon,2019,5(5):1738-1745.
[8]GHATNEKAR S D,GHALSASI D S,TAMHANE B M.The novel threetier biotechnology to convert solid waste of gelatine manufacturing unit into useful plant probiotics[J].Ind J Environ Protect,2009,29(9):767-774.
[9]MEKY N,FUJII M,IBRAHIM G M,et al.Biological hydrogen gas production from gelatinaceous wastewater via stand-alone circular dark/photo baffled fermenter[J].Energy Procedia,2019,157:670-675.
[10]李艳,孔秀琴,贾金娥,等.微好氧与厌氧水解酸化对明胶废水的预处理效果[J].土木建筑与环境工程,2017,39(5):129-134.
[11]WANG G,YU N,GUO Y.A novel process to recycle the highly concentrated calcium and chloride ions in the gelatin acidification wastewater[J].J Clean Product,2018,188(1):62-68.
[12]AGNIESZKA C,MAGDALENA Z.Bacterial communities in full-scale wastewater treatment systems[J].World J Microbiol Biotech,2016,32(4):66.
[13]许光素,刘俊新,韩云平,等.高通量测序技术应用于污水处理厂细菌气溶胶群落结构分析[J].环境科学学报,2018,38(11):4235-4242.
[14]LU X M,LU P Z.Characterization of bacterial communities in sediments receiving various wastewater effluents with high-throughput sequencing analysis[J].Microbial Ecol,2014,67(3):612.
[15]艾铄,张丽杰,肖芃颖,等.高通量测序技术在环境微生物领域的应用与进展[J].重庆理工大学学报(自然科学),2018,32(9):111-121.
[16]VIEIRA A M S,BERGAMASCO R,GIMENES M L.Microbial populations of an upflow anaerobic sludge blanket reactor treating wastewater from a gelatin industry[J].Environ Technol Lett,2001,22(12):1477-1485.
[17]SHIVSHARAN S V, WANI M, KHETMALAS B M.Isolation of microorganisms from dairy effluent[J].British Microbiol Res J,2013,3(3):346-354.
[18]许青青.反硝化法去除油脂废水恶臭物质技术及机理[D].扬州:扬州大学,2016.
[19]刘婕.除油菌的筛选鉴定及油脂废水处理研究[D].广州:华南理工大学,2010.
[20]环境保护部科技标准司.HJ 493—2009 水质样品的保存和管理技术规定[S].北京:中国环境科学出版社,2009.
[21]HAN Q Y,FANG H H P.Acidogenesis of gelatin-rich wastewater in an upflow anaerobic reactor:influence of pH and temperature[J].Water Res,2003,37(1):55-66.
[22]刘燕林,王连山.明胶废水处理系统的调试与运行[J].环境工程,2012,30(S2):64-67.
[23]刘亚利,袁一星,杜茂安,等.碱预处理污泥产酸效果及其生物群落研究[J].环境工程学报,2013,7(7):2685-2688.
[24]廖润华.胁迫条件下EGSB反应器处理高硝态氮废水及其微生物群落与功能研究[D].南京:南京大学,2014.
[25]陈兆进,丁传雨,朱静亚,等.丹江口水库枯水期浮游细菌群落组成及影响因素研究[J].中国环境科学,2017,37(1):336-344.
[26]WANG M,FAN Z J,WANG R N,et al.Nitrogen removal performance and microbial community structure of water and its association with nitrogen metabolism of an ecological engineering pond aquaculture system[J].Aquacult Rep,2022,25:101258.
[27]阴泽阳.三种不同污水生物处理系统生物信息学特征研究[D].太原:太原理工大学,2020.
[28]SENGUPTA A,DICK W A.Bacterial community diversity in soil under two tillage practices as determined by pyrosequencing[J].Microbial Ecol,2015,70(3):853-859.
[29]AMATO K R,YEOMAN C J,KENT A,et al.Habitat degradation impacts black howler monkey(Alouatta pigra) gastrointestinal microbiomes[J].ISME J,2013,7(7):1344-1353.
[30]CHEN H B,BOUTROS P C.Venn Diagram:a package for the generation of highly-customizable Venn and Euler diagrams in R[J].BMC Bioinformatics,2011,12(1):35-42.
[31]翟一帆,袁青彬,胡南.养猪废水处理系统微生物群落结构变化及影响因素研究[J].水资源保护,2018,34(1):88-94.
[32]LI H,YE D D,WANG X G,et al.Soil bacterial communities of different natural forest types in Northeast China[J].Plant and Soil,2014,383(2):38-42.
[33]刘志伟,周美修,宋俊玲,等.复合垂直流人工湿地污染物去除特征及微生物群落多样性分析[J].环境工程,2014 32(2):38-42.
[34]钟宇,杨麒,李小明,等.RBER生物降解溴酸盐及微生物群落结构分析[J].中国环境科学,2017,37(5):1945-1953.
[35]曾薇,郭京京,纪兆华,等.pH值对剩余污泥微氧水解酸化溶出物及微生物群落结构的影响[J].应用基础与工程科学学报,2018,26(3):471-482.
[36]HU M,WANG X H,WEN X H,et al.Microbial community structures in different wastewater treatment plants as revealed by 454-pyrosequencing analysis[J].Bioresource Technol,2012,117:72-79.
[37]SUN Y M, SHEN Y X, LIANG P, et al.Multiple antibiotic resistance genes distribution in ten large-scale membrane bioreactors for municipal wastewater treatment[J].Bioresource Technol,2016,222:100-106.
[38]王琦.光合细菌处理明胶废水研究[D].重庆:重庆大学,2002.
[39]徐鹏飞.光合细菌在水产养殖上的应用与使用方法[J].科学种养,2013(3):6-7.
[40]王玉堂.光合细菌及其在水产养殖业的应用(一)[J].中国水产,2009(3):48-51.
[41]ISANTA E,BEZERRA T,FERNÁNDEZ I,et al.Microbial community shifts on an anammox reactor after a temperature shock using 454-pyrosequencing analysis[J].Bioresource Technol,2015,181:207-213.
[42]刘传发,张良志,付海波,等.野牦牛和家牦牛粪便菌群与短链脂肪酸关系的研究[J].兽类学报,2019,39(1):1-7.
[43]戚科美.人工水草对污染河流中氮磷等污染物的去除效果研究[D].济南:山东师范大学,2017.
[44]姚创,刘晖,罗晓栋,等.华南地区低有机质污泥碱性厌氧产酸(VFAs)性能机理与菌群分析[J].化工学报,2016,67(4):1565-1571.
[45]周明扬.中国南海和南极菲尔德斯半岛海域沉积物中微生物、蛋白酶的多样性及有机氮的降解机制[D].济南:山东大学,2013.
[46]纪树兰,崔丹红,周明璟,等.基于城市污水好氧颗粒污泥脱氮除磷系统种群多样性研究[J].北京工业大学学报,2013,39(7):1100-1108.
[47]WANG Y,SHENG H F,HE Y,et al.Comparison of the levels of bacterial diversity in freshwater,intertidal wetland,and marine sediments by using millions of illumina tags[J].Appl Environ Microbiol,2012,78(23):8264-8271.
[48]NI K K,WANG F F,ZHU B G,et al.Effects of lactic acid bacteria and molasses additives on the microbial community and fermentation quality of soybean silage[J].Bioresource Technol,2017,238:706-715.