干酪乳杆菌(Lactobacillus casei)是一种功能性食品、发酵及酿造工业中被广泛使用的益生菌[1-3]。干酪乳杆菌能够以蔗糖、菊粉等作为营养物质快速生长,并具有调节肠道,缓解便秘,预防结直肠炎等作用。已有研究显示,干酪乳杆菌能改善动物肠道微生态环境,抑制肠道致病菌繁殖并提高机体抗病能力[4-6]。来源于干酪乳杆菌的转化酶,即β-果糖呋喃糖苷酶[EC 3.2.1.26],又称β-D-呋喃果糖苷果糖水解酶、蔗糖酶、转化素和转糖酶,它既能高效地水解菊粉生成果糖和低聚果糖,又能水解蔗糖生成果糖和葡萄糖的混合物。食品工业中,转化酶在高蔗糖浓度下,能够发挥水解作用生成葡萄糖、果糖和短链低聚果糖(fructo oligosaccharide,FOS)[7],具有制备高果糖浆的应用潜力[8]。此外,转化酶在食品、制药、化妆品、生物燃料和农业等领域也发挥着重要作用[9-10]。因此,筛选出高活性的转化酶菌株,对于将转化酶应用于工业生产中可以极大提高反应速度,缩短时间成本。OKAFOR C A等[11]从发酵饮料中分离鉴定出的植物乳杆菌、短乳杆菌和发酵乳杆菌中,均检测到转化酶活性,但活性较小。KUZUWA S等[12]将来源于干酪乳杆菌的转化酶基因进行改造,以提高转化酶的活性。KUSMIYATI N等[13]通过优化培养条件提高转化酶的活性。本研究中的转化酶蛋白分子质量较大,在37 ℃培养条件下,干酪乳杆菌中转化酶表达量有限,酶活低下。因此,开发分子质量更小的能够满足市场需求的干酪乳杆菌转化酶具有较大的理论研究和实际应用价值。
近年来随着Alpha Fold等蛋白质预测工具在蛋白质结构预测方面的重大突破,酶的理性设计已经进一步的发展为蛋白质定向进化的有力工具之一。酶的理性设计是基于目标酶结构和功能关系的已有知识,选择特定氨基酸残基进行定点突变的酶改造方法[14]。TU T等[15]通过同源建模和分子对接,在一种嗜酸真菌的内聚半乳糖醛酸酶BiPG28A中鉴定出了与底物结合相关的关键残基。ZHENG J等[16]对来源于黑曲霉的纤维素酶AnCel5A进行了理性设计改造,筛选获得了催化效率和热稳定性显著提高的突变体。
本实验采用高通量的平板显色法从千岛湖土壤样品中筛选获得了具有转化酶活性的菌株,并对其进行菌种鉴定。根据干酪乳杆菌(Lactobacillus casei)的基因组数据,设计引物,获得了相应的转化酶基因。以pET23(b+)为表达质粒,在大肠杆菌(Escherichia coli)中进行了表达。基于理性设计构建与表达了不同的截短转化酶,对截短体酶学性质进行研究,以期获得耐高温、高活性的转化酶菌株,为转化酶在酶动力学参数分析研究、酶学性质研究以及转化酶在生产实际方面的应用提供理论指导。
1 材料与方法
1.1 材料与试剂
1.1.1 样品、菌株、质粒和引物
实验样品来自千岛湖土壤;pET-23(b+)表达载体、大肠杆菌(E.coli)TOP10、大肠杆菌(E.coli)BL21(DE3):本实验室保藏;克隆质粒pMD18-T载体:大连宝生物工程有限公司;引物合成:南京金斯瑞生物公司。本实验所用引物见表1。
表1 本研究所用引物
Table 1 Primers used in this study
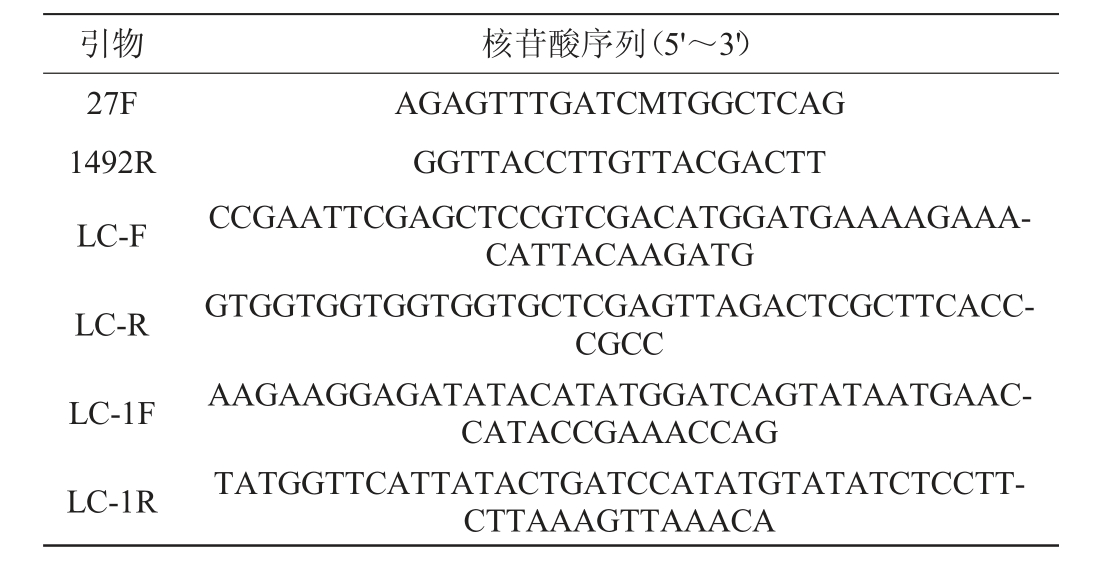
引物 核苷酸序列(5'~3')27F 1492R LC-F LC-R LC-1F LC-1R AGAGTTTGATCMTGGCTCAG GGTTACCTTGTTACGACTT CCGAATTCGAGCTCCGTCGACATGGATGAAAAGAAACATTACAAGATG GTGGTGGTGGTGGTGCTCGAGTTAGACTCGCTTCACCCGCC AAGAAGGAGATATACATATGGATCAGTATAATGAACCATACCGAAACCAG TATGGTTCATTATACTGATCCATATGTATATCTCCTTCTTAAAGTTAAACA
续表
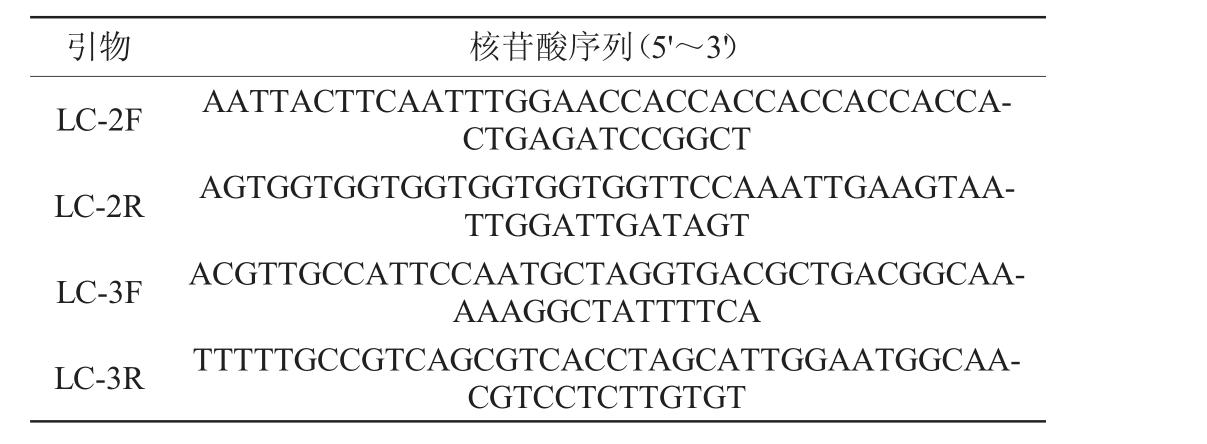
引物 核苷酸序列(5'~3')LC-2F LC-2R LC-3F LC-3R AATTACTTCAATTTGGAACCACCACCACCACCACCACTGAGATCCGGCT AGTGGTGGTGGTGGTGGTGGTTCCAAATTGAAGTAATTGGATTGATAGT ACGTTGCCATTCCAATGCTAGGTGACGCTGACGGCAAAAAGGCTATTTTCA TTTTTGCCGTCAGCGTCACCTAGCATTGGAATGGCAACGTCCTCTTGTGT
1.1.2 试剂
蛋白胨、酵母粉(均为生化试剂)、氯化钠、蔗糖(均为分析纯)、蛋白Marker:生工生物工程(上海)股份有限公司;质粒提取试剂盒、胶回收试剂盒:美国OMEGA公司;抗生素、脱氧核糖核酸(deoxyribonucleic acid,DNA)提取试剂盒、限制性核酸内切酶(Sal I、Xho I)、5×Native loading Buffer:北京全式金生物技术有限公司;Taq DNA聚合酶:大连宝生物工程有限公司;溴甲酚紫钠盐(分析纯):天津市化学试剂研究生所;3,5-二硝基水杨酸(dinitrosalicylic acid,DNS)(分析纯):上海阿拉丁生化生化科技股份有限公司;牛血清白蛋白(bovine serum albumin,BSA)(纯度>98%)、考马斯亮蓝(分析纯):合肥博美生物科技有限责任公司。
1.1.3 培养基
Luria-Bertani(LB)液体培养基:胰蛋白胨10.0 g/L,氯化钠10.0 g/L,酵母粉5.0 g/L。用1 mol/L的NaOH调节pH值至7.0,去离子水定容至1 L。121 ℃高压灭菌15 min。
LB固体培养基:LB液体培养基中添加15.0 g/L琼脂。
LB改良培养基:蛋白胨10.0 g/L,氯化钠10.0 g/L,酵母粉5.0 g/L,溴甲酚紫0.025 g/L。121 ℃高压灭菌15 min。
含卡那霉素(kanamycin,Kan)LB固体培养基:胰蛋白胨10.0 g/L,氯化钠10.0 g/L,酵母粉5.0 g/L,琼脂15.0 g/L。用1 mol/L的NaOH调节pH值至7.0,去离子水定容至1 L。121 ℃高压灭菌15 min。冷却到50 ℃左右时,每50 mL培养基中加入50 μL卡那霉素,混匀,每个平板倒入10~15 mL培养基,4 ℃凝固后养保存。
含氨苄青霉素(ampicillin,Amp)LB固体培养基:胰蛋白胨10.0 g/L,氯化钠10.0 g/L,酵母粉5.0 g/L,琼脂15.0 g/L。用1 mol/L的NaOH调节pH值到7.0,去离子水定容至1 L。121 ℃高压灭菌15 min。冷却到50 ℃左右时,每50 mL培养基中加入50 μL氨苄青霉素,混匀,每个平板倒入10~15 mL培养基,4 ℃凝固后养保存。
含5-溴-4-氯-3-吲哚-β-D-半乳糖苷(X-gal)LB固体培养基:胰蛋白胨10.0 g/L,氯化钠10.0 g/L,酵母粉5.0 g/L,琼脂15.0 g/L。用1 mol/L的NaOH调节pH至7.0,去离子水定容至1 L。121 ℃高压灭菌15 min。冷却到50 ℃左右时,每50 mL培养基中加入50 μL 5-溴-4-氯-3-吲哚-β-D-半乳糖苷,混匀,每个平板倒入10~15 mL培养基,4 ℃凝固后养保存。
1.2 仪器与设备
JY92-IIN型超声波细胞粉碎机:宁波新芝生物科技股份有限公司;Mini PROTEAN Tetra Cell型蛋白电泳系统、PowerPacTM Basic型凝胶电泳系统、T100 Thermal Cycler型聚合酶链式反应(polymerase chain reaction,PCR)仪:美国Bio-Rad伯乐公司;Tanon-3500R型凝胶成像系统:上海天能生命科学有限公司;UV-19001型紫外可见分光光度计:岛津仪器(苏州)有限公司;L535R-1型低速冷冻离心机:湖南湘仪实验室仪器开发有限公司;Spectra Max M3酶标仪:美谷分子仪器(上海)有限公司;His GraviTrap预装重力流层析柱(1 mL His GraviTrap,Ni-NTA)亲和层析柱:思拓凡生物科技(杭州)有限公司;ZQZY-78BV振荡培养箱:上海知楚仪器有限公司;Five Easy Plus pH计:梅特勒-托利多仪器(上海)有限公司。
1.3 方法
1.3.1 干酪乳杆菌的分离筛选及鉴定
2022年9月于千岛湖采集土壤,称取5 g土壤样品,并加入45 mL无菌水,37 ℃、150 r/min条件下振荡培养1 h,然后用无菌水梯度稀释为10-2、10-3浓度的悬浮液,每个梯度溶液吸取0.1 mL涂布于LB固体培养基,37 ℃培养3~4 d,挑取具有典型特征的菌落,继续在LB改良培养基上于37 ℃静置培养3~4 d获得产转化酶的菌株,然后接种到LB液体培养基于37 ℃、200 r/min恒温摇床振荡培养14~16 h,将菌液按体积比1∶1加入50%甘油中,于-80 ℃保藏备用。
利用PCR扩增单菌落的16SrRNA基因序列,引物为细菌16S rRNA基因通用引物27F、1492R。PCR扩增体系(50 μL):无菌水16 μL,2×Taq Master Mix酶25 μL,引物27F 2 μL,引物1492R 2 μL,基因组DNA 5 μL。PCR扩增条件:94 ℃预变性3 min;94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸1 min,30个循环;72 ℃终延伸7 min。PCR产物进行凝胶电泳鉴定后,利用试剂盒进行纯化回收,连接到pMDTM18-T上,转化到E.coli TOP10感受态细胞中,经含Kan、X-gal LB固体培养基筛选、菌落PCR验证后挑取阳性转化子送至上海生工生物测序,测序结果通过美国国家生物技术信息中心(national center of biotechnology information,NCBI)网站的GenBank数据库进行基本局部比对搜索工具(basic local alignment search tool,BLAST)同源序列比对[17],选取同源性较高的菌株16S rRNA,利用MEGA7.0软件中的邻接(neighbor joining,NJ)法构建菌株系统发育树,确定菌株的分类学地位,并将菌种送至中国典型培养物保藏中心室保藏。
1.3.2 重组干酪乳杆菌转化酶构建与蛋白表达
采用提取干酪乳杆菌基因组DNA,利用特异性引物LC-F、LC-R进行PCR扩增获得干酪乳杆菌转化酶基因(GeneBank登录号:AB185852.1)开放阅读框序列,扩增产物纯化后用SaLI、XhoI进行双酶切,pET-23(b+)载体用相同方式进行酶切,PCR仪上37 ℃相连接30 min之后电击转化至E.coli BL21(DE3)感受态细胞。采用菌落PCR法筛选阳性转化子,送上海生工公司测序验证。将测序正确的重组菌株命名为BL21/pET23b/LC。
从LB固体培养基上挑取BL21/pET23b/LC单菌落至5 mL含100 μg/mL的LB液体培养基中,37 ℃、200 r/min振荡培养6~8 h后转接至50 mL含10 μg/mL Kan的LB液体培养基中,37 ℃、200 r/min振荡培养至OD600 nm值为0.6~0.8时,添加终浓度为0.5 mmol/L的异丙基-β-D-硫代吡喃半乳糖苷(isopropyl-β-D-galactopyranoside,IPTG),26 ℃、200 r/min诱导培养14~16 h,于5 000 r/min、4 ℃条件下离心15 min收集细胞,用10 mL磷酸盐缓冲液(phosphate buffer solution,PBS)清洗1次,5 000 r/min条件下离心15 min。弃上清,沉淀用10 mLPBS重新悬浮,利用超声波细胞粉碎机破碎10 min。5 000 r/min、4 ℃条件下离心15 min,上清液即为重组干酪乳杆菌转化酶蛋白粗酶液。粗酶液经His GraviTrap Ni-NTA亲和层析后获得重组干酪乳杆菌转化酶蛋白纯化液,命名为重组干酪乳杆菌转化酶(LevH1-LC)。
1.3.3 分子对接
由于PDB数据库中没有LevH1-LC蛋白晶体结构的记录,同源性最高的蛋白序列为1Y4W,序列一致性为32%。采用Swiss-Model对LevH1-LC进行同源建模获得其高级结构模型。采用Vector NTI和Pymol对两者进行序列对比和蛋白空间结构分析,设计不同截短突变体。采用AutoDock对亲本及突变体进行分子对接,采用Pymol和Ligplot+对分子对接的结果进行可视化及分析。
1.3.4 重组干酪乳杆菌转化酶截短体
将重组质粒pET23b-LC作为模板利用特异性引物LC-1F、LC-1R,LC-2F、LC-2R和LC-3F、LC-3R分别进行PCR扩增。PCR扩增体系(50 μL):无菌水32 μL,10×KOD Buffer缓冲液5 μL,25 mmol/L MgSO4 5 μL,2 mmol/L脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)5 μL,引物F 0.75 μL,引物R 0.75 μL,DNA模板0.5 μL,KOD DNA聚合酶1 μL。PCR扩增条件:98 ℃预变性3 min;98 ℃变性1min,62℃退火45s,68℃延伸3min 30 s,30个循环;68 ℃终延伸10 min。纯化回收的PCR产物电转化至E.coliTOP10感受态细胞,经菌落PCR和测序鉴定,获得截短转化酶表达完整质粒pET23b-LC-N、pET23b-LC-C和pET23b-LC-M。然后以重组质粒pET23b-LC-N为模板,LC-2F和LC-2R作引物,采用全质粒PCR扩增的技术,获得截短转化酶表达完整质粒pET23b-LC-NC。最后将构建有不同转化酶的质粒转化至大肠杆菌BL21(DE3),获得4株截短的重组工程菌株BL21/pET23b-LC-N、BL21/pET23b-LC-C、BL21/pET23b-LCM、BL21/pET23b-LC-NC。参照1.3.2的方法对4株截短的重组工程菌株进行诱导表达、细胞破碎和His GraviTrap Ni-NTA亲和层析。纯化后的不同转化酶截短体分别标记为LevH1-LC-N、LevH1-LC-C、LevH1-LC-M 和LevH1-LC-NC,并采用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecylsulphate polyacrylamide gel electrophoresis,SDS-PAGE)进行分析。
1.3.5 重组干酪乳杆菌转化酶截短体分子质量测定
取40 μL亲本转化酶LevH1-LC和其不同截短体LevH1-LC-N、LevH1-LC-CL和LevH1-LC-NC的纯化酶液与10 μL 5×Native loading Buffer,混合煮沸15 min,静置室温,混匀,取7 μL 用于十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(SDSPAGE)(15%分离胶,4%浓缩胶)分析。
1.3.6 转化酶酶活及蛋白浓度测定
转化酶酶活测定采用3,5-二硝基水杨酸(DNS)法[18]。以葡萄糖含量(x)为横坐标,OD515 nm值(y)为纵坐标建立标准曲线,得到标准曲线回归方程y=1.073 7x+0.246 1,相关系数R2=0.991 5,按照回归方程计算转化酶酶活。转化酶酶活定义:在20 ℃下反应30 min转化1 mg蔗糖为葡萄糖和果糖为一个酶活单位(U/mL)。
蛋白浓度测定采用Bradford法[19]。以BSA含量(x)为横坐标,OD595nm值(y)为纵坐标建立标准曲线,得到标准曲线回归方程y=7.551 3x+0.031 4,相关系数R2=0.996 4,按照回归方程计算转化酶的蛋白浓度。
1.3.7 酶学性质
将单菌落接种于LB(含氨苄)液体培养基的摇瓶,经IPTG诱导表达,培养14~16 h后,5 000 r/min、4 ℃条件下离心15 min收集细胞,用10 mL磷酸盐缓冲液(PBS)清洗1次,5 000 r/min条件下离心15 min。弃上清,沉淀用10 mL PBS重新悬浮,利用超声波细胞粉碎机破碎10 min。5 000 r/min、4 ℃条件下离心15 min,上清液即为重组干酪乳杆菌转化酶蛋白粗酶液。
(1)最适反应pH
将1 mL粗酶液分别与5 mL pH值为3.0、4.0、5.0、6.0、7.0、8.0、9.0、10.0、11.0、12.0的6.5%蔗糖底物混合,于20 ℃条件下反应30 min。取1.5 mL反应液和3.5 mL DNS试剂混合,99 ℃加热10 min,待冷却至室温(25 ℃)后加入20 mL水。静置10 min后利用酶标仪于波长515 nm下测定吸光度值。设置3组平行实验。以最高酶活为100%,计算各温度条件下相对酶活。
(2)最适反应温度
将1 mL粗酶液与5 mL 6.5%蔗糖底物混合,分别置于20 ℃、30 ℃、40 ℃、50 ℃、60 ℃,pH 5.0条件下反应30 min。按1.3.6所述设置对照组并测定酶活。以最高酶活为100%,计算各温度条件下的相对酶活,设置3组平行试验。
(3)热稳定性
将粗酶液分别置于20 ℃、30 ℃、40 ℃、50 ℃、60 ℃、70 ℃、80 ℃、90 ℃下孵育0.5 h。静置至室温,在20 ℃下检测酶活性。以反应起始点酶活为100%,计算各温度条件下的残余酶活。
(4)pH稳定性
将粗酶液分别置于室温下不同pH缓冲液(pH 3.0、4.0、5.0、6.0、7.0、8.0、9.0、10.0、11.0、12.0)中孵育0.5 h,分别检测酶活性。以反应起始点的酶活为100%,计算各pH条件下的残余酶活。
1.3.8 不同截短转化酶的酶促反应动力学参数
分别配制0.1~10 mg/mL的蔗糖底物,取不同转化酶截短体LevH1-LC、LevH1-LC-N、LevH1-LC-C、LevH1-LC-M和LevH1-LC-NC的纯化后的酶液1 mL在pH5.0、20 ℃下反应5 min,测定OD515nm值,设置3组平行试验。计算米氏常数Km、Kcat、Km/Kcat及比酶活。
2 结果与分析
2.1 产转化酶菌株的分离筛选及鉴定
从土壤样品中分离筛出一株产转化酶的菌株,编号为LC23,其菌落及细胞形态见图1。由图1a可知,干酪乳杆菌菌落通常为圆形,直径约1~2 mm,呈乳白色且不透明,表面湿润光滑,微隆起,边缘整齐。由图1b可知,细胞形态呈现杆菌、长条状。对菌株LC23的16S rRNA基因序列进行PCR扩增和测序,并将所获得的序列提交至美国国家生物技术信息中心(NCBI)网站的GenBank数据库进行BLAST同源序列比对,登录号为MT473618.1。采用MEGA 7.0构建系统发育树,结果见图2。由图2可知,产转化酶菌株LC23与Lacticaseiba casei Lp900亲缘关系最近。因此,结合其形态学观察结果,可鉴定产转化酶菌株LC23为干酪乳杆菌(Lacticaseiba casei)。KAUR M等[20]从土壤中筛选分离出一株产转化酶的枯草芽孢杆菌,并通过物理和化学诱变,实现菌株转化酶活性的增强。本研究所得天然干酪乳杆菌转化酶活性仅为(0.06±0.003)U/mL,后续将结合理性设计提高其活性。
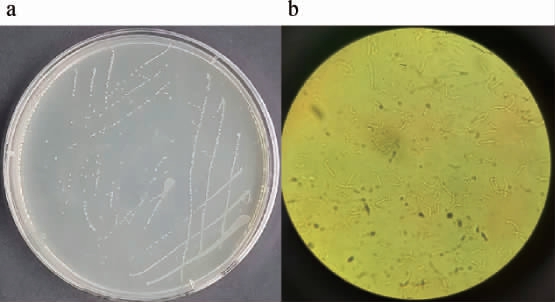
图1 菌株LC23的菌落(a)及细胞(b)形态
Fig.1 Colony (a) and cell (b) morphology of strain LC23
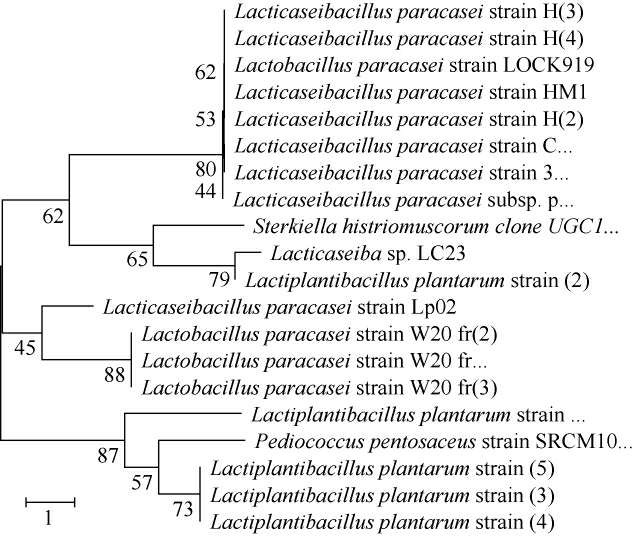
图2 基于16S rRNA基因序列构建的菌株LC23的系统进化树
Fig.2 Phylogenetic tree of strain LC23 constructed based on 16S rRNA gene sequences
2.2 截短转化酶的理性设计
用Pymol软件将亲本转化酶LevH1-LC及其截短体LevH1-LC-N、LevH1-LC-C、LevH1-LC-M和LevH1-LC-NC分别与同源性最高的蛋白1Y4W进行蛋白叠合,对比分析其蛋白的空间结构差异,结果见图3。
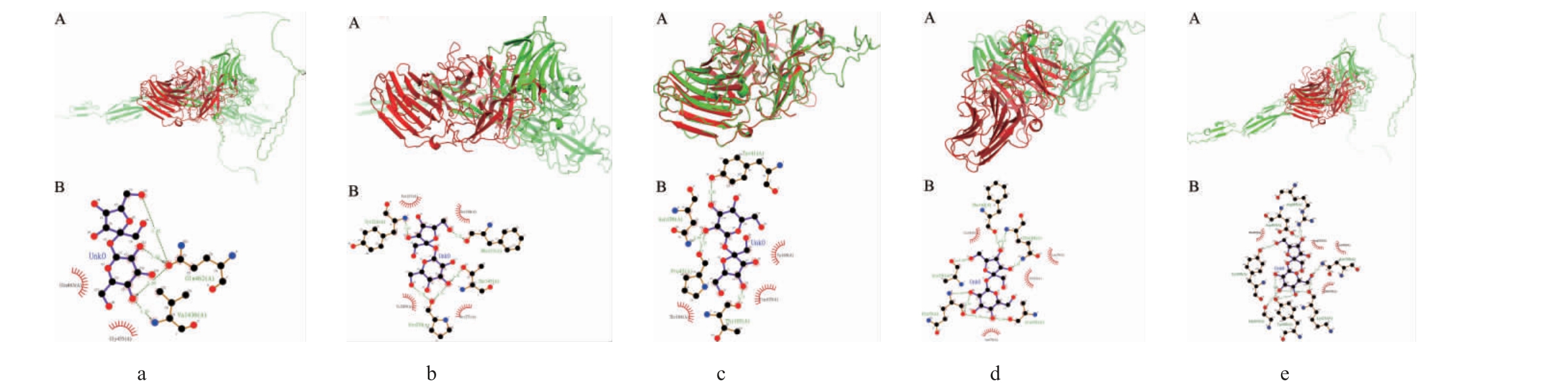
图3 LevH1-LC(a)、LevH1-LC-N(b)、LevH1-LC-C(c)、LevH1-LC-NC(d)和LevH1-LC-M(e)与蛋白1Y4W的叠合及分子间作用力
Fig.3 Superposition and intermolecular force of LevH1-LC (a), LevH1-LC-N (b), LevH1-LC-C (C), LevH1-LC-NC (d), and LevH1-LC-M (e)and protein 1Y4W
A-叠合,B-分子间作用力。
氨基酸序列对比结果表明:LevH1-LC包含938个氨基酸,蛋白1Y4W包含518个氨基酸。LevH1-LC比蛋白1Y4W在序列N端多出174个氨基酸,C端多出420个氨基酸,中间序列248L~282G不具有高度同源性。由图3可知,相对于蛋白1Y4W,LevH1-LC存在不同的空间结构,主要是氮端(N端)包含一段较长肽链,碳端(C端)则由多个折叠片组成。其N端和C端不同部分与关键活性残基D198和活性口袋(D198、R388、D389和E440)相隔甚远,基本可预测不参与催化活动。但推测N端和C端的这部分序列可能与酶学性质相关,有可能影响比活性。由此设计干酪乳杆菌转化酶基因LevH1 3个截短位点,分别为截短N端174个氨基酸的截短体LevH1-LC-N,即1M~174N;截短C端420个氨基酸的截短体LevH1-LC-C,即740S~938D;截短中间序列的截短体LevH1-LC-M,即248L~282G。同时截短1M~174N和740S~938D获得的截短体LevH1-LC-NC。
用Swiss-Model软件对不同截短体进行同源建模,用AutoDock软件以小分子蔗糖为底物进行分子对接,蔗糖和LevH1-LC的462Q和436V之间形成4个氢键作用,与435G和465Q形成两个疏水作用;蔗糖和LevH1-LC-N的218Y、270P、337F和345T形成5个氢键,与217V、269V、271M和3384S形成4个疏水作用;蔗糖和LevH1-LC-C的41Y、43P、105T和106N形成4个氢键,与104T、107W和169V形成3个疏水作用;蔗糖和LevH1-LC-NC的75Q、182F、184Q、191A和192A形成8个氢键,与72P、74L、76N和183I形成4个疏水作用;蔗糖和LevH1-LC-M的202Y、207G、209Y、483D、485R和760N形成7个氢键作用,与234D、482P、537E和762L形成4个疏水作用。根据同源分子对接结果可以发现不同转化酶截短体与底物的相互作用都有所增强,比活性可能会得到提高。
2.3 转化酶及不同截短体的分子质量测定
由于截短体LevH1-LC-M未正常表达出转化酶活性,不作为继续研究的对象。亲本转化酶LevH1-LC和其不同截 短 体LevH1-LC-N、LevH1-LC-C 和LevH1-LC-NC 通 过SDS-PAGE检测分析,结果见图4。由图4可知,亲本转化酶LevH1-LC 及 其 不 同 截 短 体LevH1-LC-N、LevH1-LC-C、LevH1-LC-NC 分子质量分别为103 kDa、84 kDa、81 kDa、62 kDa,这与其理论分子质量和载体序列翻译的肽段分子质量之和相一致。LevH1-LC-M未能正常表达和检测到活性。
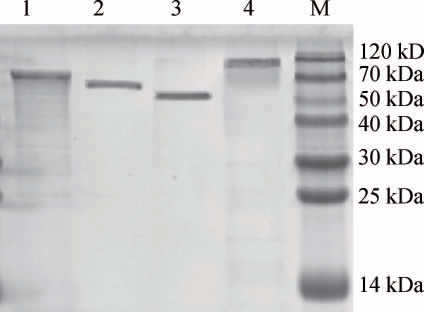
图4 转化酶LevH1-LC及其截短体LevH1-LC-N、LevH1-LC-C、LevH1-LC-NC的SDS-PAGE分析
Fig.4 SDS-PAGE analysis of invertase LevH1-LC and its truncates LEVH1-LC-N, LEVH1-LC-C, and LevH1-LC-NC
M-蛋白Marker;1-LevH1-LC-N;2-LevH1-LC-C;3-LevH1-LC-NC;
4-LevH1-LC。
2.4 转化酶及其不同截短体的酶促反应动力学参数
亲本转化酶LevH1-LC及其截短体LevH1-LC-N、LevH1-LC-C、LevH1-LC-NC分别纯化后,测定其酶动力学常数,结果见表2。由表2可知,酶活变化最显著的是截短体LevH1-LC-M,可能由于转化酶结构被破坏,结构不能够保持稳定,从而未检测到表达和活性。截短体LevH1-LC-NC和LevH1-LC-N的发酵酶活性为55.42 U/mL和34.11 U/mL,分别是亲本转化酶LevH1-LC的6.6倍和4.1倍,截短体LevH1-LC-C酶活性较LevH1-LC下降近50%至4.77 U/mL。其中酶活最高的是截短体LevH1-LC-NC,是天然干酪乳杆菌转化酶活性的923倍,其催化效率最高。该结果与分子对接的结果吻合度较高,进一步提高了理性设计在指导提高酶活性和酶热稳定性方面的可靠性。
表2 转化酶及其突变体酶动力学参数分析
Table 2 Analysis of kinetic parameters of invertase and its mutants
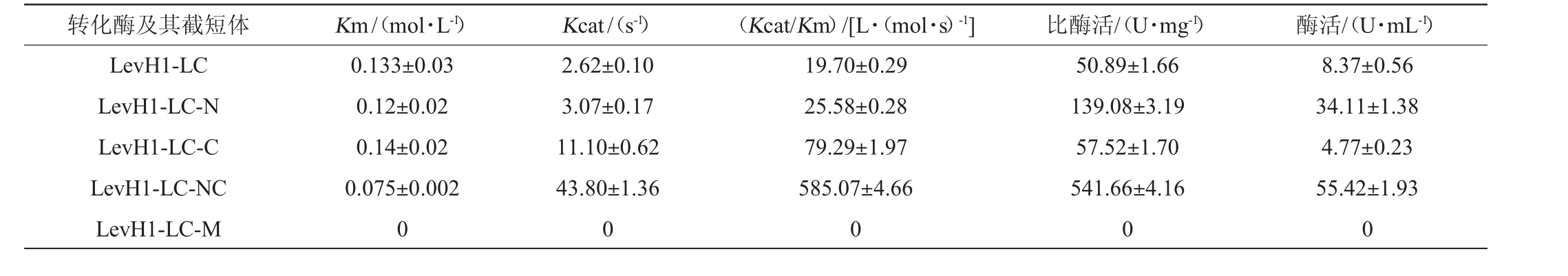
转化酶及其截短体 Km/(mol·L-1) Kcat/(s-1) (Kcat/Km)/[L·(mol·s)-1] 比酶活/(U·mg-1) 酶活/(U·mL-1)LevH1-LC LevH1-LC-N LevH1-LC-C LevH1-LC-NC LevH1-LC-M 0.133±0.03 0.12±0.02 0.14±0.02 0.075±0.002 0 2.62±0.10 3.07±0.17 11.10±0.62 43.80±1.36 0 19.70±0.29 25.58±0.28 79.29±1.97 585.07±4.66 0 50.89±1.66 139.08±3.19 57.52±1.70 541.66±4.16 0 8.37±0.56 34.11±1.38 4.77±0.23 55.42±1.93 0
比酶活是指每毫克酶蛋白所含的酶活力单位数,是衡量酶纯度的重要指标,对于同一种酶,比酶活越高,表明其催化活性越高。截短体LevH1-LC-NC比酶活最大[(541.66±4.16)U/mg],较亲本LevH1-LC[(50.89±1.66)U/mg]提高了10.6倍,说明截短体LevH1-LC-NC具有更好的水解效率。
Km反映酶与底物之间的亲和力,其值越大,表示酶对底物的亲和力越小。截短体LevH1-LC-NC的Km最小,为0.075 mol/L,表明同时截断C端和N端可以增大酶与底物的亲和力。截短体LevH1-LC-C的Km值最大,为0.14 mol/L,表明单独截断C端降低了酶与底物的亲和力。
有研究报道,改造后的酵母转化酶和天然转化酶的Km值分别为0.058 92 mol/L和0.125 45 mol/L[21],AVILA T L等[22]研究结果表明,假丝酵母转化酶在25 ℃和50 ℃下蔗糖的Km值分别产生30.5 mmol/L和28.7 mmol/L。OSIEBE O等[23]研究结果表明,酿酒酵母胞内转化酶的米氏常数Km为(5.85±1.72)mmol/L。和本实验结果相似。
Kcat是在酶被底物饱和的状态下,每秒钟每个酶分子转换底物的分子数,单位为1/s。Kcat值越大,表明酶的催化速率越高。截短体LevH1-LC-NC的Kcat最大,为43.80/s,表明其转化底物生成产物的转化率最大。
Kcat/Km称为催化效率,是衡量酶催化效率的一个重要指标。一个酶的Kcat/Km值越大,说明它的催化效率越高。截短体LevH1-LC-NC的Kcat/Km最大,为585.07 L/(mol·s),表明其催化效率最高,截短体LevH1-LC-C的Kcat/Km最小,为19.7 L/(mol·s),表明其催化效率最低。
综上,截短体LevH1-LC-NC为基于理性设计和截短工程所得到的优质转化酶突变体,为其工业化应用奠定了基础。
2.5 转化酶及其不同截短体的酶学性质
2.5.1 转化酶及其不同截短体的最适温度及热稳定性
为进一步考察转化酶突变体的最适温度,研究以蔗糖为底物模型,分别测定了亲本转化酶LevH1-LC及其截短体LevH1-LC-N、LevH1-LC-C、LevH1-LC-NC在20~60 ℃条件下转化酶活性及其20~90 ℃条件下的热稳定性,结果见图5。由图5a可知,LevH1-LC、LevH1-LC-N、LevH1-LC-NC的最适反应温度均为50 ℃,LevH1-LC-C的最适反应温度是40 ℃,较亲本降低了10 ℃。表明截短C端会引起转化酶的最适反应温度降低。由图5b可知,转化酶LevH1-LC及其截短体LevH1-LC-N、LevH1-LC-C、LevH1-LC-NC分别在20~90 ℃温浴30 min后,相对酶活均在5%以上。在60 ℃温浴30 min后,截短体LevH1-LC-N、LevH1-LC-C和LevH1-LCNC的相对酶活分别为70%、60%、60%,低于亲本转化酶LevH1-LC(80%)。结果表明,截短工程导致了转化酶热稳定性的降低,尤其是C端截短影响更为突出。
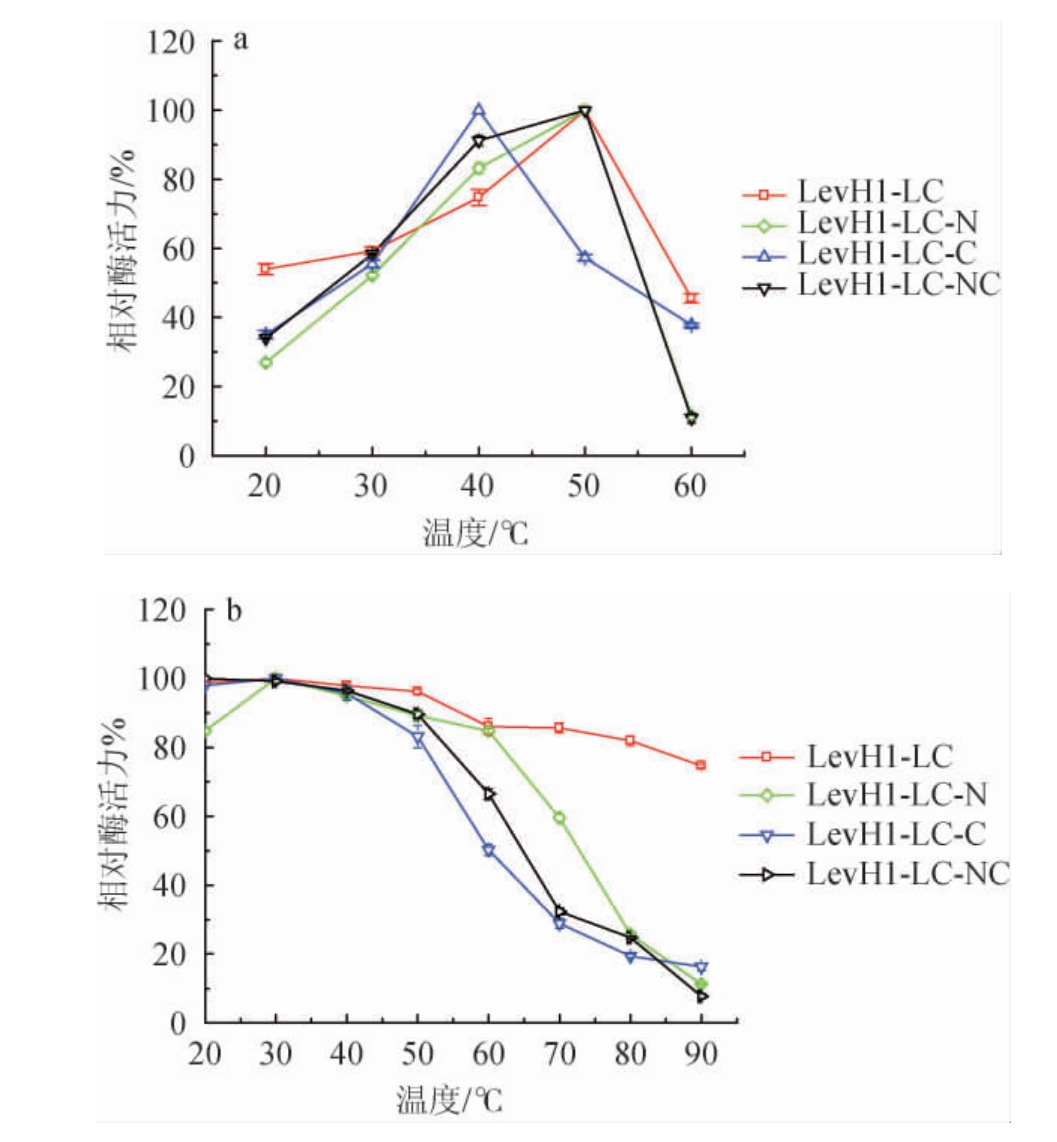
图5 转化酶及其突变体的最适反应温度(a)及热稳定性(b)
Fig.5 Optimum reaction temperature (a) and thermal stability (b) of invertase and its mutants
2.5.2 转化酶及其不同截短体的最适pH及pH耐受性
为进一步考察转化酶突变体的最适pH,研究以蔗糖为底物模型,分别测定了亲本转化酶LevH1-LC及其截短体LevH1-LC-N、LevH1-LC-C、LevH1-LC-NC 在pH3.0 ~12.0条件下转化酶活性及pH耐受性,结果见图6。由图6a可知,亲本转化酶LevH1-LC和LevH1-LC-N最适反应pH为5.0,LevH1-LC-C和LevH1-LC-NC最适反应pH为6.0,较亲本提高1个单位。由图6b可知,亲本转化酶LevH1-LC在pH 5.0~7.0时活性较为稳定,LevH1-LC-C在pH8.0时活性保持最好,但整体对环境酸碱性质更为敏感。LevH1-LC-NC在pH 7.0~8.0时相对酶活在80%以上,LevH1-LC-N在pH 8.0~10.0的环境中相对活性保持>80%。表明LevH1-LC为酸性转化酶,在酸性条件下保持稳定,而不同截短的转化酶突变体pH性质发生较大改变,都倾向在碱性条件下更为稳定。截短体LevH1-LC-NC整体上能适应更宽泛的pH条件。
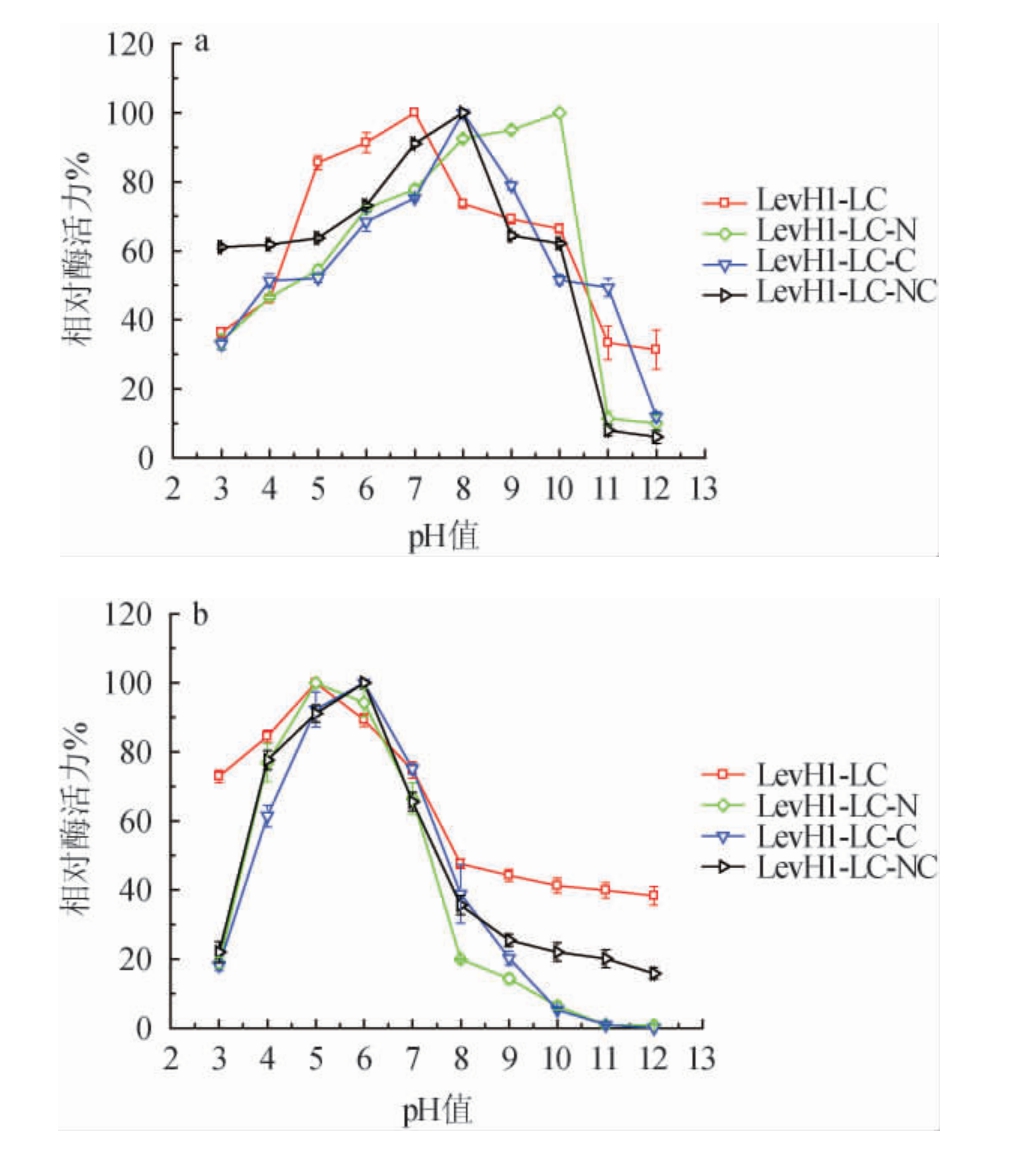
图6 转化酶及其突变体的最适反应pH(a)及pH耐受性(b)
Fig.6 Optimum reaction pH (a) and pH tolerance (b) of invertase and its mutants
由大量微生物产生的转化酶的Km值为0.063~470 mmol/L,Kcat为5.68~7.48/s,在30~48 ℃和pH2.9~6.2之间最佳[24]。DU L Q等[25]从宏基因组中克隆了耐酸性的转化酶基因,命名为Uninv2,以蔗糖为底物时,Uninv2的活性在pH4.5和45 ℃时最佳。截断Uninv2的N-末端结构域后,最佳pH值移至6.0,最适反应温度保持不变。Uninv2和截短体在37 ℃的中性pH值下保持高度稳定,在4 ℃的最佳pH值下稳定长达30 d。OSIEBE O等[23]研究结果表明,酿酒酵母转化酶50 ℃时显示出最大活性,在此温度下孵育1 h后活性没有下降。在pH值为5.0时,该酶表现出最佳活性,并且在酸性范围(pH值为3.0~6.0)内保持了60%以上的活性。AVILA T L等[22]研究酵母转化酶的最佳蔗糖水解条件,结果表明,最适反应温度和pH为50 ℃和pH 5.0,温度稳定性表明:其残余活性通常在40~70 ℃之间呈线性。最适反应温度和本研究结果一致,最适反应pH同未截短的亲本一致,较截短体小1个单位,原因可能是由于对基因进行了截短,截短体的性质发生了某些改变。
3 结论
本研究采用稀释涂布平板法和平板显色法从土壤样品中筛选分离出1株产转化酶的菌株LC23,经16S rRNA基因测序鉴定为干酪乳杆菌(Lactobacillus casei)。在此基础上,异源表达了一种新颖的转化酶LevH1-LC,通过理性设计和截短工程获得了4株截短体,截短体LevH1-LC-NC和LevH1-LC-N的酶活均高于亲本转化酶LevH1-LC和天然干酪乳杆菌表达的转化酶活性。截短体LevH1-LC-NC的最适反应温度和pH分别为50 ℃和6.0,在20~50 ℃残余酶活力在80%以上,在pH7.0~8.0残余酶活力在80%以上,其为基于理性设计和截短工程所得到的优质转化酶突变体。结果表明,同时截短N端和C端能够最大程度的提高了该转化酶的催化活性,N端对于比酶活影响更为显著,C端与温度性质关联性更强。这对于转化酶的进一步进化改造和实际应用具有积极作用。
[1]吴洪博,周丽娜,周雷进雨.干酪乳杆菌的特性及其在畜牧生产中的应用[J].现代畜牧兽医,2024(1):65-68.
[2]刘伊湄,周佳莹,韩言言,等.干酪乳杆菌YG-01株的益生特性及抑菌活性研究[J].中国畜牧兽医,2024,51(2):829-836.
[3]包秋华,王丽娜,张雨虹,等.益生菌干酪乳杆菌Zhang VBNC态和正常态的代谢组学研究[J].中国乳品工业,2022,50(6):10-15.
[4]REN C C,ZHANG Q X,DE HAAN B J,et al.Protective effects of lactic acid bacteria on gut epithelial barrier dysfunction are Toll like receptor 2 and protein kinase C dependent[J].Food Funct,2020,11(2):1230-1234.
[5]DOMINGOS-LOPES M F P,STANTON C,ROSS R P,et al.Histamine and cholesterol lowering abilities of lactic acid bacteria isolated from artisanal Pico cheese[J].J Appl Microbiol,2020,129(6):1428-1440.
[6]ZHU H,CAO C J,WU Z C,et al.The probiotic L.casei Zhang slows the progression of acute and chronic kidney disease[J].Cell metab,2021,33(10):1926-1942.
[7]MENÉNDEZ C, MARTÍNEZ D, PÉREZ R E, et al.Engineered thermostable β-fructosidase from Thermotoga maritima with enhanced fructooligosaccharides synthesis[J].Enzyme Microb Technol,2019,125:53-62.
[8]VU D P,RODRIGUES C M,JUNG B,et al.Vacuolar sucrose homeostasis is critical for plant development,seed properties,and night-time survival in Arabidopsis[J].J Exp Bot,2020,71(16):4930-4943.
[9]CHAUDET M M,AMIRI M,MARTH N,et al.Phylogenetic analysis reveals key residues in substrate hydrolysis in the isomaltase domain of sucrase-isomaltase and its role in starch digestion[J].Biochim Biophys Acta Gen Subj,2019,1863(9):1410-1416.
[10]DU F, LIU H, YIN X, et al.Potassium-mediated regulation of sucrose metabolism and storage root formation in sweet potato[J].Arch Agron Soil Sci,2021,67(5):703-713.
[11]OKAFOR C A,OKANLAWON T S,OLAGUNJU A M,et al.Invertase activities of lactic acid bacteria isolated from traditional fermented milk("nono"),agadagidi and palm wine obtained from different locations in Ile-Ife,Osun-State,Nigeria[J].GSC Biol Pharm Sci,2023,22(1):147-156.
[12]KUZUWA S, YOKOI K, KONDO M, et al.Properties of the inulinase gene levH1 of Lactobacillus casei IAM 1045; cloning, mutational and biochemical characterization[J].Gene,2012,495(2):154-162.
[13]KUSMIYATI N,SUNARTI S,WAHYUNINGSIH T D,et al.Inulinase activity of extracellular protein of Lactobacillus casei AP in different growth conditions[J].Key Eng Mater,2020,840(3):101-106.
[14]刘乙卿,俞如旺.现代酶工程技术概述[J].生物学教学,2024,49(4):10-13.
[15]TU T,LI Y Q,LUO Y,et al.A key residue for the substrate affinity enhancement of a thermophilic endo-polygalacturonase revealed by computational design[J].Appl Microbiol Biotechnol,2018,102:4457-4466.
[16]ZHENG J,LIU H Q,QIN X,et al.Identification and mutation analysis of nonconserved residues on the TIM-barrel surface of GH5_5 cellulases for catalytic efficiency and stability improvement[J].Appl Environ Microbiol,2022,88(17):e0104622.
[17]刘可玉,宁喜斌.耐盐碱性蛋白酶菌株LK-3的筛选及酶学性质[J].食品与生物技术学报,2024,43(3):46-53.
[18]LI J Y,XU X X,SHI P J,et al.Overexpression and characterization of a novel endo-β-1,3(4)-glucanase from thermophilic fungus Humicola insolens Y1[J].Protein Expr Purif,2017,138:63-68.
[19]BRADFORD M M.A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J].Anal Biochem,1976,72:248-254.
[20]KAUR M, TEOTIA P.Isolation, characterization and strain improvement of soil bacteria Bacillus subtilis for invertase production[J].Uttar Pradesh J Zoology,2021,42(18):97-104.
[21]黎春怡,黄卓烈,巫光宏,等.用壳寡糖修饰的方法提高酵母转化酶的催化活性[J].生物加工过程,2023,21(6):623-631.
[22]AVILA T L,TORALLES R P,JANSEN E T,et al.Extraction,purification and characterization of invertase from Candida guilliermondii isolated from peach solid wastes[J].Rev Bras Frutic,2022,44(2):849.
[23]OSIEBE O, ADEWALE I O, OMAFUVBE B O.Production and characterization of intracellular invertase from Saccharomyces cerevisiae(OL629078.1),using cassava-soybean as a cost-effective substrate[J].Sci Rep,2023,13:16295.
[24]NADEEM H, RASHID M H, SIDDIQUE M H, et al.Microbial invertases:a review on kinetics,thermodynamics,physiochemical properties[J].Process Biochem,2015,50(8):1202-1210.
[25]DU L Q, PANG H, WANG Z L, et al.Characterization of an invertase with pH tolerance and truncation of its N-terminal to shift optimum activity toward neutral pH[J].PLoS One,2013,8(4):e62306.