黄酒是我国的民族特产,与葡萄酒、啤酒并称为世界三大古酒,据考证中国黄酒生产至今已有近7 000年的历史[1]。我国黄酒主要以糯米、黍米等淀粉谷物作为基本原料,经过浸米、蒸煮、加曲、糖化发酵、压榨、过滤、煎酒、贮存、勾兑等工序而成[2]。其独特风味主要是由发酵剂(也称为酒曲)在发酵过程中分解谷物淀粉产生的酯类、有机酸类、氨基酸类及醇类等物质形成的。此外,黄酒还有一定的营养和保健作用,因为黄酒在酿造时还会产生很多种类的营养成分和保健活性成分[3]。
古语有云,“曲为酒之骨”,此曲即为酒曲[4]。酒曲中不仅含有黄酒酿造的主要微生物以及微生物产生的各类酶,而且对黄酒的风味和品质也有很大影响。黄酒在酿造过程中微生物群落结构复杂,酒曲中含有各种霉菌、细菌及酵母,加上开放式发酵环境中的微生物也会共同参与发酵,它们会在酿造过程中通过自身代谢合成各种酶类,这些酶能够降解淀粉等大分子物质,同时促进各类风味物质和营养成分的合成,赋予黄酒丰富的营养功能和独特的风味[5]。因此,系统研究黄酒酿造过程中的微生物组成及其特性,有利于黄酒发酵机理的深入揭示,对推动黄酒的品质提升和产业健康发展具有重要意义。
传统的培养分离是酿酒微生物群落结构研究最早采用的通用方法,该方法对于筛选出的微生物分类和鉴定非常准确,缺点是工作量很大,而且容易遗漏相对丰度较低的大部分微生物,从而不能获得完整的的微生物群落结构信息,因此研究结果具有一定的局限性[6]。现代分子生物技术的快速发展促进了基因芯片技术[7]、变性凝胶梯度电泳技术[8]及高通量测序技术[9]等新技术的发展和应用,这些新技术对于不可培养但却真实存活的菌群或细胞浓度较低菌群的检测、鉴定及定量分析有很大帮助。其中,高通量测序技术具有测序时间短、测序通量高等优点,目前已被国内外研究者广泛应用于酿酒微生物多样性的研究中[10-13]。CAI H Y等[14]采用高通量测序技术对8个发酵剂样品的微生物菌群组成进行分析,共鉴定出10个酵母属和霉菌属及11个细菌属;多样性分析结果表明,发酵剂微生物中真菌菌群主要以根霉属(Rhizopus sp.)为主。谷晓东等[15]以6种黄酒酒曲为原料,利用高通量测序技术从中共鉴定出247个细菌属和93个真菌属;其中,优势细菌属有乳酸杆菌属(Lactobacillus)、葡萄球菌属(Staphylococcus)等,优势真菌属有红曲霉属(Monascus)、曲霉菌属(Aspergillus)等。李斌等[4]利用高通量测序技术对南阳红谷黄酒酒曲中的红曲、大曲、小曲的微生物群落结构多样性进行分析,结果表明,微生物菌群多样性以大曲最高,红曲最低;红曲中优势细菌属为伯克霍尔德氏菌属(Burkholderia)、醋酸杆菌属(Acetobacter)等,优势真菌属为紫红曲霉(Monascus purpureus)、黑曲霉(Aspergillus niger)等;大曲中优势细菌属为克罗彭斯特菌属(Kroppenstedtia)、魏斯氏菌属(Weissella)等,优势真菌属为嗜热子囊菌(Thermoascus aurantiacus)、黄曲霉(Aspergillus flavus)等。目前,高通量测序技术在酿酒微生物中的应用,主要集中在揭示不同地区酒曲在主要微生物菌属的种类和比例差异,并发现不同酒曲中部分微生物菌属的种类及其与黄酒风味形成之间的相关性。
丹阳黄酒历史悠久,据考证酿造史已有1 700多年[16]。丹阳黄酒最早的文字记载见于晋代王嘉的《拾遗记》,称“云阳出美酒”,后至南北朝时期,以“曲阿美酒”闻名于世。丹阳黄酒色泽黄橙透明,口感爽口,香气醇厚。目前,关于丹阳黄酒酒曲中微生物菌群多样性研究鲜见报道。因此,本研究以丹阳某黄酒厂的酒曲为原料,利用Illumina Miseq高通量测序技术,研究酒曲微生物菌群的多样性,为进一步研究丹阳黄酒独特风味的形成与酒曲中微生物的关系奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
黄酒酒曲样本(HJQ):在江苏丹阳某黄酒酒业有限公司,从不同批次酒曲贮存容器中的内部和周围边缘区域分别取等质量的样品,样品均匀混合后,置于-20 ℃冰箱中保存备用。
1.1.2 试剂
Taq脱氧核糖核酸(deoxyribonucleic acid,DNA)聚合酶(5 U/μL):美国Thermo公司;E.Z.N.A.Soil DNA Kit提取试剂盒:美国OMEGA公司;SanPrep柱式DNA胶回收试剂盒、Qubit2.0 DNA检测试剂盒:南京擎科生物科技有限公司。其他试剂均为国产分析纯。
1.2 仪器与设备
5424R高速冷冻离心机:上海力申科学仪器有限公司;ME204E电子分析天平:瑞士梅特勒-托利多公司;T100聚合酶链式反应(polymerase chain reaction,PCR)扩增仪、GelDoc EZ电泳凝胶成像分析系统:美国伯乐公司;MX-S涡旋振荡仪:大龙兴创实验仪器(北京)股份公司;UV5Nano超微量紫外可见分光光度计:梅特勒-托利多仪器(上海)有限公司;Miseq高通量测序仪:美国Illumina公司。
1.3 方法
1.3.1 DNA的提取
采用E.Z.N.A.Soil DNA Kit试剂盒提取酒曲样品中微生物的总基因组DNA,用1%琼脂糖凝胶电泳和超微量分光光度计检测DNA的完整性和浓度。
1.3.2 PCR扩增及测序
以提取的总基因组DNA为模板,使用引物338F(5′-ACTCCTACGGG AGGCAGCA-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)对细菌的16S rDNA V3-V4高变区进行PCR扩增;使用引物ITS1F(5′-CTTGGTCATTTAGAGGAAGTAA-3′)和ITS2(5′-GCTGCGTTCTTCATCGATGC-3′)对真菌的ITS1区域进行PCR扩增[17]。PCR扩增条件:98 ℃预变性3 min;98 ℃变性30 s,50 ℃退火30 s,72 ℃延伸60 s,共30个循环;72 ℃再次延伸5 min,4 ℃保存。PCR扩增体系:10×PCR buffer 5 μL,脱氧核糖核苷三磷酸(deoxyribonucleoside triphosphate,dNTP)0.1 mmol/L,DNA 10 ng,正向及反向引物各0.5 μmol/L,Taq DNA聚合酶0.05 U,用双蒸水(ddH2O)补充至50 μL。
1.3.3 高通量测序
PCR扩增结束后,采用1.8%琼脂糖凝胶电泳检测扩增产物,再使用SanPrep柱式DNA胶回收试剂盒回收PCR扩增产物。利用Qubit2.0 DNA检测试剂盒对回收的PCR扩增产物进行精确定量,然后上机进行高通量测序。以上DNA的提取、PCR扩增及高通量测序均委托生工生物工程(上海)股份有限公司完成。
1.3.4 数据处理
测序得到的数据是双端序列,首先使用cutadapt去除引物接头序列,再使用PEAR将成对的reads拼接成一条序列,然后按照barcode标签序列和引物序列从拼接后数据中分割出各样本数据,并校正序列方向,使用PRINSEQ切除reads尾部质量值<20的碱基,设置10 bp的窗口,如果窗口内的平均质量值<20,从窗口开始截去后端碱基,过滤质控后的含N序列和短序列,最终过滤掉低复杂度的序列,得到各样本有效数据[12]。
使用Usearch软件,在97%的相似度条件下,对上述得到的优化序列进行操作分类单元(operational taxonomic units,OTUs)划分,然后绘制稀释曲线(rarefaction curves),计算样品中微生物的Alpha多样性指数,并统计分析细菌和真菌群落结构信息。
2 结果与分析
2.1 高通量测序结果分析
2.1.1 有效序列筛选
采用Illumina Miseq高通量测序平台得到黄酒酒曲样本中细菌和真菌的原始序列分别为90 664条和98 968条,其碱基平均长度分别为450.74 bp和298.13 bp,经拼接、过滤,分别得到有效序列89 697条和98 951条,其碱基平均长度分别为413.6 bp和256.14 bp。
2.1.2 OTU聚类及稀释曲线分析
在97%的相似度条件下,从黄酒酒曲样品中共划分到258个细菌OTUs和185个真菌OTUs,细菌OTU数大于真菌OTU数,表明细菌菌群丰度高于真菌菌群。随机抽取一定数量的序列,与其所代表的OTU数绘制稀释曲线,结果见图1。由图1可知,对于细菌菌群,当测序量<10 000时,OTU数快速增加,在测序量为10 000~80 000时,随着序列数的増加,OTU数目增加走势趋于平缓。对于真菌菌群,当测序量<5 000时,样本的OTU数快速增加,当测序量>10 000时,OTU数增加趋势减缓。结果表明,当前测序量足够且合理,测序结果可以反映样本中微生物多样性[2,18]。
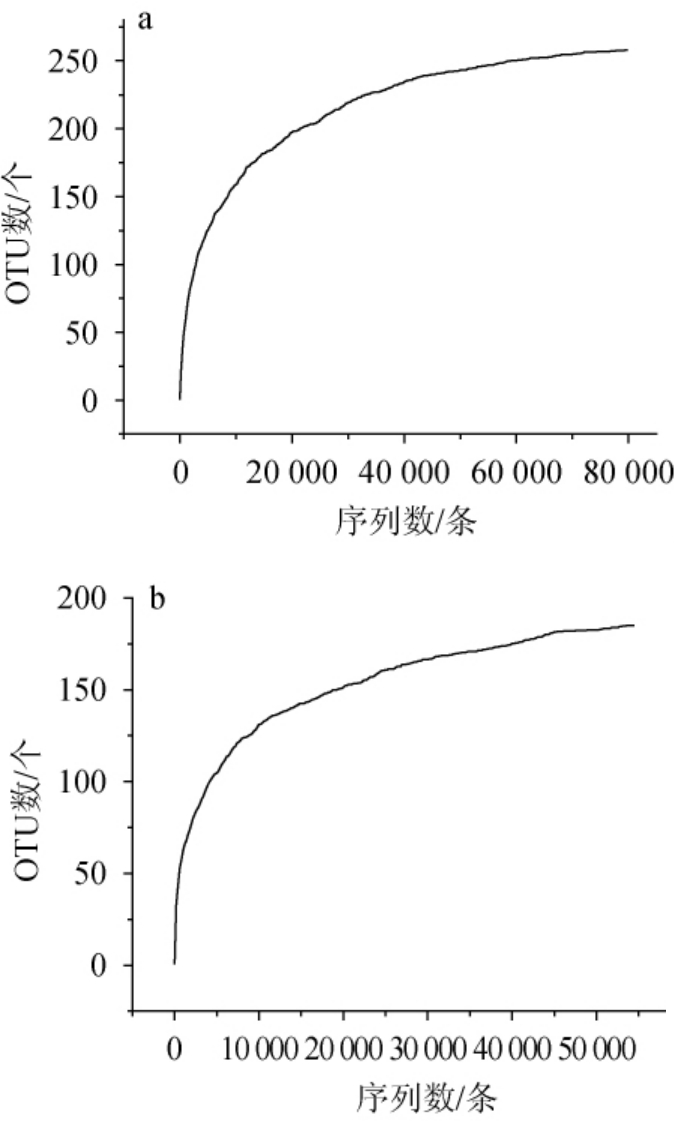
图1 基于操作分类单元黄酒酒曲样本中细菌(a)和真菌(b)菌群的稀释曲线
Fig.1 Rarefaction curves of bacteria (a) and fungi (b) in Huangjiu Jiuqu samples based on operational taxonomic unit
2.2 黄酒酒曲样本中微生物菌群多样性分析
黄酒酒曲样本中微生物菌群的Alpha多样性分析结果见表1。
表1 黄酒酒曲样品微生物菌群Alpha多样性分析结果
Table 1 Results of Alpha diversity of microbial community in Huangjiu Jiuqu samples

Shannon指数细菌真菌 真菌 真菌 真菌覆盖率/%细菌 真菌2.32 205.11 205.31 0.28 ACE指数细菌Chao1指数细菌Simpson指数细菌样品HJQ 2.06 261.86 258.57 0.24 99.99 99.95
微生物群落物种多样性通常使用Alpha多样性指数来表示,其中Shannon指数、ACE指数、Chaol指数、Simpson指数是最常用的,它们可以对一个群落的物种组成丰富度及均匀度进行综合性的评价[17]。其中,Shannon指数值越大,则表示群落中物种的多样性越高,而Simpson指数与Shannon指数意义相反,其值越大说明群落多样性越低;Chaol指数或ACE指数越大则表明群落物种丰富度越高[19]。由表1可知,黄酒酒曲样品中细菌和真菌菌群的Shannon指数分别为2.06和2.32,ACE指数分别为261.86和205.11,Chao1指数分别为258.57和205.31,Simpson指数分别为0.24和0.28,说明黄酒酒曲样品中,在群落物种多样性上真菌菌群高于细菌菌群,而在群落物种丰富度上细菌菌群则高于真菌菌群。由覆盖率的数值可知实验的取样是否合理,一般认为覆盖率≥97%时,则证明取样是合理的[20]。由表1亦可知,细菌和真菌菌群的覆盖率分别达到99.99%和99.95%,均高于97%,说明本次取样合理。
2.3 黄酒酒曲样本中微生物群落结构分析
2.3.1 细菌群落结构分析
黄酒酒曲样品中细菌群落结构分析结果见图2。由图2a可知,在门水平上,黄酒酒曲样本中共注释到7个细菌门,其中有3个优势细菌门(相对丰度≥1%),分别为厚壁菌门(Firmicutes)(38.0%)、变形杆菌门(Proteobacteria)(27.7%)、蓝细菌门(Cyanobacteria)(6.5%)。由图2b可知,在属水平上,黄酒酒曲样本中共注释到10个细菌属,其中有8个优势细菌属(相对丰度≥1.0%),分别为乳杆菌属(Lactobacillus)(39.2%)、魏斯氏菌属(Weissella)(14.6%)、葡萄球菌属(Staphylococcus)(5.9%)、肠杆菌属(Enterobacter)(4.2%)、不动杆菌属(Acinetobacter)(2.1%)、泛菌属(Pantoea)(1.2%)。有研究发现,乳杆菌属产酸能力突出,对黄酒风味的形成有举足轻重的作用,乳酸杆菌在高酸环境中还能产生大量的乳酸脱羧酶,有利于合成γ-氨基丁酸[21]。有研究表明,魏斯氏菌属与黄酒风味物质中的癸酸和异戊醇呈正相关性[22]。其他优势菌属在文献中也有报道[23-25]。
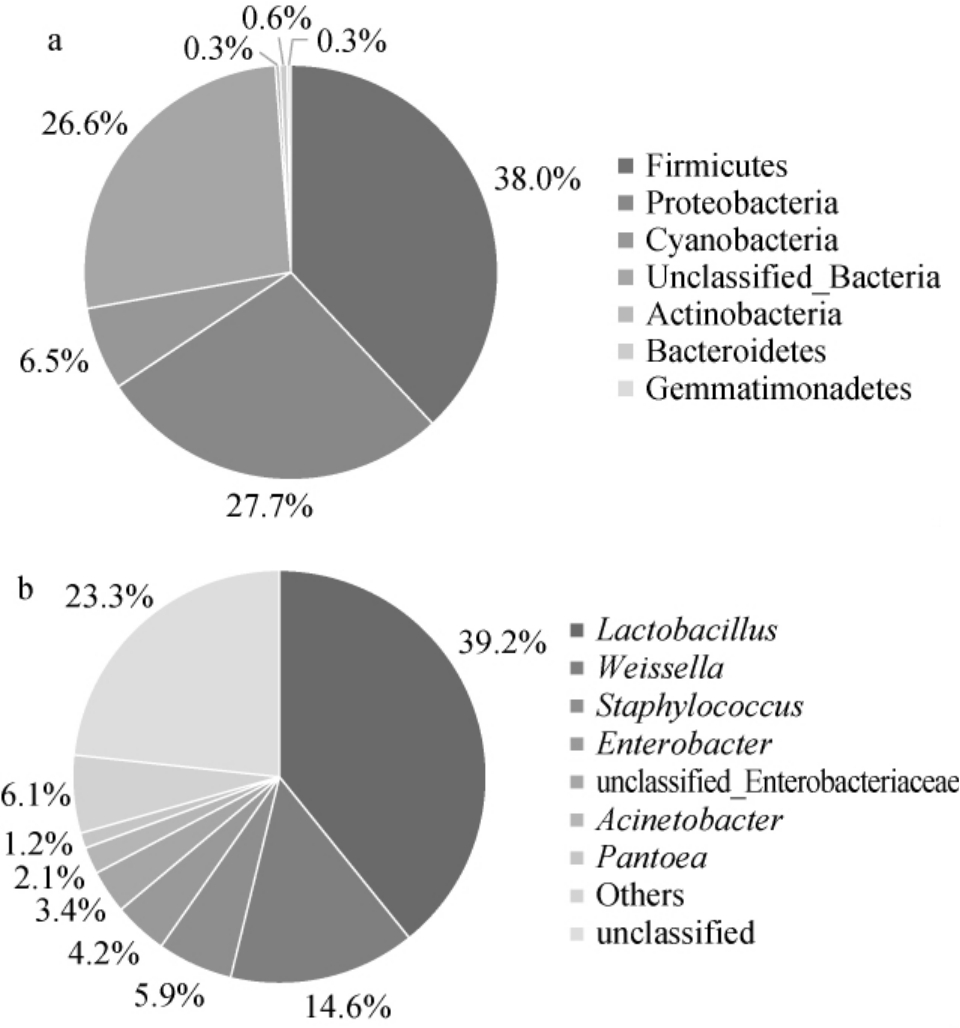
图2 基于门(a)和属(b)水平黄酒酒曲样品中细菌菌群结构
Fig.2 Bacterial community structure in Huangjiu Jiuqu samples based on phylum (a) and genus (b) levels
Others代表相对丰度<1%的其他细菌属。
2.3.2 真菌群落结构分析
黄酒酒曲样本中真菌群落结构分析结果见图3。由图3a可知,在门水平上,黄酒酒曲样本中共注释到6个真菌门,其中有3个优势真菌门(相对丰度≥1%),分别为子囊菌门(Ascomycota)(91.8%)、担子菌门(Basidiomycota)(4.7%)、毛霉门(Mucoromycota)(3.1%)。由图3b可知,在属水平上,黄酒酒曲样本中共注释到14个真菌属,其中有10个优势真菌属(相对丰度≥1.0%),分别为曲霉属(Aspergillus)(59.3%)、短梗霉属(Aureobasidium)(8.7%)、芽枝霉属(Cladosporium)(4.9%)、威克汉姆酵母属(Wickerhamomyces)(4.6%)、青霉属(Penicillium)(3.5%)、木拉克酵母属(Mrakia)(2.6%)、根霉属(Rhizopus)(2.4%)、镰刀菌属(Fusarium)(1.9%)、帚枝霉属(Sarocladium)(1.4%)、篮状菌属(Talaromyces)(1.3%)。
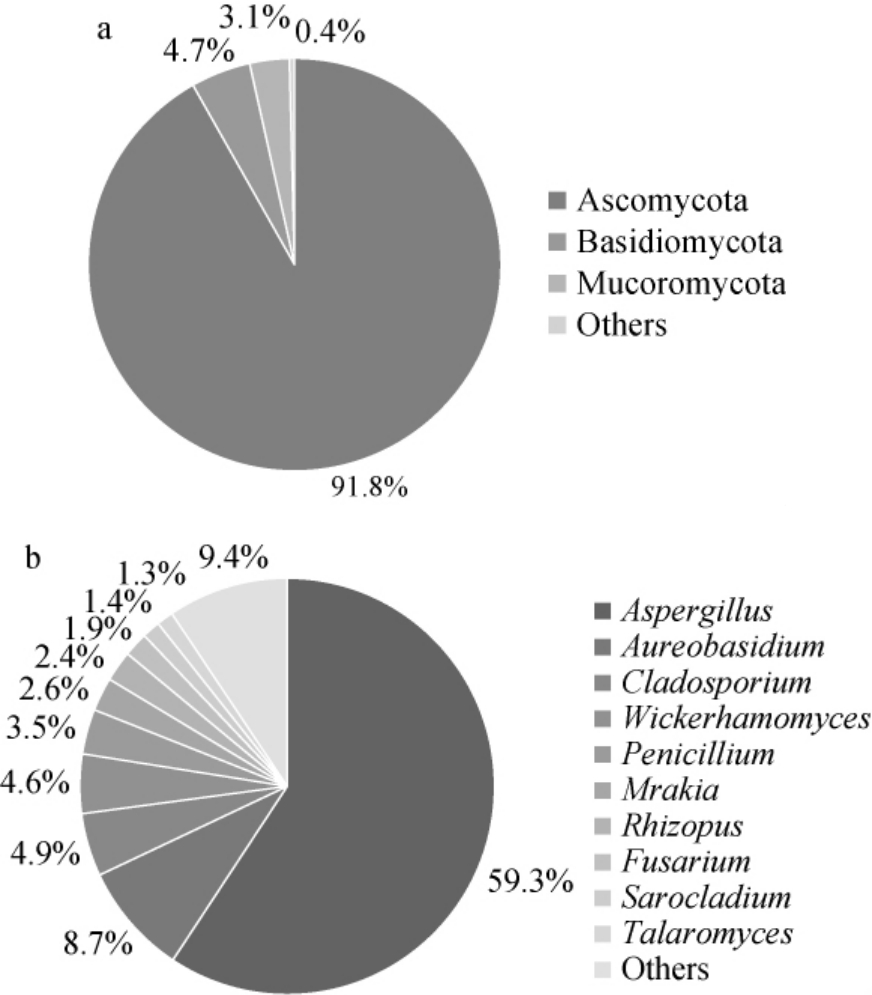
图3 基于门(a)和属(b)水平黄酒酒曲样本中真菌菌群结构
Fig.3 Fungal community structure in Huangjiu Jiuqu samples based on phylum (a) and genus (b) levels
Others代表相对丰度<1%的其他真菌门或真菌属。
本研究中子囊菌门为绝对优势菌门,其次是担子菌门与毛霉门,这与凌梦荧[26]的研究结果一致。在属水平上,曲霉属(Aspergillus)为优势菌属,其属于半知菌亚门(Deuteromycotina)、丝孢纲(Hyphomycetes)、丝孢目(Hy phomycetes)、从梗孢科(Moniliaceae),能够产生淀粉酶、蛋白酶、糖化酶等,有利于淀粉糖化、蛋白质分解[27]。此外,威克汉姆酵母(Wickerhamomyces)属于非酿酒酵母,可以合成多种酶催化各种前体物质转化成酯、酸、高级醇和醛等风味物质,有利于黄酒风味的形成[28]。本研究中发现的短梗霉属(Aureobasidium)、芽枝霉属(Cladosporium)、木拉克酵母属(Mrakia)、帚枝霉属(Sarocladium)、篮状菌属(Talaromyces)在酒曲微生物的研究报道中并不常见,有待进一步研究。
3 结论
本研究基于Illumina MiSeq高通量测序技术对江苏丹阳黄酒酒曲中的微生物菌群多样性进行研究。发现酒曲样品中真菌菌群的多样性高于细菌,而丰富度低于细菌。从样品中共注释7个细菌门,10个细菌属;6个真菌门,14个真菌属。其中,优势细菌属(相对丰度≥1%)为乳杆菌属(Lactobacillus)、魏斯氏菌属(Weissella)、葡萄球菌属(Staphylococcus)、肠杆菌属(Enterobacter)、不动杆菌属(Acinetobacter)、泛菌属(Pantoea)等;优势真菌属(相对丰度≥1%)为曲霉属(Aspergillus)、短梗霉属(Aureobasidium)、芽枝霉属(Cladosporium)、威克汉姆酵母属(Wickerhamomyces)、青霉属(Penicillium)、木拉克酵母属(Mrakia)、根霉属(Rhizopus)、镰刀菌属(Fusarium)、帚枝霉属(Sarocladium)、篮状菌属(Talaromyces)等。其中,短梗霉属、篮状菌属均为酒曲微生物中报道较少的属,其特性和功能尚不清晰,有待进一步深入的研究。本研究结果为丹阳黄酒的风味和工艺的进一步研究提供了一定的依据,具有重要的理论和实践意义。
[1]杜贞娜,单之初,沈赤,等.传统绍兴黄酒酿造真菌群落与多样性发生机制[J].应用生态学报,2022,33(4):1118-1124.
[2]谷晓东,刘怡琳,席晓丽,等.基于高通量测序技术对6种黄酒酒曲中微生物多样性的研究[J].食品工业科技,2022,43(16):148-157.
[3]杜贞娜,程斐,单之初,等.高通量测序技术及其在黄酒微生物多样性研究中的应用[J].中国酿造,2021,40(8):14-19.
[4]李斌,陈亚楠,孙旭,等.Illumina MiSeq高通量测序分析红谷黄酒酒曲微生物菌群多样性[J].中国酿造,2022,41(9):86-92.
[5]CHEN C,LIU Y,TIAN H,et al.Metagenomic analysis reveals the impact of Jiuyao microbial diversity on fermentation and the volatile profile of Shaoxing-jiu[J].Food Microbiol,2020,86:103326.
[6]陈一钒,吴余宁,徐春燕,等.基于高通量测序技术分析黄酒微生物多样性的研究进展[J].现代食品,2020(23):38-43.
[7]ANDORRA I,LANDI S,MAS A,et al.Effect of fermentation temperature on microbial population evolution using culture independent and dependent techniques[J].Food Res Int,2010,43(3):773-779.
[8]FIERER N,LAUBER C L,ZHOU N,et al.Forensic identification using skin bacterial communities[J].Proc Natl Acad Sci,2010,107(14):6477-6481.
[9]吴成,王春晓,王晓丹,等.高通量测序技术在酿酒微生物多样性研究中的应用[J].食品科学,2019,40(3):348-355.
[10]PORTILLO M D C,FRANQUÈS J,ARAQUE I,et al.Bacterial diversity of Grenache and Carignan grape surface from different vineyard at Priorat wine region(Catalonia,Spain)[J].Int Food Microbiol,2016,219:56-63.
[11]WANG X D, BAN S D, HU B D, et al.Bacterial diversity of Moutaiflavour Daqu base on high-throughput sequencing method[J].J I Brewing,2017,123(1):138-143.
[12]李斌,闫志鹏,李慧星,等.基于高通量测序技术的浓香型和芝麻香型白酒酒曲细菌群落结构分析[J].中国酿造,2018,37(8):148-152.
[13]席啦,孔祥聪,杨少勇,等.基于Illunima Miseq测序技术的黄酒麦曲真菌多样性分析[J].食品研究与开发,2022,43(12):185-190.
[14]CAI H Y,ZHANG T,ZHANG Q,et al.Microbial diversity and chemical analysis of the starters used in traditional Chinese sweet rice wine[J].Food Microbiol,2018,73:319-326.
[15]谷晓东,刘怡琳,席晓丽,等.基于高通量测序技术对6种黄酒酒曲中微生物多样性的研究[J].食品工业科技,2022,43(16):148-157.
[16]乔晓月,靳迎春,曹喜涛,等.白藜芦醇丹阳黄酒抗氧化活性和稳定性研究[J].中国酿造,2018,37(7):117-121.
[17]李斌,胡俊杰,张兰兰,等.基于高通量测序浓香型和芝麻香型白酒酒曲真菌群落结构的分析[J].中国酿造,2019,38(10):96-100.
[18]牟穰.清爽型黄酒酿造微生物群落结构及其与风味物质相关性研究[D].无锡:江南大学,2015.
[19]罗爱国,郗鑫瑞,郑同庆,等.基于Illumina MiSeq高通量测序分析清香型白酒酒糟微生物群落多样性[J].中国酿造,2022,41(1):98-102.
[20]庞春霞,李艺,虞任莹,等.基于Illumina Miseq 技术比较不同地区传统发酵大豆制品细菌多样性[J].食品工业科技,2022,43(8):133-140.
[21]杜丹,解修超,李新生,等.黄酒酒曲微生物及其代谢产物的研究进展[J].生物资源,2019,41(2):104-111.
[22]陈麒名,冯霞,张蓓蓓,等.中国黄酒的微生物多样性与风味的研究进展[J].食品与发酵科技,2021,57(6):77-82.
[23]李艳,董振玲,牟德华.羊羔美酒大曲中酵母菌多样性及分子鉴定[J].食品科学,2014,35(5):144-149.
[24]郑亚伦,赵婷,王家胜,等.数字化高温大曲发酵过程中微生物群落结构的变化[J].食品科学,2022,43(12):171-178.
[25]JIN Y, LI D Y, ZHOU R Q.Correlation between volatile profiles and microbial communities:A metabonomic approach to study Jiang-flavor liquor Daqu[J].Food Res Int,2019,121:422-432.
[26]凌梦荧.绍兴黄酒麦曲的关键指标筛选及品质评价方法研究[D].无锡:江南大学,2019.
[27]赵中开,龙可,马莹莹,等.米曲霉菌在酿酒工业中的研究进展[J].现代食品科技,2013,29(4):932-935.
[28]简晓平,简章容,余丽辰,等.1株Wickerhamomyces酵母菌对浓香型白酒发酵的影响[J].酿酒科技,2017(9):33-38.