弧菌属是一群菌体短小,弯曲成弧形的革兰氏阴性菌,其污染水产品对人体健康有极大危害[1],典型临床症状为发热、腹泻、呕吐,甚至死亡[2]。致病性弧菌随水产品进入人体消化系统侵染细胞前,需要耐受人体内生理环境的筛选和胁迫[3-5],而细菌内的应激调控因子决定了其对环境胁迫的耐受能力[6-7]。因此,调控因子中应激信号的传导对保障水产品安全有重要意义。激酶RsbT是应激信号传导通路中的开关蛋白[8-9],在受环境胁迫后会被激活并磷酸化,但在巴西弧菌(Vibrio brasiliensis)中,RsbT如何将信号传导给下游的首个蛋白,RsbU蛋白仍然未知[10]。前期研究发现,当革兰氏阳性细菌枯草芽孢杆菌(Bacillus subtilis)受到环境胁迫,枯草芽孢杆菌B因子调控蛋白U(Bacillus subtilis regulator of sigma B protein U,bsRsbU)参与的可逆磷酸化信号转导系统使细菌在复杂环境中生存下来,这是因为菌体应激后,枯草芽孢杆菌B因子调控蛋白T(Bacillus subtilis regulator of sigma B protein T,bsRsbT)被激活,在下游和bsRsbU结合形成一个具有磷酸酶活性的蛋白-蛋白复合体,胁迫信号得以传导到下游[11-14]。巴西弧菌(Vibrio brasiliensis)属于革兰氏阴性菌,其信号传导通路下游基因序列与枯草芽孢杆菌不同[15],不能确定环境胁迫激活的蛋白激酶RsbT能否通过蛋白磷酸化的形式,将应激信号传递给磷酸酶RsbU,且RsbT和RsbU间的蛋白互作机制也未知[8]。
本研究以巴西弧菌(Vibrio brasiliensis)为模式菌株,通过构建载体表达和纯化RsbU,并采用SDS-PAGE 凝胶电泳和Bradford 试剂盒测定其纯度、分子质量和浓度。依托美国国家生物技术信息中心(national center of biotechnology information,NCBI)、ProtParam、Prosite等软件分析了RsbU的结构域、基本理化性质、功能位点、二级结构和三级结构,并用Discovery studio软件将RsbU与RsbT进行分子对接,采用蛋白互作网络分析数据库String得到蛋白互作图谱。对RsbU的酶学特性进行研究,用丝/苏氨酸(Serine/Threonine,Ser/Thr)试剂盒测定RsbU的酶活性并绘制酶反应动力学曲线;设计RsbU和RsbT的体外磷酸化反应体系并通过Westernblot探究两者间的互作机制,探究两者间的信号传导,为上下游之间的应激信号通路传导提供理论依据。
1 材料与方法
1.1 材料与试剂
丝/苏氨酸试剂盒:Promega 公司;大肠杆菌(Escherichia coli)BL21(DE3)感受态细胞:北京全式生物技术股份有限公司;重组载体构建相关试剂盒:通用生物系统(安徽)有限公司;Bradford 蛋白浓度测定试剂盒、Tris-HCl 缓冲液:上海沃凯生物技术有限公司;p-硝基甲磺酸(p-nitrobenzyl mesylate,PNBM)(纯度>99%)、兔单克隆抗体[5-8]、山羊抗兔LgG H&L辣根过氧化物酶(horseradish peroxidase,HRP):英国Abcam 公司。
1.2 仪器与设备
690BR027447型全能型蛋白转印系统转膜仪:美国伯乐公司;GenoSens 1880型化学发光成像仪:上海勤翔科学仪器有限公司;JY92-11N型超声波细胞粉碎机:北京天林恒泰科技有限公司;TS-1型脱色摇床:海门市其林贝尔仪器制造有限公司;DYY-6C型电泳仪:北京市六一仪器厂;T100 型聚合酶链反应(polymerase chain reaction,PCR)仪:美国Bio-Bad公司。
1.3 方法
1.3.1 RsbU蛋白表达载体的构建
根据NCBI中rsbU基因的CDS序列设计引物。聚合酶链反应(PCR)扩增获得开放阅读框后,添加双酶切位点和正反向引物。PCR扩增引物并进行琼脂糖凝胶电泳分离产物。用PCR凝胶回收试剂盒回收目的条带,双酶切后回收产物,并与rsbU基因片段连接生成重组质粒,转化进感受态细胞中。将感受态细胞传代两次后获得重组质粒菌液,以此为模板使用鉴定引物(同扩增引物)进行PCR扩增。将产物进行凝胶电泳后用质粒提取试剂盒提取符合预期的阳性克隆。提取重组质粒后测序并进行蛋白质的翻译和序列比较,并再次双酶切鉴定。获得RsbU蛋白表达载体后,于-20 ℃保存重组质粒。
四种蛋白表达载体的酶切位点、测序引物序列和引物序列见表1。
表1 四种RsbU蛋白表达载体的酶切位点、测序引物序列及引物序列
Table 1 Enzyme digestion sites, sequencing primer sequences and primer sequences of 4 RsbU protein expression vectors
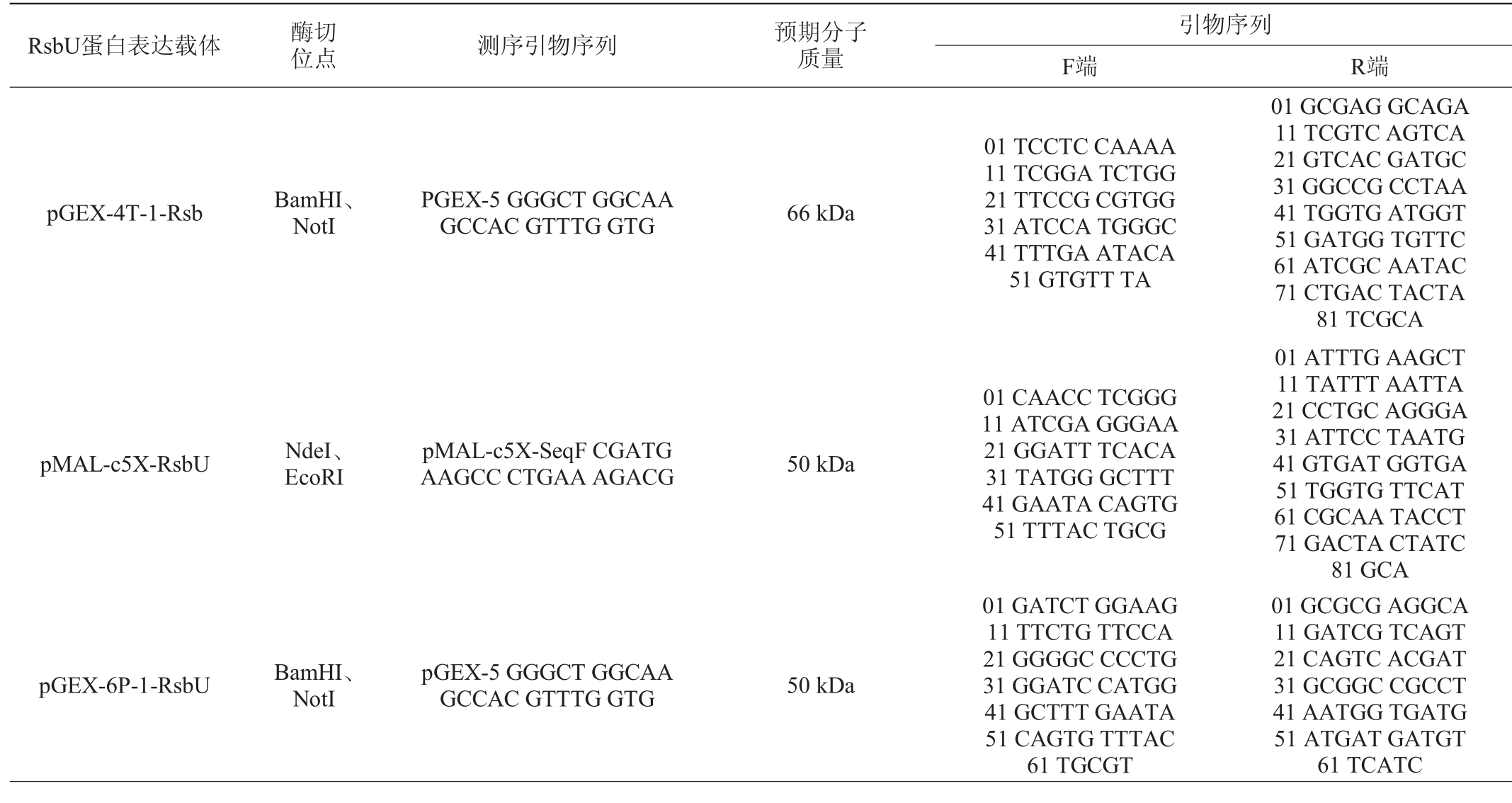
RsbU蛋白表达载体酶切位点测序引物序列预期分子质量引物序列F端R端pGEX-4T-1-Rsb BamHI、NotI PGEX-5 GGGCT GGCAA GCCAC GTTTG GTG 66 kDa 01 TCCTC CAAAA 11 TCGGA TCTGG 21 TTCCG CGTGG 31 ATCCA TGGGC 41 TTTGA ATACA 51 GTGTT TA pMAL-c5X-RsbU NdeI、EcoRI pMAL-c5X-SeqF CGATG AAGCC CTGAA AGACG 50 kDa 01 CAACC TCGGG 11 ATCGA GGGAA 21 GGATT TCACA 31 TATGG GCTTT 41 GAATA CAGTG 51 TTTAC TGCG pGEX-6P-1-RsbU BamHI、NotI pGEX-5 GGGCT GGCAA GCCAC GTTTG GTG 50 kDa 01 GATCT GGAAG 11 TTCTG TTCCA 21 GGGGC CCCTG 31 GGATC CATGG 41 GCTTT GAATA 51 CAGTG TTTAC 61 TGCGT 01 GCGAG GCAGA 11 TCGTC AGTCA 21 GTCAC GATGC 31 GGCCG CCTAA 41 TGGTG ATGGT 51 GATGG TGTTC 61 ATCGC AATAC 71 CTGAC TACTA 81 TCGCA 01 ATTTG AAGCT 11 TATTT AATTA 21 CCTGC AGGGA 31 ATTCC TAATG 41 GTGAT GGTGA 51 TGGTG TTCAT 61 CGCAA TACCT 71 GACTA CTATC 81 GCA 01 GCGCG AGGCA 11 GATCG TCAGT 21 CAGTC ACGAT 31 GCGGC CGCCT 41 AATGG TGATG 51 ATGAT GATGT 61 TCATC
续表

RsbU蛋白表达载体酶切位点测序引物序列预期分子质量引物序列F端R端pSmart-I-RsbUBamHI、HindIII pSmart-seqF TTAAG ATTCT TGTAC GACGG pET-seqF GGCCC CAAGG GGTTA TGCTA GT 35 kDa 01 GAGGC TCACA 11 GAGAA CAGAT 21 TGGTG GCCAA 31 GGATC CATGG 41 GCTTT GAATA 51 CAGTG TTTAC 61 TGCGT ACCG 01 GTGGT GGTGG 11 TGGTG CTCGA 21 GTGCG GCCGC 31 AAGCT TCTAT 41 TCATC GCAAT 51 ACCTG ACTAC 61 TATCG CAGT
1.3.2 重组蛋白的原核表达和电泳
将测序正确的RsbU蛋白表达载体转化进大肠杆菌(Escherichia coli)BL21(DE3)感受态细胞,转化完成后使用含氨苄青霉素、氯霉素的固体培养基进行一次传代鉴定,获得阳性菌株后进行二次传代。传代结束取出部分菌液作为诱导前对照样品。传代结束后加入异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG)诱导,诱导结束取出部分菌液作为诱导后样品。诱导前、后样品保存于4 ℃,其余菌液离心收集沉淀菌株,液氮速冻保存于-80 ℃。向菌株中加入蛋白稀释缓冲液、苯甲脒盐酸水合物、丝氨酸抑制剂在冰上重溶,溶解后加入脱氧核糖核酸(deoxyribonucleic acid,DNA)酶进行水解,完成后破碎细胞收集上清和沉淀。
将诱导前、后样品,上清和沉淀进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulphate-polyacrylamide gel electrophoresis,SDS-PAGE)凝胶电泳并比对蛋白条带情况。若蛋白在上清中含量高不需要复性,若蛋白在沉淀中含量高则通过包涵体蛋白复性再次收集。
1.3.3 蛋白的纯化及蛋白的纯度、分子质量及浓度检测
使用镍柱(6.35 mm×6.35 mm),设置泵压力为4.0 MPa,用浓度分别为25 mmol/L、50 mmmol/L、100 mmmol/L、150 mmmol/L、200 mmmol/L、250 mmmol/L的咪唑梯度洗脱蛋白,收集能使考马斯亮蓝变蓝的洗脱液并透析、超滤脱盐浓缩纯化蛋白。纯化的蛋白适度稀释后进行SDSPAGE凝胶电泳检测蛋白的纯度及分子质量。用Bradford试剂盒测定并计算蛋白浓度。
1.3.4 结构域预测与RsbU的基本理化性质分析
根据RsbU的氨基酸序列,采用NCBI数据库中Conserved Domain Database在线软件对巴西弧菌rsbU基因序列中的功能结构域进行预测。再采用Proparam对RsbU的基本理化性质等进行分析,并用Protscale分析RsbU的亲疏水性,选用参数Hphob./Kyte&Doolittle计算得到亲疏水打分值[15]。
1.3.5 RsbU的功能位点分析
根据RsbU的氨基酸序列,采用NetPHos 3.1和NetNGlyc 1.0分别分析RsbU蛋白潜在的磷酸化位点和N-糖基化位点;采用Prosite预测RsbU蛋白的功能位点,并分析作用。
1.3.6 RsbU的二级结构、三级结构预测
根据RsbU的氨基酸序列,采用Sompa进行RsbU二级结构的预测。
利用NCBI蛋白质结构数据库,限定E-value>e-5,筛选建模模板并进行序列比对后进行模型构建。通过DOPE值选择较优模型,优化完成后用拉氏图评价、verify 3D法对其结构进行评估。
1.3.7 RsbU蛋白的分子对接与蛋白互作分析
从Pubchem数据库下载对硝基苯磷酸二钠(p-nitrophenyl phosphate disodium,pNPP)的3D结构,通过Chamdraw3D中的MM2操作进行能量最小化预处理,运用Autodock tools 1.5.6和pymol软件对RsbU模型进行修改,使蛋白可视化,用Autodock-vina1.1.2对RsbU和pNPP进行半柔性分子对接,将RsbU设置为刚性,pNPP设置为柔性,对接算法选用拉马克遗传算法。
对得到RsbT的三维结构模型进行评价。采用Discovery Studio软件中的ZDock模块进行蛋白刚性对接,受体蛋白为RsbT,配体蛋白为RsbU,设置Angular Step Size为15,采样角度为15°,将RMSD Cutoff的cluster半径设置为10.0 Å、将Interface Cutoff 设置为10.0 Å,最大聚类数为100,对接结果产生2000 poses。运用Top Roses in Largest Clusters工具,列出打分较好的前100个构象,通过Cluster-Zdock score图,筛选ZDock score>17的关键构型,通过RDock精准对接,确定最优对接。
用String进行蛋白互作分析,选取与RsbU互作性最强的10种蛋白形成蛋白互作图谱并分析互作关系。
1.3.8 RsbU的酶学性质研究
(1)金属离子对酶活性的影响
在150μL的Tris-HCl缓冲液(pH 7.0)中加入20 μLpNPP、10 μL浓度为40 mmol/L的不同金属离子(Zn2+、Ca2+、Mg2+和Mn2+)盐溶液以及20 μL初始浓度7.5 μmol/L的RsbU-GST。在该体系中探究不同金属离子对RsbU-GST酶活性的影响。在室温下进行反应,反应过程中连续测定体系的OD405nm值。
(2)金属离子浓度对酶活性的影响
在150μL的Tris-HCl缓冲液(pH 7.0)中加入20 μLpNPP、10 μL浓度分别为20 mmol/L、40 mmol/L、80 mmol/L、100 mmol/L、120 mmol/L、160 mmol/L的最适金属离子盐溶液以及20 μL初始浓度7.5 mmol/L的RsbU-GST。在该体系中探究最适金属离子的浓度对RsbU-GST酶活性的影响。在室温下反应,测定反应60 min 的OD405nm值。
(3)最适反应pH
在150 μL pH 分别为3.0、4.0、5.0、6.0、7.0、8.0、9.0、10.0的Tris-HCl缓冲液中加入20 μL pNPP、10 μL最适摩尔浓度的最适金属离子的盐溶液以及20 μL初始浓度7.5 μmmol/L的RsbU-GST。在该体系中探究RsbU-GST酶活性的最适反应pH。在室温下反应,测定反应60 min的OD405nm值[17]。
(4)最适反应温度
在150 μL pH为最适pH的Tris-HCl缓冲液中加入20 μL pNPP、10 μL最适摩尔浓度的最适金属离子的盐溶液以及20 μL初始浓度7.5 μmmol/L的RsbU-GST。在该体系中探究RsbU-GST酶活性的最适反应温度。设置梯度反应温度为4 ℃、18 ℃、27 ℃、37 ℃、47 ℃、52 ℃、60 ℃,测定反应60 min的OD405nm值。
1.3.9 RsbU与RsbT的互作分析
将RsbT体外磷酸化并通过Western-blot(蛋白质印迹法)获得标准免疫印迹条带。设置B因子调控蛋白U-谷胱甘肽S-转移酶融合蛋白(regulator of sigma B protein U-glutathione S-transferase fusion protein,RsbU-GST)与RsbT共同体外磷酸化反应体系;设置RsbU与富集后的RsbT共同体外磷酸化反应的体系;设置B因子调控蛋白U-鸟苷酸结合蛋白(regulator of sigma B protein U-guanine nucleotide-binding protein,RsbU-G)与RsbT共同体外磷酸化反应体系;三组体系均通过Western-blot(聚丙酰胺电泳)获得相应的免疫印迹条带。应用ImageJ.JS软件对标准免疫印迹条带和各组反应的免疫印迹条带进行灰度分析,并将各组反应的灰度与标准条带的灰度进行比对,探究RsbU与RsbT的互作关系。
2 结果与分析
2.1 RsbU表达载体构建与蛋白的纯化结果
RsbU表达载体构建与蛋白的纯化结果见图1。由图1可知,四种表达载体pGEX-4T-1-RsbU、pMAL-c5X-RsbU、pGEX-6P-1-RsbU和pSmart-1-RsbU在第二次双酶切后得到的条带610 bp 和4 939 bp,663 bp 和5 581 bp,610 bp 和4 954 bp,953 bp 和5 207 bp,均符合预期长度,表明rsbU基因已成功插入克隆载体中。
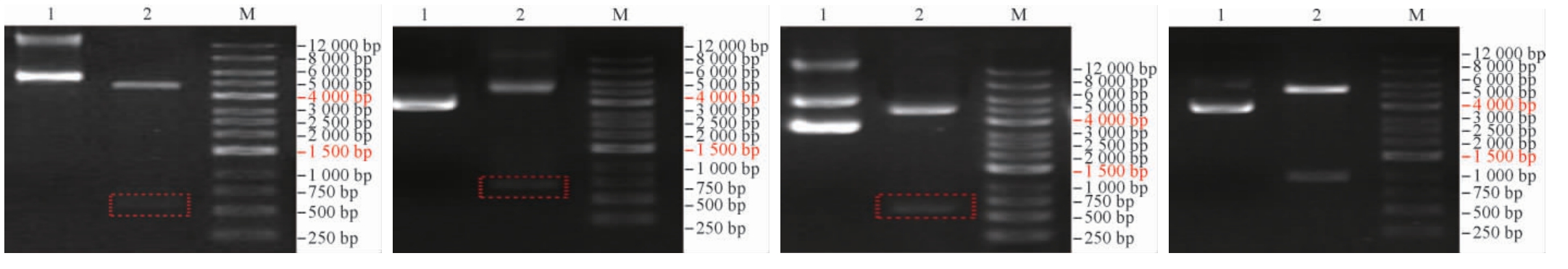
图1 重组质粒表达酶切结果
Fig.1 Enzyme digestion results of recombinant plasmid expression
M为Marker;1为重组质粒;2为酶切质粒。
四种重组表达载体pMAL-c5X-RsbU、pGEX-4T-1-RsbU、pGEX-6P-1-RsbU和pSmart-1-RsbU在诱导前、诱导后、上清、沉淀中的SDS-PAGE结果见图2。由图2可知,pMAL-c5XRsbU诱导后在上清中较好表达;pGEX-4T-1-RsbU在上清中部分表达;pGEX-6P-1-RsbU、pSmart-1-RsbU 诱导后主要存在于包涵体,但后者表达效果优于前者。

图2 重组载体诱导前后十二烷基硫酸钠-聚丙烯酰胺凝胶电泳结果
Fig.2 Results of sodium dodecyl sulphate-polyacrylamide gel electrophoresis of recombinant vector before and after induction
M为Marker;A(1~4)为pMAL-c5X 诱导前、诱导后、上清、沉淀;A(5~8)为pGEX-4T-1 诱导前、诱导后、上清、沉淀;B(1~4)为pGEX-6P-1 诱导前、诱导后、上清、沉淀;C(1~4)为pSmart-I 诱导前、诱导后、上清、沉淀。
不同IPTG 浓度诱导前后SDS-PAGE结果见图3。由图3可知,RsbU-MBP分子质量为66 kDa,与预期分子质量65.18 kDa相符,测定其浓度为190 μmol/L;RsbU-MBP分子质量为50 kDa,与预期分子质量48.18 kDa相符,测定其浓度为25 μmol/L;RsbU-MBP分子质量为35 kDa,与预期分子质量33.38 kDa相符,测定其浓度为50 μmol/L。

图3 不同IPTG质量浓度诱导前后十二烷基硫酸钠-聚丙烯酰胺凝胶电泳结果
Fig.3 Results of sodium dodecyl sulphate-polyacrylamide gel electrophoresis before and after induction with different IPTG mass concentration
M为Marker;A(1~4)为10 mg/mL IPTG 诱导前、诱导后、上清、沉淀;A(5~8)为25 mg/mL IPTG诱导前、诱导后、上清、沉淀;B(1~4)为50 mg/mL IPTG 诱导前、诱导后、上清、沉淀;C(1~4)为100 mg/mL IPTG 诱导前、诱导后、上清、沉淀;C(1~4)为250 mg/mL IPTG 诱导前、诱导后、上清、沉淀。
2.2 RsbU的生物信息学分析结果
2.2.1 RsbU的基本理化性质和结构域分析
经ProtParam分析后得到的RsbU的基本理化性质,见表2。
表2 RsbU基本理化性质
Table 6 Basic physicochemical properties of RsbU
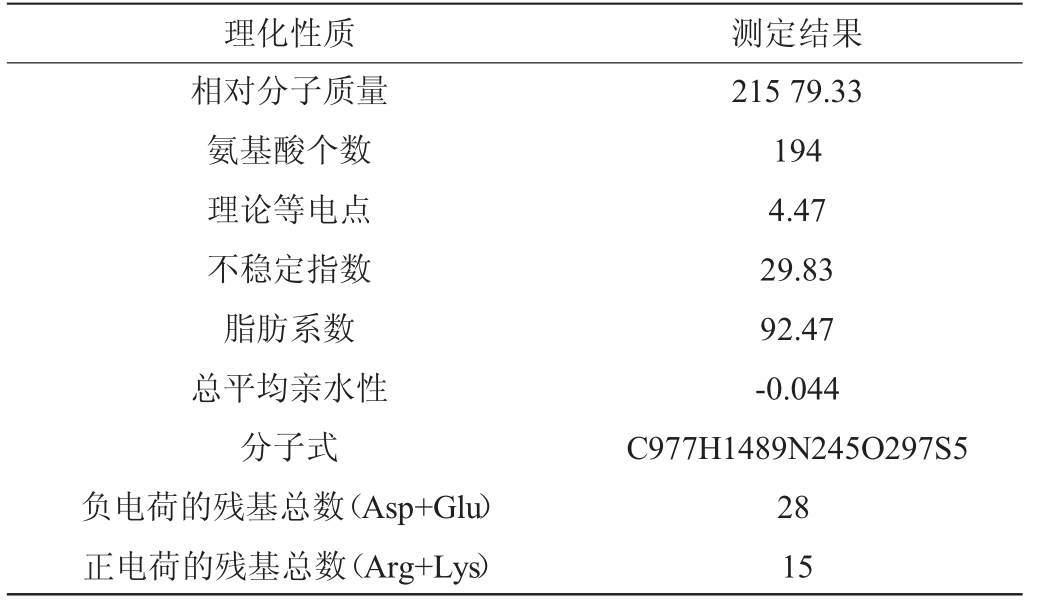
理化性质测定结果相对分子质量氨基酸个数理论等电点不稳定指数脂肪系数总平均亲水性分子式负电荷的残基总数(Asp+Glu)正电荷的残基总数(Arg+Lys)215 79.33 194 4.47 29.83 92.47-0.044 C977H1489N245O297S5 28 15
表3 RsbT与RsbU的相互作用力、作用位点及位点间距离
Table 3 Interaction forces, action sites and distance between sites of RsbT and RsbU

作用位点距离/Å 相互作用力备注T:LYS141:HZ1-U:GLU57:OE2 T:ARG118:NH1-U:ASP29:OD2 U:ARG26:NH1-T:ASP121:OD2 T:ARG118:HH11-U:ASP29:OD1 T:LYS139:HZ3-U:ARG26:O T:TRP140:HE1-U:GLU167:OE2 T:LYS141:HZ2-U:GLU57:OE1 U:PHE3:HN-T:ARG118:O U:TYR5:HN-T:ASP121:O U:TYR22:HH-T:ARG78:O U:ARG26:HN-T:GLU42:OE2 U:ARG26:HH11-T:MET119:O U:GLU53:HE2-T:GLU79:OE2 U:GLU4:CA-T:ASP121:O U:ARG26:CD-T:GLU45:OE1 U:ARG26:CD-T:MET119:O T:LYS141:OCT2-U:TYR23 U:VAL7:CG2-T:TRP140 T:TYR124-U:TYR168 T:PRO114-U:MET1 T:ARG117-U:MET1 T:TRP140-U:VAL24 T:TRP140-U:VAL7 2.001 04 3.928 58 2.958 13 2.158 39 2.744 85 2.014 46 2.264 51 2.879 08 1.966 95 1.886 66 2.739 51 2.305 87 2.546 74 3.185 91 3.681 11 3.769 75 3.766 66 3.651 31 5.637 71 4.720 16 4.875 03 5.138 34 4.154 02盐桥静电作用静电作用氢键氢键氢键氢键氢键氢键氢键氢键氢键氢键氢键氢键氢键静电作用疏水相互作用疏水相互作用疏水相互作用疏水相互作用疏水相互作用疏水相互作用氢键和静电引力有吸引力的电荷有吸引力的电荷传统氢键传统氢键传统氢键传统氢键传统氢键传统氢键传统氢键传统氢键传统氢键传统氢键C-H…O型C-H…O型C-H…O型阴离子-π σ-π π-π(T-shaped)烷基烷基π-烷基π-烷基
经NCBI数据库比对可知,RsbU属于PP2C超家族[18],含有PP2Cc和SpollE结构域。用Protscale预测亲疏水性,结果见图4。由图4可知,得分越低则表示亲水性更强,说明RsbU的N端疏水性强于C端,是弱亲水性蛋白[19]。
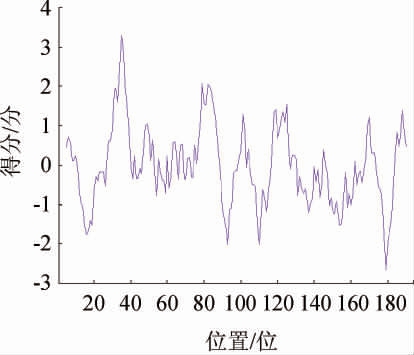
图4 RsbU亲疏水性分析结果
Fig.4 Analysis results of RsbU hydrophilicity and hydrophobicity
2.2.2 磷酸化位点、N-糖基化位点和功能位点分析
NetPHos 3.1分析发现,RsbU含有31个潜在磷酸化位点,1个潜在N-糖基化位点(为第176位NYSK[20])。Prosite分析发现,RsbU具有1个N-糖基化位点、1个酪氨酸激酶磷酸化位点、2个酪蛋白激酶Ⅱ磷酸化位点、1个蛋白激酶C磷酸化位点、5个N-肉豆蔻酰基化位点,均可用于蛋白的翻译后修饰,参与各种生物功能[21]。
2.2.3 RsbU的二及三级结构
用SOMPA预测RsbU二级结构,结果见图5A;用NCBI数据库预测并优化RsbU三级结构得到模型见图5B。由图5可知,三级结构模型中含有的二级结构与预测的二级结构能够对应。
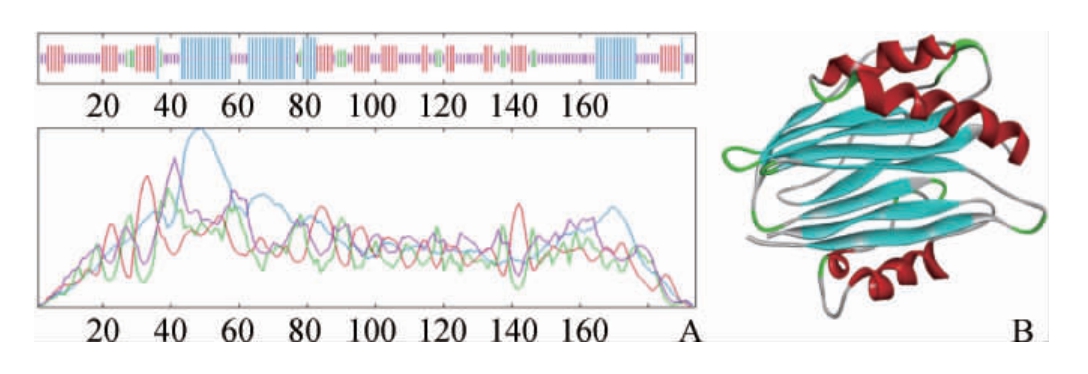
图5 RsbU蛋白的二级结构(A)及三级结构(B)
Fig.5 Secondary structure (A) and tertiary structure(B) of RsbU protein
图5A中紫色代表无规卷曲,红色代表β-折叠,绿色代表β-转角,蓝色代表α-螺旋;图5B蓝色代表β-折叠;红色代表α-螺旋;灰色代表无规卷曲;绿色代表β-转角。
2.2.4 RsbU与pNPP的分子对接
使用Autodock-vina预测RsbU与pNPP的的结合能(即对接打分值,该值介于-5.0~-7.0之间表明化合物与靶点的结合能力较好),对接打分值为-5.8 kcal/mol~-5.0 kcal/mol,说明pNPP能够与RsbU发生良好、自发的结合[22]。使用Pymol进行分子对接的可视化,结果见图6。由图6可知,pNPP 能够稳定的结合到RsbU蛋白Asp89、Gln94和Lys96形成的空腔中,主要通过氢键和疏水相互作用与周围的氨基酸发生相互作用。
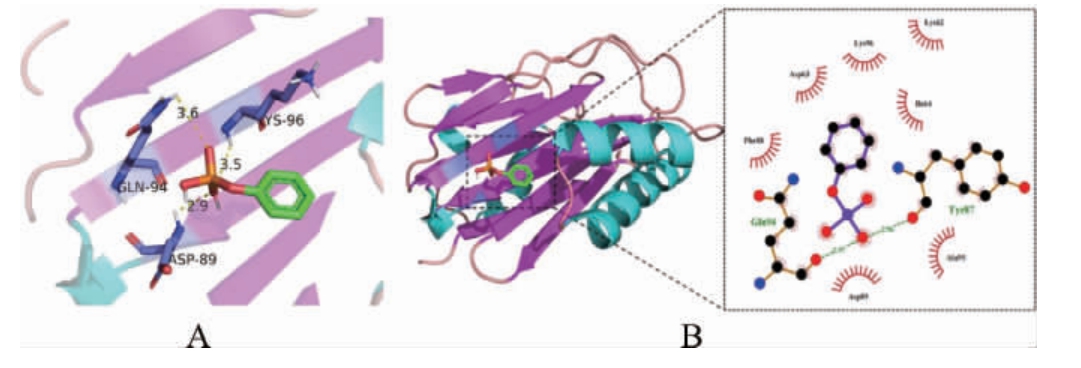
图6 RsbU与pNPP的分子对接模式
Fig.6 Molecular docking pattern of RsbU and pNPP
A为RsbU与pNPP 的对接三维局部模式图;B为RsbU与pNPP对接的二维局部模式图。
2.2.5 RsbU与RsbT的分子对接
用RDock 筛选出最优对接模式的ZDock score 为-2.980 91 kcal/mol,说明RsbU与RsbT间有结合能力,但不够强[23]。RsbT与RsbU之间的对接模式见图7。由图7A可知,分别涉及RsbU的8个氨基酸和RsbT的7个氨基酸,其中RsbU的Try22与bsRsbU的Try28对应。除此之外,RsbU的C端也有结合位点。由图7B可知,RsbU与RsbT结合界面的主要作用力包括7个氢键(绿色线条)、静电引力(红色线条)、盐桥以及疏水相互作用。
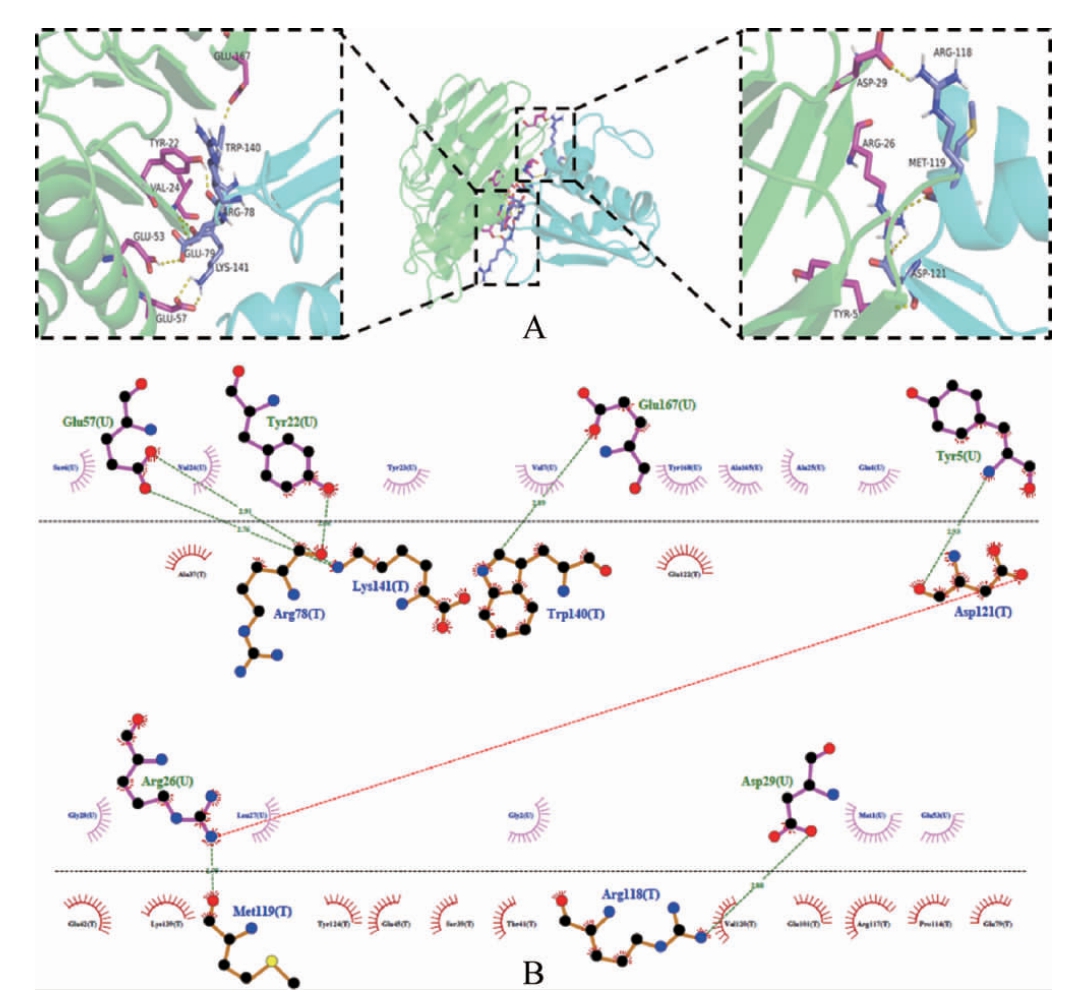
图7 RsbU与RsbT的分子对接模式
Fig.7 Molecular docking pattern of RsbU and RsbT
A为RsbU与RsbT的三维局部对接模式;B为RsbU与RsbT的二维局部对接模式。
RsbU与RsbT的分子对接各相互作用的作用类型、作用位点以及位点间距离见表7。由表7可知,RsbU与RsbT间中存在的13个氢键中有3个是长程弱C-H…O型氢键,除此之外的作用力都不强。总之,结合界面间形成的氢键、静电引力和盐桥是稳定RsbU与RsbT结合的主要作用力,但不够稳定。
2.2.6 RsbU的蛋白互作
RsbU与和其互作性最强的10种蛋白的互作图谱见图8。由图8可知,RsbU与EGA65439.1(RsbT,Ser/Thr蛋白激酶)、EGA65441.1(RsbR,甲基趋化蛋白)的互作分数分别高达0.990和0.987,表明RsbU有很大可能通过互作参与应激信号传导通路。
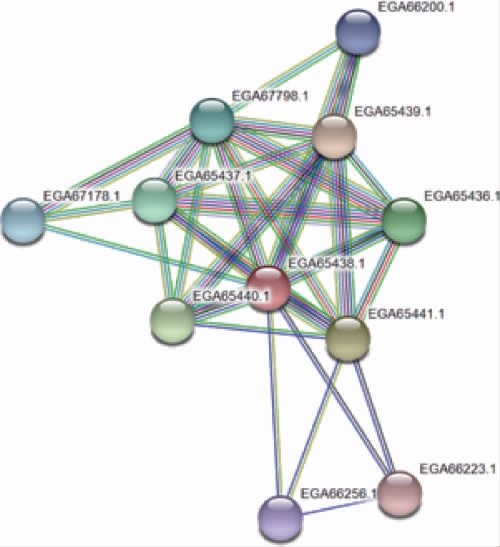
图8 RsbU蛋白互作分析
Fig.8 Interaction analysis of RsbU proteins
2.3 RsbU的酶学性质
2.3.1 RsbU-GST与pNPP反应的影响
去磷酸化后的pNPP会形成黄色的pNP,而pNP的物质的量与OD405nm值成正比,因此可用OD405 nm值直接表征磷酸酶的酶活性。金属离子、最适离子浓度、pH及温度对酶活的影响见图9。由图9A可知,Ca2+、Mg2+和Mn2+对酶活的影响均随作用时间的延长而增加,其中Mn2+对酶活性的促进作用最强。因此,最适金属离子为Mn2+。由图9B可知,随Mn2+浓度在4~10 mmol/L范围内增加,酶活性先增加后趋于平稳,并在Mn2+浓度为5 mmol/L时,酶活性最高。因此,最适Mn2+浓度为5 mmol/L。由图9C可知,在pH为3.0~6.0时,酶活性变化不大;在pH为6.0~8.0时,酶活性随之升高;在pH为8.0时,酶活性最高;在pH>8.0之后,酶活性呈下降趋势。因此,最适pH为8.0。由图9D可知,温度为0~47 ℃时,酶活性随之增高,在温度为47 ℃时,酶活性最高;在温度>47 ℃之后,酶活性迅速下降,说明RsbU对温度较为敏感。
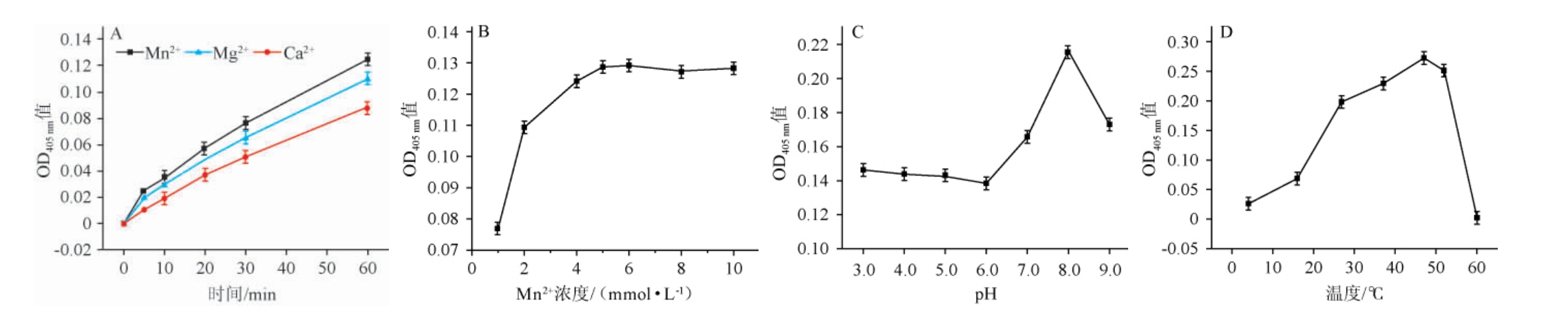
图9 金属离子(A)、最适离子浓度(B)、pH(C)及温度(D)对酶活的影响
Fig.9 Effect of metal ions (A), optimal ion concentration (B), pH (C) and temperature (D) on enzyme activity
2.3.2 RsbU的酶反应动力学曲线
丝/苏氨酸磷酸肽反应的标准曲线、酶活性变化曲线和酶反应动力学曲线见图10。由图10A可知,丝/苏氨酸磷酸肽反应的标准曲线相关系数R2=0.999 1,OD590 nm值和PO42-之间线性拟合较好,可以用OD590 nm值表征酶活性。由图10B可知,随磷酸酶RsbU物质的量增加,PO42-浓度之间增加,酶活性增强,这符合Ser/Thr磷酸酶在丝/苏氨酸磷酸肽反应后的表现,因此可以验证RsbU是Ser/Thr磷酸酶。由图10C可知,RsbU水解Ser/Thr磷酸酶时,酶反应动力学方程为y=57.81838x+1.17504,最大反应速率(Vm)为0.85μmol/(L·min),米氏常数(Km值)为49.21 μmol/L。
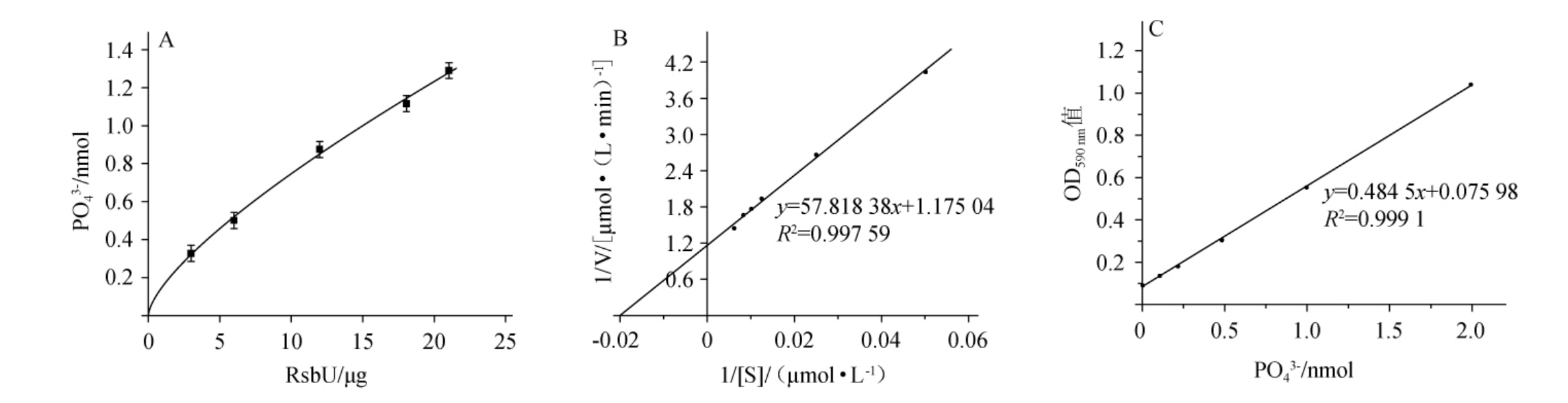
图10 丝/苏氨酸磷酸肽反应的标准曲线(A)、酶活性变化曲线(B)和酶反应动力学曲线(C)
Fig.10 Standard curve (A), enzyme activity variation curve (B) and enzyme reaction kinetics curve (C) of filament/threonine phosphopeptide reaction
2.4 RsbU与RsbT的蛋白互作
Western Blot可通过分析蛋白条带及灰度以确定其磷酸化程度,Western Blot采用的是聚丙烯酰胺凝胶电泳。由图11A和图11B可知,pSmart-1-RsbU加入后,条带灰度变化不大,表明其对RsbT磷酸化程度影响不显著。由图11C、11D可知,RsbU-RsbT的条带灰度大于RsbT,因此RsbU-MBP的加入使条带灰度增加。但RsbU和RsbT相加的条带灰度(图中蓝色线条)与RsbU-RsbT的条带灰度相似,因此两者之间可能没有互作,灰度变化可能来自MBP标签与抗体间的非特异性结合。由此可得RsbU-SUMO、RsbU-MBP与RsbT之间并没有明显互作。
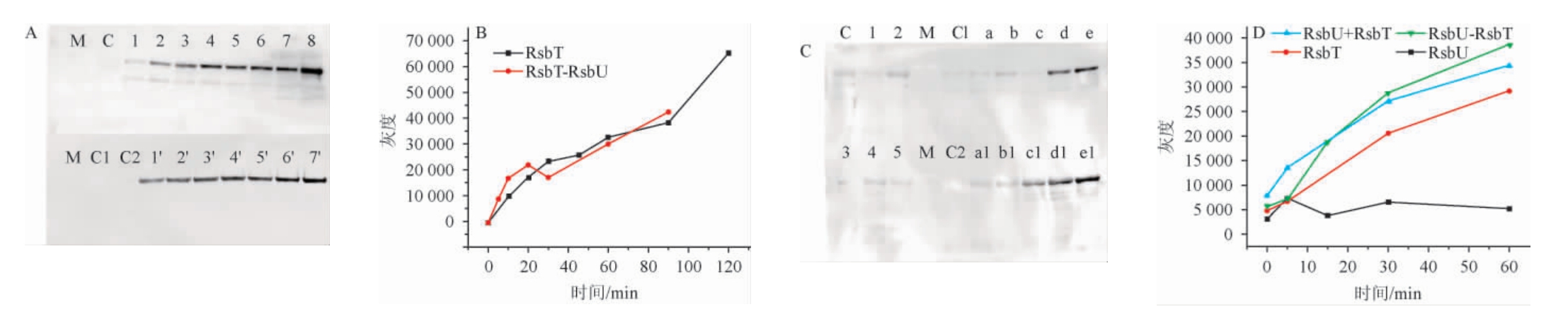
图11 pSmart-1-RsbU与RsbT、RsbU-MBP与RsbT的聚丙酰胺电泳及灰度分析
Fig.11 Polyacrylamide gel electrophoresis and grayscale analysis of pSmart-1-RSBU and RsbT, RsbU-MBP and RsbT
图A、C为聚丙酰胺电泳:M为Marker;C、C1、C2为对照;1~8为0、10 min、20 min、30 min、45 min、60 min、90 min、120 min;1'~7'为0、5 min、10 min、
20 min、30 min、60 min、90 min;RsbU-MBP(1:0 min、2:5 min、3:15 min、4:30 min、5:60 min;RsbT(a:0 min、b:5 min、c:15 min、d:30 min、e:60 min);
RsbT-RsbU(a1:0 min、b1:5 min、c1:15 min、d1:30 min、e1:60 min);图B、D为灰度分析图。
由图12A和图12B可知,加入RsbU-GST后,RsbT的自磷酸化活性显著降低,说明RsbU在一定程度上抑制RsbT的自磷酸化。相比之下,在自磷酸化30min后的RsbT中加入RsbUGST继续反应,RsbT的磷酸化受抑制程度明显减小,说明RsbU-GST对还未磷酸化的RsbT的抑制作用更明显。
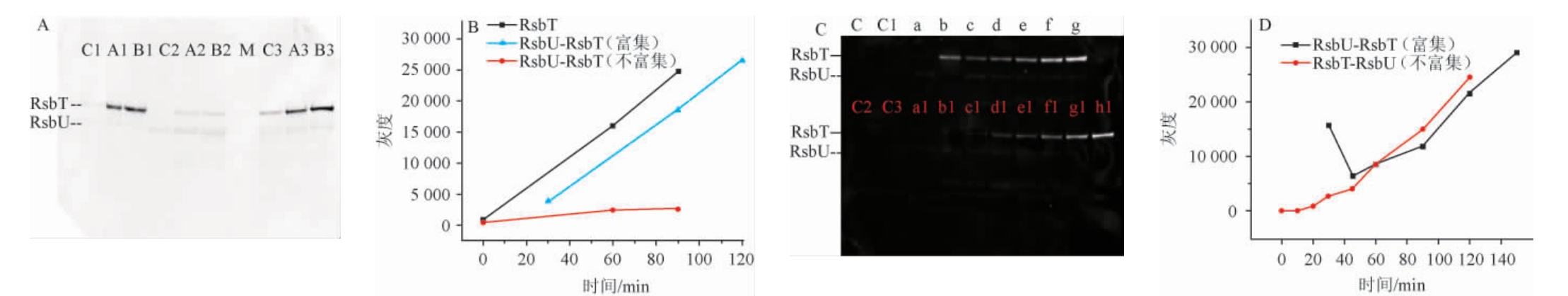
图12 RsbU-GST与RsbT的聚丙酰胺电泳、灰度分析和互作分析
Fig.12 Polyacrylamide gel electrophoresis, grayscale analysis and interaction analysis of RsbU-GST and RsbT
图A为聚丙酰胺电泳;C1为RsbT 0 min;A1为RsbT 60 min;B1为RsbT 90 min;C2为RsbU-RsbT 0 min;A2为RsbU-RsbT 60 min;B2为RsbU-RsbT 90 min;
C3为RsbU-RsbT 富集30 min后0 min;A3为RsbU-RsbT富集30 min后60min;B3为RsbU-RsbT富集30 min后90 min;图B为灰度分析;图C为聚丙酰胺电泳(C/C1/C2/C3:对照、a~g:RsbU-RsbT(聚集30 min);a:0 min、b:30 min、c:45 min、d:60 min、e:90 min、f:120 min、g:150 m;a~g:RsbU-RsbT(不聚集);
a1:0 min、b1:10 min、c1:20 min、d1:30 min、e1:45 min、f1:60 min、g1:90 min、h1:120 min);图D为灰度分析。
为研究RsbU-GST对RsbT的磷酸化抑制作用是通过去磷酸化RsbT-P还是抑制RsbT的磷酸化,进行进一步实验。由图12C和图12D红色曲线可知,在RsbU存在时,RsbT的磷酸化从30 min左右才开始,说明RsbU蛋白具有去磷酸化的作用,但在图12C中RsbU的灰度值极低,且灰度值不随时间变化,说明其不具有携带磷酸基团的能力,因此推断RsbU在去磷酸化过程中不会同时自身携带磷酸基团。由黑色曲线可知,在RsbU的存在下,RsbT自磷酸化程度先短暂减弱但之后仍呈现上升趋势,表明RsbU的去磷酸化活性低于RsbT自身磷酸化活性。因此可推断,RsbU-GST对RsbT的磷酸化抑制作用是通过去磷酸化RsbT-P。
3 讨论
通过RsbU与RsbT的蛋白互作研究可知RsbU-GST具有阻碍RsbT自身磷酸化和去磷酸化RsbT-P的能力[24],但去磷酸化能力较弱且主要在前期起作用,去磷酸过程中不会自身携带磷酸基团。结合RsbU与RsbT的分子对接预测,RsbU-GST和RsbT之间有弱结合作用。在这一步预测的结合界面上涉及的RsbU的8个氨基酸中有7个靠近N端,因此猜测RsbU的N端主要负责与RsbT结合来接收信号[25],而C端与RsbT的微弱结合用于部分去磷酸化RsbT-P。
在RsbU-GST的C端去磷酸化的过程中,其对还未磷酸化的RsbT的抑制作用比对已磷酸化的抑制作用更明显,且去磷酸化能力弱。这可能是因为RsbU与RsbT的结合具有短时性,达到信号传导作用后通过静电力的消除等解除互作,二者属于弱结合,这与前文的对接结果一致。在过去对枯草芽孢杆菌的研究中,发现bsRsbU与bsRsbT的互作也具有短时性,在结合20 min后信号传导最强,30 min后二者不再互作[26]。因此,该结论与预测对接模式、枯草芽孢杆菌中的互作模式一致,具有较高参考价值。
RsbU在去磷酸化过程中不会同时自身携带磷酸基团,这表明RsbU不是RsbT的直接作用底物,二者主要结合在RsbU的N-端,并在结合界面形成复合物。这与枯草芽孢杆菌传递信号的机理一致,bsRsbT传递信号的方式不是磷酸化bsRsbU,而是与RsbU结合互作[27]。在革兰氏阳性菌的信息传导过程中,蛋白的N端结构域也被用于环境信号的接收,在改变构象后传递信号,N端对其与RsbT的结合有重大的意义[23],与本文结论一致。
经本研究发现,在巴西弧菌中RsbU和RsbT二者间存在互作关系,RsbU的N端与RsbT结合来接收应激信号,而C端与RsbT的微弱结合用于部分去磷酸化RsbT-P。综上,认为RsbT可以将应激信号传导给RsbU,并激活RsbU,将上游应激信号传导至下游通路。
4 结论
本研究成功构建了四种表达载体,分别是pGEX-4T-1-RsbU、pMAL-c5X-RsbU、pGEX-6P-1-RsbU、pSmart-1-RsbU,并纯化了RsbU-SUMO、RsbU-MBP、RsbU-GST三种标签蛋白。String的结果显示,RsbU与RsbT之间的互作分数超过0.98,酶学性质结果表明,RsbU是Ser/Thr磷酸酶,去磷酸化能力较弱且主要在前期作用,自身不携带磷酸基团。因此推断出以下结论:①预测的结合界面上涉及的RsbU的8个氨基酸中有7个靠近N端,RsbU的N端主要负责与RsbT结合来接收信号,而C端与RsbT的微弱结合用于部分去磷酸化RsbT-P。②RsbU与RsbT的结合具有短时性,达到信号传导作用后解除互作,二者属于弱结合。③RsbU在去磷酸化过程中不会自身携带磷酸基团,这表明RsbU不是RsbT的直接作用底物,其传递信号的方式不是直接磷酸化RsbU,而是结合在RsbU的N-端,并在结合界面形成复合物。
综上,在巴西弧菌(Vibrio brasiliensis)受环境胁迫后,激酶RsbT被激活并磷酸化,通过与下游蛋白RsbU进行短时结合来传递应激信号至下游。在两者结合时,RsbU的N端主要接收来自RsbT的信号,而C端用于部分去磷酸化RsbT,其通过结合在RsbU的N-端传递信号。以上结论与枯草芽孢杆菌中两蛋白的互作模式有较高相似度,可信度较高。但影响两者形成复合物的能力与趋势的因素,以及预测的结合位点是否正确还需更多研究证明。应激信号在传递给RsbU后,在下游通路中的传导也有待研究。
[1]ESCHBACH E,MARTIN A,HUHN J,et al.Detection of enteropathogenic Vibrio parahaemolyticus,Vibrio cholerae and Vibrio vulnificus:performance of real-time PCR kits in an interlaboratory study[J].Eur Food Res Technol,2017,243(8):1335-1342.
[2]滕勇勇,王琪,吴雷,等.致病性弧菌的生物学特性和致病因子研究进展[J].热带医学杂志,2014,14(10):1396-1398.
[3]DONALDSON G P,LEE S M,MAZMANIAN S K.Gut biogeography of the bacterial microbiota[J].Nat Rev Microbiol,2016,14(1):20-32.
[4]张艺蓓.EnvZ/OmpR感应Fe3+调控副溶血性弧菌毒力因子的分子机制研究[D].杨凌:西北农林科技大学,2021.
[5]欧杰,王秋钰,黄奥迪,等.水产品中弧菌生物被膜的形成及调控机制[J/OL].水产学报:1-11[2024-04-07].http://kns.cnki.net/kcms/detail/31.1283.S.20220618.1834.002.html.
[6]郭玉坤,朱遇安,范晓明,等.创伤弧菌致病机制的研究进展[J].中华损伤与修复杂志(电子版),2022,17(1):81-84.
[7]CHI P S, WONG H C.Variation of genomic islands and flanking fragments in Vibrio parahaemolyticus isolates from environmental and clinical sources in Taiwan[J].Int J Food Microbiol,2017,259(1):68-76.
[8]LIEBAL U W, MILLAT T, MARLES-WRIGHT J, et al.Simulations of stressosome activation emphasize allosteric interactions between RsbR and RsbT[J].BMC Syst Biol,2013,7(1):3.
[9]WOODBURY R L,LUO T,GRANT L,et al.Mutational analysis of RsbT,an activator of the Bacillus subtilis stress response transcription factor,σB[J].J Bacteriol,2004,186(9):2789-2797.
[10]PANÉ-FARRÉ J,LEWIS R J,STÜLKE J.The RsbRST stress module in bacteria: A signalling system that may interact with different output modules[J].Microb Physiol,2005,9(2):65-76.
[11]冯莹颖,张晓莉,罗勤,等.Sigma B因子活性的调节及其在几种革兰氏阳性食源性致病菌中的作用[J].微生物学报,2008,48(6):839-843.
[12]HARDWICK S W, PANÉ-FARRÉ J, DELUMEAU O, et al.Structural and functional characterization of partner switching regulating the environmental stress response in Bacillus subtilis[J].J Biol Chem, 2007,282(15):11562-11572.
[13]PANÉ-FARRÉ J,JONAS B,HARDWICK S W,et al.Role of RsbU in controlling SigB activity in Staphylococcus aureus following alkaline stress[J].J Bacteriol,2009,191(8):2561-2573.
[14]ROTHSTEIN D M,LAZINSKI D,OSBURNE M S,et al.A mutation in the Bacillus subtilis rsbU gene that limits RNA synthesis during sporulation[J].J Bacteriol,2017,199(14):e00212-17.
[15]JIA X,WANG J B,RIVERA S,et al.An O2-sensing stressosome from a Gram-negative bacterium[J].Nat Commun,2016,7(1):12381-12381.
[16]马骏骏,王旭初,聂小军.生物信息学在蛋白质组学研究中的应用进展[J].生物信息学,2021,19(2):85-91.
[17]LI M,MENG X G,DU J,et al.Metallomicellar catalysis:Hydrolysis of PNPP catalyzed by copper(II),zinc(II),cerium(IV)complexes with long alkyl pyridine ligands in CTAB micellar solution[J].J Disper Sci Technol,2009,30(8):1182-1187.
[18]ROSARIO C,TAN M.Chlamydia trachomatis RsbU phosphatase activity is inhibited by the enolase product,phosphoenolpyruvate[J].J Bacteriol,2022,204(10):e00178-22.
[19]BARÁK I,BEHARI J,OLMEDO G,et al.Structure and function of the Bacillus SpoIIE protein and its localization to sites of sporulation septum assembly[J].Mol Microbiol,1996,19(5):1047-1060.
[20]任伟夫,边琪.蛋白质N-糖基化的生物学功能、分析方法及其在肾脏疾病中作用的研究进展[J].临床肾脏病杂志,2021,21(11):935-941.
[21]谭永聪,王启军,赵国屏,等.原核生物的蛋白质翻译后修饰[J].生物化学与生物物理进展,2011,38(3):197-203.
[22]TANG S,CHEN R,LIN M,et al.Accelerating AutoDock Vina with GPUs[J].Molecules,2022,27(9):3041-3041.
[23]CHEN R,LI L,WENG Z.ZDOCK: an initial-stage protein-docking algorithm[J].Proteins,2003,52(1):80-87.
[24]CHATURONGAKUL S,BOOR K J.RsbT and RsbV contribute to σB-dependent survival under environmental, energy, and intracellular stress conditions in Listeria monocytogenes[J].Appl Environ Microbiol,2004,70(9):5349-5356.
[25]MARLES-WRIGHT J,GRANT T,DELUMEAU O,et al.Molecular architecture of the "Stressosome," a signal integration and transduction Hub[J].Science,2008,322(5898):92-96.
[26]DELUMEAU O,DUTTA S,BRIGULLA M,et al.Functional and structural characterization of RsbU,a stress signaling protein phosphatase 2C[J].J Biol Chem,2004,279(39):40927-40937.
[27]KANG C M,VIJAY K,PRICE C W.Serine kinase activity of a Bacillus subtilis switch protein is required to transduce environmental stress signals but not to activate its target PP2C phosphatase[J].Mol Microbiol,1998,30(1):189-196.