传统发酵乳制品是新疆地区牧民的传统特色食品,具有丰富的乳酸菌资源[1]。传统发酵乳制品中的微生物组成和数量受该地的海拔高度、气候变化、发酵温度和时间以及个人制作方式等因素的影响[2]。传统发酵乳制品中的乳酸菌主要包括乳杆菌属(Lactobacillus)、链球菌属(Streptococcus)、乳球菌属(Lactococcus)、肠球菌属(Enterococcus)和明串珠菌属(Leuconostoc),并呈现出丰富的生物多样性[3]。新疆地域广阔,不同地区少数民族均有利用牛乳、马乳、驼乳、羊乳等发酵自然乳制品的习俗,其中蕴含着丰富的乳酸菌资源[4]。
黄坚等[5]从传统牦牛发酵酸奶中筛选出4株具有提高动物生长性能和改善免疫力功能的耐久肠球菌(Enterococcus durans),其中耐久肠球菌SWUN5857能很好地适应胃肠环境压力,有效提高动物的生长性能和免疫功能,可以作为候选的益生性菌株。董翎逸等[6]研究发现,屎肠球菌(Enterococcus faecium)WEFA23和海氏肠球菌(Enterococcus hirae)WEHI01可增强荷叶的抗氧化能力,且具有较强拮抗致病菌的能力。相关研究发现[7]从我国传统发酵乳制品分离得到的如粪肠球菌(Enterococcus faecalis)、屎肠球菌(Enterococcus faecium)和耐久肠球菌(Enterococcus durans)等肠球菌分离株,具有维持肠道稳态等良好的益生特性,上述物种的分离株已经用于食品发酵和临床微生物学相关研究[4]。
目前,我国市场上销售的发酵乳制品所使用的发酵剂大多依赖进口,缺少具有自主知识产权的菌株[8]。新疆地区传统发酵乳制品中乳酸菌资源丰富,挖掘新疆地区传统发酵乳制品中乳酸菌资源具有重要意义。本研究在新疆南疆和田地区采集了新疆地区传统发酵乳制品,对其中的乳酸菌进行分离、培养、鉴定,并将分离筛选肠球菌进行16SrRNA基因测序及API 50 CHL糖发酵实验,以期揭示筛选菌株的碳水化合物代谢多样性,为后续肠球菌资源开发利用奠定基础。本研究揭示了新疆南疆和田地区传统发酵乳制品中肠球菌的多样性,为肠球菌资源开发利用奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 材料
本研究从在新疆南疆和田地区采集了3份新疆牧民家庭传统工艺制作的自然发酵酸牛奶,用于乳酸菌分离鉴定。样品采集时,用无菌吸管吸取2~5 mL传统发酵乳于加有保护剂(碳酸钙∶淀粉=50∶1)的无菌采样管中,用冰袋降温运送至实验室,立即进行乳酸菌的分离和鉴定。
1.1.2 化学试剂
琼脂粉(生化试剂):北京康倍斯科技有限公司;TIANamp细菌脱氧核糖核酸(deoxyribonucleic acid,DNA)试剂盒:天根生化科技(北京)有限公司;DneasyPowerFood Kit:德国QIAGEN公司;聚合酶链式反应(polymerase chain reaction,PCR)试剂:上海派森诺生物科技有限公司;API 50 CHL糖发酵鉴定试剂盒:法国梅里埃公司。
1.1.3 培养基
MRS 培养基:青岛高科技工业园海博生物技术有限公司。
1.2 仪器与设备
SX-500型全自动高压灭菌锅:日本TomyDigitalBiology公司;ND-1000型微量紫外分光光度计:美国Nanodrop公司;ZHJH-C1214C超净工作台:上海智城仪器制造厂;9902型PCR仪:美国Life Technologies公司;CX33光学显微镜:日本OLYMPUS公司;5810R台式高速离心机、KDC-1044立式低速离心机:德国Eppendorf 公司。
1.3 方法
1.3.1 乳酸菌的分离
采用十倍梯度稀释法[9]将传统发酵乳样品进行稀释后,选取合适的浓度梯度(10-4、10-5、10-6)涂布于MRS固体培养基,置于37 ℃恒温培养箱需氧培养48~72 h。待菌落形成后,挑取单个菌落在MRS固体培养基上采用平板划线法进行2~3次的划线纯化[10]。将镜检结果为单一菌落且形态为球菌的菌落连续培养至第三代。
将革兰氏染色阳性、过氧化氢酶试验阴性的纯培养物确定为疑似乳酸菌[11],加入脱脂乳作为保护剂,均匀分置于安瓿管进行抽真空冷冻干燥保藏。
1.3.2 乳酸菌的鉴定
将培养三代菌液于8 000×g条件下离心5 min,用磷酸盐缓冲液(phosphate buffer solution,PBS)洗涤2次,革兰氏染色后,使用光学显微镜对细胞形态进行观察记录。
分子生物学鉴定:利用细菌基因组DNA提取试剂盒提取乳酸菌DNA,并用ND-1000型微量紫外分光光度计进行检测,若OD260 nm值/OD280 nm值范围保持在1.8~2.0即视为纯DNA样品。利用PCR扩增仪进行16S rRNA基因扩增[12]。PCR扩增体系为:2×EasyTaq PCR SuperMix 25 μL,上、下游引物(10 μmol/L)各1.5 μL,模板DNA 2 μL,加双蒸水(ddH2O)补充至50 μL。16S rRNA基因序列引物为27F(5'-AGAGTTTGATCMTGGCTCAG-3')和1492R(5'-ACCTTGTTACGACTT-3')。PCR扩增条件为:95 ℃预变性5 min;95 ℃变性1 min,58 ℃退火45 s,72 ℃延伸1 min,30个循环;72 ℃再延伸7 min。将PCR扩增成功的PCR产物送至上海桑尼公司进行16S rRNA基因测序,测序结果在美国国家生物技术信息中心(National Center of Biotechnology Information,NCBI)的GenBank数据库中进行基本局部比对搜索工具(Basic Local Alignment Search Tool,BLAST)同源性比对,选择同源性较高模式菌株序列为参考,采用TreeBeST 1.9.2软件基于邻接法(neighbor joining,NJ)进行系统发育树构建,Bootstrap值为1 000。通过iTol在线软件(https://itol.embl.de/)构建系统发育树用于菌种鉴定[13-14]。
1.3.3 筛选乳酸菌糖发酵实验
为探究不同菌株的碳水化合物代谢能力,参考李东宇等[15]的方法,严格按照API 50 CHL试剂盒说明书进行试剂的制备及实验操作。使用37 ℃恒温培养箱培养24 h。分别在12 h和24 h各记录1次数据。
2 结果与分析
2.1 乳酸菌的分离
采用培养组学方法,从新疆牧民家庭传统工艺制作的自然发酵酸牛奶中通过分离培养得到乳酸菌24株,通过镜检筛选出球菌7株,分别编号为622331、G22381、YL-CX-2-1、23150、916021、130和AQH21-3。
2.2 乳酸菌的鉴定
2.2.1 形态学观察
筛选7株菌的菌落及细胞形态见图1。由图1a可知,7株菌在MRS培养基中菌落呈圆形,乳白色不透明,表面光滑湿润,边缘整齐,无晕环,菌落中央有凸起,直径1~2 mm。结果显示,7株菌的菌落形态与罗成莹等[16]报道的乳酸菌菌落形态相似。由图1b可知,7株菌革兰氏染色为阳性,菌体形态为圆形或椭圆形,呈单个、成对或短链状排列,无芽孢,无鞭毛,直径约为0.5~1.0 μm。7株菌的镜检结果与陈晓慧等[17]报道粪肠球菌相似,因此,初步推测本研究筛选这7株菌为肠球菌(Enterococcus)。
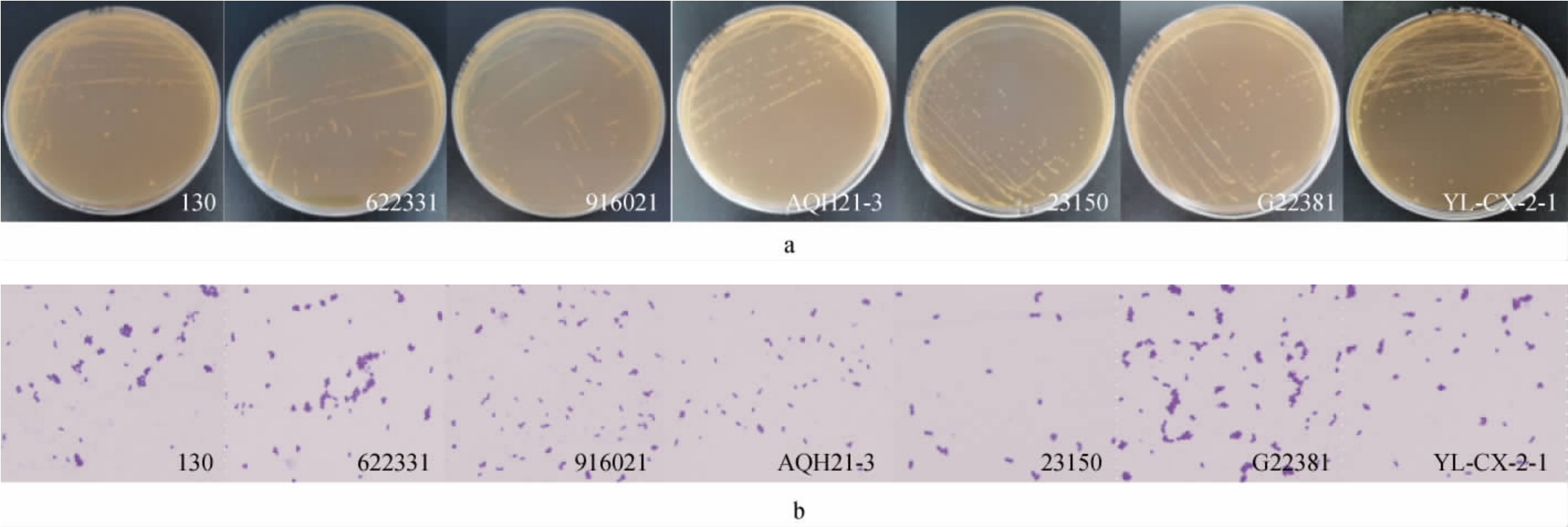
图1 筛选菌株的菌落(a)及细胞(b)形态
Fig.1 The colony (a) and cell (b) morphology of screened strains
2.2.2 分子生物学鉴定
细菌16S rRNA基因序列被称为细菌鉴定的黄金标准,在细菌学和分类学方面常被用于细菌的检测和鉴定[18]。本研究基于7株分离株模式株菌Enterococcus faecalis LMG 7937T、Enterococcus faecium LMG 11423T、Enterococcus durans DSM20633T、Enterococcus canintestini LMG 13590T及Enterococcus faecalis WMS RS15905、Enterococcus durans CJZ72 RS04065、Enterococcus faecium SRR24-E6A31 RS13515的16SrRNA基因序列构建系统发育树,结果见图2。
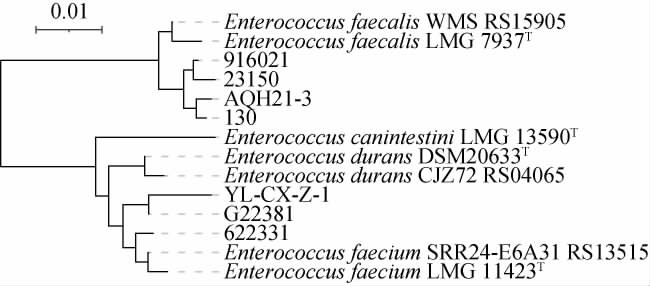
图2 基于16S rRNA基因序列筛选菌株的系统发育树
Fig.2 Phylogenetic tree of screened strains based on 16S rRNA gene sequences
由图2可知,分离菌株916021、23150、AQH21-3和130与粪肠球菌Enterococcus faecalis LMG 7937T、Enterococcus faecalis WMS RS15905聚在同一分支,分离菌株916021、23150、AQH21-3和130与Enterococcus faecalis LMG 7937T和Enterococcus faecalis WMS RS15905亲缘关系较近。622331、G22381和YL-CX-2-1与Enterococcus faecium LMG 11423T和Enterococcus faecium SRR24-E6A31 RS13515聚在同一分支,结果显示,分离菌株622331、G22381和YL-CX-2-1与屎肠球菌Enterococcus faecium LMG 11423T、Enterococcus faecium SRR24-E6A31 RS13515亲缘关系较近。而作为外群的Enterococcus canintestini LMG 13590T独自被分为一支,表明其与其他菌株亲缘关系较远。因此,菌株916021、23150、AQH21-3和130被鉴定为为粪肠球菌(Enterococcus faecalis),菌株622331、G22381和YL-CX-2-1被鉴定为为屎肠球菌(Enterococcus faecium)。
2.3 筛选菌株糖发酵实验
基于API 50 CHL试剂盒对菌株23150、916021、130、AQH21-3、622331、G22381、YL-CX-2-1进行糖发酵实验,分别在12 h和24 h进行信息采集,结果见表1。由表1可知,7株分离菌株糖发酵实验在12 h和24 h的结果一致。比较粪肠球菌23150、916021、130、AQH21-3糖酵解能力发现,菌株AQH21-3相较其他3株粪肠球菌不可利用丙三醇、山梨醇、苦杏仁苷、D-塔格糖,可利用L-阿拉伯糖。菌株916021、130可以利用D-蔗糖、D-松三糖,菌株916021还可利用L-鼠李糖,无法利用D-乳糖。比较屎肠球菌622331、G22381、YL-CX-2-1糖酵解能力发现,菌株G22381、YL-CX-2-1无法利用丙三醇、山梨醇、苦杏仁苷、葡萄糖酸钾,与其他两菌株622331、YL-CX-2-1相比G22381可利用L-阿拉伯糖,无法利用D-塔格糖。
表1 7株肠球菌糖发酵实验结果
Table 1 Results of sugar fermentation experiments of 7 strains of Enterococcus

注:“+”表示结果阳性;“-”表示结果阴性。
项目菌株23150菌株916021菌株130菌株AQH 21-3菌株622331菌株G22381菌株YL-CX-2-1阴性对照丙三醇赤藻糖醇D-阿拉伯糖L-阿拉伯糖D-核糖D-木糖L-木糖D-核糖醇甲基-β-D毗喃木糖苷D-半乳糖D-葡萄糖D-果糖D-甘露糖L-山梨糖L-鼠李糖卫矛醇肌醇甘露醇山梨醇甲基-a-D-毗喃甘露糖甘甲基-a-D-毗喃葡萄糖甘N-乙酰葡萄糖胺苦杏仁苷熊果甘七叶树柠檬酸铁水杨苷D-纤维二糖D-麦芽糖D-乳糖D-密二糖D-蔗糖D-海藻糖菊粉D-松三糖D-棉子糖淀粉糖原木糖醇D-龙胆二糖D-土伦糖D-来苏糖D-塔格糖D-岩藻糖L-岩藻糖D-阿拉伯醇L-阿拉伯醇葡萄糖酸钾2-酮基葡萄糖酸钾5-酮基葡萄糖酸钾-+---+——++++——++--++++++++--+——--+--+——+---+---+——++++-+--++--+++++++--++-+——+--+——+---+---+——++++——++--++++++++-++-+——+--+——+——--++——++++——+---+-++++++--+——--+——---+---+---+——++++——++--++++++++--+——--+--+——+——--++——++++————+-+++++++-+——--+——————---+——++++————+-++++++--+——--+--+——---
结果表明,实验所用4株粪肠球菌和3株屎肠球菌对碳水化合物利用不存在明显差异,即不存在某一碳水化合物仅能被全部粪肠球菌或屎肠球菌菌株利用。此外,相同物种内不同菌株对碳水化合物利用也存在差异。上述结果表明,肠球菌在碳水化合物利用上存在多样性。
3 讨论
新疆传统发酵乳制品中微生物多样性丰富[19],其中蕴含丰富的乳酸菌。这些乳酸菌经过长期的自然选择和驯化,不乏具有优良益生特性和生产特性的菌株[20]。肠球菌常被用作奶酪等乳制品的生产,是良好的乳酸菌发酵剂菌种[21],因此收集保护自然发酵乳制品中的肠球菌具有重要意义。本研究从新疆传统发酵乳制品中分离鉴定了7株肠球菌,主要是粪肠球菌、屎肠球菌和耐久肠球菌,并对发现不同肠球菌在碳水化合物利用上存在多样性。
屎肠球菌和粪肠球菌是最常见的肠球菌物种,其是人类和动物肠道的正常菌群[22-23]。因具有“强生命力”的特性,肠球菌广泛存在于大多数食品中[24]。MARTINO G P等[25]于阿根廷奶酪中分离出一株肠球菌。在肠球菌可作为发酵剂物种加速发酵进程[26],并可通过蛋白水解系统和脂类水解赋予发酵乳制品良好的稳定性和风味[27]。此外,肠球菌还被应用于发酵肉制品的制作[28]和果蔬罐头的储藏[29]。此外,相关研究发现部分肠球菌菌株可以产生细菌素,从而一直致病菌和腐败菌[30]。相关研究发现,肠球菌活菌还具有刺激免疫、抗炎活性、降胆固醇作用、预防和治疗某些疾病等有益特性[31]。有研究发现,含有粪肠球菌的新鲜奶酪在体外实验中对血管紧张素转换酶具有很高的活性抑制作用[32]。肠球菌作为重要的食品微生物,具有良好的应用前景。
不同菌株特性往往存在较大差异。李东宇等[15]研究发现,动物双歧杆菌乳亚种Probio-M8、V9、BB-12及HN019对L-阿拉伯糖、D-木糖、甘露醇、D-松三糖及D-塔格糖的代谢存在差异。宋宇琴[33]研究发现,不同德氏乳杆菌保加利亚亚种菌株可利用的糖的种类不同,表现出碳水化合物利用较高的多样性。相关研究发现编码碳水化合物利用系统的质粒对屎肠球菌适应环境提供帮助[34],而不同肠球菌的菌株其携带质粒的数量可能存在明显差异。此外,研究人员从肠球菌基因组检测到了低聚果糖代谢基因簇,进一步强调了肠球菌碳水化合物利用的菌株异质性[35]。
4 结论
本研究对分离自新疆地区传统发酵乳制品中的肠球菌进行分离、培养和保藏,将分离筛选的7株肠球菌进行16S rRNA基因测序,通过构建系统发育树确定菌株916021、23150、AQH21-3和130的分类学地位为粪肠球菌(Enterococcus faecalis),菌株622331、G22381和YL-CX-2-1的分类学地位为屎肠球菌(Enterococcus faecium)。通过API 50 CHL糖发酵实验,发现不存在某一碳水化合物仅能被粪肠球菌或屎肠球菌利用,表明粪肠球菌或屎肠球菌在碳水化合物利用上存在多样性。本研究分离保藏了7株肠球菌菌株,并揭示了肠球菌在碳水化合物利用上存在多样性,为后续肠球菌资源进一步开发研究奠定基础。
[1]蔡宏宇.新疆地区乳制品中乳酸菌多样性研究[D].呼和浩特:内蒙古农业大学,2021.
[2]SAKANDAR H A,ZHANG H.Curious case of the history of fermented milk:tangible evidence[J].Sci Bull,2022,67(16):1625-1627.
[3]耿瑶,曹颖,崔烓堃,等.食品中乳酸菌的种类及应用前景研究进展[J].食品工业,2023,44(3):234-238.
[4]刘玮,曹威,刘浩,等.益生屎肠球菌RS3 的分离鉴定及增殖条件的优化[J].食品研究与开发,2022,43(13):156-163.
[5]黄坚,童京京,岳华,等.牦牛发酵酸奶中耐久肠球菌的筛选鉴定和益生特性[J].食品科学,2017,38(12):43-49.
[6]董翎逸,刘正奇,裘梁,等.屎肠球菌WEFA23 和海氏肠球菌WEHI01发酵荷叶上清的抑菌及抗氧化评价[J].南昌大学学报(理科版),2018,42(4):375-381.
[7]韩慧玲,李华,包振江,等.内蒙古传统发酵乳制品中乳酸菌的分离及其耐药性研究[J].中国酿造,2022,41(2):40-47.
[8]万金敏.西藏传统发酵乳制品中优良乳酸菌的筛选及发酵性能研究[D].杨凌:西北农林科技大学,2017.
[9]YU J,WANG W H,MENGHE B L G,et al.Diversity of lactic acid bacteria associated with traditional fermented dairy products in Mongolia[J].J Dairy Sci,2011,94(7):3229-3241.
[10]GOOCH J.Streak Plate Method[M].New York:Springer,2011:926.
[11]张秋月,黎谢飞,曾小群,等.产超氧化物歧化酶乳酸菌的筛选及发酵条件优化[J].中国食品学报,2021,21(10):143-150.
[12]国笑情,武岳,李瑜,等.新疆地区母乳中乳酸菌的分离鉴定及潜在益生菌的筛选[J].食品与发酵工业,49(18):275-281.
[13]VILELLA A J,SEVERIN J,URETA-VIDAL A,et al.EnsemblCompara genetrees:Complete,duplication-aware phylogenetic trees in vertebrates[J].Genome Res,2009,19(2):327-335.
[14]DELBÈS C, ALI-MANDJEE L, MONTEL M-C.Monitoring bacterial communities in raw milk and cheese by culture-dependent and-independent 16S rRNA gene-based analyses[J].Appl Environ Microbiol,2007,73(6):1882-1891.
[15]李东宇,李伟程,康小红,等.母乳源乳双歧杆菌Probio-M8的遗传特征及基因组差异比较分析[J].微生物学报,2022,62(7):2768-2781.
[16]罗成莹,何秀,徐美余,等.五株乳酸菌和三株芽孢杆菌的生物学特性和功能[J].微生物学通报,2024,51(1):172-188.
[17]陈晓慧,徐淑琴,马祥兆,等.欧拉型藏羊源粪肠球菌的分离鉴定及药敏实验[J].食品工业科技,2021,42(22):133-139.
[18]刘琛,丛琳,刘文俊,等.塔吉克斯坦地区自然发酵牛乳中乳酸菌的分离鉴定及多样性分析[J].中国食品学报,2022,22(12):247-256.
[19]李伟程,侯强川,于洁,等.传统发酵乳制品中微生物多样性研究[J].食品工业科技,2018,39(1):131-136.
[20]张和平.自然发酵乳制品中乳酸菌的生物多样性[J].生命科学,2015,27(7):837-846.
[21]GIRAFFA G.Functionality of enterococci in dairy products[J].Int J Food Microbiol,2003,88(2-3):215-222.
[22]谢小芳,周惠琴,沈海英,等.632 株肠球菌属的临床分布及耐药分析[J].临床输血与检验,2012,14(2):116-118.
[23]宗春光,李军,杨亚平,等.老年患者肠球菌属感染的临床分布及耐药性分析[J].中华医院感染学杂志,2012,22(17):3868-3870.
[24]孙雅如.肠球菌发酵食品分离株的抗生素抗性及相关耐药基因的研究[D].呼和浩特:内蒙古农业大学,2018.
[25]MARTINO G P,QUINTANA I M,ESPARIZ M,et al.Draft genome sequences of four Enterococcus faecium strains isolated from Argentine cheese[J].Genome Announc,2016,4(1):e01576-15.
[26]夏玉,郑华,林捷,等.屎肠球菌发酵特性及其功能性研究[J].食品工业科技,2014,35(12):123-126.
[27]MORENO M F,SARANTINOPOULOS P,TSAKALIDOU E,et al.The role and application of enterococci in food and health[J].Int J Food Microbiol,2006,106(1):1-24.
[28]CALLEWAERT R, HUGAS M, DE VUYST L.Competitiveness and bacteriocin production of Enterococci in the production of Spanish-style dry fermented sausages[J].Int J Food Microbiol,2000,57(1-2):33-42.
[29]李雷花,孙俊红,马青素.尿路感染老年患者的病原菌分布及耐药分析[J].国际检验医学杂志,2012,33(12):1450-1451,1453.
[30]DE VUYST L,VANDAMME E J.Antimicrobial potential of lactic acid bacteria[M].New York:Springer,1994:91-142.
[31]BRAÏEK O B, SMAOUI S. Enterococci: Between emerging pathogens and potential probiotics[J].BioMed Res Int,2019,2019(1):5938210.
[32]TARANGO-HERNÁNDEZ S,ALARCÓN-ROJO A,ROBLES-SÁNCHEZ M, et al.Potential of Fresco-style cheese whey as a source of protein fractions with antioxidant and angiotensin-I-converting enzyme inhibitory activities[J].J Dairy Sci,2015,98(11):7635-7639.
[33]宋宇琴.德氏乳杆菌保加利亚亚种的群体遗传学和功能基因组学研究[D].呼和浩特:内蒙古农业大学,2018.
[34]BOUMASMOUD M,DENGLER HAUNREITER V,SCHWEIZER T A,et al.Genomic surveillance of vancomycin-resistant Enterococcus faecium reveals spread of a linear plasmid conferring a nutrient utilization advantage[J].MBio,2022,13(2):e03771-21.
[35]COMERLATO C B, PRICHULA J, SIQUEIRA F M, et al.Genomic analysis of Enterococcus durans LAB18S, a potential probiotic strain isolated from cheese[J].Genet Mol Biol,2022,45:e20210201.